Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Universidad y Salud
Print version ISSN 0124-7107On-line version ISSN 2389-7066
Univ. Salud vol.16 no.2 Pasto July/Dec. 2014
Sedea: Prototipo de sistema experto para el diagnóstico de enfermedades autoinmunes de órgano basado en internist
Sedea: Prototype expert system for the diagnosis of autoimmune diseases organ based internist
Christian Humberto Cabrera-Jojoa1
1 Ingeniero de sistemas, Magíster Ingeniería de sistemas y computación. Centro de Innovación en Tecnología y Educación, Conecta-TE Universidad de los Andes. Bogotá, Colombia. e-mail: ch.cabrera38@uniandes.edu.co
Fecha de recepción: Julio 15 - 2013 / Fecha de aceptación: Octubre 15 - 2014
Resumen
Realizar un buen diagnóstico es vital para el éxito en el tratamiento de una enfermedad, por ello, las herramientas que apoyan el proceso de diagnóstico son de gran interés. Particularmente, los especialistas en inmunología no cuentan con herramientas que apoyen el diagnóstico de enfermedades autoinmunes específicas de órgano. Esto hace que en dicho proceso los especialistas deban acudir a su experiencia y al conocimiento formalizado de esta área de la medicina. Pero cuando dicho conocimiento no está a la mano o simplemente no se cuenta con la experiencia, el diagnóstico presenta complicaciones que seguramente repercutirán en la salud del paciente. Desde las TI se han realizado diferentes intentos por colaborar en la tarea de diagnóstico, generalmente con la construcción de Sistemas Expertos que modelan el conocimiento de los especialistas ante circunstancias determinadas. Este trabajo plantea la creación de un prototipo de Sistema Experto para el Diagnóstico de Enfermedades Autoinmunes específicas de órgano SEDEA, el cual integra el conocimiento clínico con el modelo descriptivo ofrecido por Internist, a través de una ontología que permite manejar los diferentes conceptos por medio de reglas declaradas en el motor de inferencia de JESS, ofreciendo además interfaces que permiten ingresar y procesar datos con facilidad.
Palabras clave: Enfermedades autoinmunes, diagnóstico, Internist, ontología, sistema experto. (Fuente: DeCS, Bireme).
Abstract
To make a good diagnosis is vital to the successful in the treatment of a disease. Therefore, tools that contribute to more accurate diagnosis are of great interest. Particularly, immunology specialists do not have tools to support the organ-specific autoimmune diseases diagnosis process. This makes that during this process, specialists must resort to their experience and to the formalized knowledge of this medicine area. But when the knowledge is not at hand or simply no one has the experience, the diagnosis presents complications which will surely impact on the patients health. Different efforts to collaborate in diagnostic task has been made from the IT field; generally in the building of Experts Systems that model the specialist knowledge to certain conditions. This paper proposes the creation of an Expert System prototype for the Diagnostic of organ-specific Autoimmune diseases SEDEA, which not only integrates the clinical knowledge with the Internist descriptive model through an ontology that allows to handle the different concepts using rules declared in the JESS inference engine, but alsomoffers interfaces toeasilyinsert and process data.
Key words: Autoimmune diseases, diagnosis, expert system, Internist, ontological model. (Source: DeCS, Bireme).
Introducción
Las enfermedades autoinmunes, son un conjunto de enfermedades en las cuales la patología es causada por las respuestas inmunitarias adaptativas a los antígenos propios del cuerpo.1 Físicos y científicos han identificado más de 80 enfermedades autoinmunes distintas en las cuales el sistema inmune, por error, ataca sus propios órganos, tejidos y células.2
Estas enfermedades se han clasificado en dos tipos: Enfermedades autoinmunes sistémicas y específicas de órgano.
Las enfermedades autoinmunes sistémicas se presentan cuando existen reacciones del sistema inmune contra diferentes órganos o tejidos del cuerpo, mientras que las específicas de órgano involucran un órgano o tejido en particular.2
Estas enfermedades son un grupo heterogéneo de desórdenes con un gran rango extenso de síntomas clínicos. Individuos con la misma enfermedad autoinmune pueden presentar diferentes fenotipos clínicos y variabilidad en el curso de su enfermedad. A pesar de estas diferencias, muchas enfermedades autoinmunes, al parecer, comparten mecanismos inmunológicos para responder a los tratamientos con agentes terapéuticos similares. El diagnóstico de la mayoría de estas enfermedades es complicado porque la enfermedad puede presentar la ausencia de signos y síntomas. Además la falta de estándares o criterios validados dificultan el proceso en etapas tempranas de la enfermedad.2
Teniendo en cuenta la dificultad del diagnóstico y la falta de definición de mecanismos que permitan identificar estas patologías, este trabajo de investigación presenta un prototipo de sistema experto, que pretende ayudar en el proceso de diagnóstico de enfermedades autoinmunes específicas de órgano, este tipo de sistemas son programas diseñados para emular el trabajo de expertos en áreas específicas del conocimiento.3 Para su construcción se tomó como referencia Internist, un sistema experto médico que utiliza un método descriptivo para modelar el conocimiento y que permite encontrar las enfermedades que más se ajusten a los hallazgos encontrados en un paciente.4
Este sistema experto, previamente utilizado en proyectos como QMR (Quick Medical Reference), se integra junto al conocimiento clínico a través de un modelo ontológico, representación formal y explicita de una conceptualización compartida, que permite administrar los conceptos envueltos en el diagnóstico y sus relaciones, logrando así establecer un modelo conceptual que contempla las diferentes entidades y variables que participan en los procesos salud/enfermedad de este tipo.5-7 Para complementar la funcionalidad de diagnóstico del prototipo, el modelo conceptual se integra a un proceso de razonamiento mediante reglas declaradas en JESS, el motor de inferencia para Java.8
En primera instancia este artículo presenta la metodología utilizada para construir el sistema, haciendo énfasis en la forma de integrar el conocimiento clínico y el modelo descriptivo de Internist.9 Luego se muestran los resultados obtenidos, las interfaces ofrecidas al usuario y un ejemplo, con datos simulados, de cómo se trabajaría con el prototipo. Finalmente se presentan los trabajos futuros que nacen de este trabajo y las conclusiones obtenidas.
Materiales y métodos
La metodología utilizada para la creación del prototipo de sistema experto se basa en la propuesta por Milton.10 Dicha metodología, utilizada en el desarrollo de sistemas de administración de conocimiento en diferentes dominios, está compuesta por una serie de pasos divididos en 4 fases.11-13
Fase 1: Alcance y plan del proyecto
Para crear el prototipo se establecieron las siguientes tareas:
• Determinar fuentes de conocimiento
• Análisis del dominio
• Recolectar el conocimiento
• Formalizar el conocimiento
• Desarrollar interfaz para el prototipo
En esta fase del proyecto se abordaron las dos primeras tareas. Las fuentes identificadas fueron: los médicos encargados de realizar el diagnóstico, los cuales poseen el conocimiento fruto de su formación profesional y experiencia de forma tácita en su mente y debe ser capturado mediante entrevistas. Por otro lado se identificó el conocimiento que está en la literatura existente sobre enfermedades autoinmunes específicas de órgano, el cual está explícito en libros, revistas, artículos, etc., y debe ser recolectado a partir de revisión de literatura. En la tabla 1 se relacionan los tipos de conocimiento involucrados en el desarrollo del prototipo y sus fuentes.
El análisis del dominio se realizó a través de entrevistas a expertos sobre enfermedades autoinmunes, las cuales fueron construidas de acuerdo al modelo descriptivo que propone Internist.4 Las preguntas de la entrevista inicial sobre enfermedades autoinmunes fueron:
• ¿Qué es una enfermedad autoinmune?
• ¿Cuál es la diferencia entre una enfermedad autoinmune específica de órgano y una sistémica?
• ¿Cómo se identifican las enfermedades específicas de órgano?
• ¿Cómo se puede determinar si un paciente padece o no una Enfermedad?
• ¿Cómo se clasifican los síntomas?
• ¿Qué son los exámenes clínicos y paraclínicos?
Además se realizó una revisión de literatura, a partir de lo cual se escogieron las siguientes enfermedades como referencias para construir el prototipo:

Con base en esto se realizó una segunda entrevista, con el fin de determinar un conocimiento más específico para este grupo de enfermedades. Para cada enfermedad se preguntó:
• ¿Cuál es su definición?
• ¿Cuáles son sus síntomas?
• ¿De estos cuáles son determinantes y exclusivos?
• ¿Qué exámenes clínicos y paraclínicos se deben tener en cuenta?
• ¿Cómo se deben interpretar los resultados de los exámenes?
Fase 2: Captura inicial y modelado
Con base en el conocimiento adquirido en la fase anterior se da paso al proceso de captura y modelado inicial para ellos se formalizo modelo descriptivo utilizado por Internist, se estableció que este modelo trabaja con una base de conocimiento que comprende dos elementos fundamentales:4,14
• Manifestaciones
• Enfermedades
Las manifestaciones están compuestas por: historia clínica, síntomas, signos físicos y hallazgos de laboratorio los cuales asociados a una enfermedad definen su perfil. Las enfermedades se agrupan en categorías las cuales también tienen asociado un grupo de manifestaciones.
Cada manifestación define un conjunto de enfermedades o categorías por medio de una estructura de datos EVOCA, la cual registra para cada manifestación la lista de enfermedades en las que la manifestación tiene alguna posibilidad de ocurrencia. La fuerza o posibilidad, un entero entre 0 y 5, con la que la manifestación está vinculada a la enfermedad se registra en la estructura de datos. Esta relación modela la probabilidad de que una manifestación esté presente a causa de una determinada enfermedad.
Por otro lado cada enfermedad se asocia con una lista de manifestaciones y un estimativo de frecuencia de cada manifestación que es un número entre 1 y 5, esta relación tiene el nombre de MANIFIESTA, y modela la frecuencia con que una enfermedad ha sido causa de una manifestación a través del tiempo. Finalmente Internist propone la relación IMPORT, la cual es expresada por un valor entre 1 y 5 y modela la necesidad de que una manifestación sea parte del perfil de una enfermedad.4,14
Los valores para evocación, frecuencia e importancia para las manifestaciones de una enfermedad o categoría se establecen a través de observaciones históricas del comportamiento de las enfermedades, en hospitales y centros de salud. Debido a que son probabilidades y frecuencias, para efectos de este trabajo dichos valores se asignaran sin validación experta, lo cual no es un impedimento para construir el sistema de información porque el enfoque descriptivo permite separar los procesos, de la base de conocimiento. Esta base de conocimiento puede ser actualizada o corregida en cualquier momento, sin tener que cambiar el proceso de deducción del sistema.
A partir de esto se creó un modelo entidad relación que integra la arquitectura de Internist con los conceptos del prototipo construido en este trabajo.9
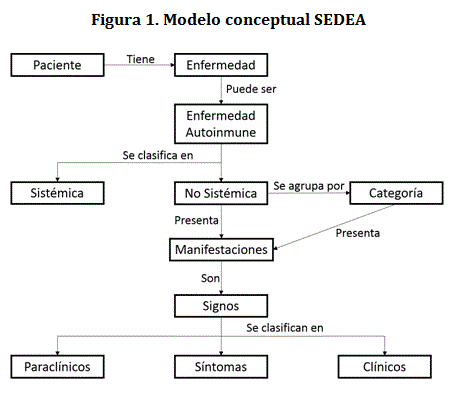
En este modelo se puede observar la entidad paciente que tiene una enfermedad, la cual puede ser de tipo autoinmune y a la vez puede ser de órgano. Esta enfermedad pertenece a una categoría. La enfermedad y su categoría presentan manifestaciones o signos que pueden ser paraclínicos, síntomas o clínicos.
A partir de lo anterior se construyó una ontología, en formato OWL con la herramienta Protégé.15 Este modelo de conocimiento, permite modelar y administrar el conocimiento que el prototipo requiere, además provee la estructura sobre la cual, el sistema debe realizar sus inferencias.
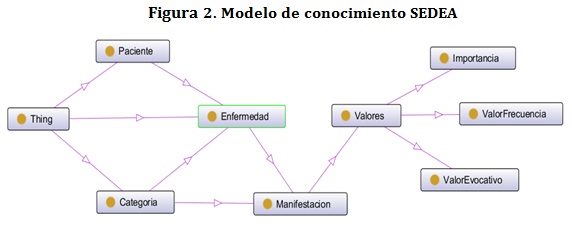
En el modelo ontológico propuesto, se puede observar la entidad paciente el cual tiene una enfermedad que pertenece a una determinada categoría. Tanto la categoría como la enfermedad causan manifestaciones, las cuales tienen asociados unos valores de evocación, frecuencia e importancia. De esta forma se determina que los datos que deben ser capturados de las fuentes expertas, corresponden a las enfermedades, categorías, manifestaciones, valores de evocación, frecuencia e importancia a partir de los cuales el sistema realizará su proceso de inferencia.
Continuando con el modelo descriptivo, el siguiente paso es determinar el diagnóstico de las enfermedades alternando dos formas deductivas:
Encadenamiento hacia adelante: Inicialmente se introducen los datos del paciente y sus síntomas, un síntoma ingresado evoca una categoría de enfermedad, y las enfermedades en esta categoría tienen el síntoma común, pero con eventuales diferencias en valor evocativo, frecuencia e importancia. De esta manera se origina un conjunto de enfermedades como posibles diagnósticos para el paciente. A medida que se ingresa un síntoma, este puede evocar una u otra categoría, y puede volver a evocar una categoría evocada por un síntoma anterior. Cuando se finaliza de ingresar los síntomas, el sistema procede a dar un puntaje a cada categoría evocada. Este puntaje establece un orden dentro de las categorías posibles, para proceder a caracterizar una de las varias enfermedades dentro de la categoría, por encadenamiento hacía atrás.
Para lograr que el sistema experto realice este proceso, aprovechando el modelo de conocimiento planteado, se definieron los siguientes hechos en JESS:
• Causa Valor: Establece el valor de causa de una categoría.
• Manifestación Categoría: Establece el valor de causa de una categoría a una a una categoría.

Además, fue necesario expresar una regla para agrupar las categorías determinando el valor de una categoría como causa para las manifestaciones seleccionadas. En JESS se expresa de la siguiente forma:

Como primer paso se llena la base de hechos con hechos Causa Valor, tomando todos las categorías de la base de conocimiento como las causas, cada una con valor 0. Posteriormente, de acuerdo a la selección de síntomas realizada por el usuario, se llenan los hechos Manifestación Categoría, especificando el síntoma, la categoría y el valor de la categoría como causa de la manifestación. Finalmente, con la regla de agrupación se establece el puntaje o valor de cada categoría o causa de acuerdo a las manifestaciones seleccionadas, obteniendo una lista ordenada, por peso y por categorías.
Encadenamiento hacia atrás: La estructura de representación de la base de conocimiento, permite que las enfermedades de la categoría con mayor puntaje estén dispuestas para un proceso de encadenamiento hacía atrás. Para realizar este procedimiento, se pregunta al usuario los síntomas particulares de cada enfermedad de la categoría, para ir afirmando la hipótesis o enfermedad. Por cada respuesta afirmativa del síntoma, se agrega un hecho Manifestación Enfermedad, el cual establece un valor para cada síntoma como causa de la enfermedad. En JESS se expresa así:8

Posteriormente, se aplica la regla Agrupar Enfermedad para determinar el valor para cada enfermedad de la categoría, obteniendo nuevamente un listado ordenado de acuerdo al puntaje. Esta regla se expresa en JESS de la siguiente manera:

Si por encadenamiento hacía atrás no puede concluirse ninguna enfermedad de la categoría, se procede a realizar el mismo proceso con las categorías subsiguientes.
El puntaje para cada categoría y enfermedad, en los procesos de encadenamiento hacía adelante y hacía atrás, se calcula de acuerdo a la siguiente expresión:15,16
S(Hi) = P(Hi) - N(Hi)
Donde S es puntaje, P es el indicativo de síntomas comunes encontrados y está dado por:15,16
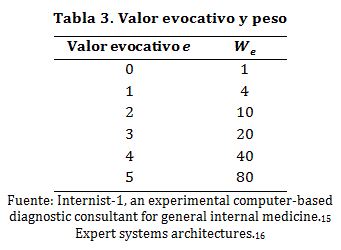
P(Hi) = Σ We(Ej)
Donde Ej representa la fuerza o valor evocativo del síntoma sub-j encontrado en la categoría y We está dado por la tabla:
N, es el indicativo de síntomas comunes no encontrados en la categoría y viene dada en los trabajos.13,14
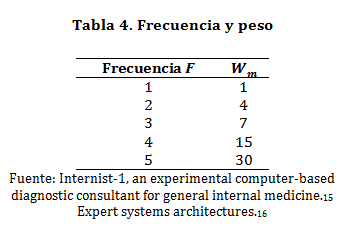
N(Hi)=Σ WmFj + WuIj
Donde Fj representa la frecuencia e Ij la importancia del síntoma sub-j no encontrado en la categoría, Wm y Wu están dados por la tabla:
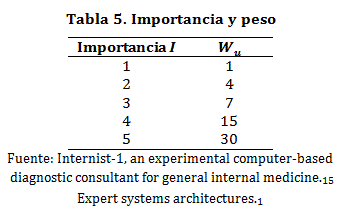
Para el prototipo, los valores de las tablas descritas anteriormente se almacenan en la base de conocimiento y la formula se expresa mediante JESS mediante la siguiente función:

Fase 3: Captura detallada y modelado
En el proceso de captura detallada se consultaron las fuentes expertas con el fin de determinar las categorías y manifestaciones para las enfermedades listadas en la Fase 1.
Con el fin de realizar pruebas al prototipo construido se asignaron valores para evocación, importancia y frecuencia para las diferentes manifestaciones, los cuales deben ser validados en un trabajo posterior, después de realizar la respectiva investigación y observación del comportamiento de este tipo de enfermedades en un determinado ambiente, esto con el fin de dotar al prototipo de valores confiables que permitan obtener diagnósticos reales.
Estas enfermedades, categorías, manifestaciones y valores constituyen la base de conocimiento sobre la cual trabajará el prototipo.
Por otro lado se estableció el proceso que debe llevar a cabo el prototipo para realizar el diagnóstico de una enfermedad, el cual determina el flujo de eventos e interfaces que requiere el prototipo.
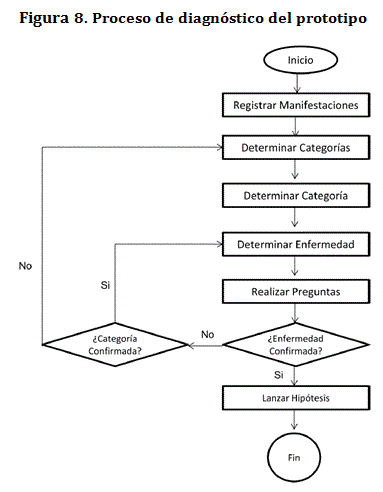
En primera instancia se solicita ingresar las manifestaciones encontradas en el paciente, con las cuales se determinan las categorías en donde se puede encontrar la enfermedad. Se escoge la enfermedad de mayor puntaje dentro de la categoría de mayor puntaje y se realizan preguntas para confirmar la enfermedad, si se realiza la confirmación se lanza la hipótesis. De lo contrario, se repite el proceso con la siguiente enfermedad en puntaje de la categoría escogida. Si las enfermedades de la categoría escogida se acaban sin obtener un diagnóstico, se prosigue con las enfermedades de la categoría que en puntaje le sigue a la escogida inicialmente. Este proceso se repite mientras no se haya lanzado una hipótesis o existan categorías y enfermedades candidatas sin evaluar. De la misma forma el usuario final puede rechazar la hipótesis, en ese caso el sistema debe descartar e intentar buscar otra que satisfaga las condiciones dadas.
Fase 4: Compartir el conocimiento almacenado
Con el fin de permitir que la información almacenada en la base de conocimiento del sistema pueda ser manipulada por los usuarios, se construyeron interfaces que permiten administrar categorías, enfermedades y manifestaciones:
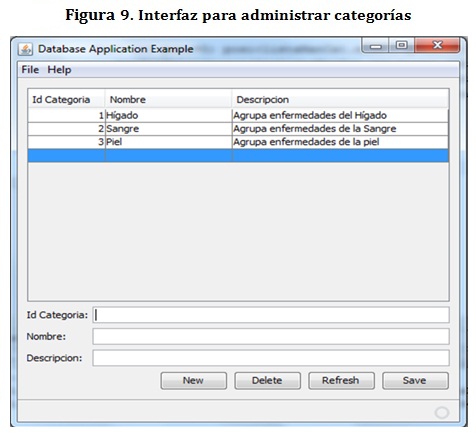
Además de los datos que identifican las categorías, enfermedades o manifestaciones, se requiere la descripción la cual puede ofrecer una mejor explicación a la hora de mostrar el diagnóstico.
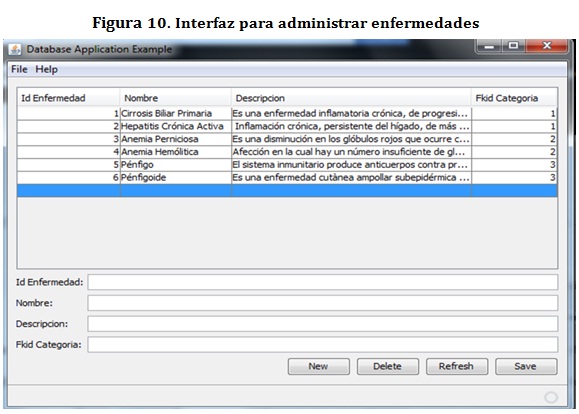
En el caso de las enfermedades, además de la descripción, se debe especificar la categoría a la cual pertenece como se puede apreciar en el gráfico anterior.
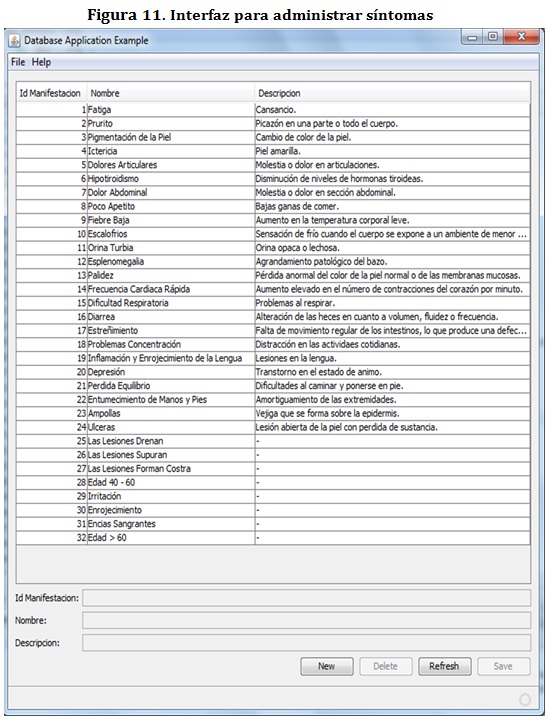
Por otro lado, se implementaron las interfaces que permitan manipular los valores de evocación, importancia y frecuencia para manifestación/categoría y manifestación/enfermedad:
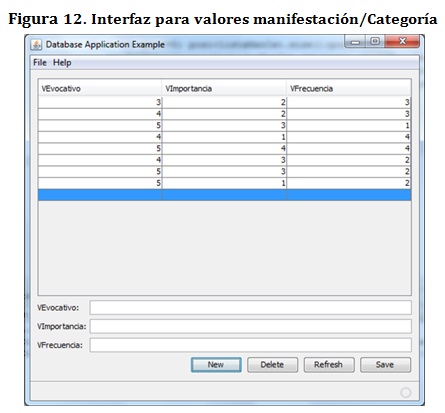
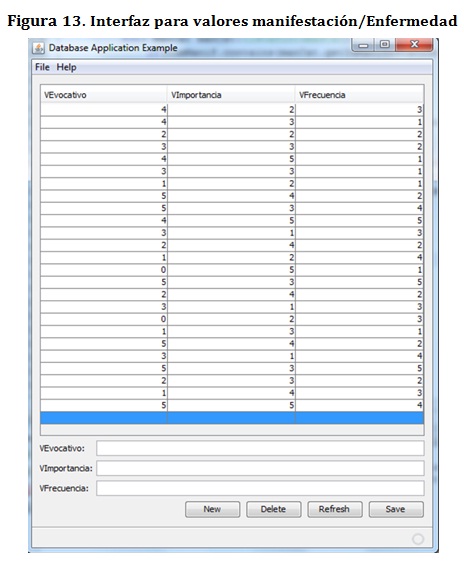
Estas interfaces ofrecen un acceso directo a la base de conocimiento sobre el cual trabaja el prototipo, esta es una gran ventaja ya que además de dotar al sistema de flexibilidad para manipular el conocimiento, ofrecen extensibilidad hacía otros dominios del diagnóstico médico, al permitir la inserción de nuevas categorías, síntomas, enfermedades y relaciones entre ellas.
Resultados y discusión
Se construyó un prototipo de sistema experto para el diagnóstico de enfermedades específicas de órgano. Este prototipo se obtuvo después de 4 fases de trabajo, partiendo desde la determinación de alcance y planeación, siguiendo por la captura de conocimiento, modelado del proceso de deducción y terminando con la creación de interfaces que permitan la interacción con el usuario final. En este proceso, se tuvieron en cuenta a los expertos en diagnósticos médicos para enfermedades autoinmunes, literatura del dominio y la arquitectura de Internist.
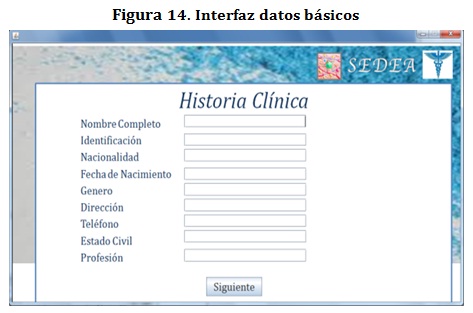
A continuación, se presentará un flujo de ejecución del prototipo. Como primer paso, el sistema, pide unos datos básicos del paciente: nombre, identificación, nacionalidad, fecha de nacimiento, género, dirección, teléfono, estado civil y profesión a través de la siguiente interfaz:
En el siguiente paso, se presentan los síntomas generales que identifican a las diferentes categorías. Los cuales, al ser seleccionados son la entrada para el encadenamiento hacía adelante explicado anteriormente(Figura 15):
Con base en la selección anterior, se presenta un listado de síntomas específicos que permiten concluir, a través del encadenamiento hacía atrás, la enfermedad del diagnóstico. Cabe mencionar, que en este ejemplo, al trabajar con valores de evocación, importancia y frecuencia simulados, el resultado puede variar de lo que en un escenario real se debería obtener(Figura 16).
Finalmente, se presenta el diagnóstico obtenido con su respectiva descripción (Figura 17).
En principio el prototipo trabaja con un subgrupo de enfermedades específicas de órgano, el cual puede fácilmente escalar a un mayor número de enfermedades gracias a las interfaces construida que permiten la inserción, eliminación y actualización de datos sobre la base de conocimientos. En ese sentido, se puede resaltar que como resultado de este trabajo, se tiene un sistema que puede trabajar con conocimiento médico en diferentes dominios ya que el modelo descriptivo sobre el cual trabaja, separa los datos de los procesos de deducción.
Por esta misma razón el hecho de asignar valores de evocación, frecuencia e importancia a las diferentes manifestaciones sin validarlos con expertos no afecta el objetivo de este trabajo, en cuanto a la construcción y funcionamiento del sistema de información. Será tema de una investigación posterior la validación e ingreso de dichos valores, para obtener diagnósticos acordes a la realidad.
Trabajo futuro
Como se ha mencionado a lo largo de este documento, el primer trabajo futuro es la validación con los expertos de los datos de evocación, frecuencia e importancia sobre los cuales trabaja el sistema. Dicho trabajo no se realizó en esta oportunidad ya que estos valores, son el resultado de trabajos de observación de eventos históricos en hospitales y centros de salud, donde llegan las personas con este tipo de enfermedades.
Por otro lado, se plantea alimentar la base de conocimientos con datos en diferentes dominios del diagnóstico médico con el fin de observar el comportamiento del sistema de información y la arquitectura utilizada.
Conclusiones
Los sistemas expertos de diagnóstico médico han ido cobrando importancia a través del tiempo en el trabajo de los profesionales de la salud y e incluso en la vida cotidiana de las personas. Este tipo de sistemas, dada la complejidad y amplitud del dominio, tienden a crearse de forma cerrada para un grupo de enfermedades específicas. En este sentido, la implementación de modelos descriptivos para capturar y formalizar el conocimiento médico hace posible la creación de sistemas de información cuyo proceso se separa de los datos, lo cual permite que estos sistemas tengan una gran flexibilidad para trabajar en diferentes dominios, el cambio de un dominio a otro solo necesita de una actualización de la base de conocimientos, esta es la gran ventaja que ofrece este tipo de implementaciones.
En el caso particular de este trabajo, se logró integrar el modelo descriptivo, ofrecido por Internist, con el conocimiento clínico a través de una ontología que modela la base de conocimiento del sistema a través de sus conceptos y relaciones, lo cual permite separar los diferentes componentes de conocimiento que integran un diagnóstico. Por otro lado, para obtener el diagnóstico o integrar los componentes mencionados anteriormente, se aprovechó el motor de inferencia de Java, JESS, en el cual a partir de reglas se codifico el núcleo que permite al sistema dar una respuesta al problema planteado.
En cuanto al funcionamiento del prototipo, como se pudo evidenciar en la sección de resultados, se creó una aplicación que cuenta con las interfaces necesarias para, ingresar datos o síntomas de un paciente, interactuar con el usuario llevándolo por los diferentes procesos de razonamiento y mostrar los resultados finales del proceso, a través de elementos que permiten ver sus diferentes hipótesis o sugerencias de diagnóstico.
En el caso particular de Internist, se puede concluir que simplifica el proceso de construcción del sistema experto ya que es de gran utilidad para determinar la información requerida, en la construcción de la base de conocimientos.
Referencias
1. Schwab M. Ed. Encyclopedia of Cancer. Berlin, Heidelberg: Springer Berlin Heidelberg; 2012. [ Links ]
2. National Institutes of Health, Autoimmune Diseases Coordinating Committee. US: Department of Health and Human Services. (Internet). Maryland: NIH (2013 Oct 17; citado 2013 May 15). Disponible en: http://www.nih.gov/about/contact.htm [ Links ]
3. Lemaire JB, Schaefer JP, Martin LA, Faris P, Ainslie MD, Hull RD. Effectiveness of the quick medical reference as a diagnostic tool. CMAJ.1999. [ Links ]
4. Gruber TR. A translation approach to portable ontology specifications. Knowl. Acquis. 1993;5(1):199-220. [ Links ]
5. Kendal S, Creen M. An introduction to knowledge engineering. Springer Link, London; 2007. [ Links ]
6. Todd BS. An introduction to expert Systems. Technical Monograph; 1992. [ Links ]
7. Miller RA. Quick medical reference. Open Clinical; Londres, 2004. [ Links ]
8. "Jess, the Rule Engine for the Java Platform." Online. Available: http://herzberg.ca.sandia.gov/. (Accessed: 16-Sep-2014) [ Links ]
9. Myers JD. The background of Internist - I and QMR. A History of medical informatics. ACM Press, 1990. [ Links ]
10. Milton NR. Knowledge acquisition in practice. A step-by-step guide. Springer; 2007. [ Links ]
11. Luo X, Miao Ch, Jennings N, He M, Shen Z, Zhang M. KEMNAD: A knowledge engineering methodology for negotiating agent development. Computational intelligence. 2012;28(1). [ Links ]
12. Cardillo E, Serafini L, Tamilin A. A hybrid methodology for consumer-oriented Halthcare 1Knwoledge Acquisition. AIME - International Conference on Knowledge Representation for Health - Care. LNCS, Springer-Verlag Berlin, Heidelberg; 2010. [ Links ]
13. Angeli C. Diagnostic expert systems: From expert´s knowledge to real - Time systems. Advanced knowledge based systems: Model, Applications & Research. 2010. [ Links ]
14. "Protégé." (Online). Available: http://protege.standford.edu/. (Accessed: 16-Sep-2014) [ Links ]
15. Miller RA, Pople HE, Myers JD. INTERNIST-1, An Experimental computer-based diagnostic consultant for general internal medicine. N Engl J Med. 1982;307(8):468-76. [ Links ]
16. Myers JD, Pople HE. INTERNIST-I. In: Johnson L, Keravnou ET. Expert Systems Architectures. 1988:64-77. [ Links ]














