Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Ciencias Químico - Farmacéuticas
Print version ISSN 0034-7418On-line version ISSN 1909-6356
Rev. colomb. cienc. quim. farm. vol.36 no.2 Bogotá July/Dec. 2007
Artículo Técnico
Diseño de un modelo bioinformática para la detección, identificación y clasificación de genes codificantes de ß-lactamasas, como herramienta para el cruce de datos moleculares con datos clínicos
Bioinformatics model design for the detection, identification and classification of b-lactamases genes, as a tool for crossing molecular data with clinical data
Emiliano Barreto Hernández1, María Teresa Reguero Reza2
1, 2 Instituto de Biotecnología, Edificio Manuel Ancízar, Universidad Nacional de Colombia, Bogotá, D.C., Colombia. 1 Correo electrónico: ebarretoh@unal.edu.co
Recibido para evaluación: 12 de diciembre de 2006 Aceptado para publicación: 20 de agosto de 2007
RESUMEN
El seguimiento epidemiológico y farmacológico de la resistencia a antibióticos ß-lactámicos mediada por la presencia de ß-lactamasas requiere identificación precisa, lo cual es posible mediante la secuencia del gen, que es identificado con herramientas bioinformáticas que se incorporan a sistemas de información. Para implementar un prototipo de sistema de información que permite clasificar secuencias de genes de ß-lactamasas producidas por microorganismos resistentes aislados en hospitales y realizar su cruce con los datos clínicos, se obtuvieron las secuencias de ß-lactamasas reportadas a escala mundial en la base de datos Uniprot utilizando el sistema de recuperación de secuencias (SRS) del Instituto Europeo de Bioinformática (EBI), se diseñó un sistema de datos moleculares y clínicos usando "Unified Modeling Language" (UML), y se generó un modelo implementado en un servidor con sistema operativo UNIX, gestor de bases de datos MySQL para realizar la creación de la base de datos y lenguajes de programación Perl y PHP para implementar programas y cruzar datos. El sistema de información BLA_ID_CLINIC permite hacer el cruce de datos moleculares y clínicos, de tal forma que se puedan identificar las ß-lactamasas de microorganismos resistentes en hospitales y hacer un seguimiento del manejo de los antibióticos y el comportamiento epidemiológico, modelo bioinformático permanentemente actualizado, disponible a través de Internet en http://bioinf.ibun.unal.edu.co/BLA_ID_CLINIC.
Palabras clave: Bioinformática, beta-lactamasas, identificación, sistema de información.
SUMMARY
The epidemiology and pharmacology of the ß-lactamic antibiotics resistance at hospital level mediate by the presence of ß-lactamasas requires of their precise identification, possible through the gene sequence that can be identified using bioinformatics tools and incorporated into the information systems. Prototype of information system BAL_ID_CLINIC was implemented for allowing genes sequences classification of b-lactamases produced by isolated resistant microorganisms in hospitals and their crossing with the clinical data. The information system was development using Unified Modeling Language UML and the prototype was implemented in an Unix server, using MySQL management system for data base building and Perl and PHP as a programming languages for writing scripts that obtain cross relationship between the different kind of data inside the system and the result report presentation. BLA_ID_CLINIC allows crossing of molecular and clinical data. It can identify ß-lactamasas produced by resistant microorganisms isolated in hospital patients and can give information related with the antibiotics management and the epidemiologic behavior. The bioinformatics prototype BLA_ID_CLINIC is permanently updated and available through Internet in http://bioinf.ibun.unal.edu.co/BLA_ID_CLINIC.
Key words: Bioinformatics, beta-lactamases, classification, information system.
INTRODUCCIÓN
La gran incidencia de infecciones nosocomiales, la importancia de las enterobacterias como los agentes causales más frecuentes y el surgimiento de cepas resistentes a los antibióticos, son factores que inciden en uno de los mayores problemas de la medicina actual y del futuro: la resistencia bacteriana. Mundialmente, el índice de infecciones intrahospitalarias (IIH) es de 7,4%, en promedio; el 35% son de origen exógeno y de este el 80% corresponden a contaminaciones cruzadas. El promedio de días estancia por esta causa oscila entre 7 y 10, aunque, dependiendo del servicio, puede llegar a ser de 20 o más días. La mortalidad general puede ir de 0,5% a 2% y se estima como causa directa de muerte en pacientes hospitalizados entre el 3 y el 4% y como causa asociada el 3% (1). Este problema clínico se ha convertido en un factor de importancia epidemiológica y socioeconómica tal, que fue declarado en 1999 por la Organización Mundial de la Salud como un problema de salud pública.
En el ámbito local, en Bogotá desde el año 2000 hasta el 2003 se notificaron anualmente de 8.000 a 12.000 infecciones intrahospitalarias y han sido identificadas, por certificados de defunción, entre 300 y 400 muertes asociadas a IIH (3,75% a 4%); el índice global de IIH de la ciudad oscila entre 2,4 y 2,5 por cada 100 egresos hospitalarios (1).
La resistencia de los microorganismos a los antibióticos es la consecuencia fenotípica de la diseminación de genes portados por elementos genéticos transferibles y de la presión selectiva de cepas resistentes por el uso inadecuado de antibióticos, las cuales disminuyen la efectividad de los tratamientos y generan en ocasiones fracaso terapéutico (2).
Desde la introducción de la penicilina, hace más de 50 años, uno de los grupos de antibióticos más utilizados han sido los ß-lactámicos. El uso de estos antibióticos no ha escapado a la pérdida de sensibilidad de parte de los microorganismos, principalmente porque éstos expresan diferentes tipos de enzimas, denominadas ß-lactamasas, que les permiten hidrolizar el anillo ß-lactámico presente en estos fármacos. La aparición de resistencia a los antibióticos, atribuible a presencia de las ß-lactamasas, ha propiciado que se intensifique la investigación y el desarrollo de modificaciones químicas en la estructura de los antibióticos ß-lactámicos, y que se generen varios subgrupos: penicilinas, cefalosporinas, inhibidores de ß-lactamasas, carbapenémicos y monobactámicos.
Desde el punto de vista hospitalario, la aparición de cepas resistentes a los antibióticos ß-lactámicos implica que el comité de infecciones de la respectiva institución hospitalaria cuente con información no sólo acerca del microorganismo, sino también del tipo de ß-lactamasa involucrada, de tal forma que se puedan plantear estrategias terapéuticas alternativas que permitan controlar la infección y limite el incremento de la resistencia. Con esta información disponible sería posible hacer un seguimiento epidemiológico, detectar la forma en que se van diseminando los microorganismos causantes de los brotes, el mecanismo utilizado para transmitir la resistencia, que en muchos casos puede ser horizontal y no clonal, y la racionalidad con que han sido utilizados los antibióticos en el tratamiento de los diferentes casos.
Para adelantar la caracterización del microorganismo y el tipo de ß-lactamasa que biosintetiza se ha recurrido, por su elevada especificidad, a métodos moleculares que identifican marcadores moleculares específicos (3). Estos marcadores moleculares representan un gran volumen de secuencias (12.430 secuencias (4)), por lo general de ADN que se encuentran, en su mayoría, reportadas en bases de datos como el EMBL o GenBank, o en algunos casos en las publicaciones de los investigadores que se especializan en el estudio de este tipo de microorganismos (8.626 reportes en Medline (5)). Este hecho le dificulta a un comité de infecciones tener acceso rápido a métodos que permitan comparar las características genéticas del microorganismo que está evaluando con los reportados tanto en las bases de datos internacionales, como los datos que se pueden generar en el entorno local, y es aún más difícil evaluar el comportamiento del microorganismo frente a determinado antibiótico.
En general, los países desarrollados han implementado sistemas informáticos hospitalarios que permiten hacer un seguimiento del comportamiento de la infección intrahospitalaria y su relación con el uso de antibióticos, y han desarrollado sistemas computarizados que apoyan a los médicos en la selección de antibióticos (6) o en la realización de estudios que utilizan bases de datos locales e inferencias epidemiológicas dentro de las instituciones hospitalarias (7), o a comparar estas estadísticas con las de otros países (8).
Es evidente que este tipo de problema mundial es más crítico en Colombia, donde hay dificultades de caracterización, registro y sistematización permanente de los casos de IIH, en especial aquellos en donde se presenta resistencia a los antibióticos. En este aspecto, el Grupo para el Control de la Resistencia Antimicrobiana en Bogotá (Grebo (9)) ha cumplido una gran labor, al coordinar esfuerzos entre múltiples entidades hospitalarias locales estableciendo, como medio para compartir, estandarizar, sistematizar y analizar los datos, el programa Whonet (10), lo cual ha permitido hacer un seguimiento sistemático de la resistencia de los microorganismos en el entorno hospitalario. Sin embargo, a causa de las limitaciones en la identificación precisa de los diferentes tipos de ß-lactamasas involucradas en la resistencia, el seguimiento de los agentes causales, no es preciso.
El desarrollo de modelos sistematizados para el registro de datos clínicos, que utiliza estándares como los propuestos por la Organización Mundial de la Salud (OMS) por medio del software Whonet, combinado con estrategias adecuadas de caracterización molecular, ayuda a mejorar la precisión en el seguimiento de la resistencia frente a los antibióticos ß-lactámicos y permite identificar los tipos de agentes responsables de la infección, el tipo de resistencia, sus mecanismos de diseminación y la forma como deben manejarse racionalmente los agentes antiinfecciosos para poder controlar y, más aún, para prevenir la aparición de resistencias a éstos.
En general, puede decirse que en muchas de las instituciones hospitalarias privadas y del sistema estatal se cuenta con herramientas de cómputo que permiten sistematizar los datos asociados con las historias clínicas de los pacientes en las que se encuentra información acerca de la resistencia y de los microorganismos causantes de infección intrahospitalaria, pero es difícil encontrar, en el caso de las resistencias a antibióticos ß-lactámicos, el tipo específico de enzima causante, y más difícil aún es correlacionar estos datos para hacer evaluaciones epidemiológicas y de manejo adecuado de los antibióticos. Se han realizado trabajos de recopilación y análisis de datos relacionados con la resistencia bacteriana intrahospitalaria (11, 12) y un sistema que permite guiar a los investigadores en la caracterización molecular de un grupo particular de ß-lactamasas (13), sin que existan reportes de modelos que integren y crucen los dos tipos de datos.
Con base en el análisis anterior, es evidente la necesidad de desarrollar una herramienta bioinformática que permita:
-
Sistematizar los datos locales asociados con la aparición de microorganismos resistentes a antibióticos ß-lactámicos y sus características moleculares.
-
Sistematizar las características moleculares de ß-lactamasas reportadas en el mundo.
-
Jerarquización y sistematizar los datos clínicos y farmacológicos asociados con datos moleculares de algunos aislamientos obtenidos en el entorno hospitalario por el Grupo de Epidemiología Molecular del Instituto de Biotecnología de la Universidad Nacional de Colombia, Sede Bogotá, utilizando como referencia las indicaciones de la OMS por medio de su programa Whonet.
-
Comparar los datos moleculares obtenidos localmente con las bases de datos internacionales para hacer su identificación.
Un prototipo de sistema bioinformático con estas características constituye una herramienta de apoyo importante para los comités de infecciones de las instituciones hospitalarias, en el seguimiento del manejo y comportamiento de la resistencia de los antibióticos, ya que en la medida en que se acumulen los datos con el paso del tiempo, éstos permitirán hacer análisis tanto prospectivos como retrospectivos del comportamiento epidemiológico de la resistencia bacteriana, mediada por ß-lactamasas, en el ámbito hospitalario. Por tratarse de un problema multifactorial, el modelo propuesto pone a disposición del medio una parte importante de los datos sistematizados y jerarquizados asociados con la resistencia que presentan los microorganismos productores de ß-lactamasas, la cual se puede cruzar con las demás fuentes de datos disponibles, de tal forma que se puede llegar a tener un entendimiento integral del problema y a generar bases locales que agilicen y fortalezcan la toma de decisiones.
Este trabajo presenta la implementación de una herramienta bioinformática que permite comparar de la secuencia de una posible ß-lactamasa aislada de un microorganismo resistente con las ya reportadas, predecir su clasificación y almacenar los datos clínicos de resistencia frente a antibióticos y de localización del aislamiento, para que sirvan como base futura en el seguimiento epidemiológico y en el manejo racional de los antibióticos en los ámbitos nacional e internacional.
METODOLOGIA
Se basa en la estrategia para el diseño de sistemas de información y comprende cuatro fases fundamentales:
Búsqueda de secuencias
Se realizó la búsqueda de datos por medio del Sistema de Recuperación de Secuencias (SRS) implementado por el Instituto Europeo de Bioinformática (EBI) (10), el cual realiza la recuperación de secuencias y otros datos biológicos mediante un sistema indexado que permite hacer el cruce simultáneo de varias bases de datos y responde a consultas complejas.
Organización de las secuencias
Las secuencias fueron jerarquizadas con la finalidad de facilitar el diseño de una base de datos para almacenar estas y otros datos biológicos asociados, útiles para mantener la referencia entre la base de datos Uniprot y los datos clínicos almacenados localmente.
Selección de los datos clínicos
Tomando en cuenta las referencias bibliográficas, con la asesoría de expertos de las áreas clínica y epidemiológica, se seleccionó un conjunto de datos clínicos utilizando como referencia los campos incluidos en la base de datos del software Whonet (10). Estos datos se correlacionaron con el tipo de microorganismo, la clase de ß-lactamasa producida, su resistencia frente a los antibióticos ß-lactámicos, terapias previas y la localización fuente del aislamiento, entre otros.
Modelamiento de los datos moleculares y clínicos
Con los datos seleccionados se elaboró un modelo utilizando Unified Modeling Language UML (14), que permite cruzar datos moleculares y clínicos, de tal forma que se pueden caracterizar las ß-lactamasas de microorganismos intrahospitalarios resistentes y hacer un seguimiento del manejo de los antibióticos y el comportamiento epidemiológico. El sistema de información BLA_ID_CLINIC fue implementado en un servidor SUN Z40 del grupo de Bioinformática del Instituto de Biotecnología de la Universidad Nacional de Colombia, con sistema operativo Linux SUSE 10, servidor web Apache versión 2.0; PHP versión 4.3.4; MySQL versión 4.0.18 y lenguaje Perl versión 5.8.3 y está disponible en Internet en la dirección: http://bioinf.ibun.unal.edu.co/BLA_ID_CLINIC/.
RESULTADOS
Búsqueda de las secuencias
Teniendo en cuenta que la clasificación de las ß-lactamasas está basada en los cambios en la secuencia de la proteína, la base de datos Uniprot integra las principales bases de datos de secuencias de proteínas tales como Swissprot, Trembl y PIR, entre otras. De Uniprot se obtienen las secuencias de aminoácidos y las anotaciones relativas a las ß-lactamasas que se han identificado, secuenciado y documentado hasta el momento.
La consulta a esta base de datos se realizó utilizando del Sequence Retrive System SRS del Instituto Europeo de Bioinformática (4), restringida solo a aquellos registros que contenían la secuencia de caracteres: "beta" y "lactamase", en cualquier parte del registro y además la palabra "bacteria", con el propósito de recuperar sólo aquellas secuencias de ß-lactamasas que han sido descritas en bacterias. Esta consulta dio como resultado un total de 1.534 registros.
En la búsqueda de datos sistematizados de ß-lactamasas y con el propósito de encontrar secuencias que faciliten su identificación, se localizó en Internet el sitio web "Amino Acid Sequences for TEM, SHV and OXA Extended-Spectrum and Inhibitor Resistant β-Lactamases" (15), que intenta estandarizar la nomenclatura ß-lactamasas manteniendo en tablas actualizadas las secuencias tipo y los nombres de las ß-lactamasas de los grupos TEM, SHV and OXA clasificadas hasta el momento y cubre los tipos de estas enzimas más diseminados hasta ahora. De este sitio en Internet se descargaron las tablas, en formato texto, para utilizarlas como referencia para la identificación, por tratarse de secuencias tipo de ß-lactamasas ya identificadas. Así es posible comparar una secuencia problema, obtenida de un microorganismo resistente a los antibióticos ß-lactámicos, con las secuencias que se encuentran reportadas en estas tablas, para hacer su identificación.
Organización de las secuencias
Para recuperar las 1.534 secuencias de ß-lactamasas fue necesario seleccionar los campos, dentro de la base de datos Uniprot, que pudieran ser útiles para realizar el diseño del repositorio local de estos datos, parte fundamental del sistema de información en desarrollo. Fueron seleccionados los campos de identificación (EntryName) de la secuencia, el nombre del microorganismo (Species), la fecha en que fue reportada por última vez esta secuencia (DateSeqUpate), las referencias bibliográficas (MedlineID), la descripción de la secuencia (Description), el nombre del gen (GeneName) y sus sinónimos (Synonym) y desde luego la secuencia en formato Fasta (Sequence), como un subconjunto de campos que permiten fácilmente hacer seguimiento de la secuencia dentro de otras bases de datos, dentro de la bibliografía asociada a ella y de su clasificación como ß-lactamasa. En estas circunstancias se configuró el SRS para que extrajeran de Uniprot los datos almacenados en los campos mencionados previamente para los 1.534 registros y se pudiera generar así una tabla de texto plano llamada "wgetbla.txt", que se almacenó localmente.
Selección de los datos clínicos
Con el propósito de desarrollar un prototipo de sistema de información que integrara la información molecular de las ß-lactamasas con la información clínica relacionada con ellas, se seleccionó un conjunto de datos clínicos (Tabla 1), utilizando como referencia los campos incluidos en la base de datos del software Whonet (10). éstos se pueden dividir en cinco grupos así: Localización: permite geoposicionar la muestra de tal forma que sea fácil hacer un seguimiento de cualquier otro dato asociándolo al lugar donde se detecta la resistencia. Demografía del paciente: incluye los datos mínimos que permiten identificar y cualificar al paciente, de tal forma que se pueden correlacionar con ciertos comportamientos de los procesos infecciosos y sobre todo con su respuesta frente a los fármacos utilizados en los tratamientos respectivos, como los antibióticos. Información clínica: hace referencia a los campos relacionados con las causas iniciales de tratamiento del paciente, y los sitúa en el tiempo de la infección, la aparición de la resistencia, su tipo y el sitio. Resultados de laboratorio: estos campos permiten vincular la identificación del agente causante, el tipo de resistencia y especialmente del antibiograma realizado por métodos automatizados (discos de difusión, MIC o ETest®) o procedimientos manuales estandarizados en laboratorio responsable. Información farmacológica: los campos incluidos relacionan los tratamientos farmacológicos dados al paciente durante su tiempo de tratamiento; el tipo de antibiótico, la dosis, el tiempo de tratamiento y el resultado de éste, son correlacionados con la identificación precisa de la ß-lactamasa causante de la resistencia, lo que permite hacer seguimiento de la efectividad de los esquemas de tratamiento a través del tiempo.

Modelamiento de los datos moleculares y clínicos
Se diseñó el diagrama de casos de uso para el sistema BLA_ID_CLINIC (Figura 1) en el cual, tomando en consideración el punto de vista de un usuario, se definieron tres módulos que integran los datos moleculares y los datos clínicos, permiten la clasificación de la ß-lactamasa usando métodos bioinformáticos, reportan los datos moleculares y clínicos y, lo más importante, permiten recuperar los datos que facilitan el seguimiento de las ß-lactamasas a varios niveles, como se describe a continuación:
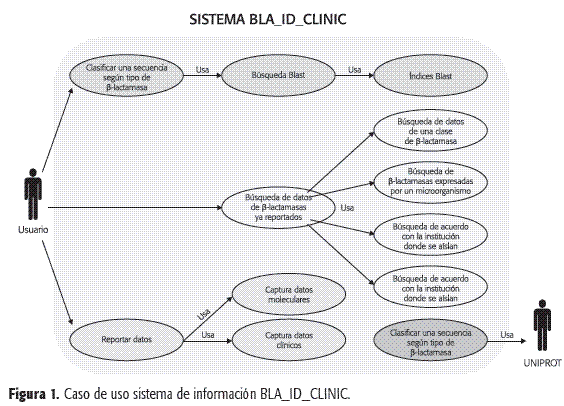
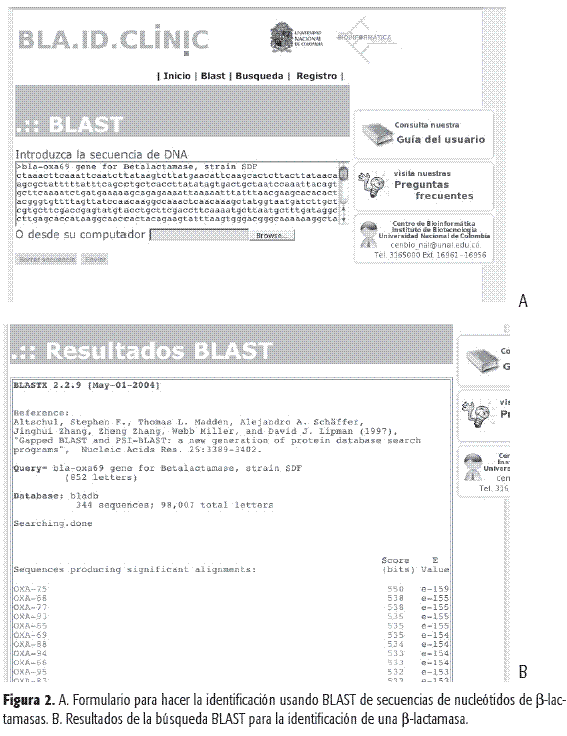
Módulo 1. Identificación de secuencias. Este módulo le permite al usuario introducir una secuencia de nucleótidos de una ß-lactamasa que quiera identificar (Figura 2A), la cual es comparada con la base de datos local de secuencias provenientes tanto de Uniprot, como de las que hayan sido aisladas y reportadas localmente y registradas directamente en el sistema mediante el módulo de reporte de datos, utilizando el programa Blast del Nacional Center for Biotechnology Information (NCBI) (16). Como se observa en la Figura 2B, el usuario recibe un listado en el cual se presenta la clasificación y el origen de las secuencias que pueden estar relacionadas con la secuencia que se va a clasificar, ordenadas por similaridad, a partir de la cual la clasificación final asignada a la secuencia corresponde a una decisión del investigador.
Módulo 2. Búsqueda de datos de ß-lactamasa. Como se ve en la figura 1, este módulo usa procedimientos que, desde el punto de vista de un usuario, van dirigidos a la búsqueda de reportes de ß-lactamasas agrupadas según: su clasificación; el microorganismo que las produce; tipo de ß-lactamasas reportadas para una determinada institución clasificadas y ordenadas de acuerdo con la localización del paciente dentro del hospital y tipo de ß-lactamasas asociadas a la resistencia de determinado antibiótico. Las consultas se realizan seleccionando la opción correspondiente en el formulario implementado para tal propósito (Figura 3A). Una vez realizada la consulta, los datos se presentan como un listado. Si el usuario selecciona acceder a datos referentes a un tipo de ß-lactamasa, el sistema buscará y presentará tanto las secuencias reportadas en Uniprot, como las que se hayan secuenciado e identificado localmente correspondientes al tipo seleccionado (Figura 3B).

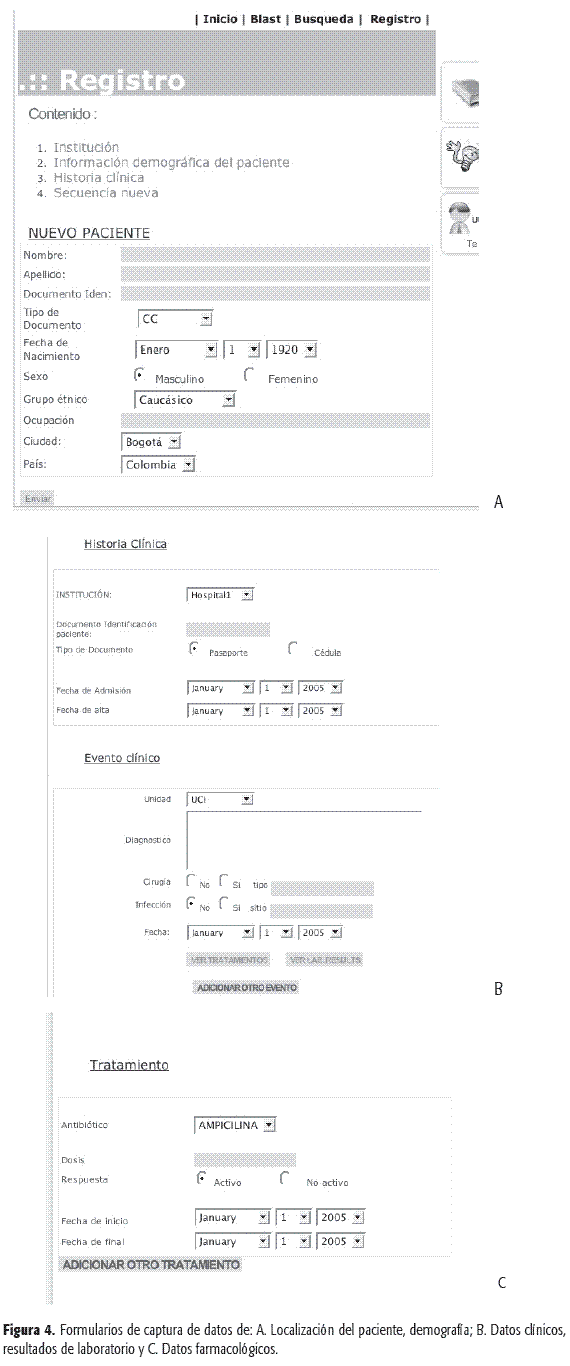
Módulo 3. Reporte de datos de ß-lactamasas. Si se toma en cuenta que uno de los mayores problemas en nuestro medio, para el seguimiento de la aparición de la resistencia bacteriana, es la falta de sistemas que permitan documentar los datos relativos a ésta, se desarrolló un conjunto de formularios para realizar el registro de la información asociada con el sitio donde se encuentra el paciente; su información demográfica; los datos clínicos; los resultados de laboratorio y los datos farmacológicos (Figura 4). Estos formularios permiten enviar la información a la base de datos, en la medida en que éstos se van produciendo en el ámbito clínico o de laboratorio. El módulo de registro también incluye el almacenamiento de secuencias de ß-lactamasas aisladas e identificadas en nuestro medio, para lo cual se diseñó un formulario que permite capturar la información molecular (Figura 5).

DISCUSIÓN
De la búsqueda en Uniprot se obtuvieron 1.534 secuencias de proteínas que permiten identificar ß-lactamasas y los respectivos microorganismos que las contienen, y además sus índices cruzados con las bases de datos Uniprot y MedLine. Cabe subrayar que aunque es posible obtener los registros completos para almacenarlos localmente, sería una redundancia innecesaria, ya que ellos pueden ser consultados directamente en cualquier momento en Uniprot, utilizando el EntryName de la ß-lactamasa.
En cuanto a los datos clínicos incorporados, éstos corresponden a los incluidos en una historia clínica completa dentro del sistema de información de una institución de salud cualquiera, el cual a su vez es compatible con otros sistemas, de tal forma que los datos puedan ser cruzados utilizando herramientas como Whonet para hacer seguimiento de la resistencia hospitalaria en Bogotá. Por tratarse de la construcción de un prototipo que debe compartir fácilmente sus datos con Whonet y que se integre sin mucha dificultad con otros sistemas de información, se incorporó la menor cantidad de datos posibles dentro del sistema desarrollado, seleccionándolos de los manejados por Whonet en su Dbase tabla FIELDLST.ENG (10), de tal forma que permitieran hacer un adecuado seguimiento del comportamiento de las ß-lactamasas en cuanto al origen de la muestra, el microorganismo resistente, lugar donde se presenta el hallazgo, los resultados del laboratorio clínico, los tratamientos con antibióticos utilizados, las características del paciente y su repuesta frente a los tratamientos utilizados.
Se implementó el sistema de información BLA_ID_CLINIC, integrando los elementos necesarios para suministrarle al usuario una herramienta actualizada, que facilita el seguimiento de la aparición de secuencias de ß-lactamasas en la base de datos Uniprot; permite hacer el almacenamiento local de secuencias aisladas e identificadas de microorganismos resistentes detectados en instituciones hospitalarias locales. Además, el sistema articula la información molecular con la clínica utilizando los estándares que facilitan su integración con herramientas como Whonet. El sistema BLA_ID_CLINIC le permite al investigador evaluar datos sobre el manejo de los antibióticos, con lo cual se facilitará la implementación de herramientas para el seguimiento epidemiológico de este tipo de infecciones en el entorno intrahospitalario y entre diferentes instituciones, ya que hace una referenciación precisa del lugar de aislamiento y de la clase de ß-lactamasa.
Para desafiarlo primero se alimentaron los datos obtenidos desde Uniprot en la tabla UniprotBLA y se introdujeron los datos de secuencias de ß-lactamasas tipo en la tabla Clasificacion. Así mismo se obtuvo una muestra de historias clínicas tomadas de casos de resitencia bacteriana a antibióticos ß-lactámicos, documentados en estudios previos realizados por el grupo de Epidemiología Molecular del Instituto de Biotecnología, con las que se alimentó la base de datos en los aspectos relacionados con localización, instituciones, eventos clinicos, tratamientos y resultados de laboratorio.
Utilizando a los expertos del grupo de Epidemiología Molecular del Instituto de Biotecnología, se probó el sistema de identificación con varias secuencias, una de ellas obtenida de un aislamiento de una presunta ß-lactamasa CTX-M. Conviene destacar que el sistema de información BLA_ID_CLINIC arrojó datos que permitieron clasificar las secuencias conocidas y que en el caso de la presunta CTX-M, el resultado dio un porcentaje de identidad menor de 100 con el grupo de secuencias clasificadas como CTX-M en el sistema, que no correspondía a ninguna de las ya clasificadas, lo cual fue corroborado por los expertos. Una vez asignada su clasificación, ésta fue incluida como una nueva secuencia tipo dentro del sistema.
Por tratarse de un prototipo, que puede ser adaptado según las necesidades de los usuarios, esta primera versión no incluyó ninguna seguridad relacionada con quienes pueden administrar, modificar o sólo ver los datos; sin embargo, estas consideraciones son importantes cuando se trate de la puesta en producción del sistema. En la etapa de producción del sistema BLA_ID_CLINIC se considerará el manejo confidencial, legal y ético que debe dárseles a los datos clínicos, que involucran decisiones de especialistas, manejo de pacientes de parte de instituciones de salud y resultados de análisis de laboratorio.
La siguiente etapa en el desarrollo realizado es poner a disposición de las entidades que hacen seguimiento de la resistencia a antibiótico en Bogotá el sistema BLA_ID_CLINIC para intentar su implementación y mejoramiento productivo en Bogotá, desarrollar una interfase que permita utilizar las herramientas de análisis del software Whonet directamente sobre los datos que se puedan almacenar en el sistema BLA_ID_CLINIC y ampliar su base de datos para que incluya otros tipos de resistencia, para que sea un modelo integral para la recopilación y análisis de datos moleculares y clínicos asociados con la aparición de cualquier tipo de resistencia a los antibióticos.
El sistema BLA_ID_CLINIC promueve la creación de bancos de datos de resistencia a antibióticos que puedan ser alimentados en línea mediante Internet, que garanticen el acceso de los datos y que permitan hacerle un seguimiento adecuado a todo nivel a la forma en que se maneja y controla la resistencia a antibióticos.
Por último, el éxito de sistemas que permitan un adecuado seguimiento de la aparición de resistencia, tanto en el aspecto epidemiológico como en el farmacológico, molecular y clínico, implica avanzar en la implementación de métodos moleculares como la secuenciación, porque son el medio más idóneo para garantizar la identificación precisa de ß-lactamasas. Este tipo de tecnologías, a raíz de la secuenciación del genoma humano, están cada día más disponibles debido a su rapidez, reproducibilidad y bajo costo.
AGRADECIMIENTOS
Los autores expresan su agradecimiento a la Universidad Nacional de Colombia y a los miembros del grupo de Epidemiología Molecular del Instituto de Biotecnología.
BIBLIOGRAFIA
1. Protocolos de vigilancia en salud pública, Secretaría Distrital de Salud, Bogotá, 2003. [ Links ]
2. P.A. Espinal, E.M. Valenzuela, C. Saavedra, A.L. Leal, C. Alpuche, J.R. Mantilla, Epidemiología molecular de infección por Klebsiella pneumoniae productora de beta-lactamasas de espectro extendido (BLEE) en un hospital de tercer nivel de Bogotá, Biomédica. 24, 252 (2004). [ Links ]
3. F.H. M´Zali, J. Heritage, D.M. Gascoyne-Binzi, et al, PCR single strand conformational polymorphism can be used to detect the gene encoding SHV-7 extended-spectrum beta-lactamase and to identify different SHV genes within the same strain, J. Antimicrob. Chemother., 41, 123 (1998). [ Links ]
4. European Bioinformatics Institute - SRS. . Fecha de actualización: 15/07/2006. Fecha consulta: 15/07/2006.http://srs.ebi.ac.uk/srsbin/ [ Links ]
5. National Center for Biotechnology Information (NCBI). Fecha de actualización: 15/07/2006. Fecha consulta: 15/07/2006. http://www.ncbi.nlm.nih.gov/. [ Links ]
6. C. J. Mullett, J.G. Thomas, C.L. Smith, A.R. Sarwari, R.A. Khakoo, Computerized antimicrobial decision support: an offline evaluation of a database-driven empiric antimicrobial guidance program in hospitalized patients with a bloodstream infection, Int. J. Med. Inform., 73, 455 (2004). [ Links ]
7. T.G. McLoughlin, Jr., M.M. Joseph, Antibiotic resistance patterns of uropathogens in pediatric emergency department patients, Acad. Emerg. Med., 10, 347 (2003). [ Links ]
8. I. Filliol, S. Ferdinand, C. Sola, J. Thonnon, N. Rastogi, Spoligotyping and IS6110-RFLP typing of Mycobacterium tuberculosis from French Guiana: a comparison of results with international databases underlines interregional transmission from neighboring countries, Microbiol., 153, 81 (2002). [ Links ]
9. Grupo para el Control de la Resistencia Antimicrobiana en Bogotá (Grebo), 2006. http://www.grebo.org. [ Links ]
10. World Health Organization, Whonet. 2006. http://www.who.int/drugresistance/whonetsoftware/en/. [ Links ]
11. C. álvarez, J. Cortés, A. Arango, C. Correa, A.L. Leal, Grupo para el Control de la Resistencia Bacteriana en Bogotá. 2006, Anti-microbial resistance in intensive care units in Bogotá, Colombia, 2001-2003, Rev. Salud Pública, 8, Suppl 1, 86 (2006). [ Links ]
12. A.L. Leal, J. Eslava-Schmalbach, C. álvarez, G. Buitrago, M. Méndez, Grupo para el Control de la Resistencia Bacteriana en Bogotá, Endemic channels and bacterial resistance markers in third-level hospitals in Bogotá, Colombia, Rev. Salud Pública, 8, Suppl 1, 59 (2006). [ Links ]
13. C. Gil, Diseño e implementación de un sistema bioinformático para la identificación de genes codificantes para ß-lactamasas de espectro extendido (BLEE). Tesis de Maestría en Microbiología, Facultad de Ciencias, Universidad Nacional de Colombia, Sede Bogotá, 2004. [ Links ]
14. J. Rumbaugh, I. Jacobson, G. Booch, The Unified Modeling Language Reference Manual, Addison-Wesley Professional, 2nd Edition, Boston, 2005, p. 496. [ Links ]
15. G. Jacoby, K. Bush, C. Lahely, Amino Acid Sequences for TEM, SHV and OXA Extended-Spectrum and Inhibitor Resistant ß-Lactamases. Fecha de actualización 21/09/06. Fecha de consulta 02/10/06. http://www.lahey.org/studies/webt.asp [ Links ]
16. S.F. Altschul, W. Gish, W. Miller, E.W. Myers, D.J. Lipman, Basic local alignment search tool, J. Mol. Biol., 215, 403 (1990). [ Links ]














