Introducción
La sífilis es una infección de importancia epidemiológica, ya que afecta a la mujer en periodo de gestación y al feto1,2. El 1% de gestantes ha sido diagnosticada con sífilis gestacional (SG) siendo la segunda causa de muerte fetal a nivel mundial3,4. En Colombia la prevalencia de SG ha pasado de 7,4 casos en 2010 a 7,9 por 1000 nacidos vivos (NV) en 2017. A junio de 2018 el Instituto Nacional de Salud reportó 2.511 casos confirmados por laboratorio, con prevalencia de 3,85. El Sivigila reportó en este mismo año para el departamento del Choco una prevalencia de SG de 10.6 y una incidencia de SC de 5.88, siendo el indicar más alto de todos los departamentos6. El protocolo de vigilancia en salud pública frente a sífilis propone como estrategia fundamental para la reducción de los casos de SG y eliminación de la sífilis congénita (SC), la optimización de la cobertura y la calidad de la atención de los servicios de salud y el fortalecimiento de la detección y tratamiento de los casos de sífilis7.
La SC se presenta cuando la gestante transmite la infección al feto por vía hematógeno-trasplacentaria o durante el parto8. La detección oportuna previene complicaciones como parto prematuro, bajo peso al nacer, muerte fetal, neonatal e infantil y enfermedades congénitas9,10. Su diagnóstico se dificulta debido a gestantes sin acceso a controles prenatales, signos inespecíficos en neonatos y resultados inconsistentes en el laboratorio11.
La serología como método diagnóstico en SC es utilizada de forma paralela con el suero de la madre y del neonato en el momento del parto7. Estas pruebas no son óptimas, ya que pueden afectarse por situaciones propias del paciente o de la técnica y no ofrecen un tamizaje diferencial. El problema central es distinguir el infante asintomático infectado del lactante no infectado, nacido de una madre con pruebas serológicas reactivas de sífilis. Esta dificultad se debe en gran parte a la incapacidad para distinguir la respuesta inmune humoral de la madre de la respuesta de anticuerpos específicos del neonato, ya que los anticuerpos tipo inmunoglobulina G pasan vía transplacentaria al feto12,13. Además, las técnicas serológicas no tienen especificidad suficiente para la detección de la enfermedad en los neonatos y su sensibilidad es baja (78%), por lo que se pueden presentar falsos positivos o falsos negativos14.
La reacción en cadena de la polimerasa (PCR) es una técnica rápida y precisa que puede descartar los falsos positivos de las pruebas no treponémicas. Se ha implementado para el diagnóstico oportuno de sífilis, tipificación de las cepas y evaluación de la resistencia antimicrobiana14,15,16. Varios genes diana de T. pallidum, agente causal, se han utilizado para la detección por PCR; estos incluyen los genes tpf-1, tmpA, tmpB, bmp, polA, 16S ADN y TpN4711,17. El objetivo del presente estudio fue estandarizar una qPCR con sonda TaqMan dirigida al gen TpN47, para la determinación de T.pallidum en muestras de suero procedentes de pacientes con SG y SC, con el objetivo de ofrecer una técnica diagnóstica eficiente, sensible y específica que aporte a la detección oportuna de esta enfermedad.
Materiales y métodos
Se realizó un estudio analítico para estandarizar y evaluar una qPCR con sonda en la detección de T. pallidum en muestras de suero de gestantes y neonatos. El estudio fue considerado sin riesgo (resolución número 8430 de 1993-MinSalud) y las muestras procesas fueron obtenidas del banco de sueros del laboratorio departamental de salud pública del Choco.
Se procesaron un total de 100 muestras clínicas, 58 muestras procedentes pacientes con resultados serológicos positivos y 42 sin evidencia clínica de sífilis es decir, sin sintomatología compatible con la enfermedad, sin eventos conductuales de riesgo y sin hallazgos de laboratorio sugestivos de infección (pruebas serológicas negativas).
A cada muestra después de su obtención se le realizo las pruebas serológicas; posteriormente se almacenaron a -20°C durante 6 meses y se descongelaron a temperatura ambiente para realizar la extracción de ADN.
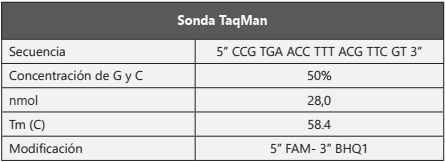
Como control positivo se empleó ADN extraído de exudado de testículo de conejo infectado con la cepa de control Nichols de T. pallidum donada por Universidad de Washington (cada exudado estaba resuspendido en Buffer de lisis 2X (Tris 10 mM pH 8,0, SDS 0,5% y EDTA 0.1M pH 8,0) y el plásmido recombinante pJET1.2-TpN47 (con un inserto de 210 pb del gen TpN47 de T. pallidum) obtenido mediante el Kit CloneJET™ PCR Cloning (Thermo Scientific™).
Extracción de ADN
El ADN del plásmido pJET1.2-TpN47 se obtuvo con el kit UltraClean 6 minutes mini plasmid prep (MOBIO). El ADN de las muestras clínicas y el exudado de testículo de conejo fueron extraídos mediante el kit UltraClean® Blood spin DNA isolation (MOBIO), con un pretratamiento de proteinasa K (0.5 mg/ml) a 65°C por 1 hora.
Sensibilidad, especificidad y eficiencia analítica de la qPCR
Para la detección del gen TpN47 por qPCR se usaron los iniciadores TpN47-sifF GCTTGTTTAAGCCGTTG y TpN47-sifR AAGAGTCAGGTGCAGTA18. Se utilizó el kit TaqMan 2X Universal PCR Master (Applied Biosystems) y oligo sonda con modificación 5” FAM- 3” BHQ diseñada (tabla 1); las condiciones para una reacción de 15 μl fueron Master mix 1X, primers sentido y antisentido 0.14 μM, sonda 0.10 μM, el volumen de ADN utilizado fue de 5ul. El programa de ciclaje comprende un ciclo de 50°C por 2 minutos (min), un ciclo de 95°C por 7 min seguido por 50 ciclos de 95°C por 30 s (segundos), 60°C por 30 s y 72°C por 5 s, la amplificación se llevó a cabo en el termociclador iQ5 (BioRad).
Para el límite de detección, se realizaron diluciones seriadas en base 10 del ADN de plásmido pJET1.2-TpN47 (concentración inicial de 113.5 nanogramos), y se elaboró curva estándar a partir de los valores de Ct obtenidos de 11 puntos de dilución por cuadruplicado, estos fueron graficados en el programa GraphPad Prism. A partir de la pendiente de la curva estándar se determinó la eficiencia de la reacción de amplificación. La especificidad analítica se evaluó empleando como molde de amplificación ADN de bacterias obtenidas de aislamientos clínicos como S. epidermidis, E. coli y Klebsiella spp y ADN de la cepa ATCC USA 300 de S. aureus.
Se evalúo la presencia de inhibidores en la qPCR escogiendo al azar muestras que no mostraron amplificación e infectándolas con 2 μl y 5 μl ADN de Treponema pallidum subsp pallidum. Adicionalmente se realizó control endógeno de la extracción amplificando en las muestras clínicas extraídas el gen β-actina humano.
Pruebas serológicas
A todas las muestras se les realizó prueba treponémica TPPA (Serodia) y no treponémica VDRL test (Wiener lab).
Resultados
Pruebas serológicas
Las muestras evaluadas fueron obtenidas de 51 gestantes entre los 14 y 45 años de edad, del total de muestras 7 pertenecían a neonatos, todos de sexo masculino.
qPCR sensibilidad y especificidad analítica
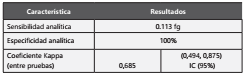
El límite de detección fue 0,113 femtogramos (fg), la desviación estándar entre las réplicas por dilución de ADN del plásmido pJET1.2-TpN47 oscilo entre 0,01 y 0,09; no se detectó señal de amplificación en ADN de otras bacterias (especificidad analítica de 100%).
Características de la qPCR
En la fig.1a se observan las curvas de amplificación de las diluciones seriadas del ADN del plásmido pJET1.2-TpN47 y en la fig.1b la regresión lineal de los valores Ct con una pendiente de -3.11, reflejando una fidelidad de 104%. Se evidenció correlación optima en la prueba, sugerida por un r2 de 0.99 y un valor p <0,0001 de la qPCR. En la tabla 3 se evidencian las características operativas de la qPCR estandarizada.
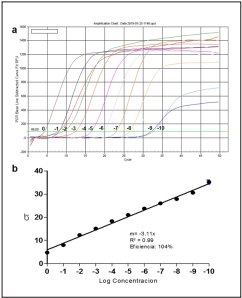
Figura 1 a. Curva de amplificación de ADN del plásmido pJET1.2-TpN47 puro (0) y diluciones 10-1 hasta 10-10. b. Regresión lineal de valores CT de 10 diluciones seriadas de ADN del plásmido pJET1.2-TpN47. En azul se observa la desviación estándar entre replicas. CT: cycle threshold. m: pendiente. R2: coeficiente de regresión.
Todas las muestras clínicas infectadas con ADN de T. pallidum mostraron amplificación del gen TpN47 lo que evidencio ausencia de inhibidores en las muestras extraídas. Adicionalmente, los sueros mostraron amplificación del gen beta actina lo que corroboró el éxito de la extracción de ADN (figura 2).
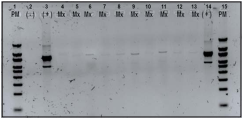
Figura 2. Electroforesis en gel de agarosa 1.8% de los productos de amplificación del gen beta actina en muestras clínicas. Carril 1. Marcador de peso molecular 100 pb; Carril 2. Control negativo; Carril 3. Control positivo gen beta actina 800 pb; Carriles 4-13. Muestras clínicas; Carril 14. Control positivo gen beta actina 800 pb; Carril 15. Patrón de peso molecular 100 pb.
Discusión
La sífilis gestacional y congénita representan un desafío para la salud pública, por su alta morbilidad y mortalidad perinatal a nivel mundial; debido a la trasmisión materno-infantil y a las secuelas en los neonatos19. La detección molecular de la espiroqueta en los fluidos y tejidos del cuerpo tiene un enorme atractivo como estrategia de diagnóstico de sífilis, ya que proporciona una evidencia de infección activa; además, de un diagnóstico precoz para aquellos pacientes que se encuentran en periodo de ventana y para aquellos que no pueden asistir a análisis serológico secuencial15,20,21.
En el presente trabajo se logró la estandarización de una metodología molecular con sensibilidad analítica de menos de una copia del genoma bacteriano de T. pallidum, en comparación con otros reportes con límites de detección entre 1 pg (equivalentes 800 copias) y 2,3 copias del genoma9,15. La especificidad analítica fue del 100% y se logró una óptima eficiencia y linealidad en la reacción de qPCR (R2 0,99), además se observó una buena reproducibilidad entre las réplicas evaluadas de ADN del plásmido pJET1.2-TpN47. La comparación entre las pruebas serológicas y moleculares mostró una concordancia adecuada (kappa 0,685) y estos resultados son reproducibles con los reportados por Pinilla et al14.
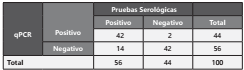
Del total de muestras presuntivas de SC y SG, en 44 (75%) fue posible amplificar el gen TpN47, lo que evidencia que la qPCR TaqMan es una alternativa de diagnóstico que permite aumentar la sensibilidad de detección al amplificar una región especifica del gen TpN47 por medio de la sonda de hidrolisis. Se ha propuesto que esta metodología permitiría evaluar la continuidad de la espiroquetemia en lactantes22,23.
Del total de muestras, 14 mostraron discrepancias (serología positiva-qPCR negativa) (tabla 2); 4 pertenecen a neonatos con pruebas serológicas reactivas (1-2 dils) esto puede estar asociado a infección antigua de la madre, por transferencia pasiva de anticuerpos a través de la placenta, ya que permanecen en circulación hasta los 15 meses de edad12,7. Respecto a las 10 muestras restantes podría tratarse de una infección muy reciente y por ende una carga treponémica indetectable; se deben contemplar factores como la presencia o no de las espiroquetas en el sitio de muestreo en el momento de toma y por otra parte el volumen utilizado en la reacción podría carecer de una copia completa del gen del antígeno de 47 KDa22,24. Se debe tener presente si el paciente recibió tratamiento previo, además de posibles reacciones cruzadas en las pruebas serológicas, puesto que su rendimiento puede disminuirse ante la presencia de infección concomitante, malignidad avanzada, embarazo y enfermedades vasculares del colágeno, en donde son frecuentes las reacciones falsas positivas asociadas a inflamación14,25.
Se evidenciaron 2 muestras con resultados serológicos negativos y qPCR positivos (tabla 2), los falsos negativos de las pruebas serológicas se pueden relacionar al efecto prozona en mujeres embarazadas, es decir las muestras evaluadas pueden ser fuertemente reactivas y producir una reacción inaparente interpretada como negativa. Además los pacientes pueden estar en periodo de ventana inmunológico o en estadio primario de la infección y por tanto los anticuerpos aun no son detectables26,27,28.
La alternativa molecular descrita tiene una buena sensibilidad y especificidad analítica, y por tanto es una herramienta molecular prometedora y con gran potencial diagnóstico para la detección y hallazgo de T. pallidum subsp. pallidum, a través de la amplificación del gen TpN47 como diana en muestras clínicas; en este trabajo nos limitamos a realizar la optimización de la técnica; sin embargo, es de gran importancia validar la técnica para el diagnóstico y confirmación clínica, por medio de su evaluación en pacientes con diagnóstico confirmado de SG y/o SC en el binomio madre e hijo. En el presente estudio no fue posible evaluar el efecto prozona en las muestras evaluadas y se recomienda realizar este análisis en futuras investigaciones que comparen pruebas serológicas y moleculares.