Comentario sobre el artículo
Klunk, J., Vilgalys, T. P., Demeure, C. E., Cheng, X., Shiratori, M., Madej, J., Beau, R., Elli, D., Patino, M. I., Redfern, R., DeWitte, S. N., Gamble, J. A., Boldsen, J. L., Carmichael, A., Varlik, N., Eaton, K., Grenier, J. C., Golding, G. B., Devault, A., Rouillard, J. M., Yotova, V., Sindeaux, R., Ye, C.J., Bikaran, M., Dumaine, A., Brinkworth, J. F., Missiakas, D., Rouleau, G. A., Steinrücken, M., Pizarro-Cerdá. J., Poinar, H. N., Barreiro, L. B. (2022). Evolution of immune genes is associated with the Black Death. Nature, 611, 312-319. https://doi.org/10.1038/s41586-022-05349-x
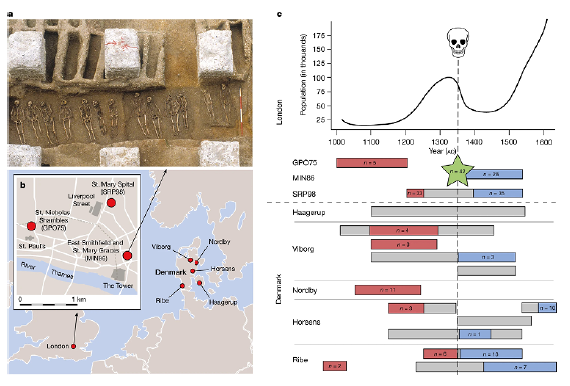
Figura 1 Lugares de entierro masivo y muestra de East Smithfield, junto con rangos de fechas y números de muestra finales utilizados para el presente estudio.
Las infecciones son una causa importante de mortalidad en poblaciones humanas y, por lo tanto, ejercen una selección natural, la fuerza creativa y regulatoria de la evolución de las especies. Descrita por Charles Darwin, la selección natural se fundamenta en el concepto de que en un medio natural sobreviven los más aptos o los más fuertes (p.ej., aquellos que sobrevivieron a pandemias como la Peste Negra). Los individuos menos aptos, o más débiles, perecen y sus genes no se transmiten a las siguientes generaciones al no reproducirse.
En algunos estudios en que se han relacionado las frecuencias de variantes genéticas, o polimorfismos, de poblaciones humanas con la diversidad de ciertos microorganismos, se ha establecido que la selección natural dirigida por patógenos es una fuerza relevante en la adaptación local de numerosos genes y, en consecuencia, de proteínas que participan en diversas vías de señalización, incluida la respuesta inmunitaria. Tal es el caso de los genes del complejo mayor de histocompatibilidad, de aquellos que codifican los antígenos de los grupos sanguíneos o de los que codifican para algunas moléculas del sistema inmunitario conocidas como citocinas, entre otros (Gugamalli et al., 2011; Nedelec et al., 2016).
En los años cincuenta del siglo pasado los científicos descubrieron cómo algunos grupos sanguíneos y anormalidades en la hemoglobina habrían surgido en áreas endémicas como protección contra el colera y la malaria, respectivamente. Algunos alelos del gen APOE4 protegieron a nuestros antepasados contra retrasos cognitivos durante la niñez cuando sufrían diarreas frecuentes y graves, pero ahora aumentan el riesgo cardiovascular y de Alzheimer (Guerrant et al., 2007).
Una investigación colaborativa internacional, publicada recientemente en la revista Nature (Klunk et al., 2022), mostró que la Peste Negra, causada por la bacteria Yersinia pestis, que acabó con 30 a 60 % de las personas en ciudades del norte de África, Europa y Asia en el siglo XIV, ejerció una significativa presión de selección sobre la población humana, incrementando la frecuencia de ciertas variantes genéticas relacionadas con el sistema inmunitario y aumentando la propensión a las enfermedades autoinmunes.
Los autores del estudio examinaron muestras de ADN de los huesos de más de 200 personas que vivieron en Londres y Dinamarca y que fallecieron antes, durante y después de la Peste Negra. Se identificaron variantes, o polimorfismos, en cuatro genes que protegían o aumentaban la sensibilidad a la infección por Yersinia. Uno de los genes, aquel que codifica la enzima aminopeptidasa del retículo endoplásmico 2 (ERAP2, por sus siglas en inglés), resultó ser fundamental para la defensa contra la infección. Las personas que poseían dos copias del polimorfismo rs2549794 en este gen produjeron más proteína funcional, en comparación con otra variante que condujo a una versión truncada y no funcional. Poseer dos copias de dicho polimorfismo habría hecho que una persona tuviera un 40 % más de probabilidades de sobrevivir a la Peste Negra que aquellos que tenían dos copias de la variante no funcional. La misma variante se ha asociado con una mayor propensión a las enfermedades autoinmunes e, incluso, se la ha caracterizado como un conocido factor de riesgo para la enfermedad de Crohn, lo que indica que una fuerza de selección natural presente durante una antigua pandemia contribuyó a aumentar la posibilidad de sufrir enfermedades autoinmunes en la era actual.
En Colombia se han desarrollado algunos estudios que sugieren un fenómeno similar con la tuberculosis. Investigadores de la Corporación para Investigaciones Biológicas (CIB) de Medellín evidenciaron en grupos de población antioqueña que una variante del gen que codifica para el factor de necrosis tumoral (TNF) y que produce una citocina inflamatoria, y otra variante del gen de la proteína tirosina fosfatasa no receptora de tipo 22 (PTPN22) que codifica para la proteína linfoide tirosina fosfatasa, importante regulador negativo de la activación de los linfocitos T (células del sistema inmunitario), son variantes genéticas de riesgo para enfermedades autoinmunes, pero protegen contra el desarrollo de la tuberculosis (Correa et al., 2005, Gómez et al., 2005, Gómez et al., 2005). No sobra mencionar que las variables medioambientales y los determinantes sociales influyen también en el desarrollo y la gravedad de las enfermedades, en general, y de las infecciones, en particular.
Recientemente, un estudio colaborativo entre investigadores estonios y rusos mostró que el 28 %> de los polimorfismos de riesgo en 21 enfermedades autoinmunes e inflamatorias presentan huellas de selección moderada y débil y que algunos de ellos son específicos de la población de estudio (Pankratov et al., 2022).
Se ha planteado que con la pandemia de COVID-19 causada por el coronavirus SARS-CoV-2 pueda ocurrir un fenómeno similar. ¿Qué tanto podrán influir la COVID-19 y la vacunación masiva en la selección de variantes genómicas de susceptibilidad a enfermedades futuras? ¿Cuál será el costo evolutivo de sobrevivir a esta pandemia? Hasta la fecha el virus se ha asociado con el desarrollo de autoinmunidad, tanto latente (presencia de múltiples autoanticuerpos) como clínicamente manifiesta, en particular artritis reumatoide, vasculitis y diabetes mellitus tipo 1 (Anaya et al., 2022), lo que refleja la necesidad de estudiar la interacción entre infección y autoinmunidad para la comprensión de la supervivencia de la especia humana.















