Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biotecnología en el Sector Agropecuario y Agroindustrial
versão impressa ISSN 1692-3561
Rev.Bio.Agro vol.15 no.2 Popayán jul./dez. 2017
https://doi.org/10.18684/BSAA(15)49-56
DOI: http://dx.doi.org/10.18684/BSAA(15)49-56
CARACTERIZACIÓN DE LA DIVERSIDAD GENÉTICA DE UNA COLECCIÓN DE QUINUA (Chenopodium quinoa Willd)
CHARACTERIZATION OF GENETIC DIVERSITY OF A COLLECTION OF QUINUA (Chenopodium quinoa Willd)
CARACTERIZAÇÃO DA DIVERSIDADE GENÉTICA DE UMA COLEÇÃO DE QUINUA(Chenopodium quinoa Willd)
ANA CRUZ MORILLO CORONADO1 , MÓNICA ANDREA CASTRO ROBERTO2, YACENIA MORILLO-CORONADO3
1Universidad Pedagógica y Tecnológica de Colombia, Facultad de Ciencias Agropecuarias, Programa Ingeniería Agronómica, Grupo Manejo Biológico de Cultivos. PhD. Fitomejoramiento. Tunja, Colombia.
2Universidad Pedagógica y Tecnológica de Colombia, Programa Ingeniería Agronómica, Grupo Competitividad, Innovación y Desarrollo Empresarial (CIDE). Estudiante Ingeniería Agronómica.Tunja, Colombia.
3Universidad de Caldas, Programa Ingeniería Agronómica. Biotecnología y Mejoramiento Vegetal. Manizales, Colombia.
Correspondencia: ana.morillo@uptc.edu.co
Recibido para evaluación: 15 de Agosto de 2016. Aprobado para publicación: 8 de Marzo de 2017.
RESUMEN
La quinua (C. quinoa W.) es considerada un cereal con excelentes propiedades nutricionales, entre las que se destacan su alto contenido proteíco, ya que tiene todos los aminoácidos, elementos traza y cantidades significativas de vitaminas. En Colombia, más exactamente en el departamento de Nariño, Cauca, Cundinamarca y Boyacá ha tenido actualmente un gran impulso debido a sus potencialidades agronómicas y diferentes beneficios que se derivan de la producción, industrialización y comercialización de sus productos. Por lo anterior, el objetivo de esta investigación fue caracterizar la diversidad genética de una colección de 55 materiales de quinua con siete marcadores microsatélites RAMs. El análisis mediante el coeficiente de Nei-Li a un nivel de similitud de 0,65 dividió a la población en cuatro grupos de acuerdo al sitio de procedencia de los materiales. El valor de heterocigosidad promedio fue de 0,34 el cual se considera bajo en comparación con otros estudios de diversidad genética en Chenopodium. El Análisis de Varianza Molecular (AMOVA) y el Fst muestran la existencia de variabilidad genética a nivel intraespecífico. Los parámetros de diversidad genética encontrados en este estudio muestran que los materiales de quinua de la colección de la gobernación de Boyacá son muy homogéneos, sin embargo la variabilidad que existe debe ser aprovechada en los programas de mejoramiento genético de la especie que conduzcan hacia la obtención de nuevos y mejores materiales de quinua.
PALABRAS CLAVE: Diversidad genética, Microsatélites RAMs, Cereal andino.
ABSTRACT
Quinua (C. quinoa W.) is considered a cereal with excellent nutritional properties, including high protein contein, because it has all amino acids, trace elements and significant amounts of vitamins. In Colombia more accurately in the department of Nariño, Cauca, Cundinamarca y Boyacá currently has had a huge boost due to their agronomic potential and different benefits derived from the production, processing and marketing of its products. Therefore, the objective of this research was to characterize the genetic diversity of a collection of 55 materials quinoa with seven microsatellite markers RAMs. The analysis by the coefficient of Nei-Li at the level of similarity of 0,65 divided the population into four groups according the site origin materials. The value of average heterozygosity was 0,34 which is considered low compared to other studied of genetic diversity in Chenopodium. Molecular Analysis of Variance (AMOVA) and Fst demonstrate the existence of genetic variability at the intraspecific level. The parameters of genetic diversity found in this study show that the quinoa materials of the Boyacá governance collection are very homogeneous, however the variability that exists must be exploited in the breeding programs of the species leading to the production of new and better quinoa materials.
KEY WORDS: Genetic diversity, Microsatellites RAMs, Andean cereal.
RESUMO
Quinua (C. quinoa W.) é considerado um cereal com excelentes propriedades nutricionais, entre os quais seu alto teor de proteína, uma vez que tem todos os aminoácidos, oligoelementos e quantidades significativas de vitaminas. Na Colômbia, mais precisamente no departamento de Nariño, Cauca, Cundinamarca e Boyacá ele já teve um grande impulso devido ao seu potencial agronômico e diferentes benefícios derivados da produç ão, transformaç ão e comercializaç ão dos seus produtos. Portanto, o objetivo deste trabalho foi caracterizar a diversidade genética de uma coleç ão de 55 materiais com sete marcadores microssatélites RAMs quinoa. A análise pelo coeficiente de Nei-Li em um nível de similaridade de 0,65 dividiu a populaç ão em quatro grupos de acordo com o local de origem dos materiais. O valor de heterozigosidade média foi de 0,34, que é considerado baixo em comparaç ão com outros estudos de diversidade genética em Chenopodium. Análise molecular de variância (AMOVA) e Fst mostram a existência de variabilidade genética no nível intra-específica. Os parâmetros de diversidade genética encontrados neste estudo mostram que a coleta de materiais quinoa governadoria Boyacá s ão muito homogénea, no entanto, a variabilidade que existe deve ser utilizada em programas de melhoramento das espécies que levam à obtenç ão de novos e melhores materiais de quinoa.
PALAVRAS-CHAVE: Diversidade genética, Microssatélites RAMs, Cereal andina
INTRODUCCIÓN
La quinua (Chenopodium quinua Willd.) es una especie anual, dicotiledónea que pertenece a la familia Chenopodiaceae, cultivada desde el nivel del mar hasta los 4000 m, con amplia adaptación agroecológica y a diferentes tipos de suelos. Originaria de Sur América, sigue siendo cultivada en diferentes regiones de ese continente, especialmente en países como Colombia, Chile, Bolivia, Ecuador y Perú, siendo recientemente introducida en Europa, América del Norte, Asia y África. Se estima que más del 80% de su producción mundial se concentra en países como Perú, Bolivia y Ecuador [1] . También se produce en las zonas costeras del sur de Chile y los valles andinos del sur de Colombia, más exactamente en el departamento de Nariño, Cauca, Boyacá y Cundinamarca [2], en donde ha tenido actualmente un gran impulso debido a sus potencialidades agronómicas y diferentes beneficios que se derivan de la producción, industrialización y comercialización de sus productos [1].
Es considerada un cereal con excelentes propiedades nutricionales entre las que se destacan su alto contenido proteíco, ya que tiene todos los aminoácidos, elementos traza y cantidades significativas de vitaminas C, E (tocoferoles) y B (B1, B2 y B3) junto con importantes minerales (Ca, K, Fe, Mg, Mn, P), e isoflavonas que pueden contribuir a sus propiedades antioxidantes. La quinua no tiene gluten y sus ácidos grasos de alta calidad [3]. La testa de sus semillas presenta saponinas, considerada anteriormente como un antinutriente debido a su sabor amargo, actualmente se extrae para fines industriales y biomédicos [4]. También se le considera una especie con alta adaptabilidad agroecológica, como resultado de un largo proceso de domesticación, y una alta variabilidad genética. Es una especie que puede tolerar diferentes tipos de estrés como salinidad, frío, alta radiación solar, temperaturas de congelamiento nocturnas, así como tolerancia a factores fitosanitarios [1,5]. Debido a su potencial económico y a que representa un cultivo de seguridad alimentaria para las comunidades andinas, en la última década se ha incentivado su producción entre agricultores, empresas agroindustriales e instituciones [6].
La quinua (C. quinoa) constituye uno de los cultivos andinos con escasa investigación en el área de genética y fitomejoramiento, a pesar de que presenta una alta variabilidad en caracteres como color de la planta, flores, contenidos nutricionales y de metabolitos de interés [7]. Estudios de colecta, conservación y caracterización son necesarios para el planteamiento de estrategias de mejoramiento de esta especie, en este sentido, a nivel internacional se reportan alrededor de 16.263 accesiones de Chenopodium colectadas en todo el mundo, las cuales han sido conservadas y caracterizadas parcialmente por instituciones principalmente de Bolivia, Perú, Estados Unidos e India [6]. En Colombia, Corpoica Tibaitatá reporta un banco de germoplasma de 28 accesiones de quinua [6], sin embargo se conoce de la existencia de pequeñas colecciones en los principales departamentos productores. En cuanto a la caracterización de ese recurso fitogenético en el país, solo se tienen los estudios morfoagronómicos desarrollados por Torres et al., (2000) [8] en la Sabana de Bogotá.
Por lo anterior, se hace necesario la implementación de investigaciones que conduzcan al conocimiento de la genética de esta especie, ya que solo existen pocas variedades comerciales, lo cual representa un riesgo fitosanitario y una pérdida de diversidad genética.
El desarrollo de herramientas de biología molecular, biotecnología, secuenciación y bioinformática han permitido el estudio genético de diferentes especies del género Chenopodium; entre éstas el mapeo y la caracterización de miles de microsatélites, marcadores de polimorfismo en un simple nucleótido (SNP), la creación de librerías de secuencias expresadas, librerías BAC como también la generación de un segundo mapa de ligamiento, deberían facilitar la introgresión de nuevos alelos en el genoma de la quinua [6, 9]. Para la identificación de la diversidad genética en quinua se han utilizado diferentes sistemas marcadores, entre ellos los más utilizados son los microsatélites, que son de herencia codominante, presentan buena cobertura del genoma, y son de naturaleza multialélica [10]. Entre los microsatélites, los RAMs (Microsatélites Amplificados al Azar) han sido utilizados en diferentes plantas para medir la variación intra e interespecífica, su metodología es factible para pequeños laboratorios y permite la determinación de parámetros genéticos confiables [11,12].
Uno de los principales problemas que afronta el departamento de Boyacá en el sistema productivo de la quinua, es la falta de identificación de sus materiales de siembra, ya que son los mismos agricultores quienes seleccionan su propia semilla de sus cultivos ciclo tras ciclo; esto ha llevado actualmente a que en los campos se vea una mezcla varietal fruto de ese proceso de selección. Por lo anterior, se necesitan establecer los principios básicos para la obtención de materiales de siembra 'puros' de buenos rendimientos, con tolerancia a factores bióticos y abióticos, y adaptados a las condiciones locales. Dentro de este contexto, el presente trabajo de investigación busca caracterizar con marcadores RAMs la colección de quinua de la Gobernación de Boyacá como una primera aproximación hacia el conocimiento, conservación y uso eficiente de este recurso fitogenético, lo cual redundaría en ventajas para la economía, la salud humana y la seguridad alimentaria de los agricultores andinos.
MÉTODO
Material Vegetal
Se evaluaron un total de 54 materiales genéticos de quinua (C. quinoa), que hacen parte de la colección que tiene el Laboratorio de Biotecnología Vegetal de la Dirección de Desarrollo Agropecuario, dependiente de la Secretaria de Fomento Agropecuario de la Gobernación de Boyacá, en la ciudad de Tunja ubicada a una altura de 2.820 m.s.n.m, con una temperatura promedio de 13°C (Cuadro 1).
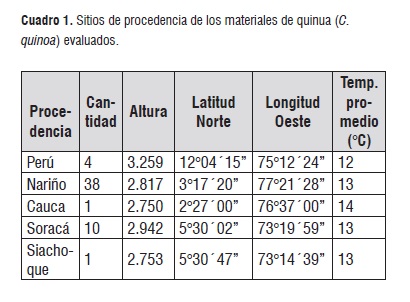 Cuadro 1
Cuadro 1Caracterización molecular
Se realizó en los laboratorios de investigación en Biología Molecular, BIOPLASMA y GEBIMOL, de la Universidad Pedagógica de Colombia (Tunja). Para la extracción de ADN se utilizó el protocolo de Dellaporta modificado por Muñoz et al., (2008) [13]. Los ADN totales se visualizaron en geles de agarosa al 0,8%, en una cámara Maxicell Primo EC-340 Electroforesis Gel System. Para determinar la concentración de ADN se utilizó el fluorómetro Hoefer Dyna Quant 200 y se diluyó en agua tipo HPLC a un volumen total de 100 µl a 10 ng/µl y se almacenó a -20 °C. Para el análisis RAMs se utilizaron siete cebadores sintetizados por Technologies Inc. Bioneer (Cuadro 2).
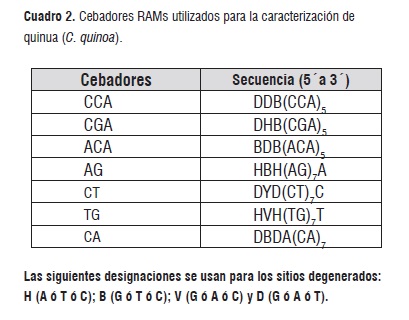 Cuadro 2
Cuadro 2La amplificación se llevó a cabo en un termociclador PTC 100 Programmable Termal Controller (MJ. Research, Inc). La desnaturalización inicial fue a 95°C durante 5 min; desnaturalización a 95°C por 30 seg, hibridación a una temperatura de 50°C (cebador AG y CA)° 55°C (cebador CCA-TG-CT) y 58°C (cebador CGA) durante 45 seg, una extensión de 72°C por 2 min, 37 ciclos desde la desnaturalización a extensión y por último una extensión a 72°C durante 7 min. Los productos amplificados fueron separados por electroforesis en geles de poliacrilamida 37:1 (acrilamida: bisacrilamida) al 7 % a 150 v por 1 h en una cámara pequeña de DNA Sequencing System, FB-SEQ-3545 de Fisher Biotechnologies. La tinción se realizó usando sales de plata.
Análisis Estadístico
Se generó una matriz binaria de ausencia (cero) y presencia (uno). La similitud genética entre los individuos se calculó utilizando el coeficiente de similitud de Nei y Li (1979) [14]. El análisis de agrupamiento se realizó por el método UPGMA y se generó un dendrograma utilizando el paquete estadístico NTSYS (Numerical Taxonomy System for personal Computer, versión 2.02® PC, New York, 1997) [15]. Para evaluar la diversidad genética se estimó la heterocigosidad insesgada y el porcentaje de loci polimórficos utilizando el paquete estadístico TFPGA (Tools For Population Genetic Analices, versión 1,3, Arizona 1997) [16]. Se determinó el f estadístico insesgado con un intervalo de confianza del 95 % y se realizó el Análisis de Varianza Molecular (AMOVA) con el programa GEnALex 6,5® [17].
RESULTADOS
El análisis mediante el coeficiente de Nei-Li [14] a un nivel de similitud de 0,65 diferenció a los materiales en cuatro grupos (Figura 1). En el grupo I es encuentran los materiales de quinua procedentes del departamento de Nariño, los cuales presentaron distancias genética comprendidas entre 0,80 a 0,92, lo cual muestra que son materiales altamente homogéneos, lo cual puede atribuirse a los procesos de domesticación a que está sometida la especie, el constante intercambio de semillas entre los agricultores de la misma vereda u otras cercanas e incluso con otras zonas productoras del país, el sistema de apareamiento y los eventos de cuello de botella por los cuales ha pasado la especie, lo cual ha llevado a la pérdida de diversidad genética [1].
A 0,60 de similitud encontramos el grupo II con materiales que fueron colectados en las zonas productoras del departamento de Nariño, que se encuentran a una distancia genética de 0,91, con respecto al grupo I, lo cual muestra el alto grado de parentesco que existen entre las quinuas procedentes de una misma región. Ya en el grupo II y grupo IV se puede observar una distribución mucho más laxa de los individuos procedentes de los diferentes sitios de evaluación, poniendo de manifiesto el flujo genético entre ellos, con distancias genéticas entre los diferentes grupos mayores a 0,80, lo cual puede ser beneficioso para los programas de mejoramiento que implementen estrategias de hibridación [9]. Por otro lado, también se puede observar el grado de consanguinidad que existe entre la quinua (C. quinua) y otras especies relacionadas como la Kiwicha (Amaranthus spp.) e incluso con los materiales peruanos, reafirmando la existencia de un continuo intercambio de semilla entre agricultores o investigadores. En términos generales los agrupamientos correspondieron con el sitio geográfico donde fueron colectados los materiales de quinua, lo cual ya había sido reportado en otros estudios de diversidad genética usando diferentes tipos de marcadores [9, 18]. Estudios realizados en otros países andinos, ya habían reportado también baja variabilidad en las variedades locales de quinua, lo cual era de esperarse por los procesos de selección llevados a cabo por los mejoradores. Así la diversidad genética disminuye después de los procesos sistemáticos de selección llevados a cabo por agricultores o mejoradores [7].
Los siete cebadores RAMs utilizados para la caracterización molecular de los materiales de quinua (C. quinoa) generaron un total de 178 bandas (Cuadro 3). El número de bandas por cebador varió entre 20 para el CGA y 35 para ACA, con pesos moleculares entre 300 y 1300 Kb, lo cual se considera adecuado para la estimación de los parámetros genéticos [9,19]. El porcentaje de loci polimórficos para los siete cebadores estuvieron en un rango comprendido entre el 86,36% al 100% (CCA y AG-CCA, respectivamente), con valores de heterocigosidad entre 0,28 para el cebador CT y 0,39 para CGA. Teniendo en cuenta la definición de Ott (1992) [20], la cual considera a un marcador como polimórfico si la He es mayor o igual a 0,1 y altamente polimórfico si esta es mayor o igual a 0,7; se determina así que los cebadores RAMs son un grupo de marcadores polimórficos que pueden ser útiles para la discriminación de individuos de quinua estrechamente relacionados [9,21] . El cebador CA fue el que mayor aporte hizo a la variación genética encontrada, Fst de 0,32, lo cual significa que puede ser útil para la diferenciación de materiales del género Chenopodium en estudios de diversidad genética intra o interespecífica.
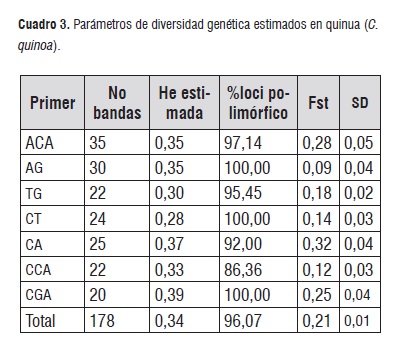 Cuadro 3
Cuadro 3Asimismo se identificaron que las repeticiones CA, CGA y TG son las secuencias repetidas más frecuentes en el genoma de la quinua a comparación de CCA y AG las cuales parecen estar en uma menor frecuencia en los genotipos evaluados y está relacionado con los procesos evolutivos que ha sufrido la especie; resultados similares a los encontrados por Jarvis et al., 2008 [22], quienes desarrollaron un mapa de ligamiento usando marcadores SSR, AFLPs, la región de almacenamiento de proteína en la semilla (11S) y la región organizadora nucleolar (NOR) y en otras especies relacionadas como el amaranto [19].
Para la población total el porcentaje de loci polimórficos y la heterocigosidad promedio esperada (He) fueron de 96,07% y 0,34 respectivamente. El coeficiente de diferenciación genética obtenido (Fst) al evaluar los materiales de quinua de la colección, con los siete microsatélites RAMs, fue de 0,21 con una desviación estándar de 0,01 (Cuadro 3). Según Wright (1978) [23], valores comprendidos entre 0,15 y 0,25 muestran diferenciación genética, la cual está asociada con el nivel de estructuración de la población que tiende a estabilizarse.
En estudios de caracterización de la diversidad genética en Chenopodium, con marcadores moleculares dominantes y/o codominantes, se han obtenido altos porcentajes de loci polimórficos y altos valores de heterocigosidad lo cual podría atribuirse a la alotetraploídia, al sistema de apareamiento autopolinización/polinización cruzada, ginomonoecia, autoincompatibilidad y la esterilidad masculina [24]; al intercambio de información genética constante a nivel intra o interespecífico con parientes silvestre y ancestrales [9,25]. Con estas herramientas moleculares ha sido posible identificar diferencias a nivel del genoma y similitudes que están asociadas con características morfológicas tales como color del grano y de la panícula, fenología, y distribución geográfica [6]. Por el contrario, los parámetros de diversidad genética encontrados en este estudio muestran que los materiales de quinua de la colección de la gobernación de Boyacá son muy homogéneos, lo cual corrobora los resultados obtenidos en el dendrograma y en la estimación de las distancias genéticas y coincide con estudios realizados por los programas de mejoramiento Chilenos y en la caracterización de 172 accesiones de la colección nacional de Perú mediante marcadores microsatélites, lo que permite inferir teóricamente que hay un déficit de heterocigotos en la población y por tanto, una mayor cantidad de individuos homocigotos debido a los altos porcentajes de autofecundación [7,9].
El análisis de estructura genética mostró una diferenciación entre los individuos evaluados, mientras se pone de manifiesto una deficiencia de heterocigotos como resultado de los procesos de la dinámica espacio-temporal a la que están sometidos estos materiales en su entorno natural, como al sistema de apareamiento tan complejo que presentan las especies de quinua; también podría ser el reflejo de prácticas culturales y ancestrales de las comunidades andinas [7, 9, 25]. El análisis de varianza molecular mostró que el 77% de la variabilidad genética observada en los materiales de quinua evaluados, es debida al componente dentro de grupos y que el 23% restante se atribuye a las diferencias entre ellos, lo cual indica que hay que hacer estudios microgeográficos a nivel intra grupos que es donde se pueden identificar individuos con las mejores combinaciones alélicas para las características de producción, industrialización y mercado. Sin embargo el 23% de variación entre grupos es significativa y también puede incorporarse dentro de las estrategias de mejoramiento de la especie.
La diversidad genética de la quinua (C. quinoa) ha logrado mantenerse en el tiempo gracias a las tradiciones y conocimientos ancestrales de las comunidades campesinas, por lo cual es considerado un cultivo de herencia familiar. Sin embargo, en los últimos años con el incremento de su demanda tanto en mercados nacionales como internacionales, ha conducido a que haya una sustitución de las variedades nativas por las comerciales, lo cual genera pérdida de la diversidad genética e incremento de la homocigosis en las poblaciones haciéndolas más vulnerables a los problemas fitosanitarios y a una reducción en su capacidad para adaptarse a las ambientales tan cambiantes ?26?. Por otro lado, el desarrollo industrial también ha provocado el abandono de las áreas rurales por la migración masiva hacia las ciudades, esta situación social y económica está cambiando el uso de la tierra y afectando la variabilidad genética de los cultivos andinos.
Por tanto, es fundamental preservar a pequeña escala la diversidad genética de los materiales locales de quinua, la cual se encuentra fuertemente asociada principalmente a su cultura. Conservar la agrobiodiversidad significa entonces preservar también la cultura asociada a los campesinos que viven en la región andina [7].
Colombia tiene el potencial para incrementar la productividad de este cultivo, por una parte se pueden obtener hasta dos cosechas por año, lo cual no es posible en Bolivia (principal país productor) y por otra, el cultivo se puede someter al esquema de rotación en zonas frías. Boyacá es uno de los departamentos de Colombia con gran vocación agrícola y con las condiciones agroecológicas favorables para el desarrollo de la quinua, en donde se cuenta con más de once municipios productores y algunos se encuentran asociados (Fedequinua); además, como lo pone de manifiesto este trabajo existe variabilidad genética que puede ser aprovechada en los programas de mejoramiento encaminados a la obtención de nuevos y mejores materiales, adaptados a las condiciones locales y que respondan a las necesidades del agricultor, productor y consumidor.
CONCLUSIONES
Los marcadores RAMs permitieron determinar la variabilidad genética existente en los materiales de quinua evaluados agrupándolos de acuerdo al sitio geográfico de procedencia.
Los parámetros de diversidad genética encontrados en este estudio muestran que los materiales de quinua de la colección de la gobernación de Boyacá son muy homogéneos, lo cual corrobora los resultados obtenidos en el dendrograma y en la estimación de las distancias genéticas y coincide con estudios realizados por los programas de mejoramiento Chilenos y Peruanos.
Sin embargo existe variabilidad genética que puede ser aprovechada en los programas de mejoramiento encaminados a la obtención de nuevos y mejores materiales, adaptados a las condiciones locales y que respondan a las necesidades del agricultor, productor y consumidor.
AGRADECIMIENTOS
Los autores expresan su más cordial agradecimiento a los Laboratorios de Investigación en Biología Molecular, GEBIMOL y BIOPLASMA de la Universidad Pedagógica y Tecnológica de Colombia. Al grupo de Investigación Competitividad, Innovación y Desarrollo Empresarial (CIDE) y al Grupo Manejo Biológico de Cultivos (GMBC).
REFERENCIAS
[1] ZURITA, A., FUENTES, F., ZAMORA, P., JACOBSEN, S. and SCHWEMBER, A. Breeding quinoa (Chenopodium quinoa Willd.): potential and perspectives. Molecular Breeding, 34(1), 2014, p.13-30 [ Links ]
[2] AGRONET. Anuario estadístico del sector agropecuario [online]. 2013. Disponible http://www.agronet.gov.co. [Citado enero de 2016] [ Links ].
[3] FUENTES, F. y PAREDES, X. Perspectivas nutracéuticas de la Quínoa: Propiedades biológicas y aplicaciones funcionales, In: Bazile D, Bertero HD, Nieto C (eds.). Estado del arte de la quinua en el mundo en 2013. Santiago (Chile): FAO-CIRAD, 2014, p. 341-357. [ Links ]
[4] MIRANDA, M., VEGA, A., MARTÍNEZ, E., LÓPEZ, J., MARÍN, R., ARANDA, M. and FUENTES, F. Influence of contrasting environments on seed composition of two quinoa genotypes: nutritional and functional properties. Chilean Journal Agricultural Research, 73(2), 2013, p.108-116. [ Links ]
[5] YANG, A., AKHTAR, S., AMJAD, M., IQBAL, S. and JACOBSEN, S. Growth and Physiological responses of quinoa to drought and temperatures stress. Journal Agronomy and Crop Science, 30(1), 2016, p. 1-9. [ Links ]
[6] ROJAS, W., PINTO, M., ALANOCA, C., GÓMEZ, L., LEÓN, P., ALERCIA, A., DIULGHEROFF, S., PADULOSI, S. and BAZILE, D. Quinua Genetics Resources and ex situ conservation. Chapter 1.5. Roma (Italia): FAO and CIRAD, State of the Art Report of Quinoa in the World in 2015, p. 56-82. [ Links ]
[7] BAZILE, D. Contesting Blossoming Treasures of Biodiversity article 42: 'Quinoa - is the United Nations featured crop of 2013 bad for biodiversity?' - Quinoa, a model crop to examine the dynamics of biodiversity within agricultural systems. Biodiversity, 15(1), 2014, p.3-4. [ Links ]
[8] TORRES, J., VARGAS, H., CORREDOR, G. y REYES, L. Caracterización morfoagronómica de diecinueve cultivares de quinua (Chenopodium quinoa Willd) en la sabana de Bogotá. Agronomía Colombiana, 17(1-3), 2000, p. 61-68. [ Links ]
[9] VÍA, R. y FERNÁNDEZ, R. Determinación de la diversidad genética de 172 accesiones de la colección nacional de Chenopodium quinoa Willd. 'Quinua' mediante marcadores microsatélites [Tesis Licenciada en Biología]. Lima (Perú): Universidad Ricardo Palma, Facultad de Ciencias Biológicas, 2015, p. 97. [ Links ]
[10] JUYÓ, D., SARMIENTO, F., ÁLVAREZ, M., BROCHERO, H., GEBHARDT, C. and MOSQUERA, T. Genetic diversity and population structure in diploid potatoes of group Phureja. Crop Science, 55(2), 2015, p. 760-769. [ Links ]
[11] MORILLO, A., MORILLO, Y., PINZÓN, E. and ÁVILA, I. Molecular characterization of the plum collection (Prunus domestica (L.) Borkh) of the Pedagogical and Technological University of Colombia. African Journal of Biotechnology, 14(3), 2015, p. 257-263. [ Links ]
[12] DOTOR, M., GONZÁLEZ, L., CASTRO, A., MORILLO, A. y MORILLO, Y. Análisis de la diversidad genética de mora en el departamento de Boyacá. Biotecnología en el Sector Agropecuario y Agroindustrial, 14(2), 2016, p. 10-17. [ Links ]
[13] MUÑOZ, J, MORILLO, A. y MORILLO, Y. Microsatélites Amplificados al Azar (RAM) en estudios de diversidad genética vegetal. Acta Agronómica, 57(4), 2008, p. 219-226. [ Links ]
[14] NEI, M. and LI, W.H. Mathematical model for studying genetic variation in terms of restriction endonuclease. Proceedings of the National Academy of Sciences, 76(10), 1979, p. 5269-5273. [ Links ]
[15] ROHLF, F. Numerical Taxonomy and Multivariate Analysis System (NTSYS-pc Version. 2.02. New York (USA): Applied Biostatistics Inc., Exeter Software, 1997. [ Links ]
[16] MILLER, M. Tools for Population Genetic Analyses (TFPGA). A windows program for the analyses of allozyme and molecular population genetic data. Arizona (USA): Department of Biological Sciencies, Northern Arizona University, 1997. [ Links ]
[17] PEAKAL, R. and SMOUSE, P. GenAlex 6.5: Genetic analysis in Excel. Population genetic software for teaching and research. Bioinformatics, 28(19), 2012, p. 2537–2539. [ Links ]
[18] MAUGHAN, P., SMITH, S., ROJAS, J., ELZINGA, D., RANEY, J., JELLEN, E., BONIFACIO, A., UDALL, J. and FAIRBANKS, D. Single nucleotide polymorphism identification, characterization, and linkage mapping in quinoa. Plant Genome 5(3), 2012, p.114–125. [ Links ]
[19] ODUWAYE, A., BARÁNEK, M., CECHOVÁ, J. and RADDOVÁ, J. Reliability and comparison of the polymorphism revealed in amaranth by amplified fragment length polymorphism (AFLPs) and Inter Simple Sequence Repeats (ISSRs). Journal of Plant Breeding and Crop Science, 6(4), 2014, p. 48-56. [ Links ]
[20] OTT, J. Strategies for characteriziting highly polymorphic markers in human gene mapping. American Journal of Human Genetics, 51(2), 1992, p. 283-290. [ Links ]
[21] TAMAYO, H. Caracterización molecular inter e intragenotípica de 16 accesiones de Chenopodium quinoa (quinua) mediante la técnica de ISSR [Tesis de Maestría en mejoramiento genético de plantas]. Lima (Perú): Universidad Nacional Agraria La Molina, 2010, p. 76. [ Links ]
[22] JARVIS, D., KOPP, O., JELLEN, E., MARLLORY, M., PATTEE, J., BONIFACIO, A., COLEMAN, C., STEVENS, M., FAIRBANKS, D. and MAUGHAN, P. Simple sequence repeat marker development and genetic mapping in quinoa (Chenopodium quinoa Willd.). Journal of Genetics, 87(1), 2008, p. 39–51 [ Links ]
[23] WRIGHT, S. Evolution and the genetics of populations, variability within and among natural populations. Chicago (USA): University of Chicago Press, 1978, 566 p. [ Links ]
[24] SPEHAR, C. and ROCHA, J. Exploiting genotypic variability from low-altitude Brazilian savannah-adapted Chenopodium quinoa. Euphytica, 175(1), 2010, p. 13-21. [ Links ]
[25] COSTA, S., MANIFIESTO, M., BRAMARDI, S. and BERTERO, H. Genetic structure in cultivated quinoa (Chenopodium quinoa Willd.) a reflection of landscape structure in Northwest Argentina. Conservation Genetics, 13(4), 2012, p. 1027-1038. [ Links ]
[26] FUENTES, F., BAZILE, D., BHARGAVA, A. and MARTÍNEZ, E. Implications of farmers seed exchanges for on-farm conservation of quinoa, as revealed by its genetic diversity in Chile. The Journal of Agricultural Science, 150(6), 2012, p. 702-716. [ Links ]














