Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Química
Print version ISSN 0120-2804On-line version ISSN 2357-3791
Rev.Colomb.Quim. vol.34 no.1 Bogotá June 2005
Luz Dary Riaño A.1Humberto Miguel Zamora E.2
1Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia, Sede Bogotá.
2Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia, Bogotá, Colombia. Correo electrónico:hmzamorae@unal.edu.co
RESUMEN
Se aislaron y caracterizaron parcialmente proteínas antifúngicas de los espacios intercelulares de hojas de tomate Lycopersicon esculentum cerasiforme, variedad que ha mostrado resistencia en campo a Phytophthora Infestans; se observó que después de inoculación con el patógeno dichas proteínas se acumularon sistémicamente en la planta. Las proteínas identificadas mostraron características de Defensinas de plantas, una nueva familia de proteínas con bajo peso molecular, carga positiva a pH fisiológico y actividad antifúngica evaluada in vitro contra P. infestans. Los análisis electroforéticos en geles de poliacrilamida con SDS-Tricina en condiciones reductoras y no reductoras, sugirieron que están asociadas en trímeros y tetrámeros y poseen pesos moleculares de 5,2 kDa.
Palabras clave:espacios intercelulares, hojas, tomate, proteínas PR (relacionadas con patogenicidad), Defensinas de plantas, actividad antifúngica.
ABSTRACT
Antifungal Proteins from intercellular space of Tomato leaves Lycopersicon esculentum cerasiforme were isolated and partially characterized; this variety had shown resistance against Phytophthora infestans, after inoculating plants with the pathogen it was observed that these proteins were accumulated systemically. The isolated proteins be haved as plant defensins, a novel protein family, with low molecular weight, cationics at physiological pH and Antifungal Activity in vitro evaluated against P. infestans. Electrophoretic analyses in SDS Tricine-PAGE, under reducing and non-reducing conditions suggested that, they were associated as trimers and tetramers; and have molecular weights of 5,2 kDa.
Key words: Intercellular space, leaves, tomato, PR (pathogenesis related) proteins, Plant Defensins, Antifungal activity.
INTRODUCCIÓN
En la periferia de la membrana plasmática ocurren eventos moleculares útiles para estudiar las interacciones planta- patógeno, de allí que se considere el espacio intercelular (1, 2) como el primer sitio de respuesta de la planta al ataque de microorganismos, además de construir una fuente importante de proteínas PR (3, 4, 5). Se han aislado diversas proteínas PR de plantas de tomate, tabaco y papa encontrando en algunas de ellas actividad antifúngica (6) contra microorganismos como Phytophthora infestans (7), Clavibacter michiganensi, Botrytis cinerea y Fusarium oxisporum, los cuales atacan generalmente estos cultivos (8, 9).
Aunque las plantas no poseen un sistema de defensa como el nuestro (10), en condiciones de estrés y en presencia de agentes patógenos producen péptidos catiónicos (11), llamados defensinas con características tan similares a los que poseemos, que se han considerado como si formasen parte de un sistema inmune en ella (10, 12). Las defensinas son péptidos ricos en cisteína, los cuales difieren en longitud, número de cisteínas, enlaces y patrón de plegamiento (13); se han considerado como elementos ancestrales y esenciales de defensa en vertebrados (13), insectos (14) y plantas (15-17).
En plantas las defensinas (18) son péptidos pequeños (5kDa), básicos, con un patrón de plegamiento 3D característico estabilizado por 8 cisteínas enlazadas por disulfuros; su estructura, determinada por RMN, comprende una triple hebra β-laminar con una α-hélice en paralelo (19). En este trabajo se identificó la presencia de posibles defensinas en los espacios intercelulares de hojas de tomate luego de la inducción de resistencia sistémica en las plantas, y se determinó actividad antifúngica contra Phytophthora infestans.
La mayoría de las defensinas de plantas que han sido aisladas provienen de semillas, principalmente de rábano, de tejidos de flores y otras especies, siendo ésta la primera vez que se identifican en hojas de tomate (20-23). En nuestro laboratorio se han identificado y caracterizado para esta variedad de tomate enzimas como β1,3-glucanasas y quitinasas, las cuales fueron consideradas como parte del sistema de defensa de la planta al ataque de P. infestans (24-26).
MATERIALES Y MÉTODOS
Material vegetal
Las semillas de L. esculentum cerasiforme, variedad resistente, fueron suministradas por Corpoica del Banco de Germoplasma Nacional, número de acceso L1568. Las plantas crecieron en condiciones de invernadero; al cabo de 18 semanas se obtuvieron 220 plantas adecuadas para inducir resistencia sistémica en 110 de ellas empleando Phytophthora infestans, las 110 restantes se utilizaron como control.
Los análisis se realizaron en el líquido de lavado intercelular (LLI) obtenido a partir de las hojas.
Propagación del patógeno
El Oomycete P. infestans aislamiento M49, fue suministrado por la profesora Celsa García, directora del Laboratorio de Fitopatología (Universidad Nacional de Colombia). éste se cultivó en medio agar, con arveja y sacarosa 20, 60, 20 g/litro (24-26) y se mantuvo a 20 °C en la oscuridad. Los esporangios de P. infestans se suspendieron en agua destilada desionizada a temperatura ambiente y se ajustó la concentración a 1x104 zoosporas/mL, y así se utilizó para la inoculación de las plantas y para la determinación de actividad antifúngica.
Inducción de resistencia sistémica
Para la inducción (27, 28) de proteínas PR se utilizaron plantas de 4 meses las cuales se inocularon con la suspensión de esporangios de P. infestans aplicándolas sobre algunas de las hojas en forma de goteo y aspersión, finalmente se dejaron incubando por 3 días en oscuridad con una humedad relativa entre 80-90%, y después de 15 días aparecieron los primeros síntomas (28).
Obtención del LLI
Se examinó el líquido de lavado intercelular (29, 30) utilizando dos sistemas de infiltración diferentes: agua destilada a 4 °C y buffer fosfato pH 7 10 mMNaHPO4, 15 mM NaH2PO4, 100 mM KCl, 2 mM EDTA, 0,15% PVP, 5 mMácido ascórbico a 4 °C(31-33) tanto para hojas de plantas sanas como para inoculadas.
Se utilizó la siguiente nomenclatura para la identificación de los extractos: 11A= LLI planta sana extraído con agua, 11B= LLI planta sana extraído con buffer, 21A= LLI planta inoculada extraído con agua, 21B= LLI planta inoculada extraído con búfer.
Determinación de proteína
La proteína se determinó según el método de Bradford modificado por Zor y Selinger (34).
Aislamiento de proteínas de los LLI (35, 36)
La proteína de los extractos (11A, 11B, 21A, 21B) se precipitó con sulfato de amonio hasta un 30% de saturación, a 4 °C, se centrifugó a 7000 g durante 30 minutos a 4 °C; el sobrenadante obtenido se llevó hasta un 75% de saturación y se dejó 2 horas a 4 °C, se centrifugó nuevamente a 7000 g durante 30 minutos a 4 °C, el precipitado se resuspendió con 5 o 6 mL de buffer fosfato de sodio 50 mM, pH 6 se dializó contra éste durante 2 días a 4 °C con cambios cada 8 horas; finalmente se cuantificó la proteína, se obtuvo un patrón electroforético, se realizó el ensayo de actividad antifúngica, se fraccionó en alícuotas y se almacenó a –20 °C.
Purificación y caracterización parcial de las proteínas en 21A y 21B
Cromatografía de tamiz molecular
Las proteínas de los extractos obtenidos a partir de las hojas, y que mostraron en el perfil electroforético la presencia de proteínas con pesos moleculares inferiores a 6,5 kDa (último marcador patrón), se separaron por cromatografía de tamiz molecular empleando una columna de 120 x 1,5 cm empacada con Sephadex G25, utilizando como buffer de equilibrio y de elución fosfato de sodio 50 mM, pH 6; la separación se realizó con un flujo de 18 mL/h, siguiendo la proteína con lecturas de absorbancia a λ=280 nm, λ=250 nm y λ=214 nm, longitudes de onda a las que también absorben cisteínas y los enlaces disulfuros (32). Se determinó proteína para cada ’’pool’’, así como un perfil electroforético para identificar las fracciones con proteínas de bajo peso molecular, y se evaluó la actividad antifúngica en cada caso.
Cromatografía de intercambio aniónico (38)
Las proteínas de las fracciones obtenidas despúes de cromatografia de tamiz molecular (CTM) de los extractos 21A y 21B, que en el perfil electroforético presentaban pesos moleculares bajos, se separaron de acuerdo con su carga empleando una columna de intercambio aniónico Pharmacia 10/16, con un lecho de 10 ml de DEAE Fraktogel TSK 650M®, equilibrada en buffer fosfato de sodio 50 mM pH 6, y las proteínas retenidas se eluyeron con un gradiente lineal NaCl 0-1M a una velocidad de flujo de 16 mL/h.
Las fracciones catiónicas de cada cromatografía se concentraron mediante precipitación con sulfato de amonio hasta 75% de saturación, luego de centrifugación a 7000 g durante 30 minutos a 4 °C, se resuspendieron y dializaron contra buffer fosfato de sodio 50 mM pH 6 a 4 °C durante 2 días cambiándola cada 8 horas, utilizando membranas de 3500 PM; se cuantificó proteína total, se determinó el peso molecular mediante electroforesis SDS-tricina-PAGE en condiciones reductoras y no reductoras, y se evaluó la actividad antifúngica. Las proteínas aniónicas que se eluyeron con el gradiente salino se dejaron para análisis posteriores.
Electroforesis SDS-Tricina-PAGE (38)
La purificación parcial de las proteínas se controló mediante la determinación de los pesos moleculares relativos, empleando un sistema de electroforesis discontinuo con geles de poliacrilamida, en condiciones denaturantes adecuado para péptidos y proteínas en el rango entre 1-100kDa.
Fijación, tinción y decoloración de proteínas
Las bandas de proteínas se fijaron con una solución de ácido tricloroacético 5% por 30 min; la tinción de las bandas se realizó con azul de comassie R en etanol- ácido acético 2:1 por 2 horas. Los geles se decoloraron utilizando etanol: ácido acético: agua 5:5:2 (38).
Determinación de peso molecular
Se empleó el sistema de electroforesis SDS-Tricina-PAGE en geles T 10% C 3%. Las muestras se prepararon en condiciones reductoras empleando β-mercaptoetanol para estimar el peso molecular relativo; las electroforesis en condiciones no reductoras se realizaron para establecer la posible formación de trímeros y tetrámeros (38).
Ensayo de antifungicidad
Ensayo microespectrofotométrico
La actividad antifúngica se midió espectrofotométricamente de acuerdo con trabajos anteriores (39) empleando cajas para microtitulación de 96 pozos y un lector de micro placas Biorad modelo 680.
El método consiste básicamente en tomar medidas de absorbancia a 595 nm de microcultivos del organismo que se va a evaluar y una dosis de la proteína de la que se presuma posee actividad inhibidora de crecimiento para el microorganismo. Los microcultivos de control contenían 20 µL de la muestra protéica para ensayo en condiciones estériles y 80 µL de la suspensión de esporangios, inactivados por el calentamiento durante 10 minutos en baño de agua en ebullición. Las placas de microcultivos se incubaron a temperatura ambiente durante 48 horas en la oscuridad. El porcentaje de inhibición de crecimiento se definió así (39): Ec. 1
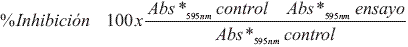
Bioensayo con azul de Lactofenol (40)
La actividad de los extractos crudos y de las fracciones proteicas obtenidas durante el crecimiento de P. infestans se evaluó empleando placas de microtitulación de 96 pozos con 20 µL de muestra y 80 µLde una suspensión de esporangios del microorganismo, en medio de cultivo líquido con arveja y sacarosa; luego de 5 días de incubación se tiñeron los micelios con azul de lactofenol y se observaron al microscopio de luz.
RESULTADOS Y DISCUSIÓN
Inducción de resistencia sistémica
Del número de plantas inoculadas, el 93% presentó sintomatología de infección en hojas, no se observaron daños ni señales de infección en flores, tallo ni frutos en ninguna de las plantas tratadas, evidenciándose la tolerancia de la variedad de tomate al ataque del patógeno, teniendo en cuenta que no hubo destrucción del cultivo.
Extracción del LLI
Los dos tipos de extractos del LLI obtenidos,uno empleando agua destilada a 4 °C y otro con buffer fosfato de sodio pH 7+, se realizaron teniendo en cuenta las características catiónicas y altamente básicas de las defensinas a pH fisiológico, se utilizó una fuerza iónica alta para solubilizarlas.
Las condiciones iniciales de los cuatro LLI obtenidos se resumieron en la Tabla 1, donde se aprecia que la cantidad de LLI de plantas inoculadas extraído con buffer es mayor que aquel de plantas sanas, lo mismo sucedió con los extraídos con agua; de igual forma la cantidad proteína total fue mayor en plantas inoculadas que sanas.
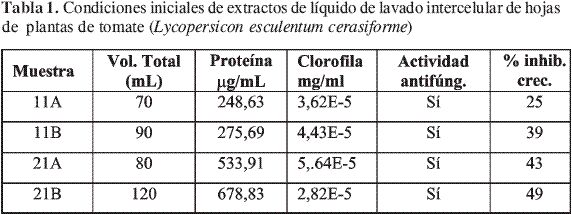
En los LLI obtenidos con buffer el porcentaje de inhibición fue mayor en 21B (49%) que en 11B (39%), y en los extraídos con agua fue mayor en 21A (45%) que en 11A (25%), sugiriendo que en las plantas inoculadas la actividad aumentó luego de la infección, comparada con la de las plantas sanas; se presentó mayor actividad en los extractos con buffer, indicando que con este sistema se logró extraer mayor cantidad de ‘‘compuestos antifúngicos’’ que con agua (Figura 1).
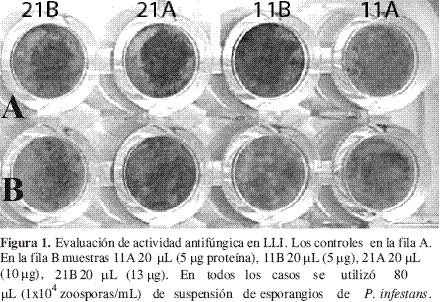
Aislamiento de proteína
La cantidad de proteína en los LLI, durante la precipitación con sulfato de amonio hasta un 30% de saturación no fue considerable, comparada con la del 70%; tampoco hubo actividad antifúngica ya que los porcentajes de inhibición presentaron valores negativos en todos los casos, indicando que no hubo inhibición de crecimiento y que por el contrario, P. infestans continuó creciendo en el medio; éstos resultados permitieron asegurar la continuación de las proteínas antifúngicas en la fracción del 70%, como se indica en la Tabla 2.
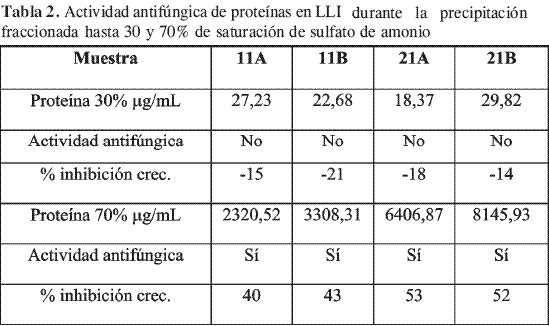
Los porcentajes de inhibición de crecimiento más altos correspondieron a los extractos 21A y 21B, confirmando una inducción de respuesta de defensa de las plantas al ataque de Phytophthora infestans luego de inoculadas.
La actividad antifúngica que presentaron los extractos 11A y 11B, correspondientes a las plantas sanas, sugirió que la planta posee su propio mecanismo de defensa y que en condiciones de patogenicidad éste se refuerza, confiriéndole mayor resistencia a la enfermedad (resistencia sistémica).
Los porcentajes de inhibición provienen de la aplicación de la ecuación 1 con las lecturas realizadas a 595 nm, de los controles y las muestras a los 30 minutos y a las 48 horas después de iniciado el ensayo, así: los valores negativos y menores de 10% indican no antifungicidad, y los valores inferiores a 20% y superiores a 10% indican actividad deficiente.
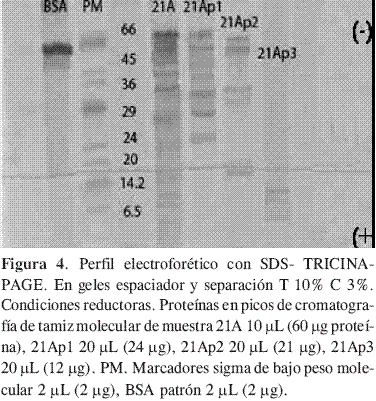
El perfil electroforético en la Figura 2 mostró zonas enriquecidas de proteína en todos los casos; los carriles correspondientes a las plantas inoculadas, 21A y 21B presentaron proteínas de peso molecular inferiores a 6,5 kDa (asignado para el patrón de PM más bajo en el marcador), mientras que en 11A y 11B no se apreciaron; del mismo modo el número y la intensidad de las bandas fue menor, aun cuando la cantidad de proteína dispuesta en todos los pozos fue similar. (Pie de Figura 2). Teniendo en cuenta que se buscaban proteínas de bajo PM se decidió purificar las que estaban presentes en los extractos 21A y 21B.

Purificación parcial de proteínas en 21A
Cromatografía de tamiz molecular
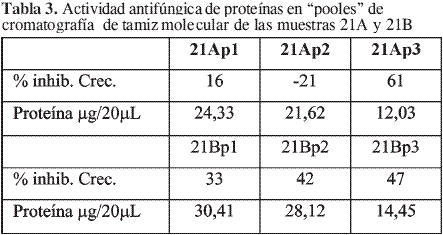
En el perfil cromatográfico en la Figura 3, las proteínas en el extracto 21A se separaron en tres picos, denominados 21Ap1, 21Ap2 y 21Ap3 respectivamente. Se observó (Figura 4) que en el primero se aislaron proteínas con pesos moleculares entre 24 y 66 kDa en relación con los patrones de PM. El segundo pool 21Ap2 contenía proteínas superiores a 14kDa diferentes a 21Ap1, y en la fracción denominada 21Ap3 proteínas de pesos moleculares relativos inferiores a 14kDa en las cuales se identificó inhibición de crecimiento de P. infestans superior al 50%. Mientras que en las otras dos fracciones no se apreció este tipo de actividad (Tabla 3).
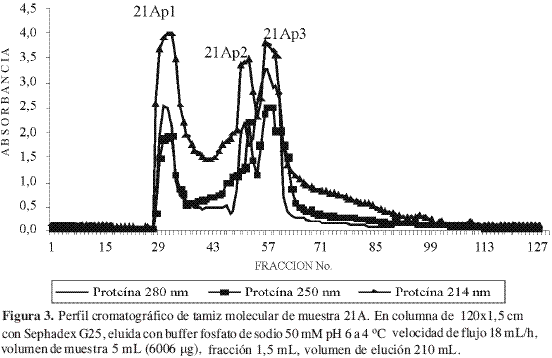
Cromatografía de intercambio aniónico de 21Ap3
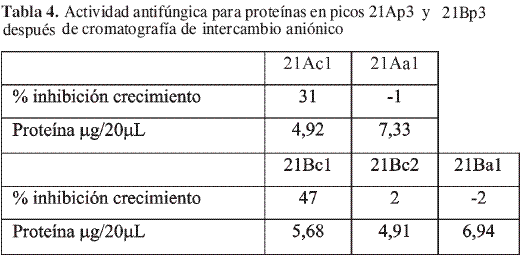
La separación de las proteínas de acuerdo con su carga en la fracción 21Ap3 proveniente de CTM mostró 3 picos; el primero representa un grupo de proteínas básicas y se denominó 21Ac1, las proteínas ácidas se designaron 21Aa1 y 21Aa2, respectivamente, se evaluó la actividad antifúngica para las fracciones 21Ac1 y 21Aa2 de acuerdo con el interés del estudio hacia las proteínas catiónicas de bajo peso molecular, y se establecieron diferencias en cuanto a actividad antifúngica entre los dos tipos de proteína (21Ac1 y 21Aa2) de diferente carga (Figura 5 y Tabla 3).
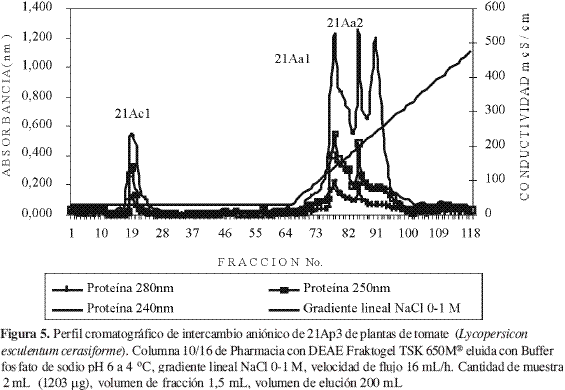
Hubo inhibición de crecimiento del 31% en la fracción 21Ac1, en la aniónica (21Aa2) se obtuvo un porcentaje negativo (-1%) indicando actividad nula. De aquí se concluyó que en el extracto 21A existían proteínas catiónicas, de pesos moleculares relativos inferiores a 14kDa y con actividad antifúngica in vitro contra Phytophthora infestans del orden de 31% de inhibición con cerca de 5 g de proteína.
Purificación parcial de proteínas en 21B
Cromatografía de tamiz molecular
Luego de la separación por tamiz molecular (Figura 6) se encontraron proteínas con pesos moleculares inferiores a 14 kDa en 21 Bp3; en las fracciones 21Bp2 y 21Bp1 se separaron moléculas con PM superiores a 20000 Da, presentándose el mayor número de éstas en 21Bp1 (Figura 7). Nuevamente, dado que se buscaban proteínas de bajo peso molecular, se tomó el pool 21 Bp3 para realizar la separación de proteínas por carga y su posterior evaluación de actividad antifúngica.
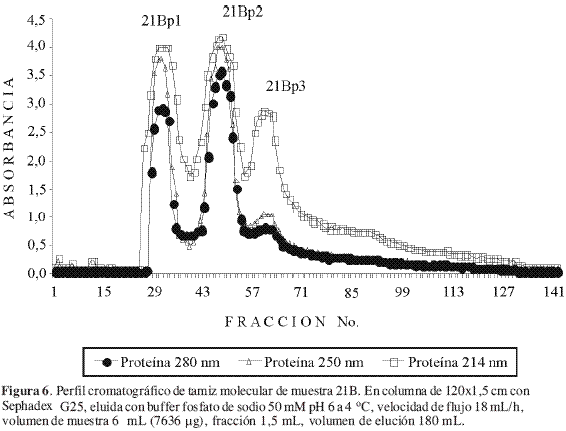

En todos los ‘‘pooles’’ hubo inhibición de crecimiento visto como una disminución en la intensidad de color de micelios de P. infestans teñidos con el azul de lactofenol; el porcentaje de inhibición fue mayor para 21Bp3 (Tabla 3), por lo tanto, se pudo confirmar que la extracción de las proteínas con actividad antifúngica y bajos pesos moleculares, en el LLI de hojas de tomate resistente, en las que se había inducido resistencia sistémica, fue mejor con buffer fosfato pH 7 que aquella realizada con agua destilada únicamente.
Cromatografía de intercambio aniónico de 21Bp3 (Figura 8)
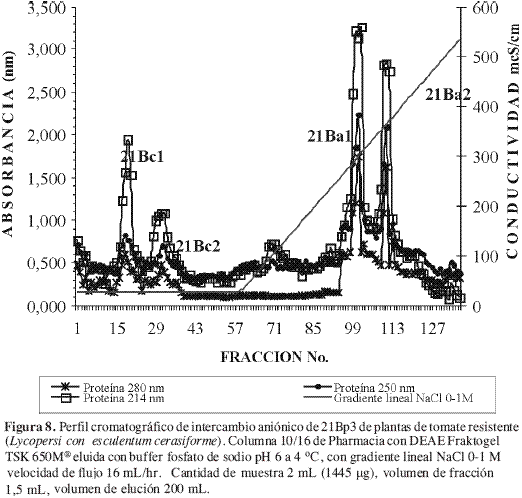
Se observaron 4 picos correspondientes a cuatro tipos de proteínas separadas de acuerdo con su carga; en ellos se encuentran 2 grupos básicos, 21Bc1, 21Bc2, y dos ácidos 21Ba1, 21Ba2.
Al evaluar la actividad se encontró que 21Bc1 inhibió el crecimiento in vitro de P. infestans, con un 47,2%, en 21Bc2 se esperaba un comportamiento similar, pero no presentó actividad antifúngica (Tabla 4). Con la fracción 21Ba1 se obtuvo un valor negativo en porcentaje, por lo que se pudo afirmar que en el LLI de hojas de tomate inoculadas extraído con buffer fosfa0to pH 7 (21B), se encontraron 2 grupos de proteínas catiónicas (21Bc1 y 21Bc2) de bajo peso molecular, de las cuales sólo uno poseía actividad antifúngica.
Luego de aislar las proteínas catiónicas de bajo peso molecular con actividad antifúngica de los extractos 21Ay 21B, se procedió a evaluar otras características propias de las defensinas que sirvieron como método de identificación parcial. Durante todo el estudio se realizaron electroforesis en condiciones denaturantes en geles de poliacrilamida, con el sistema SDS-Tricina- PAGE, con el fin de hacer un seguimiento de las proteínas de bajo peso molecular; en esta sección se utilizaron dichas electroforesis para determinar el peso molecular de las proteínas presentes en las fracciones obtenidas a partir de las cromatografías de intercambio aniónico analizadas anteriormente.
Determinación de peso molecular en condiciones reductoras (Figura 9)

Se encontraron proteínas catiónicas de peso molecular 5,2 kDa en los extractos 21A y 21B, algunas otras con masas de 7,7 kDa de las cuales estas últimas tienen menor posibilidad de ser defensinas (proteínas de 5 kDa).
De acuerdo con los resultados que se muestran en la Tabla 5, en la fracción proteíca denominada 21Ac1 del LLI de plantas inoculadas extraído con agua (21A), se encontró una proteína catiónica de peso molecular relativo de 5,2 kDa, la cual posee actividad antifúngica in vitro contra P. infestans. Por otro lado, en la fracción 21Aa1 del mismo LLI, se encontraron 3 proteínas aniónicas con pesos moleculares diferentes; 13,8, 15,2 y 16,8 kDa, las cuales no poseen actividad antifúngica y su porcentaje de inhibición de crecimiento fue negativo.
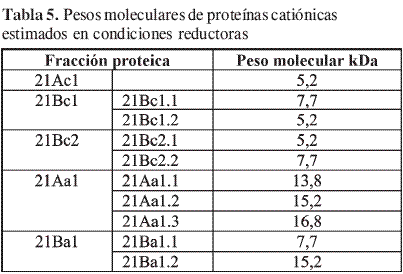
En las fracciones 21Bc1 y 21Bc2 del LLI de plantas inoculadas extraído con bufffer (21B), se encontraron dos proteínas de pesos 5,2 y 7,7 kDa, sugiriendo que éstas pertenecen a ambas fracciones; es probable que cada fracción sea una proteína y contenga trazas de la otra, ya que de acuerdo con la actividad antifúngica evaluada, 21Bc2 no presentó dicha actividad mientras que 21Bc1, sí. ¿Cuál de las dos proteínas confiere la actividad? éste es un cuestionamiento por resolver, habría que separarlas totalmente para evaluar su actividad individual y establecer la diferencia.
Determinación de peso molecular en condiciones no reductoras (Figura 10)

En cuanto a la fracción 21Ba1 perteneciente al mismo extracto anterior se encontraron 2 proteínas aniónicas con pesos moleculares de 7,7 y 15,2 kDa; de acuerdo con las observaciones anteriores dicha fracción no presentó actividad antifúngica.
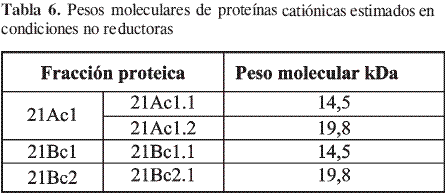
Las proteínas no reducidas proporcionaron bandas finas; para 21Ac1 y 21Bc2 (Tabla 5 y Tabla 6) se apreciaron dos bandas con una masa molecular aparente de 14,2 y 19,8 kDa en ambos casos. En 21Bc1 y en 21Bc2 se encontró una banda con peso molecular estimado de 14,2 y otra de 19,8 kDa respectivamente. Los pesos moleculares de las proteínas evaluadas antes en condiciones reductoras sugirieron que estas proteínas surgieron como oligómeros constituidos por protómeros de aproximadamente 5 kDa; de ser así estas estructuras involucraron los puentes disulfuro entre cisteínas intactas por la ausencia del agente reductor. Las bandas de 14,5 kDa representarían un trímero (en 21Ac1, 21Bc1), mientras que las de 19,8 kDa (en 21Ac1, 21Bc2) un tetrámero.
Morfología de inhibición de crecimiento de P. infestans
Se tomaron microfotografías de algunas de las pruebas realizadas en las placas de microtitulación empleando un microscopio de luz con objetivo 100X y se observó el deterioro del saco esporángico de P. infestans; éstas fueron tomadas 96 horas después de la incubación de la suspensión de esporangios de Phytophthora infestans de 1x104 células/ml en medio líquido de arveja y sacarosa, en presencia de 20l 21Ac1 (4,92g), 20l 21Bc1 (5,68g), 20l 21Bc2 (4,91 g), 10 minutos antes de tomar la microfotografía, se adicionó azul de metileno 0,01% a los microcultivos para hacer contraste. Se observaron fragmentos de micelio cenocítico (no septado) característico de P. infestans luego de una degradación (Figura 11).
Las observaciones anteriores permitieron establecer el efecto antifúngico contra el crecimiento de Phytophthora infestans, producido por las proteínas catiónicas de bajo peso molecular, aisladas de los espacios intercelulares, de hojas inoculadas de tomate resistente (Lycopersicon esculentum cerasiforme) encontradas en este trabajo.
CONCLUSIONES
Se obtuvieron tres fracciones con cuatro proteínas catiónicas provenientes de los espacios intercelulares de hojas de plantas de tomate resistente con peso molecular bajo y actividad antifúngica con un alto porcentaje de inhibición in vitro frente a P. infestans, características que permiten denominarlas como defensinas de plantas.
Se establecieron las condiciones para aislar y purificar parcialmente posibles defensinas de los espacios intercelulares de plantas de tomate, ya que las investigaciones realizadas hasta este momento habían involucrado tejidos como semillas y el endospermo de diversas especies, y no existía ningún trabajo publicado que mostrara la presencia de tales proteínas en hojas de tomate.
Las proteínas localizadas en los espacios intercelulares –y caracterizadas como posibles defensinas en este estudio, mostraron actividad antifúngica frente a P infestans, lo cual muestra que dichas proteínas están implicadas en el mecanismo de defensa de la planta y en la resistencia en campo que se ha observado en esta variedad de tomate. Es la primera vez que luego de inducción de resistencia sistémica se tienen indicios de la presencia de defensinas en plantas de tomate, especialmente en las hojas y aún más en una variedad perteneciente a nuestro Germoplasma Nacional.
Agradecimientos: Los autores agradecen a la Universidad Nacional de Colombia – Facultad de Ciencias por facilitar las instalaciones para el desarrollo de este trabajo, a la Deutschen Gesellschaf für Technische Zusammenarbeit (GTZ) y a la World University Service (WUS) por la donación de algunos de los equipos y reactivos, y a la División de Investigaciones Básicas (DIB) por la financiación este proyecto.
BIBLIOGRAFÍA
1. Cervone, F.; Hahn, M. G.; De Lorenzo,G.; Albersheim, P. (1989). Host-Pathogen Interactions. Plant Physiol. 90, 542-548. [ Links ]
2. Barón, C.; Zambryski C. (1995).The Plant response in Pathogenesis, symbiosis and wounding variations on a common theme. Ann. Rev. Gen. 29, 107-129. [ Links ]
3. Bowles, D. J. (1990). "Defense-related proteins in higher plants". Ann. Rev. Biochem. 59, 873-907. [ Links ]
4. Ryals, J. A.; Uknes, S.; Ward, E. (1994). Systemic Acquired Resistance. Plant Physiol. 104, 1109-1112. [ Links ]
5. Kessmann, H.; Staub, T.; Hofmann, C.; Maetske, T.; Herzog, J. (1994). Induction of Systemic acquired disease resistance in plants by chemicals. Ann. Rev. Phytopathol. 32, 439-459. [ Links ]
6. Woloshuk, C. P.; Meulenhoff, J. S.;Sela-Buurlage, M.; Cornelissen, B. J. C. (1991). Pathogen-Induced Proteins with Inhibitory Activity toward Phytophthora infestans. Plant Cell. 3, 619-628. [ Links ]
7. Niderman, T.; Genett, I.; Bruyere, T. (1995). Pathogenesis-related PR-1 proteins are antifungal (isolation and characterization of three 14 kDa proteins of tomato and of a basic PR-1 of tobacco with inhibitory activity against Phytophthora infestans. Plant. Physiol. 108, 17-27. [ Links ]
8. Nielsen, K. K.; Nielsen, J. E.; Madrid, S. M.; Mikkelsen, J. A. (1990). New antifungal proteins from sugar beet (Beta vulgaris L.) showing homology to non- specific lipid transfer proteins. Plant Mol. Biol. 31, 539-552. [ Links ]
9. Kristensen, A. K.; Brusted, J.; Nielsen, J. E.; Mikkelsen, J. D.; Nielsen, K. K. (2000). Partial characterization and localization of a novel type of antifungal proteins isolated from sugar beet Leaves. Plant Sci. 159, 29-38. [ Links ]
10. Salisbury, F. B.; Ross, C. W. (1994). Fisiología Vegetal. Grupo editorial Iberoamericana, pp. 395-449. [ Links ]
11. Selitrennikoff, C. P. (2001). Antifungal Proteins. Appl. Env. Microb. 67, 2883-2894. [ Links ]
12. Van Loon, L. C.; Strien Van, E. A. (1999). The families of pathogenesis- related proteins, their activities, and comparative analysis of PR-1 type proteins. Plant. Pathol. 55, 85-97. [ Links ]
13. Ganz, T.; Lehrer, R. I. (1994). Defensins. Curr. Opin. Immunol. 6, 584-589. [ Links ]
14. Lamberty, M.; Ades, S.; Uttenweiler,S.; Brookhart, G.; Bushey, D.; Hoffmann, A. (1999). Insect immunity. Isolation from the Lepidopteran Heliothis virescens a novel insect defensin with potent antifungal activity. J. Biol. Chem. 274, 9320-9326. [ Links ]
15. Broekaert, W. F.; Terras, F. R. G.; Cammue, B. P. A.; Osborn, R.W. (1995). Plant defensins: Novel antimicrobial peptides as components of the Host defense system. Plant Physiol. 108, 1353-1358. [ Links ]
16. Thomma, B. P. H. J.; Cammune, B. P. A.; Thevissen, K. (2002). Plant Defensins. Planta. 216, 193-202. [ Links ]
17. Terras, F. R. G.; Torrekens, S.; Van Leuven, F.; Osborn, R. W.; Cammune, B. P. A.; Broekaert, W. F. (1993). A new family of basic cysteine- rich plant antifungal proteins from Brassicaceae species. FEBS Letter 316, 233-240. [ Links ]
18. Terras, F. R. G.; Schoofs, H. M. E.; De Bollet, M. F. C.; Van Leuven, F.; Cammune, B. P. A.; Broekaert, W. F. (1992). Analysis of two novel classes of Plant Antifungal proteins from Radish (Raphanus sativus L.) seeds. J. Biol. Chem. 267, 15301-15309. [ Links ]
19. Almeida, M. S.; Cabral, K. M.; Kurtenbach, E. (2002). Solution structure of Pisum sativum defensinby high resolution 1NMR: plant defensins, identical backbone with different mechanisms of action. J. Mol. Biol. 315, 749-757. [ Links ]
20. Terras, F. R. G.; Eggermont, K.; Kovalera, V.; Ralkhel, N. V.; Osborn, R. W.; Kester, A.; Torrekens, S. (1995). Small Cysteine- Rich antifungal proteins from Radish: Their role in host defense. Plant Cell. 7, 573-588. [ Links ]
21. Osborn, R. W.: De Samblanx, G. W.; Thevissen, K.; Goderis, I.; Van Leuven, F.; Broekaert, W. F. (1995). Isolation and characterisation of plant defensins from seeds of Asteraceae, Fabaceae, Hipp.ocastanaceae and Saxifragaceae.FEBS Letters 368, 257-262. [ Links ]
22. Cammune, B. P. A.; De Bollet, M. F. C.; Terras, F. R. G.; Proost, P.; Van Damme, J.; Broekmert, W. F. (1992). Isolation and characterization of a Novel Class of Plant Antimicrobial Peptides from Mirabilis jalapa L. Seeds. J. Biol. Chem. 267,2228-2233. [ Links ]
23. Van den Bergh, K. P. B.; Proost, P.; Van Damme, J.; Cooseman, J. (2002). Five disulfide bridges stabilize a hevein-type antimicrobial peptide from the bark of spindle tree (Euonymus europeus L.). FEBS letter. 530, 181-185. [ Links ]
24. Moreno, Y. (2000). Análisis diferencial de las 1,3-glucanasas presentes en plantas de tomate Lycopersicon esculentum cerasiforme y su patógeno Phytophthora infestans. Trabajo de grado. Químico. Universidad Nacional de Colombia - Bogotá. [ Links ]
25. Ceron, L. (2000). Determinación y análisis comparativo de proteínas relacionadas con la patogénesis (PR) de los espacios intercelulares de plantas de tomate resistente (Lycopersicon esculentum cerasiforme) y susceptible (Lycopersicon esculentum Mill) antes y después de la infección con Phytophthora infestans (Mont de Bary. Trabajo de grado. Químico. Universidad Nacional de Colombia- Bogotá. [ Links ]
26. Velásquez, D. E. R. (2003). Caracterización parcial de Quitinasas en los espacios intercelulares de plantas de tomates resistentes (Lycopersicon esculentum cerasiforme) y susceptibles (Lycopersicon esculentum Mill) a Phytophthora infestans. Trabajo de grado. Químico Universidad Nacional de Colombia - Bogotá. [ Links ]
27. Youn Yi, S.; Kook, H. B. (1996). Diferenctial induction and accumulation of -1,3-glucanase and chitinase isoforms in soybean hypocotyls and leaves after compatible and incompatible infection with Phytophthora megasperma f.sp. glycinea. Physiol. Mol. Plant. Pathol. 48, 179-192. [ Links ]
28. Anfoka, G.; Buchenauer, H. (1997). Systemic acquired resistance in tomato against Phytophthora infestans by pre-inoculation with tobacco necrosis virus Physiol. Mol. Plant. Pathol. 50, 85-101. [ Links ]
29. Rohringer, R.; Ebrahim-Nesbat, F. (1983). Proteins in intercellular Washing Fluids from Leaves of Barley (Hordeum vulgare L.). J. Exp. Bot. 34, 1589-1605. [ Links ]
30. Hammond-Kosack, K. E. (1983). Preparation and analysis of intercellular fluid. Plant Physiol. 72, 326-332. [ Links ]
31. Kroll, K. A.; Brunstedt, J.; Nielsen, J. E.; Nielsen, K. K. (2000). Partial characterization and localization of a novel type of antifungal protein in IWF from sugar beet leaves. Plant Sci. 159, 29-38. [ Links ]
32. Cammue, B. P. A.; De Bolle, M. F. C.; Terras, F. R. G.; Proost, P.; Van Damme, J.; Broekaert, W. F. (1992). Isolation and characterization of a novel class of Plant antimicrobial Peptides from Mirabilis jalapa L. seeds. J. Biol. Chem. 267, 2238-2233. [ Links ]
33. Terras, F. R. G.; Torrekens, S.; Van Leuven, F.; Osborn, R. W.; Vanderleyden, J.; Cammue, B. P. A.; Broekaert, W. F. (1993). A new family of basic cysteine-rich plant antifungal proteins from Brassicaceae species. FEBS letter 316, 223-240. [ Links ]
34. Zor, T.; Selinger, Z. (1996). Linearization of the Bradford Protein assay increases its sensitivity: Theoretical and experimental Studies. Anal. Biochem. 236, 302-308. [ Links ]
35. Van Jagow, G.; Schägger, H. (1994). A practical guide to membrane protein purification. Academic Press, San Diego, USA, pp. 3-21. [ Links ]
36. Linn, S. (1990). Strategies and considerations for Protein Purifications, in Methods in Enzimology, vol. 142, Academic Press, pp. 9-83. [ Links ]
37. Morris, C. J. O. R.; Morris, P. (1976). Ion-Exchange chromatography, in Separation Methods in Biochemistry, Pitman Publishing, pp. 251-357. [ Links ]
38. Schagger, H.; Von Jagow, G. (1987). Tricine-Sodium dodecyl Sulfate- Polyacrylamide Gel Electrophoresis for the separation of proteins in the Range from 1 to 100 kDa. Anal. Biochem. 166, 368-379. [ Links ]
39. Terras, F. R. G.; Schoofs, H. M. E.; De Bollet, M. F. C., Van Leuven, F.; Vanderleyden, J.; Cammue, B. P. A.; Broekaert, W. F. (1992). Analysis of two novel classes of plant antifungal proteins from radish (Raphanus sativus L.) Seeds. The J. Biol. Chem. 267, 15301-15309. [ Links ]
40. Woloshuk, C. P.; Meulenhoff, J. S.; Sela-Buurlage, M.; Van den Elzen, P. J.; Cornelissen, B. J. C. (1991). "Pathogen-induced proteins with Inhibitory Activity toward Phytophthora infestans" Plant cell. 3: 619-628. [ Links ]














