Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Vitae
Print version ISSN 0121-4004
Vitae vol.17 no.2 Medellín May/Aug. 2010
FARMACOLOGÍA Y TOXICOLOGÍA
PÉPTIDOS CON ACTIVIDAD ANTIMICROBIANA PRODUCIDOS POR MICROORGANISMOS NATIVOS
PEPTIDES WITH ANTIMICROBIAL ACTIVITY PRODUCED BY ISOLATED NATIVE MICROORGANISMS
Ligia L. CORRALES-GARCÍA.1*; Gelmy L. CIRO G.1
1Facultad de Química Farmacéutica. Universidad de Antioquia. AA 1226. Medellín Colombia. 2Programa de Ofidismo/Escorpionismo. Universidad de Antioquia. Carrera 53 # 61 – 30, Lab. 631 Torre 2. Medellín Colombia.
RESUMEN
Al igual que las proteínas, los péptidos antimicrobianos (PAM) son moléculas versátiles sintetizadas por microorganismos siguiendo rutas enzimáticas, y con interesantes propiedades alimentarias y farmacéuticas, entre ellas la capacidad antibiótica sobre patógenos. La búsqueda convencional de biomoléculas provenientes de microorganismos consiste en analizar químicamente su extracto crudo, lo cual es engorroso y prolongado. El método de aislamiento usado en esta investigación permitió localizar microorganismos con capacidad para producir PAM por interacción de cargas entre moléculas generadas en el metabolismo microbiano y un colorante cargado presente en el medio de cultivo. Se aislaron 20 muestras de suelos de varios pisos térmicos en medios selectivos. Se purificaron 35 cepas con base en la interacción con un colorante básico y se identificaron microorganismos del género Streptomyces sp. y Bacillus sp. Se obtuvieron proteínas de los extractos crudos de fermentaciones, identificando péptidos y aminoácidos por cromatografía de capa fina y electroforesis. Aquellos extractos con alto contenido proteico se evaluaron por bioautografía, y se encontraron 2 extractos de 35 con actividad inhibitoria sobre E. coli ATCC 8739 (halos de 8 mm). Se demuestra la efectividad del método para aislar y purificar microorganismos productores de péptidos cargados y que poseen actividad biológica e industrial de gran interés.
Palabras Clave:péptidos catiónicos antimicrobianos, microbiología del suelo, cromatografía en capa delgada.
ABSTRACTLike proteins, antimicrobial peptides (AMP) are versatile molecules synthesized by microorganisms using enzymatic pathways with no genetic code instruction. AMP have interesting properties in the food and pharmaceutical industries, like their antimicrobial ability against pathogens. Looking for biomolecules from microorganisms requires hard and time consuming chemical analysis of each microorganism extract. The microorganism isolation method proposed in this research allowed us to find antimicrobial peptides produced by bacteria, through interaction between a charged dye mixed with selective agar and metabolites produced by microorganisms. Twenty soil samples from different zones were isolated in selective media; thirty five strains were purified based on interaction between basic dye and charged molecules from bacteria. Streptomyces sp. y Bacillus sp. both genera were identified. Protein extracts were obtained from the isolated microorganisms cultivated in liquid media; peptides and amino acids were identified by thin layer chromatography and electrophoresis. Those extracts with high protein level were used to evaluate bioautography. Two extracts from 35 showed inhibitory activity against E. coli ATCC 8739 (8 mm halo). Method effectiveness for the isolation and the purifying of microorganisms able to produce charged molecules, of industrial interest is demonstrated.
Keywords: antimicrobial cationic peptides, soil microbiology, thin layer chromatography.
INTRODUCCIÓN
Los péptidos antimicrobianos, frecuentes en plantas, insectos y mamíferos (1, 2), son una familia de péptidos antimicrobianos catiónicos endógenos, con un amplio espectro de actividad sobre bacterias Gram positivas, Gram negativas y varios hongos patógenos (incluyendo los resistentes a azoles) (3, 4). Desde la pasada década se han descubierto cientos de péptidos antimicrobianos (PAM) naturales, generados por microorganismos, y con un amplio rango de acción contra otros microorganismos (5, 6). Se caracterizan por tener de 15 a 34 residuos de aminoácidos en su secuencia y un considerable número de lisinas o argininas que los convierten en péptidos antimicrobianos catiónicos (5, 7). En este grupo pueden encontrarse moléculas como defensinas, cecropinas, magaininas, melitinas, e-poli-L-lisina, polimixinas, g-poli-L-glutamato y cianoficina (6, 8-10). Su función es permitirle al organismo productor defenderse activamente del ataque de patógenos, haciendo parte así de su inmunidad innata (6, 11).
Los PAM pueden ser sintetizados de dos maneras. La primera sigue la traducción del mensaje génico por medio de ribosomas (6) y se presenta en todas las especies; ejemplos de estas proteínas antibióticas son la lisozima (12) y las β-defensinas (3, 13, 14). La segunda forma de síntesis no sigue las instrucciones directas del código genético para producir el péptido antimicrobiano, sino que usa rutas enzimáticas biosintéticas, como es el caso de algunos PAM de origen bacteriano, por ejemplo, la e-poli-L-lisina (6, 15-17). A éstos últimos péptidos también se les conoce como poliamidas o poliaminoácidos, para diferenciarlos de las proteínas, debido a sus características diferentes de biosíntesis, e incluyen una gran diversidad de compuestos poliméricos que tienen en común la presencia del enlace tipo amida (18). Ciertos microorganismos llevan a cabo la síntesis de los péptidos no ribosomales o poliaminoácidos, realizando ensambles consecutivos de aminoácidos por medio de un gran complejo enzimático multifuncional tipo sintetasa (catalizan la unión carbono–nitrógeno, E.C. 6.3.2.). La enzima puede contener de 4 a 6 dominios, y cada dominio tiene la capacidad de reconocer un residuo, activarlo, modificarlo si es necesario y agregarlo a la cadena peptídica naciente. Este mecanismo básico da como resultado una gran variedad de productos peptídicos y puede llegar a contener residuos inusuales, L– ó D–aminoácidos (6, 18).
Se conocen tres tipos de poli-aminoácidos producidos específicamente por microorganismos aislados de diferentes ambientes: ε-poli-Lglutamato (producido por: Bacillus amyloliquefaciens, B. licheniformis, B. megaterium, B. subtilis) (18, 19), ε-poli-L-lisina (producido por Streptomyces albulus) (20-23), y la cianoficina (producido por especies de cianobacterias) (18); sin embargo, pueden existir otros poli-aminoácidos que aún no han sido hallados (18-21).
Actualmente, la ε-poli-L-lisina es producida y comercializada a nivel biotecnológico (Chisso Corparation, Japón) y usada como aditivo en alimentos por su acción antibacteriana sobre un amplio espectro de microorganismos (bacterias Gram positivas, Gram negativas, levaduras y mohos) (24). Es inocua para los humanos (25), soluble en agua, estable a altas temperaturas y tiene bajo impacto ambiental por ser biodegradable (20, 21, 26). Por tales propiedades, es usada como conservante natural de alimentos tales como pescados, vegetales, sopas, arroz cocido, pastas, leche, entre otros (26). En 2004, la FDA declara, en referencia a ε-poli-Llisina, no tener cuestionamientos a las investigaciones y resultados del fabricante y, por tanto, la declara como GRAS (25).
La búsqueda de microorganismos con capacidad para generar biomoléculas antibacterianas de interés industrial consiste en evaluar individualmente las sustancias excretadas por el microorganismo en el medio. Pero si el objetivo es realizar la búsqueda en microorganismos del suelo, el método de detección sería un proceso engorroso y llevaría gran cantidad de tiempo. Para el proceso de detección de microorganismos productores de compuestos de naturaleza proteica se utiliza comúnmente el método de precipitación del reactivo Dragendorff en cada extracto libre de células de cada uno de los microorganismos aislados (21). En esta investigación se pretendió detectar la presencia de biomoléculas de naturaleza peptídica y catiónica, generadas por microorganismos presentes en suelos del Departamento de Antioquia, Colombia, por medio de la interacción entre dichas moléculas excretadas al medio por el microorganismo y colorantes ácidos o básicos adicionados en el medio (20). Gracias a esta metodología es posible realizar amplios screenings en medios sólidos y evaluar la capacidad antibacteriana de las biomoléculas detectadas, de naturaleza peptídica, sobre microorganismos considerados como patógenos
MATERIALES Y MÉTODOS
Microorganismos
Los siguientes microorganismos fueron usados para evaluar la actividad antimicrobiana de los extractos de naturaleza peptídica obtenidos en esta investigación: Escherichia coli ATCC 8739; Staphylococcus aureus ATCC 6538 y Candida albicans ATCC 10231; sus curvas de crecimiento se determinaron empleando el medio Müeller-Hinton (MH), (Difco®, U.S.A.).
Medios de cultivo
Para el aislamiento de los microorganismos productores de moléculas de naturaleza peptídica se utilizaron medios selectivos a los que se adicionó un colorante de naturaleza ácida o básica, de manera que la selección se llevó a cabo con base en la interacción electrostática entre las moléculas generadas por el microorganismo y el colorante. Los medios usados fueron: (a) medio glicerol (composición por litro: glicerol 50 g (J.T. Baker®, U.S.A.); sulfato de amonio 10 g (J.T. Baker®, U.S.A.); extracto de levadura 5 g (J.T. Baker®, U.S.A.); sulfato de magnesio heptahidratado 0,5g ( J.T. Baker®, U.S.A.); sulfato ferroso heptahidratado 0,03 g (J.T. Baker®, U.S.A.); sulfato de zinc heptahidratado 0,04 g (J.T. Baker®, U.S.A.) (21); (b) medio glicerol sintético (SG) (composición por litro: glicerol 10g (J.T. Baker®, U.S.A.); sulfato de amonio 0,66g (J.T. Baker®, U.S.A.); extracto de levadura 0,1g; sulfato de magnesio heptahidratado 0,25 g (J.T. Baker®, U.S.A.); dihidrógeno fosfato sódico 0,68 g (J.T. Baker®, U.S.A.) (20); (c) Medio International Streptomyces Project ISP (Difco®, U.S.A.). Todos los medios, líquidos o sólidos, se prepararon con buffer KH2PO4/Na2HPO4 20mM pH 6,8. Para la elaboración de medios sólidos se adicionó 1,5% de agar (Difco®, U.S.A.).
Aislamiento y purificación de microorganismos
La búsqueda e identificación de microorganismos productores de péptidos con actividad biológica se efectuó sembrando las diferentes muestras en diluciones seriadas, hasta 10-4 en los medios selectivos (glicerol, ISP y SG), con la adición del colorante básico, azul de metileno (Sigma®), en una proporción de 0,002%. Para la presente investigación se analizaron muestras provenientes de suelos de diferentes pisos térmicos de regiones del Departamento de Antioquia, Colombia. Los aislados se incubaron en los medios selectivos durante 1 semana a 28ºC. Aquellos microorganismos que formaban colonias con interacción específica con el colorante, ya sea de atracción o de repulsión del mismo, fueron seleccionados para posterior purificación por resiembras en los medios selectivos. El péptido antimicrobiano catiónico ε-poli-L-lisina, obtenido a partir de la fermentación de S. albulus, fue provisto por Chisso Corporation, Japón, y utilizado como control.
Detección de compuestos de naturaleza peptídica
Para obtener los péptidos con posible actividad biológica, los microorganismos que mostraron interacción con el colorante se llevaron a crecimiento en medios líquidos (medios Glicerol y SG: sin colorante). La presencia de compuestos de naturaleza proteica se monitoreó a lo largo del crecimiento del microorganismo, evaluando la reacción cualitativa con el reactivo de Dragendorff, y la precipitación oscura del colorante indicó la presencia de compuestos de naturaleza aminada y proteica, de acuerdo con lo reportado por Munier et al., 1951 (28) y Shima y Sakai, 1981 (22). Sólo aquellos microorganismos que mostraron durante su crecimiento la formación de productos de naturaleza peptídica, fueron seleccionados para los procesos siguientes. Con el fin de determinar la naturaleza química de las sustancias excretadas por los microorganismos, se obtuvieron los extractos libres de células o extractos crudos, por centrifugación de las células (6500 x g) (29). Posteriormente se precipitaron las proteínas con ácido acético (1 volumen de muestra por 4 volúmenes de ácido acético), o sulfato de amonio (saturado) (30). Los extractos microbianos libres de células, los compuestos proteicos precipitados y los compuestos proteicos, sometidos a hidrólisis con HCl 1N durante 1 hora y 6N durante 2 horas en ebullición, fueron evaluados por cromatografía de capa fina (sílica gel 60 F264 (Merk®, Alemania)), siguiendo la metodología característica para determinar la presencia de compuestos proteicos y aminoácidos; la reacción se desarrolló con n-butanol – ácido acético – piridina– agua (4:1:1:2) y se hizo la detección con el reactivo ninhidrina (Merk®, Alemania) (20). Para la determinación aproximada del peso molecular de los compuestos de naturaleza proteica se utilizó electroforesis en gel de poliacrilamida en condiciones reductoras (4% y 15%, tinción CBB-G250), en presencia de SDS 0,1% (marcador de peso molecular de 2 a 212 kD New Englands Biolabs®, U.S.A.). Se utilizó como control ε-Poli Lisina (ε-PL solución 50%, suministrado por Chisso Corporation, Japón). La concentración de proteína en las muestras se determinó por el kit Quick Start Bradford Protein Assay (BioRad®, U.S.A.) y Lowry (31). Todos los experimentos se efectuaron por triplicado.
Evaluación de actividad antimicrobiana por el método de bioautografía
La actividad antimicrobiana se evaluó por el método de bioautografía directa (32), en el que 10 µg/ mL de una muestra purificada, con posible actividad biológica, se depositó en placas de cromatografía de capa fina y se fijó por desecación de la muestra. Posteriormente se sumergió en un medio líquido Müeller-Hinton (MH), en el que microorganismos patógenos (Escherichia coli ATCC 8739, Staphylococcus aureus ATCC 6538 y Candida albicans ATCC 10231) crecieron hasta alcanzar una densidad óptica entre 0,4 a 0,5 unidades (longitud de onda de 600 nm). Esta placa, ya impregnada de la muestra de interés y del microorganismo patógeno, se dejó incubando durante 1 hora a 37ºC. Pasado este tiempo, se sumergió en una solución de sal de tetrazolium, también conocida como MTT (3-{4, 5-dimetiltiazol-2- il}-2, 5-difenil tetrazolium bromuro) (Alfa Aesar®, Alemania) y se dejó incubar 30 minutos, de manera que se permitió que la deshidrogenasa generada por el microorganismo durante su crecimiento, reaccionara con la sal de tetrazolium, convirtiéndola en formazan, que es detectado por un fuerte color azul. El color no se produce en las zonas donde está presente un componente antimicrobiano. Aquellos microorganismos que mostraron una respuesta positiva a la actividad antimicrobiana se evaluaron en sus características morfológicas y de tinción por coloración de Gram. Todos los experimentos se llevaron a cabo por triplicado. Como blanco para la bioautografía, y siguiendo su mismo procedimiento, se utilizó un inyectable de Gentamicina 80 mg/mL, que se diluyó a diferentes concentraciones, desde 2 hasta 18 µg/mL.
RESULTADOS Y DISCUSIÓN
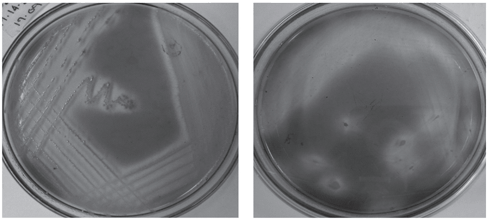
Cada una de las 20 muestras recolectadas a partir de suelo de diferentes pisos térmicos del Departamento de Antioquia, Colombia, se sometió a diluciones seriadas. Se logró aislar 35 cepas que mostraron interacción con el colorante (azul de metileno 0,002%). Es preciso aclarar que la cantidad de microorganismos aislados a partir de tales muestras fue mucho mayor (>100 cepas) que la reportada en este documento; sin embargo, sólo aquellas colonias que mostraron una interacción electrostática de 5 mm radio o mayor con el colorante presente en el medio, fueron seleccionadas para posterior purificación y búsqueda de bioactividad, de acuerdo con la figura 1.
Figura 1. Microorganismos aislados y purificados de suelos nativos antioqueños.
El rastreo y la visualización de la interacción entre colorante y colonia aislada se apreciaron con mayor claridad en el medio glicerol sintético (SG), reportado por Nishikawa et al., 2002 (20), que resultó ser el más adecuado para la búsqueda de microorganismos productores de moléculas con carga neta, y fue escogido como el medio para los subsiguientes procedimientos. El medio glicerol, reportado por Shima et al., 1977 (21), pioneros en la búsqueda de moléculas cargadas, no permitió hacer una identificación usando un volumen de muestras de trabajo tan grande como el de esta investigación, puesto que su método propone la identificación de sustancias de naturaleza proteica para cada una de las cepas purificadas. El medio ISP no arrojó resultados que condujeran al aislamiento de microorganismos de interés, aunque su uso sí fue relevante para la identificación de microorganismos como los Streptomices sp., los cuales crecen en dicho medio de manera característica.
Los microorganismos aislados y purificados de suelos nativos antioqueños excretan metabolitos que demuestran una fuerte interacción electrostática con el colorante básico presente en el medio. Considérese que antes de sembrar el microorganismo, el medio era uniformemente azul oscuro y, en la medida que el microorganismo crecía, repelía el colorante, dejando una zona incolora a su alrededor. Los microorganismos productores de moléculas extracelulares cargadas podían ser identificados por la interacción con el colorante azul de metileno directamente sobre el medio de cultivo sólido. Zonas de aproximadamente 5 mm de diámetro se tomaron como base para la elección de los microorganismos productores. El diámetro refleja la capacidad de difusión de la molécula y está relacionado con el peso molecular de la misma; además, la exclusión del colorante de la zona de crecimiento del microorganismo indica la naturaleza básica de los componentes excretados al medio por parte de éste. Tal resultado se corrobora gracias a la comparación con la exclusión del colorante que crea la muestra de ε-poli-L-lisina, proporcionada por Chisso Corporation (Japón).
Estos resultados indican que colorantes cargados pueden ser usados para detectar y aislar microorganismos que tienen la capacidad de excretar sustancias cargadas en el medio, como lo han reportado Nishikawa et al., 2002 (20). Cabe mencionar que el colorante usado en esta investigación no inhibió el crecimiento de los microorganismos, fenómeno que puede explicarse asumiendo que las células producen una cantidad apreciable de las moléculas cargadas que interactúan con el colorante indicador cargado, y que lo retiran de la zona inmediata a aquella donde se encuentra el microorganismo, dejando así el medio limpio y permitiendo la asimilación de nutrientes (20, 21, 29).
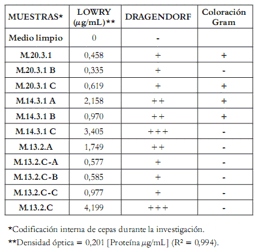
Por las características del azul de metileno, muchos microorganismos logran reducirlo, generar un cambio en su coloración y, en ocasiones, hasta hacerlo desaparecer. Por esta razón, los microorganismos que presentaron característica de decoloración en el medio de cultivo eran posteriormente evaluados en su capacidad para generar componentes proteicos y actividad antimicrobiana. Muchos microorganismos presentaran decoloración del medio pero no mostraran actividad biológica. Por consiguiente, el método puede considerarse como un paso previo a la identificación de microorganismos productores de biomoléculas que tienen carga neta y que pueden llegar a tener actividad antimicrobiana. De las 35 cepas aisladas y purificadas con base en la interacción con el colorante, sólo 11 exhibieron la presencia de proteína en condiciones apreciables y medibles por los métodos de Dragendorff, Bradford y Lowry, como se evidencia enla tabla 1, lo que permitió evaluarlas posteriormente en su actividad antimicrobiana por el método de bioautografía directa.
Tabla 1. Concentración de proteína en las cepas seleccionadas con base en la interacción electrostática con colorante del medio de cultivo.
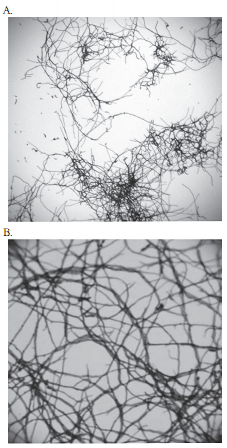
Entre las cepas purificadas que mostraron potencial bioactividad se encontraron Bacillus sp. y Streptomyces sp. Estos últimos microorganismos poseen morfología de colonia, con un micelio grisáceo y una estructura microscópica característica de este género de acuerdo a la figura 2 (22). Los Streptomyces sp. producen una coloración melanoidea en medios como ISP (27) y Sabouraud (Merck®, Alemania), así como un olor característico a tierra húmeda, proveniente de un compuesto químico llamado goesmina, producto de su metabolismo (33). Por la importancia industrial que han tenido los Streptomyces en el desarrollo de los antibióticos a nivel mundial, el hallazgo de microorganismos de éste género, en las condiciones de trabajo y con los objetivos trazados en esta investigación, constituye un descubrimiento promisorio que da cuenta de la biodiversidad de los suelos del Departamento de Antioquia y la importancia del estudio de la ecología microbiana en nuestro país.
Figura 2. Microfotografías del aislado del género Streptomyces sp. M.13.2.C. A. Vista a 40X. B. Vista a 100X.
Los microorganismos Streptomyces sp. son habitantes naturales del suelo y gozan de una cualidad destacada, su gran producción de antibióticos, más de cincuenta moléculas, entre ellas estreptomicina, neomicina, tetraciclina, clindamicina y nistatina. La búsqueda prosigue en pos de otros antibióticos producidos por los organismos de este género, máxime al considerar que la resistencia a los antibióticos por parte de los microorganismos patógenos exige el descubrimiento permanente de nuevos agentes, y que los péptidos antimicrobianos forman parte de este grupo. Lo valioso de la presente investigación es que se hallaron microorganismos del género Streptomyces sp. con capacidad de interactuar electrostáticamente con el colorante básico adicionado al medio de cultivo, constituyendo un microorganismo con potencial capacidad para producir moléculas con actividad biológica (20, 34).
A partir de las cepas que demostraron interacción con el colorante, y que además produjeron una cantidad medible de proteína por el método de Lowry et al., 1951 (31), se procedió a realizar pruebas confirmativas de la presencia de grupos aminados, tanto por cromatografía de capa fina como por precipitación con el reactivo de Dragendorff, el cual es usado para detectar la presencia de compuestos nitrogenados, entre ellos aminas simples y aminoácidos (22).
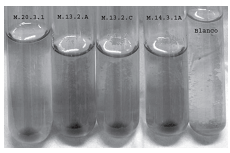
Se encontró que de las 11 cepas purificadas, sólo 4: M.20.3.1, M.14.3.1 A, M.13.2.A y M.13.2.C mostraron resultados positivos a las pruebas realizadas, como se aprecia en la figura 3. Los datos obtenidos son similares a los reportados por Shima et al. (21, 22, 35) sobre experimentos pioneros en los que se detectó la producción del péptido antimicrobiano ε-poli-L-lisina, generado por Streptomyces albulus, usado actualmente en la conservación de leche y de otros productos alimenticios porque soporta presiones y temperaturas de esterilización sin sufrir degradación (18, 25).
Figura 3. Precipitación de compuestos nitrogenados producto de la reacción entre el reactivo de Dragendorff y los compuestos nitrogenados presentes en los extractos crudos libres de células (Foto original en color, precipitado naranja).
La prueba cualitativa de la precipitación de compuestos nitrogenados por reacción con el reactivo de Dragendorff permite establecer una relación directa entre la cantidad de proteína presente en la muestra y la cantidad de precipitado que se obtiene al final de la reacción de Dragendorff, como es el caso de la muestra procedente de los extractos crudos de las cepas M.13.2.C y M.13.2.A, ambas provenientes de microorganismos del género Streptomyces sp. Los resultados obtenidos con los ensayos de cromatografía de capa fina corroboran la presencia de compuestos nitrogenados en las muestras. Al realizar la cromatografía de capa fina en las muestras de extractos microbianos libres de células, compuestos proteicos precipitados y compuestos proteicos sometidos a procesos de hidrólisis con HCl 6N, todas ellas reveladas con ninhidrina, se encontró que los extractos crudos no presentaron manchas que indicaran la presencia de compuestos nitrogenados, al igual que el medio limpio sin microorganismos inoculados; las muestras de compuestos proteicos precipitados exhibieron la presencia de éstos en forma de manchas características, pero con factores de retención (Rf) muy bajos debido, posiblemente, al acomplejamiento y aglomeración de la muestra; los compuestos proteicos hidrolizados presentaron manchas con Rf, similares a las del control del aminoácido lisina, lo que da cuenta de la hidrólisis de los enlaces peptídicos y de la liberación de aminoácidos (imágenes no mostradas).
La presencia de proteína por el método de Lowry, y de compuestos nitrogenados por los métodos de Dragendorff y cromatografía de capa fina para las cepas M.20.3.1, M.14.3.1 A, M.13.2.A, M.13.2.C demostró que la molécula es de naturaleza peptídica (21, 35) y permitió continuar la búsqueda de bioactividad antimicrobiana expresada por alguna de las cepas aisladas en esta investigación. Así pues, cada una de las cepas seleccionadas se llevó a procesos de fermentación y a la evaluación de la producción de proteína a lo largo del crecimiento microbiano. Así mismo, con cada una de las muestras obtenidas a lo largo del crecimiento microbiano, se hizo la prueba biológica para la determinación de la actividad antibacteriana sobre microorganismos considerados patógenos por medio de bioautografía.
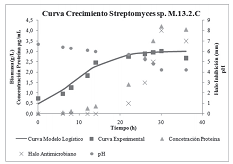
Se encuentra, entonces, que el microorganismo de la cepa M.13.2.C exhibe una curva de crecimiento normal (ajustada con el modelo Logístico) y un aumento progresivo de la concentración de proteína en el medio a lo largo del tiempo. La disminución del pH durante el proceso de fermentación es una condición esencial para la acumulación de polímeros con capacidad antibacteriana (22); ello se logró constatar con un aumento progresivo en la concentración de componentes peptídicos en la medida que el pH disminuía a lo largo del crecimiento microbiano, comenzando el proceso con un pH de 6,8; aportado por el buffer del medio, hasta un pH de 4,2; que era el que finalmente se reportaba, y que aparece en la figura 4.
Figura 4. Curva de crecimiento de biomasa de la cepa Streptomyces sp. M.13.2.C (n), concentración de proteína (), tamaños de los halos de inhibición sobre el patógeno E. coli (×) y cambio de pH () con respecto al tiempo del desarrollo del microorganismo. La curva experimental de crecimiento microbiano () se ajusta siguiendo parámetros del modelo logístico.
Al evaluar la actividad antibacteriana por el método de la bioautografía se logró obtener halos de inhibición sobre el crecimiento del microorganismo patógeno E. coli ATCC 8739. El halo crecía en la medida que la concentración de la proteína aumentaba, como puede verse en las figuras 4 y 5. A mayor diámetro, mayor es la inhibición de crecimiento del patógeno E. coli. Se obtiene, entonces que, tanto la cantidad de proteína que se encontraba a lo largo de la fermentación como el tamaño de los halos de inhibición sobre el crecimiento del patógeno, iban en aumento en la medida que el crecimiento del Streptomyces sp. llegaba a la fase de latencia, como se puede apreciar en la figura 4, lo que permite pensar que el compuesto que da origen a la capacidad antibiótica del extracto crudo es un metabolito secundario, lo cual es característico de muchas de las sustancias de interés que esta especie de microorganismos genera, como lo reportan otras investigaciones (18, 20, 29, 36).
Figura 5. Bioautografía efectuada con los componentes proteicos de los extractos crudos de la cepa M.13.2.C de Streptomyces sp. a diferentes horas de su crecimiento. Diámetro de inhibición: 20 horas: 0,5 mm (±0,01); 22 horas: 1 mm (±0,04); 26 horas: 3 mm (±0,04); 28 horas: 5 mm (±0,03); 30 horas 8 mm (±0,04).
El blanco utilizado fue Gentamicina inyectable 80 mg/mL, llevada a concentraciones de 2, 6, 10, 14, 18 µg/mL y con diámetros de inhibición: 1mm; 4 mm; 7,5 mm; 10 mm; 13,5 mm (±0,02) respectivamente. Por tanto, puede decirse que el halo de inhibición de 8 mm, creado por el extracto proteico de la cepa M.13.2.C tiene una concentración relativa al del inyectable de gentamicina de 10,67 µg/mL.
Respecto a la bioautografía realizada sobre los microorganismos S. aureus y C. albicans con los extractos peptídicos de la cepa M.13.2.C, los resultados no son totalmente claros. Posiblemente, para contrarrestar el crecimiento de S. aureus, sea necesario aumentar la concentración del compuesto peptídico. Esto se ve corroborado por ciertas investigaciones que demuestran que la concentración de un péptido antimicrobiano con capacidad de actuar sobre E. coli, va a ser menor que la de un péptido que actúe sobre S. aureus (26). Se obtuvo, entonces, para S. aureus ATCC 6538, un halo de 2mm de diámetro en la interacción con el péptido antimicrobiano, dato también de Nishikawa y colaboradores (14). En cuanto a la interacción con C. albicans ATCC 10231, no se dio inhibición en el crecimiento de esta levadura, lo cual obedece, posiblemente, a que la composición de la membrana celular de la levadura no es susceptible a la acción de los péptidos antimicrobianos, como sí lo pueden ser las células bacterianas (3)
Para explicar el mecanismo de acción por el cual un péptido antimicrobiano puede actuar sobre un microorganismo, se postula la posibilidad de que las porciones cargadas positivamente se unan a los componentes cargados negativamente en la membrana celular de una célula bacteriana, tales como fosfatidilglicerol o cardiolipinas, dando origen a la formación de poros que conducen al microrganismo a la muerte (6, 37). Tal modelo explica los estados iniciales de la actividad antimicrobiana propuesta para péptidos con actividad antibiótica, como nisina, magainina y defensinas (20, 37).
Con base en los anteriores resultados se procedió a determinar la distribución de pesos moleculares usando gel de electroforesis y comparando con un marcador de peso molecular. Las muestras fueron los compuestos de naturaleza peptídica precipitada con ácido acético, provenientes de las cepas M.13.2.C, M.13.2.C-A, M.13.2.A, M.20.3.1 B, M.14.3.1.A, M.14.3.1.C, M.20.3.1, M.14.3.1. B, M.13.2.C-B. Se encontraron bandas ubicadas entre 1,2 y 2,4 kDa, al igual que lo informan Nishikawa y colaboradores (20). Además, es semejante a la banda que muestra el blanco, que en este caso es el e-poli-L-lisina, donado por Chisso Corporation del Japón, que puede observarse en la figura 6. A pesar de encontrarse bandas que daban cuenta de la presencia del péptido, no todos los extractos proteicos mostraron bioactividad antibacteriana. Es el caso del extracto proteico de la cepa M.20.3.1 que, aunque exhibía una banda característica de la presencia del péptido de interés, no mostró actividad alguna, lo cual puede obedecer a la baja concentración del compuesto en la muestra.
Figura 6. Gel de electroforesis. Bandas características de los compuestos proteicos de las cepas: 1. Marcador de peso molecular; 2. M.13.2.C; 3. M.13.2.C-A; 4. M.13.2.A; 5. M.20.3.1 B; 6. M.14.3.1.A; 7. M.14.3.1.C; 8. M.20.3.1; 9. M.14.3.1. B. 10. M.13.2.C-B; 11. e-poli-L-lisina; 12. Medio limpio, sin inoculo.
CONCLUSIONES
Aún no se ha descubierto todo lo que la naturaleza puede brindar, pero es necesario continuar la búsqueda. Utilizando un método simple y sensible de screening, reportado anteriormente por Nishikawa et al., 2002 (20), se logró la detección de microorganismos productores de metabolitos cargados. Con la presente investigación se comprueba que es posible encontrar microorganismos que, por su metabolismo, creen biomoléculas que interaccionen electrostáticamente con colorantes cargados adicionados en el medio. El método informado en la presente investigación puede aplicarse en medios sólidos con una gran cantidad de microorganismos de búsqueda; además puede usarse sobre la generalidad de los microorganismos neutrófilos y mesófilos, porque las condiciones de cultivo son aplicables a la mayoría, y puede afirmarse que cuando el microorganismo no muestra la zona característica de inhibición alrededor de la colonia, la secreción depende de la cepa del microorganismo como tal.
Los resultados positivos a las reacciones con el reactivo de Dragendorff, y en cromatografía de capa fina con la ninhidrina, demuestran que el extracto obtenido es de naturaleza peptídica (21, 35), a partir de un microorganismo del género Streptomyces sp., aislado de suelos antioqueños, el cual, por el método de bioautografía, resultó tener actividad antimicrobiana, una concentración relativa de 10,67 µg/mL, un halo de inhibición de 8 mm sobre microorganismos considerados como patógenos, y un peso molecular de aproximadamente 2 kDa, semejante al del control.
Los resultados indican que la técnica que usa un colorante que interaccione con el metabolito es útil para el aislamiento de microorganismos productores de moléculas cargadas.
AGRADECIMIENTOS
Esta investigación se desarrolló gracias al apoyo económico para proyectos de menor cuantía CIDF- 094, del CODI, Universidad de Antioquia, Colombia, y al Grupo de Ofidismo/Escorpionismo de la misma institución. Los investigadores agradecen la colaboración de Paulina Marulanda León y Marcela Álvarez Montañez, estudiantes de Ingeniería de Alimentos y Química Farmacéutica respectivamente, también de la Universidad de Antioquia.
REFERENCIAS BIBLIOGRÁFICAS
1. Lehmann J, Retz M, Harder J, Krams M, Kellner U, Hartmann J, et al. Expression of Human ß-Defensins 1 and 2 in Kidneys with Chronic Bacterial Infection. BMC Infect Dis. [Internet]. 2002 Sept 18 [updated 2002 Sept 22 cited 2010 Feb 07]; 20 (2): 1-10. Available from: http://www.biomedcentral.com/1471-2334/2/20 [ Links ]
2. Lehrer RI, Ganz T. Defensins of Vertebrate Animals. Curr Opi Immunol. 2002 Feb ; 14 (1): 96 - 102. [ Links ]
3. Schneider JJ, Unholzer A, Schaller M, Schäfer-Korting M, Korting HC. Human Defensins. J Mol Med. 2005 Aug; 83 (8): 587 - 595. [ Links ]
4. Schwaab M, Hansen S, Pearson MD, Shagdarsuren S, Dazert S. Human ß-Defensinsat the Front Line of the Peritonsillar Abscess. Eur J Clin Microbiol Infect Dis. 2009 jul; 28 (7): 745755. [ Links ]
5. Piers KL, Brown MH, Hancock REW. Recombinant DNA Procedures for Producing Small Antimicrobial Cationic Peptides in Bacteria. Gene. 1993 Nov 30; 134 (1): 7 - 13. [ Links ]
6. Hancock REW, Chapple DS. Peptide Antibiotics. Antimicrob Agents Chemother. 1999 Jun; 43 (6): 1317 - 1323. [ Links ]
7. Peschel A, Sahl H-G. The co-Evolution of Host Cationic Antimicrobial Peptides and Microbial Resistance. Nature. 2006 Jul; 4: 529 - 536. [ Links ]
8. Ganz T. Versatile Defensins. Science. 2002 Nov 1; 298 (5595): 977-979. [ Links ]
9. Zasloff M. Antimicrobial Peptides of Multicellular Organisms. Nature. 2002 Jan 24; 415 (6870): 389 -395. [ Links ]
10. He Z, Kisla D, Zhang L, Yuan C, Green-Church KB, Yousef AE. Isolation and Identification of a Paenibacillus polymyxa Strain That Coproduces a Novel Lantibiotic and Polymyxin. Appl Environ Microbiol. 2007 Jan; 73 (1): 168-178. [ Links ]
11. Aerts AM, Thevissen K, Bresseleers SM, Sels J, Wouters P, Cammue BPA, et al. Arabidopsis thaliana Plants Expressing Human ß-Defensin-2 are More Resistant to Fungal Attack: Functional Homology Between Plant and Human Defensins. Plant Cell Rep. 2007 Aug; 26 (8): 1391-1398. [ Links ]
12. Fleming A. On a Remarkable Bacteriolytic Element Found in Tissues and Secretions. Proc R Soc Lond. 1922 May 1; 93: 306317. [ Links ]
13. Papanastasiou EA, Hua Q, Sandouk A, Son UH, Christenson AJ, Van Hoek ML, et al. Role of acetylation and charge in antimicrobial peptides based on human beta-defensin-3. APMIS. 2009 Jul; 117 (7): 492-499. [ Links ]
14. Corrales-Garcia LL, Possani LD, Corzo G. Expression Systems of Human ß-Defensins: Vectors, Purification and Biological Activities. Amino Acids. 2010; Mar 20. [Epub ahead of print]. [ Links ]
15. Stein T, Vater J, Kruft V, Otto A, Wittmann-Liebold B, Franke P, et al. The Multiple Carrier Model of Nonribosomal Peptide Biosynthesis at Modular Multienzymatic Templates. J Biol Chem. 1996 Jun 28; 271 (26): 15428-15435. [ Links ]
16. Yamanaka K, Maruyama C, Takagi H, Hamano Y. e-Poly-LLysine Dispersity is Controlled by a Highly Unusual Nonribosomal Peptide Synthetase. Nat Chem Biol. 2008; 4 (12): 766-772. [ Links ]
17. Yamanaka K, Maruyama C, Takagi H, Hamano Y. e-Poly-L-lysine Dispersity is Controlled by a Highly Unusual Nonribosomal Peptide Synthetase. Nat Chem Biol. 2008 Dec; 4 (12): 766-772. [ Links ]
18. Oppermann-Sanio FB, Steinbüchel A. Ocurrence, Functions and Biosynthesis of Polyamides in Microorganisms and Biotechnological Production. Naturwissenschaften. 2002 Jan; 89 (1): 11-22. [ Links ]
19. Kunioka M. Biosynthesis of poly (g-glutamic acid) from Lglutamine, citric acid and ammonim sulfate in Bacillus subtilis IF03335. Appl Microbiol Biot. 1995 Dec; 44 (3-4): 501-506. [ Links ]
20. Nishikawa M, Ogawa K. Distribution of Microbes Producing Antimicrobial e-Poly-L-Lysine Polymers in Soil Microf lora Determined by a Novel Method. Appl Environ Microb. 2002 Jul; 68 (7): 3575-3581. [ Links ]
21. Shima S, Sakai H. Polylysine Produced by Streptomyces. Agr Biol Chem. 1977; 41 (9): 1807-1809. [ Links ]
22. Shima S, Sakai H. Poly-L-Lysine Produced by Streptomyces. Part II. Taxonomy and Fermentation Studies. Agr Biol Chem. 1981; 45 (11): 2497-2502. [ Links ]
23. Hamano Y, Nicchu I, Shimizu T, Onji Y, Hiraki J, Takagi H. e-Poly-L-lysine Producer, Streptomyces albulus, Has FeedbackInhibition Resistant Aspartokinase. Appl Microbiol Biot. 2007 Sep; 76 (4): 873-882. [ Links ]
24. Yoshida T, Nagasawa T. e-Poly-L-Lysine: Microbial Production, Biodegradation and Application Potential. Appl Microbiol Biot. 2003; 62 (1): 21-26. [ Links ]
25. Food and Drugs Administration (U.S.) F. Agency Response Letter GRAS Notice No. GRN 000135. 2004 [updated 2009 May 22 cited 2010 Feb 07]; Available from: http://www.fda.gov/Food/ FoodIngredientsPackaging/GenerallyRecognizedasSafeGRAS/ GRASListings/ucm153957.htm [ Links ]
26. Geornaras I, Yoon Y, Belk KE, Smith GC, Sofos JN. Antimicrobial Activity of e-Polylysine Against Escherichia coli O157:H7, Salmonella Typhimurium and Listeria monocytogenes in Various Food Extracts. J Food Sci. 2007 Oct; 72 (8): M330-4. [ Links ]
27. Shirling EB, Gottieb D. Methods for Characterization of Streptomyces Species. Int J Syst Bacteriol. 1966 Jul; 16 (3): 313-340. [ Links ]
28. Munier R, Machebouf M. Paper partition microchromatography of alkaloids and of various biological nitrogenous bases. III. Examples of the separation of various alkaloids by the acid solvent phase technic (atropine, cocaine, nicotine, sparteine, strychnine and corynanthine families). Bull Soc Chim Biol. 1951; 33 (7): 846-856. [ Links ]
29. Kawai T, Kubota T, Hiraki J, Izumi Y. Biosynthesis of e-poly-Llysine in a cell-free system of Streptomyces albulus. Biochem Bioph Res Co. 2003 Nov; 311 (3): 635-640. [ Links ]
30. Bollag D, Rozycki M, Edelstein S. Protein Methods. 2 ed. New York: Wiley-Liss, Inc.; 1996. [ Links ]
31. Lowry OH, Rosebrough NJ, Farr AL, Randall RJ. Protein Measurement with Folin Phenol Reagent. J Biol Chem. 1951 Nov 1; 193: 265-275. [ Links ]
32. Colorado J, Galeano E, Martinez A. Desarrollo de la autobiografia directa como método de referencia para evaluar la actividad antimicrobiana de la gentamicina contra Escherichia coli. Vitae. 2007 Jan-Jun; 14 (1): 67 - 71. [ Links ]
33. Boone CJ, Pine L. Rapid Method for Characterization of Actinomycetes by Cell Wall Composition. Appl Microbiol. 1968 Feb; 16 (2): 279-284. [ Links ]
34. Hancock REW. Cationic Peptides: Effectors in Innate Immunity and Novel Antimicrobials. Lancet Infect Dis. 2001 Oct; 1 (3): 156-164. [ Links ]
35. Shima S, Sakai H. Poly-L-Lysine Produced by Streptomyces. Part III. Chemical Studies. Agr Biol Chem. 1981; 45 (11): 2503-2508. [ Links ]
36. Jagnow G, Dawid W. Biotecnología. Introducción con Experimentos Modelo. 1 ed. Zaragoza, España: Acribia; 1991. [ Links ]
37. Reuter M, Schwieger C, Meister A, Karlsson G, Blume A. PolyL-Lysines and Poly-L-Arginines Induce Leakage of Negatively Charged Phospholipid Vesicles and Translocate Through the Lipid Bilayer Upon Electrostatic Binding to the Membrane. Biophys Chem. 2009; 144 (2): 27-37. [ Links ]
Recibido: Agosto 04 de 2009; Aceptado: Mayo 21 de 2010
* Autor a quien debe dirigir la correspondencia: ligialu@ibt.unam.mx