Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268On-line version ISSN 1909-0544
Rev.MVZ Cordoba vol.13 no.1 Córdoba Jan./May 2008
ORIGINAL
CARACTERIZACIÓN GENÉTICA DE LOTES DE Brycon orbignyanus UTILIZADOS EN PROGRAMAS DE REPOBLAMIENTO
GENETIC CHARACTERIZATION OF Brycon orbignyanus STOCKS USED IN RESTOCKING PROGRAMS
Nelson Lopera B,1* Ph.D, Ricardo Ribeiro P,1 Ph.D, Rodolfo Sirol N,2 Ph.D, Jayme Povh,1 Ph.D, Patricia Gomes,1M.Sc, Danilo Streit Jr,3 Ph.D, Lauro Vargas M,1 Ph.D.
1Universidade Estadual de Maringá, Grupo de Pesquisa PeixeGen, Maringá, PR, Brasil.
2Estaçäo de Aqüicultura e Hidrologia da Duke Energy International, Salto Grande, SP, Brasil.
3Departamento de Zootecnia, Universidade Federal de Rio Grande do Sul, Av. Bento Gonçalves, 7712, CEP 91540000, Porto Alegre/RS, Brasil.
3Universidade Federal de Mato Gross, Campus de Rondonópolis, Departamento de Zootecnia, Av. Fernando Corrêa, s/n Coxipó, CEP 78060 - 900, Cuiabá, MT.
*Correspondencia: nelson.peixegen@gmail.com
Recibido: Agosto 24 de 2007; Aceptado: Enero 10 de 2008
RESUMEN
Objetivo. Caracterizar genéticamente dos lotes y una progenie de Brycon orbignyanus destinados para programas de repoblamiento, utilizando la técnica molecular de RAPD (Random amplified polymorphic DNA).
Materiales y métodos. Se analizaron 58 reproductores originarios de dos piscícolas ubicadas en las ciudades de Castillo (A:30 individuos) y Porto Ferreira (C:28 individuos), mantenidos en cautiverio hace seis años en la estación de acuicultura e hidrología de la Duke Energy Internacional (Geração Paranapanema) (São Paulo-Brasil). Treinta larvas de la progenie del lote A (B) también se analizaron.
Resultados. Los 14 primers usados produjeron 87 fragmentos de los cuales 70.11% fueron polimórficos. Fueron observadas diferencias (p≤0.05) en la frecuencia de 31 fragmentos, con tres exclusivos para el lote A. Los valores de divergencia, distancia e identidad genética mostraron que la diversidad genética del lote A fue mantenida en la progenie y que existe una baja diferenciación entre los lotes de reproductores. El análisis de variancia molecular mostró que la mayor parte de la variación está dentro de cada lote (87.45%) y no entre ellos (12.55%). Este resultado se corroboró con los valores de FST (0.125) y con el dendrograma, que indicaron una moderada diferenciación genética, sin la formación de agrupamientos.
Conclusiones. La diversidad genética fue preservada en la progenie debido al manejo eficiente de la reproducción. No hubo una diferenciación genética entre los lotes de reproductores, debido posiblemente a que el origen natural de ambos fue el río Paraná.
ABSTRACT
Objective. To genetically characterize two Brycon orbignyanus stocks and one progeny intended for restocking programs using the molecular technique RAPD (Random Amplified Polymorphic DNA).
Materials and methods. Fifty eight broodstocks native to two fish farms in the cities of Castilho (A:30 individuals) and Porto Ferreira (C:28 individuals) were analyzed. The fish had been maintained for six years in captivity in the aquaculture and hydrology station of Duke Energy International (Geração Paranapanema. São Paulo - Brazil); thirty larvae of progeny from the stock A (B) were also analyzed.
Results. The fourteen primers used produced 87 fragments of which 70.11% were polymorphic. Differences were observed (p≤0.05) in the frequency of 31 fragments, with three exclusive from stock A. The values for divergence, distance and genetic identity showed that genetic diversity of stock A was maintained in progeny and that a low differentiation exists among reproductive stocks. Analysis of Molecular variance showed that most of the variation is inside each stock (87.45%) and not between them (12.55%). This result was corroborated with FST (0.125) values and with the dendrogram indicating a moderate genetic differentiation, without cluster formation.
Conclusions. Genetic variability was maintained in progeny, possibly because both were native to the Paraná river.
Key words: Brycon orbignyanus, polymorphism, genetic, RAPD, fishes.
INTRODUCCIÓN
Las alteraciones ambientales causadas por acciones humanas (deforestación, construcción de hidroeléctricas, contaminación de los ríos, etc.), han provocado diversas alteraciones en los ecosistemas acuáticos, perturbando patrones fisiológicos, biológicos y simbióticos de diversos organismos (1). Entre esos organismos, los peces vienen enfrentando condiciones cada vez más adversas que impiden su sustentación y supervivencia, reduciendo drásticamente el número de poblaciones naturales y consecuentemente, llevando muchas de ellas a la extinción.
Brycon orbignyanus (Valenciennes, 1849), conocido regionalmente en el Brasil como piracanjuba o bracanjuba (Orden Characiformes, familia Characidae, subfamilia Bryconinae), es una especie migratoria nativa de las cuencas hidrográficas formadas por los ríos Uruguay y Paraná (2). Esta especie es apreciada por el elevado valor nutricional de su carne, crecimiento rápido (3) y desempeño en la pesca deportiva. Este pez ha despertado un gran interés por investigadores y productores, ya que en los últimos años se ha observado una reducción drástica de sus poblaciones naturales, (2) lo que la cataloga como especie en vía de extinción (4).
Por ser una especie de alto valor comercial y cultural, varias acciones enfocadas a su conservación se estan realizado. Los programas de repoblamiento son los más usados actualmente en el Brasil, gracias al apoyo de la sociedad, empresas gubernamentales y privadas (5). Sin embargo, la falta de orientación multidisciplinaria que permita la objetividad de las estrategias utilizadas y de mayores informaciones genéticas, biológicas y fisiológicas de las comunidades acuáticas y de las poblaciones de peces, puede hacer de los programas de repoblamiento una amenaza mayor a los ecosistemas ya impactados.
De esta forma, el monitoreo de la diversidad genética de poblaciones naturales y de lotes de peces mantenidos en estaciones piscícolas es de fundamental importancia para la conservación de las especies, donde los marcadores moleculares, como el RAPD (Random Amplified Polymorphic DNA), pueden contribuir para este propósito (6).
El objetivo del presente estudio fue caracterizar genéticamente por RAPD dos lotes y una progenie de B. orbignyanus destinados para programas de repoblamiento.
MATERIALES Y MÉTODOS
Material biológico.Se recolectaron aletas caudales de dos lotes de peces originários de piscícolas ubicadas en las ciudades de Castillo (A:30 individuos) y Porto Ferreira (C:28 individuos), mantenidos en cautiverio hace seis años en la Estación de Acuicultura e Hidrología de la Duke Energy Internacional - Geração Paranapanema (49°13’W y 23°10’S - São Paulo-Brasil).
Después de la inducción hormonal (extracto de hipófisis de carpa) de los reproductores del lote A (15 hembras y 15 machos) utilizando el sistema reproductivo por extrusión (7), fueron recolectadas 30 larvas de tres días de edad.
Extracción, cuantificación e integridad del ADN. Para la extracción de ADN, se utilizó la metodología descrita por Bardakci y Skibinski (8), modificada por Povh et al (9). Fragmentos de aleta caudal de aproximadamente 0.5 cm2 y larvas enteras, preservadas a -20°C con etanol 70%, se colocaron en micro-tubos con 550 µL de tampón de lisis (50 mM de Tris-HCl pH 8.0, 50 mM de EDTA, 100 mM de NaCl y 1% de SDS) y 200 µg/mL de proteinasa K, y se incubaron en baño-maría a 50°C por 12 horas. Posteriormente, el ADN se purificó con dos extracciones con fenol y tres de cloroformo y se precipitó con dos veces y media de volumen de etanol absoluto y un décimo de volumen de acetato de sodio en relación al volumen recuperado. Después se incubó por dos horas a -20°C. El ADN fue centrifugado (12000 rpm por cinco minutos), lavado con 2 mL de etanol 70%, y resuspendido en 60 µL de tampón TE 10 mM de Tris pH 8.0 y 1 mM de EDTA) y tratado con 30 µg/mL de RNAsa. Las muestras permanecieron por 40 minutos en baño-maría a 37°C, y luego conservadas en congelador a -20°C inmediatamente.
El ADN se cuantificó en espectrofotómetro Shimadzu (UV-1601, USA), en la amplitud de onda de 260 nm y se diluyó en tampón TE para una concentración de 10-5 ng/µL (aletas y larvas respectivamente). La integridad del ADN se verificó en electroforesis horizontal utilizando un gel de agarosa 1%, con 70 voltios por 60 minutos, en tampón 1 XTBE (500 mM de Tris-HC1, 60 mM de ácido bórico y 83 mM de EDTA). Para verificar la integridad del ADN, un control negativo con todos los reactivos excepto el ADN molde fue usado.
Amplificación y electroforesis. Las condiciones de amplificación fueron basadas en la metodología descrita por Williams et al (10), con algunas modificaciones. El ADN se amplificó en un volumen de reacción de 25 µL, en el cual se utilizó tampón Tris-KCl 1X (Tris-HCl 20 mM pH 8.4 y KCl 50 mM), 2 mM de MgCl2, 100 ng de primer, 0.2 mM de cada dNTP, una unidad de Taq ADN Polimerasa y 20 ng de ADN molde. Las reacciones de RAPD se amplificaron en un termociclador de PCR, programado con 40 ciclos, con un paso inicial de desnaturalización a 94°C por cuatro minutos y un paso final de extensión a 72°C por cinco minutos. Cada ciclo consistió de un minuto a 94°C, un minuto y treinta segundos a 40°C y dos minutos a 72°C.
Se evaluaron 19 primers del Kit Operon (Operon Technologies Inc. Alameda, CA, USA). Para analizar genéticamente los lotes y la progenie, se seleccionaron aquellos primers que presentaron mejor patrón de amplificación y reproducibilidad. Los productos de amplificación se separaron en gel de agarosa 1.5%. Se utilizaron 15 µL del producto amplificado y 2 µL de tampón de muestra (40% de sacarosa y 0.25% de azul de bromofenol) en electroforesis horizontal. La electroforesis fue conducida en 70 voltios por cuatro horas en una cubeta horizontal usando tampón TBE 1X (500 mM de Tris-HCl, 60 mM de ácido bórico y 83 mM de EDTA). Los geles de cuantificación, integridad y de amplificación del ADN se visualizaron sobre radiación UV, después de su exposición a bromuro de etídio (0.5 ug/mL) por una hora. Posteriormente la imagen se fotografió usando el programa Kodak EDAS-290 (Kodak 1D Image Analysis 3.5).
Análisis estadístico. El tamaño de los fragmentos fue estimado por comparación con el patrón ADN Ladder de 100 pb (15 bandas con tamaño entre 100 y 2072 pb - Invitrogen®, USA.). La presencia o ausencia de fragmentos de tamaños moleculares idénticos fue usada para la construcción de una matriz de similaridad con base en el cálculo del coeficiente de similaridad de Jaccard, codificando 1 como presencia de fragmento y 0 como su ausencia. Con base en esta matriz, la divergencia genética fue estimada por el Test de Mantel, utilizando el método de Monte Carlo, por medio del programa Mantel-Struct (11). El programa TFPGA 1.3 (12) se utilizó para determinar la frecuencia de los fragmentos por el test exacto. La identidad y la distancia genética basadas en Nei (13) fueron calculadas usando el programa PopGene 1.31 (14).
El programa Arlequín 3.0 (15) se utilizó para determinar la diferenciación genética entre los lotes por medio de las estimativas de FST y para el análisis de variancia molecular (AMOVA). La significancia de estas estimativas fue verificada por el método de permutaciones aleatorias con 1000 y 10000 permutaciónes. La magnitud de diferenciación genética entre los lotes fue determinada según la definición de Wright (16), donde valores de FST entre 0.00 a 0.05; 0.05 a 0.15; 0.15 a 0.25 y > 0.25 indican respectivamente pequeña, moderada, alta y elevada diferenciación genética.
La matriz del coeficiente de similaridad de Jaccard y el método de agrupamiento UPGMA se usaron para elaborar un dendrograma, mediante el programa estadistico NTSYS 1.7 (17).
RESULTADOS
Por el análisis en el gel de agarosa, se observó que no ocurrió degradación del ADN en ninguna de las muestras y que no hubo exceso de proteína que pudiese perjudicar la amplificación. De los 19 primers evaluados, 14 se seleccionaron para ser analizados por presentar patrones de amplificación claros y reproducibles (OPA01, OPA02, OPA05, OPA17, OPA18, OPW01, OPW02, OPW03, OPW04, OPW05, OPX01, OPX02, OPX03 y OPX04). Los resultados de la amplificación por el marcador RAPD, mostraron que el número total de fragmentos varió de cuatro (obtenidos con los primers OPA01, OPA02, OPW03 y OPX03) a 11 (OPW02). Las amplificaciones con el primer OPA02 mostraron un patrón monomórfico de amplificación, con la presencia de cuatro fragmentos. El mayor fragmento (2072 pb) y el menor (250 pb) fueron obtenidos por la amplificación de los primers OPA18 y OPW05. Fueron encontrados un total de 87 fragmentos de los cuales 61 fueron polimórficos (70.11%). Se observó diferencia (p≤0,05) en la frecuencia de 31 de los 87 fragmentos entre los lotes de reproductores y la progenie. En el lote A se observaron seis fragmentos exclusivos con relación a la progenie. No se observaron fragmentos exclusivos entre los dos lotes de reproductores (Tabla 1, Figura 1).
Tabla 1. Caracterización, tamaño (en pares de bases) y frecuencia de los fragmentos con valores significativos
por el test exacto (p≤0,05) para los lotes de reproductores (A y C) y la progenie (B) de Brycon orbignyanus.
Figura 1. Análisis de los fragmentos de RAPD del lote de Castillo, producidos a partir de la amplificación con el primer OPW02,
separados en gel de agarosa 1.5%. "M" corresponde al patrón DNA Ladder de 100 pb. Muestras de 1 a 30 corresponde a cada individuo del lote.
Los valores de divergencia genética (p<0.01) encontrados en los lotes de reproductores (A y C) y la progenie (B) se muestran en la figura 2.
Figura 2. Divergencia genética dentro (A, B, C) y entre (AxB, AxC y BxC) los lotes de
reproductores (A y C) y la progenie (B) de B. orbignyanus.
De acuerdo con los resultados de la AMOVA, la mayor parte de esa variación estuvo dentro de cada lote y no entre los lotes de reproductores de B. orbignyanus. Estos resultados fueron confirmados con los valores de FST, que mostraron una moderada diferenciación genética (Tabla 2).
Tabla 2. Análisis de la variancia molecular (AMOVA) de los lotes de B. orbignyanus.
Los valores de identidad genética y de distancia genética encontrados para los lotes de reproductores y la progenie se muestran en la tabla 3.
Tabla 3. Valores de identidad genética (arriba de la diagonal) y de distancia genética (debajo de la diagonal)
para los lotes de reproductores (A y C) y para la progenie (B) de B. orbignyanus.
El dendrograma de similitud genética generado con el coeficiente de Jaccard es presentado en la figura 3.
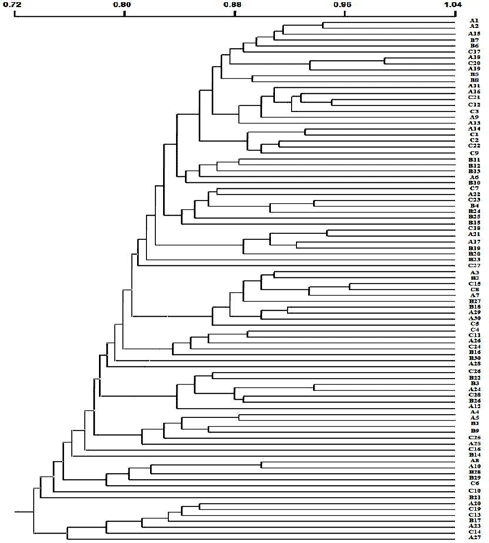
Figura 3. Dendrograma de similitud genética generado con el coeficiente de Jaccard y el algoritmo UPGMA
para los lotes de reproductores (A y C) y para la progenie (B) de B. orbignyanus.
DISCUSIÓN
La caracterización genética de dos lotes de reproductores y una progenie de B. orbignyanus utilizados en programas de repoblamiento se determinó utilizando la técnica molecular de RAPD. La amplificación de los 14 primers seleccionados mostró que la diversidad genética del lote A fue preservada en la progenie (B), evidenciada en los resultados de divergencia genética (AxB: 0.196) y de distancia (0.938) e identidad genética (0.064). Estos resultados fueron corroborados con la obtención de fragmentos exclusivos en el lote A relacionados a su progenie y la reducción de la frecuencia de 11 fragmentos en la primera generación (Tabla 1).
La preservación de la variabilidad genética en la progenie posiblemente fue debida al manejo eficiente de la reproducción. El manejo reproductivo utilizado en las piscícolas puede ser un factor importante en la preservación de la diversidad genética, ya que una pérdida de la variación siempre es esperada cuando no hay un buen manejo genético de los lotes de reproductores (18), el cual es ocasionado por la utilización de sistemas reproductivos inadecuados (19) o por el cruzamiento de individuos emparentados, lo que consecuentemente, puede aumentar la homocigosis de los lotes (20). Este cruzamiento ocurre normalmente en lotes de reproductores pequeños y limitados (con bajo número efectivo de reproductores - Ne) donde después de tres generaciones (21), la variación genética tiende a disminuir (9) limitando el potencial genético, disminuyendo la supervivencia y aumentando la tasa de anormalidades (22), afectando de esta forma la productividad.
El Ne son aquellos animales en edad de reproducción que están en capacidad de dejar una descendencia viable y que según Yokota et al (23) debe ser como mínimo 50 (25 Hembras y 25 Machos). Frost et al (24), relataron que el sistema de reproducción y fluctuaciones en el tamaño de los lotes en piscicultura pueden reducir el Ne. Por eso, lotes de reproductores usados en piscicultura y en programas de repoblamiento deben ser fundados a partir de un número grande de individuos (25) y así evitar problemas relacionados a endogamia. Los resultados encontrados en el presente estudio, demuestran que hubo un buen manejo reproductivo del lote A durante la reproducción (formación del lote con amplia variabilidad genética, cruzamientos entre individuos no emparentados, utilización de sistemas de cruzamiento y reproducción adecuados), lo que según Miller y Kapuscinski (26) resulta en la preservación de la variabilidad genética entre los reproductores y sus progenies y en la disminución de la correlación negativa entre la similitud genética y el número de generaciones (27), que en estaciones de piscicultura es normalmente irreversible (23).
Por otro lado, al analizar la diversidad genética de los lotes de reproductores (A y C), fue encontrada una baja divergencia genética entre si (0.199), corroborada con los resultados de identidad (0.950) y de distancia genética (0.057). De acuerdo con los resultados de AMOVA, la mayor parte de esta variación está dentro de cada lote (87.45%). Estos resultados hacen presumir un mismo origen, razón por la cual pueden formar un grupo homogéneo. Igualmente, esta tendencia es evidenciada en la falta de fragmentos exclusivos entre los dos lotes (Tabla 1) y en el dendrograma de similitud genética (Figura 3), donde no se observó la formación de agrupamientos que evidenciaran una separación de los lotes de reproductores y la progenie.
Los dos lotes de reproductores (A y C) mantenidos en cautiverio en las instalaciones de la Duke Energy Internacional (Geração Paranapanema) hace seis años, son originarios de dos piscícolas ubicadas en las ciudades de Castilho y Porto Ferreira respectivamente, en el Estado de São Paulo (Brasil). La formación de los lotes fue efectuada a partir de individuos adultos capturados directamente en el río Paraná. Sin embargo, a pesar de la separación geográfica de ambas piscícolas y consecuentemente de los diferentes locales de captura de los reproductores, es posible que entre los individuos de cada población existiese un flujo génico en el ambiente natural, lo que explicaría su similitud genética, circunstancia confirmada con el resultado de FST (0.125) que mostró una moderada diferenciación genética. Esta situación es común en poblaciones naturales, como fue verificado por Leuzzi et al (28), quienes al analizar poblaciones de Astyanax altiparanae de dos reservorios del río Paranapanema encontraron que hubo flujo génico entre poblaciones separadas geográficamente.
A partir de estas evidencias, se puede sugerir que el manejo reproductivo, genético y de mejoramiento de los lotes de reproductores de B. orbignyanus debe ser realizado de manera homogénea y no como lotes separados, especialmente en programas de conservación de poblaciones naturales a través del repoblamiento. Otros estudios evaluando lotes de peces utilizados en programas de repoblamiento han sido realizados. Lopera-Barrero et al (1) analizaron cuatro lotes de curimba (Prochilodus lineatus) utilizados en programas de repoblamiento encontraron una baja variabilidad genética entre ellos, resultado de la utilización de estrategias reproductivas incorrectas (uso de pocos animales en la formación del lote y cruzamiento de individuos emparentados) y falta de informaciones genéticas. Entretanto, Povh (19) comparó un lote de reproductores y una población natural de pacu (Piaractus mesopotamicus) encontró una menor diversidad genética en el lote mantenido en cautiverio, debido posiblemente a un deficiente manejo reproductivo.
Los programas de repoblamiento son estrategias comúnmente usadas en la conservación de especies de peces en peligro de extinción o de aquellas que han presentado una reducción significativa de sus poblaciones naturales. Sin embargo, un acompañamiento científico tiene que ser paralelamente usado. Por esta razón, la primera providencia a ser tomada en la implantación de una piscícola o de programas de repoblamiento y conservación, es verificar la diversidad genética de los lotes y posteriormente de las poblaciones naturales, ya que el cruzamiento entre individuos emparentados puede conducir a un aumento de la homocigosis y a una pérdida de la variabilidad genética, provocando una disminución de la supervivencia de las progenies en la piscícola (22) y de aquellas usadas en programas de repoblamiento. Wasko et al (21), analizaron progenies de Brycon cephalus obtenidas a partir de lotes mantenidos en piscícolas, encontraron que el cruzamiento de individuos emparentados provocó la disminución de la variabilidad genética en el transcurso de tres generaciones, lo que según Freitas y Galetti (27) puede ser disminuido al incrementar el número efectivo de reproductores o mediante el cruzamiento entre individuos de diferentes orígenes.
Otro factor que debe ser analizado es que en programas de repoblamiento todos los peces liberados tienen que representar genéticamente a las poblaciones naturales. Según Sønstebø et al (29), el cruzamiento de individuos genéticamente distintos a aquellos encontrados en una población natural puede promover la pérdida de genes importantes de adaptabilidad al ambiente que pueden influir sobre la supervivencia de progenies en el ambiente natural (28, 30). Por esta razón, en la implantación de programas de repoblamiento es necesario el monitoreo genético de los lotes de reproductores que serán usados, de sus progenies que serán liberadas en los ríos y de las poblaciones naturales. La conservación de peces amenazados se ha convertido en una prioridad en el mundo entero en las últimas décadas, ya que fue entendido que existe una fuerte interacción entre los peces, el ecosistema y la realidad socio-cultural de comunidades aledañas que basan su alimentación en estas fuentes de alimento.
En conclusión, la diversidad genética en la progenie del lote A de B. orbignyanus fue preservada debido al adecuado manejo reproductivo. No se encontró diferenciación genética entre los lotes de reproductores. Esa caracterización es de gran importancia en la piscicultura, ya que la base genética de las estaciones piscícolas son las que determinan el éxito de los programas de repoblamiento. Se debe también considerar la necesidad de mayores investigaciones y de más asistencia a los productores, para orientarlos correctamente en los aspectos relacionados al manejo genético y reproductivo, de forma de obtener un mayor retorno económico y una protección del ecosistema.
Agradecimientos
A la empresa Duke Energy International (Geração Paranapanema) y al Conselho Nacional de Desenvolvimento Científico e Técnológico (CNPq)(Brasil).
REFERENCIAS
1 Lopera-Barrero NM, Ribeiro RP, Jacometo CB, Oliveira SN, Streit Jr DP, Blanck DV. Análise genética de estoques de curimba (Prochilodus lineatus) destinados a programas de repovoamento. Memórias 44a Reunião Anual da Sociedade Brasileira de Zootecnia. 2007, Jaboticabal, Brasil. A556. [ Links ]
2 Zaniboni-Filho E, Reynalte-Tataje D, Weingartner M. Potencialidad del género Brycon en la piscicultura brasileña. Rev Col Cienc Pec 2006; 19: 233-240. [ Links ]
3 Borba MR, Fracalossi DM, Pezzato LE. Dietary energy requirement of piracanjuba fingerlings, Brycon orbignyanus, and relative utilization of dietary carbohydrate and lipid. Aquac Nut 2006; 12: 183-191. [ Links ]
4 Machado ABM. Lista da fauna brasileira ameaçada de extinção: incluindo as espécies quase ameaçadas e deficientes em dados. In: ABM Machado, CS Martins, GM Drummond (eds.). Belo Horizonte, Brasil, Fundação Biodiversitas, 2005. [ Links ]
5 Sirol RN, Britto SG. Conservação e manejo da ictiofauna: repovoamento. In: MG Nogueira, R Henry, A Jorcin (eds.). Ecologia de reservatórios: impactos potenciais, ações de manejo e sistemas em cascatas. São Carlos, Brasil, RiMA, 2006. [ Links ]
6 Gomes PC. Diversidade genética de três populações de piapara (Leporinus elongatus), utilizando marcadores moleculares. Dissertação (Mestrado em Zootecnia), Programa de Pós-Graduação em Zootecnia, Universidade Estadual de Maringá, Maringá, Brasil 2007. [ Links ]
7 Zaniboni-Filho E, Nuñer APO. Fisiologia da reprodução e propagação artificial dos peixes. In: Cyrino JEP, Urbinati EC, Fracalossi DM, Castagnolli N (eds.). Tópicos Especiais em Piscicultura de água Doce Tropical Intensiva. São Paulo, Brasil, TecArt, 2004, p. 533. [ Links ]
8 Bardakci F, Skibinski DOF. Application of the RAPD technique in tilapia fish: species and subspecies identification. Heredity 1994; 73: 117-123. [ Links ]
9 Povh JA, Moreira HLM, Ribeiro RP, Prioli AP, Vargas L, Blanck DV, Gasparino E, Streit Jr DP. Estimativa da variabilidade genética em tilápia do Nilo (Oreochromis niloticus) com a técnica de RAPD. Acta Sci An Sci 2005; 27: 1-10. [ Links ]
10 Williams JG, Kubelik AR, Livak J, Rafalski JA, Tingey SV. ADN polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res 1990; 18: 6531-6535. [ Links ]
11 Miller M. MANTEL-ESTRUCT: a program for the detection of population structure via mantel tests. J Hered 1999; 90: 258-259. [ Links ]
12 Miller MP. Tools of population genetic analysis (TFPGA) 1.3: a Windows program for the analysis of allozyme and molecular population genetic data. Utah State University, 1997. [ Links ]
13 Nei M. Analysis of gene diversity in subdivided populations. Proc Nat Acad Sci 1973; 70: 3321-3323. [ Links ]
14 Yeh FC, Boyle TYZ, Xiyan JM. POPGENE Version 131: Microsoft Window-based freeware for population genetic analysis. University of Alberta and Center for International Forestry Research, 1999. [ Links ]
15 Excoffier L, Laval G, Schneider S. Arlequin Ver. 3.0: an integrated software package for population genetics data analysis. Evol Bioinf Online 2005; 1: 47-50. [ Links ]
16 Wright S. Evolution and genetics of population. Chicago: University of Chicago Press, 1978, 580 p. [ Links ]
17 Rohlf FJ. NTSYS-Pc: Numerical Taxonomy and Multivariate Analysis System. New York: Exeter Publishers, 1989. [ Links ]
18 Moreira AA, Hilsdorf AWS, Silva JV, Souza VR da. Variabilidade genética de duas variedades de tilápia nilótica por meio de marcadores microssatélites. Pesqui Agropecu Bras 2007; 42: 521-526. [ Links ]
19 Povh JA. Avaliação da diversidade genética e do manejo reprodutivo do pacu, Piaractus mesopotamicus. Tese (Doutorado em Zootecnia), Programa de Pós-Graduação em Zootecnia, Universidade Estadual de Maringá, Maringá, Brasil 2007. [ Links ]
20 Kang JH, Noh JK, Kim JH, Lee JH, Kim HC, Kim KK, Kim BS, Lee WJ. Genetic relationship between broodstocks of olive flounder, Paralichthys olivaceus (Temminck and Schlegel) using microsatellite markers. Aquac Res 2006; 37: 701-707. [ Links ]
21 Wasko AP, Martins C, Oliveira C, Senhorini Já, Foresti F. Genetic monitoring of the Amazonian fish matrinchã (Brycon cephalus) using RAPD markers: insights into supportive breeding and conservation programmers. J Appl Ichthyol 2004; 20: 48-52. [ Links ]
22 Melo DC, Oliveira DAA, Ribeiro LP, Teixeira CS, Sousa AB, Coelho EGA, Crepaldi DV, Teixeira EA. Caracterização genética de seis plantéis comerciais de tilápia (Oreochromis) utilizando marcadores microssatélites. Arq Bras Med Vet Zootec 2006; 58: 87-93. [ Links ]
23 Yokota M, Harada Y, Iizuka M. Genetic drift in a hatchery and the maintenance of genetic diversity in hatchery-wild systems. Fish Sci 2003; 69: 101-109. [ Links ]
24 Frost LA, Evans BS, Jerry DR. Loss of genetic diversity due to hatchery culture practices in barramundi (Lates calcarifer). Aquaculture 2006; 261: 1056-1064. [ Links ]
25 Aho T, Rönn J, Piironen J, Björklund M. Impacts of effective population size on genetic diversity in hatchery reared Brown trout (Salmo trutta L.) populations. Aquaculture 2006; 253: 244â248. [ Links ]
26 Miller LM, Kapuscinski AR. Genetic guidelines for hatchery supplementation programs. In: EM Hallerman (ed.). Population Genetics: Principles and Applications for Fisheries Scientists, American Fisheries Society, Bethesda, USA, 2003. [ Links ]
27 Freitas PD, Galetti Jr PM. Assessment of the genetic diversity in five generations of commercial broodstock line of Litopenaeus vannamei shrimp. Afr J Biotechnol 2005; 4: 1362-1367. [ Links ]
28 Leuzzi MSP, Almeida FS, Orsi ML, Sodré LMK. Analysis by RAPD of the genetic structure of Astyanax altiparanae (Pisces, Characiformes) in reservoirs on the Paranapanema River, Brazil. Genet Mol Biol 2004; 27: 355-362. [ Links ]
29 Sønstebø JH, Borgstrøm R, Heun M. Genetic structure of brown trout (Salmo trutta L.) from the Hardangervidda mountain plateau (Norway) analyzed by microsatellite ADN: a basis for conservation guidelines. Conserv Genet 2007; 8: 33-44. [ Links ]
30 Almeida FS, Sodre LMK, Contel EPB. Population structure analysis of Pimelodus maculatus (Pisces, Siluriformes) from the Tietê and Paranapanema Rivers (Brazil). Genet Mol Biol 2003; 26: 301-305. [ Links ]














