Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268On-line version ISSN 1909-0544
Rev.MVZ Cordoba vol.16 no.2 Córdoba May/Aug. 2011
REVISIÓN DE LITERATURA
Principales marcadores moleculares utilizados para la identificación de Babesia bovis y Babesia bigemina
Principal molecular markers used to identify Babesia bovis and Babesia bigemina
Sandra Ríos T,1 M.Sc, Leonardo Ríos O,1* Ph.D.
1Universidad de Antioquia, Escuela de Microbiología, Grupo de Investigación en Microbiología Veterinaria, Calle 67 # 53-108, AA 1226. Medellín, Colombia.
*Correspondencia: mleonardo@udea.edu.co
Recibido: Junio de 2010; Aceptado: Febrero de 2011.
Resumen
Este artículo describe los principales marcadores moleculares utilizados para la identificación de B. bovis y B. bigemina reportados en la literatura científica. Para ello se diseñó una revisión sistemática a partir de la aplicación de la estrategia metodológica PICO modificada con el objetivo de definir las secuencias nucleotídicas detectadas en los diferentes sitios geográficos y su utilidad diagnóstica. Se realizó una búsqueda avanzada con los términos "Babesia bovis" y "DNA" y "Babesia bigemina" y "DNA" en las bases de datos ScienceDirect, SpringerLink y PubMed que después de ser filtradas permitieron obtener un resultado total de 68 artículos originales. Tanto los artículos incluidos como los excluidos fueron almacenados en tablas, en las cuales se presenta la justificación de su condición dentro del estudio. A los 68 artículos seleccionados se les aplicó una evaluación con criterios de inclusión y exclusión previamente definidos, de este modo, 21 artículos originales cumplieron con los criterios de inclusión y se incluyeron en el estudio. Se describe la utilidad de los marcadores moleculares referenciados en la literatura científica desde 1995 hasta el 2010: la subunidad pequeña RNAr, el gen citocromo b, gen msa-1 and msa-2c, el gen Bv, el factor de elongación alfa (EF-1α), el gen de la beta-Tubulina, SBP 1-2-3, y los RAP; su aplicación diagnóstica y su utilización en los diferentes sitios geográficos. Los marcadores moleculares utilizados para la detección de las Babesias bovinas varían dependiendo de la región geográfica, grado de conservación genética y resultados de estudios previos que concluyen su utilidad diagnóstica.
Palabras clave: Citocromo b, DNA, marcadores genéticos, secuencias nucleotídicas, tubulina. (Fuente: CAB).
Abstract
This paper describes the principal molecular markers used to identify B. bovis and B. bigemina reported in the scientific literature. A systematic review was designed based on the application of the PICO methodologic modified strategy to define the nucleotide sequences detected in different geographical locations and their diagnostic utility. An advanced search was made in data bases ScienceDirect, SpringerLink and PubMed. Using the terms "Babesia bovis" and "DNA" and "Babesia bigemina" and "DNA". A total of 68 original articles were selected after the information was filtered. Both included and excluded articles were registered in tables, where its position of their status, within the study, was represented. The 68 articles were evaluated using a previously defined criteria of inclusion or exclusion. A total of 21 articles met the inclusion criteria and were included in this study. It was described the usefulness of molecular markers referenced in the scientific literature from 1995 to 2010: small-subunit ribosomal rna, cytochrome b gene, msa-1 and msa-2c gene, bv gene, elongation factor -1 alpha (ef-1α), beta-tubulin gene, sbp 1-2-3, and rap genes, its diagnostic application and its use in different geographical locations. Molecular markers used for detection of bovine Babesia vary by geographic region, degree of genetic conservation and results of previous studies that conclude their diagnostic utility.
Key words: Cytochrome b, DNA, genetic markers, nucleotide sequences, tubulin. (Source: CAB).
Introducción
La Babesiosis bovina, una enfermedad causada por un protozoo intraeritrocítico del orden Piroplasmida, phylum Apicomplexa, género Babesia, se encuentra ampliamente distribuida en países tropicales y subtropicales (1-3). Babesia bigemina y Babesia bovis, son las especies mas frecuentes causantes de esta enfermedad en bovinos y son transmitidas por garrapatas del género Rhipicephalus (Boophilus); otros vectores incluyen Ixodes sp, y Haemaphysalis sp. (4).
B. bigemina es la especie que presenta la distribución más amplia, sin embargo, B. bovis es la más patógena. Las infecciones se caracterizan por presentar fiebre alta, ataxia, anorexia, shock circulatorio general y en ocasiones, síntomas nerviosos como resultado del secuestro de eritrocitos infectados en los capilares cerebrales (5). Los síntomas en las infecciones por B. bigemina incluyen fiebre, hemoglobinuria y anemia, pero no tiene lugar el secuestro intravascular de eritrocitos. (6).
Esta enfermedad es responsable de grandes pérdidas económicas en términos de mortalidad y morbilidad de los bovinos; generando un impacto significativo en la producción de carne y leche y por consiguiente en el manejo de la ganadería. (7).
En la literatura científica, desde la década de los noventa se han reportado secuencias nucleotídicas de cepas circulantes de B. bovis y B. bigemina en diferentes regiones, reportándose genotipos idénticos en Asia, áfrica, América, Europa y Oceanía (8,9); sin embargo, se han reportado cepas con variaciones genéticas asociadas a modificaciones endógenas y exógenas inducidas por tratamientos antiparasitarios, mecanismos de respuesta inmune del hospedador y procesos evolutivos que han generado mecanismos de resistencia y patogenicidad que garantizan su permanencia en el tiempo (7).
Las investigaciones recientes sobre babesiosis bovina se han centrado en la determinación de su taxonomía, el desarrollo de técnicas diagnósticas sensibles y específicas, aspectos de la fisiopatología y búsqueda del mejoramiento de métodos de quimioterapia e inmunoprofilaxis (10). Sin embargo, no existen estudios que recopilen y analicen la literatura existente acerca de los marcadores moleculares más utilizados para la detección de estos hemoparásitos de acuerdo con su distribución geográfica. Por lo anterior, el objetivo de esta revisión sistemática fue describir los principales marcadores moleculares utilizados para la identificación de B. bovis y B. bigemina, reportados en la literatura científica con el fin de definir las secuencias nucleotídicas detectadas en los diferentes sitios geográficos y su utilidad diagnóstica.
Se diseñó un estudio teórico bajo la metodología PICO (11), modificada, donde el paciente se relaciona con la especie parasitaria (B. bovis y B. bigemina), Intervención, con los diferentes marcadores moleculares, Comparación con la ubicación geográfica de los estudios evaluados, y Outcome con el resultado de la descripción de los diferentes estudios sobre los marcadores y la utilidad de cada uno de ellos.
A partir de la aplicación de esta estrategia metodológica se buscó responder a la pregunta de investigación: ¿Cuáles son los principales marcadores moleculares utilizados para la identificación de Babesia bovis y Babesia bigemina reportados en la literatura científica?
Se seleccionaron artículos originales reportados en la literatura científica en los cuales se amplificaron cepas de B. bovis y B. bigemina circulantes en zonas endémicas del mundo; se incluyeron artículos en inglés y español publicados entre enero de 1995 y enero de 2010.
Se excluyeron aquellos artículos que amplificaron DNA de otros parásitos o que utilizaron el DNA de B. bovis y/o B. bigemina como control para el diagnóstico de otras hemoparasitosis, artículos de revisión, protocolos y capítulos de libro.
La búsqueda directa de artículos en las bases de datos Pubmed, Springerlink, Science Direct se realizó utilizando los términos de búsqueda seleccionados con base en los sistemas Mesh (Medical subject Heading) y DecS (Descriptores de Ciencias de la Salud) donde se encuentra el vocabulario de términos biomédicos. Los términos no Mesh (Babesia bigemina) fueron adoptados del sistema DecS. Estos fueron: "Babesia bovis", "Babesia bigemina" y "DNA", unidos por el conector AND, con búsquedas diferentes "Babesia bovis" AND "DNA" y "Babesia bigemina" AND "DNA".
De los 68 artículos seleccionados, 21 artículos originales cumplieron con los criterios de inclusión (Tabla 1); y la ubicación geográfica de los estudios en los cuales se incluyen los marcadores moleculares se presenta en la figura 1.
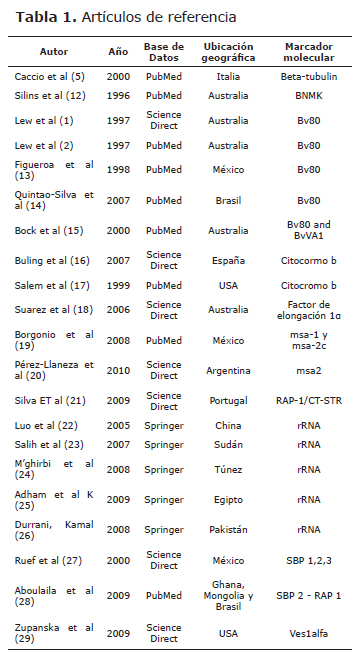
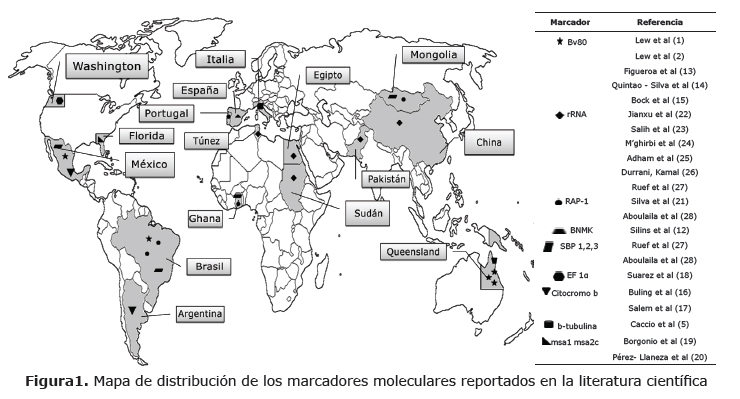
Principales marcadores moleculares utilizados para la identificación de B. bovis y B. bigemina en diferentes regiones del mundo.
Gen BNMK, L35 (b/35). Es una región del genoma nuclear de B. bovis que codifica para una proteína ribosomal homóloga L35 (b/35) y un nucleósido monofosfato quinasa (BNMK). Silins (12) presenta la primera evidencia de secuencias intrónicas, así como de RNAm heterogéneos 5 'y 3' de un miembro del género Babesia. El examen de las estructuras de los genes indicó que la codificación de regiones que contienen pequeñas secuencias de intervención, obedecen a la regla GT-AG de eucariotas o intrones. El único intrón separa el codón de iniciación B135 del resto de la región codificante y el exón (gen BNMK), no parece estar complementado de manera diferente. Ambos genes utilizan sitios de poliadenilación múltiples similares a los de los mamíferos. Los análisis revelan que la extensión del primer en el gen BNMK utiliza un conjunto de puntos de inicio de la transcripción, uno de los cuales se utiliza con más frecuencia (12).
Basándose en estos resultados, se especula que el gen BNMK tiene un papel dentro de la mitocondria de la Babesia sp. La presencia de intrones de tamaño pequeño en el genoma de Babesia sp. sugiere que desempeñan un papel importante dentro de este parásito que podría incluir complementación diferencial (12).
No se identificaron en esta región elementos de repetición, secuencias de transcripción eucariota y otros elementos semejantes de control. La ausencia más notable incluyen la típica señal de mamíferos TATAA / TA (TATA-box), CCAAT (CAAT-box) y GGGCGG (SPL).
Los datos encontrados en los estudios revisados sugieren que se empleó preferencialmente uno de los sitios de poliadenilación por el gen B135, aunque los resultados no fueron tan claros para BNMK. Los múltiples sitios de poliadenilación podrían representar una mezcla de transcripciones dependiendo de la especie de Babesia, que tal vez corresponden a variaciones morfológicas de la especie. Alternativamente, los extremos heterogéneos 3' se pueden generar sin un propósito específico, como está reportado en las plantas. Por otro lado, la heterogeneidad del 3' puede estar relacionada con la presencia de una población de parásitos genéticamente mixta; sin embargo, la escasa variación en los nucleótidos, de acuerdo con lo observado en las secuencias de ADN, sugiere que Babesia sp. no utiliza la señal de los mamíferos AATAAA para dirigir el sitio de poli A. (12).
Subunidad pequeña RNAr. Es el componente pequeño de los ribosomas del citoplasma eucariótico, y por lo tanto uno de los componentes básicos de todas las células eucariotas (22).
Los datos del DNAr 18S son ampliamente utilizados en el análisis molecular y reconstrucción de la historia evolutiva de los organismos; como su ritmo evolutivo es lento, lo hace adecuado para reconocer los cambios en lugar y tiempo (25).
Luo et al (22) compararon y analizaron las secuencias del gen de la subunidad pequeña del RNA ribosomal de seis poblaciones Chinas de Babesias bovinas, incluyeron Babesia sp., B. bigemina, B. bovis, B. ovata, B. major. Los resultados mostraron que la transmisión B. ovata se da por Haemaphysalis longicornis y los aislados de Babesia sp., se limitan al mismo grupo que B. ovata en Corea, con una identidad entre ellos mayor a 96.5%, mientras que B. major que es transmitida por H. punctata fue situado en otra rama, y la identidad con otras especies bovinas de Babesia fue inferior a 92.5%. B. ovata debe, por tanto, ser una especie válida para bovinos, a diferencia de B. major, de acuerdo con la secuencia del gen 18S RNAr.
Salih et al (23), realizaron un estudio epizootiológico de las enfermedades transmitidas por garrapatas al sur de Sudán, usando como gen blanco la subunidad pequeña del RNAr, y como primers, secuencias reportadas en Estados Unidos, que amplificaron perfectamente las cepas circulantes en Sudán, mostrando ser una secuencia conservada en diferentes regiones del mundo.
Por otro lado, M'ghirbi et al (24), en Túnez, realizaron una encuesta molecular de hemoparásitos en Babesia sp y Theileria sp, utilizando para su identificación por PCR la región hipervariable del V4 de este mismo gen. Luego de la secuenciación de los productos amplificados se demostró que es un gen muy conservado para cada una de las especies; esto se confirmó al someter las secuencias para la evaluación de su identidad (BLAST) en comparación con las previamente reportadas en el GenBank (EF643472; AY524666) resultando identidad del 96 al 100% con aislados de México y Turquía.
En Egipto, Adham et al (25) realizaron el análisis de la secuencia del producto de PCR de B. bovis y B. bigemina, encontrando un alto porcentaje de identidad en comparación con las especies similares encontradas en el GenBank. La comparación se hizo mediante un BLAST para cada una de las especies, encontrando identidad desde el 93 al 100% con aislados provenientes de diferentes ubicaciones geográficas. (Tamaulipas, EF642472; Israel, EF601930; Veracruz, EF643474, AY150059; Quintana Roo, EF643469) para B. bovis; y para B. bigemina (DQ785311; AY533147; DQ159075, DQ159072); se reporta así como un gen conservado entre aislados de diferentes continentes, susceptible de ser amplificado en estudios posteriores.
Gen del citocromo b. Gen de la proteína mitocondrial y componente obligatorio en la cadena respiratoria del microorganismo, fue utilizado por Buling et al (16), en España, para detectar y cuantificar Babesias bovinas, debido a que este presenta una mayor cantidad de copias que los genes ribosomales.
Los autores compararon secuencias amplificadas de ambos genes de aislados de España, Portugal, Argentina y, para el gen de la subunidad 18S RNAr, se encontró el 100% de coincidencia con otras secuencias depositadas en la base de datos GenBank, con la única excepción de un aislado de China (AY603402) que mostró diferencias muy leves. Se observó una mayor similitud entre muestras procedentes de América (Argentina, México [obtenidos en el GenBank]) o de Europa (España, Portugal) que entre muestras de diferentes continentes. Mientras que aislados de las ubicaciones geográficas referenciadas mostraron exactamente la misma secuencia de la proteína citocromo b, lo que demuestra su conservación en las diferentes regiones geográficas.
Se sugiere además que la amplificación del gen episomal es más sensible que la del gen ribosomal, porque con ensayos realizados con diluciones seriadas de DNA de muestras positivas se generan productos de amplificación con cantidades más pequeñas de DNA que con el gen ribosomal.
Gen msa-1 y msa-2c. Estos genes presentes en Babesia bovis codifican para las proteínas antigénicas presentes en la superficie del merozoito (msa) y están involucrados en la invasión del parásito a los eritrocitos de la especie bovina. Los estudios realizados en msa-1 han puesto en evidencia la variación alélica en B. bovis en aislados de regiones endémicas similares, así como en los aislados de diferentes regiones geográficas del mundo (Argentina, Australia, Israel). Sin embargo, estudios realizados sobre msa-2c, han demostrado que este antígeno se conserva ampliamente en los aislados de diferentes regiones geográficas (19).
Borgonio et al (19) en México plantearon la hipótesis de que los antígenos msa-1 y msa-2c contienen epítopes comunes a pesar de las diferencias en las secuencias de nucleótidos en 13 aislamientos de B. bovis y cepas recolectadas en regiones geográficamente distantes de México. Se realizó un análisis bioinformático de la estructura primaria del DNA de los fragmentos derivados de la amplificación por PCR, la clonación y secuenciación de los genes msa-1 y msa-2c de las 13 poblaciones de B. bovis, reveló que el producto de genes msa-1 presentes en las diversas cepas probadas es poco conservado entre los aislados obtenidos en una región geográfica similar en México (51-99.7% identidad de secuencia). Los resultados obtenidos del análisis por inmunoblot de extractos proteicos de B. bovis, que reaccionaron con un anticuerpo monoclonal para el antígeno msa-1 de 42 kDa, mostraron de manera concluyente una reacción cruzada con epítopes comunes sólo en aislados de México con una alta identidad en la secuencia (≥ 99%, en ocho aislamientos).
Análisis de secuencia y de alineamiento múltiple demostraron un alto grado de identidad de secuencia en el msa- 2c (90-100%) entre los aislamientos de B. bovis de México y las cepas de referencia. Al utilizar un anticuerpo policlonal de msa-2c, este reaccionó contra los extractos de proteínas reconocidas y conservadas en al menos nueve de los aislamientos de B. bovis. Los resultados obtenidos en este estudio coinciden con los previamente reportados por otros investigadores y confirman que, con base en la conservación de la secuencia identificada en México de B. bovis y las cepas aisladas hasta ahora, msa-2c representa un antígeno ideal que vale la pena evaluar en estudios moleculares como una posible vacuna.
Familia génica Bv. La familia de Bv corresponde a proteínas de las roptrias del merozoito que presentan un polimorfismo limitado dentro de las especies de Babesia; sin embargo, la comparación de las secuencias de las proteínas equivalentes y la organización de los genes sugiere que los miembros de esta familia tienen la posibilidad de adquirir y de tolerar polimorfismos sustanciales en la secuencia de aminoácidos. Se han utilizado sondas polimórficas de DNA para demostrar la heterogeneidad entre aislamientos de Babesia bovis (30-32). Estudios de hibridación de sondas han demostrado que los aislados de B. bovis comprenden subpoblaciones fenotípicamente diferentes y que por procesos de atenuación puede revertir a un fenotipo virulento (30,33-35).
Una de las secuencias conservadas entre especies es el gen BvVA1 de Babesia bovis, y ha sido aplicado con éxito para comparar las subpoblaciones de parásitos (36). Lew et al (37) demostraron que el número de alelos BvVA1 detectado refleja el número de subpoblaciones genéticamente distintas; Dalrymple et al (36) sugirieron que este gen es un marcador genético ideal para la discriminación de las cepas, por ser específico y útil para la caracterización molecular de los otros parásitos apicomplexa. Los genes de Babesia bovis Bv80 y BvVA1 tienen características similares, y las regiones conservadas se encuentran separadas por conjuntos de secuencias repetidas en tándem, que varían en longitud (36), sin embargo, por su alta variabilidad no es recomendable para realizar estudios de frecuencia en diferentes poblaciones del mundo.
Gen del factor de elongación alfa (EF-1α). Codifica para una proteína expresada de forma constitutiva de manera abundante y es un elemento clave en la traducción de proteínas eucariotas. Debido a su alto nivel de la transcripción, el promotor EF-1α ha sido utilizado para dirigir la expresión de genes exógenos en células transfectadas. Otras funciones conocidas de EF-1α en las células eucariotas son la proteólisis dependiente de microtúbulos y la ruptura de la ubiquitina. Además, EF-1α es regulador de la apoptosis programada (22).
En un estudio realizado por Suárez et al (18) identificaron y caracterizaron el EF-1α de Babesia bovis, y evaluaron la actividad transcripcional de los promotores de esta proteína. En Babesia bovis el EF-1α, contiene dos genes EF-1α idénticos ('A' y 'B') dispuestos en orientación cara a cara y separados por 1,4 kb intergénicas (IG), región que contiene una repetición terminal de 260 pb de arriba abajo. Los genes EF-1α codifican proteínas idénticas con 448 aminoácidos y un peso molecular calculado de 49 kDa. Mientras que la secuencia IG de B. bovis de EF-1α se conserva entre las múltiples cepas de B. bovis; no está significativamente relacionado con ninguna secuencia reportada de las bases de datos de DNA. La región IG promueve la expresión de los dos genes EF-1α. El análisis de las transcripciones por EF-1α confirma que ambos genes EF-1α son transcritos en los merozoitos y son muy conservados en esta especie.
Gen de la beta-tubulina. La familia de las tubulinas alfa (α), beta (β) y gamma (γ) son un grupo de proteínas que comparten una identidad entre sus cadenas de aminoácidos de 35-40%, aunque su similitud con cualquier otra proteína conocida es mínima. Las tubulinas α y β son las subunidades esenciales de los microtúbulos, mientras que la tubulina-γ es un componente fundamental del centrosoma. Los fragmentos N-terminales de las tubulinas α y β están notablemente conservados con variaciones mínimas. La alta tasa de conservación dentro de la familia de las tubulinas implica que las propiedades funcionales de estas proteínas imponen unas limitaciones enormes a cualquier diversificación de la secuencia, de manera que las mutaciones sólo pueden acomodarse en unas pocas posiciones sin producir un efecto deletéreo (38)
Caccio et al (38), amplificaron un fragmento del gen de la β-tubulina, para la identificación de las especies patógenas más comunes de Babesia y Theileria. El gen de β-tubulina es uno de los pocos genes apicomplejos que son interrumpidos por uno o varios intrones. Además, la posición de estos intrones se conserva en todas las especies investigadas hasta ahora, lo que permite un diseño fácil de los cebadores en torno a esta región. Por último, los intrones asociados con el gen β-tubulina muestran grandes variaciones tanto en longitud como en secuencia. Estas características hacen de esta región del genoma, un buen candidato para el desarrollo de marcadores informativos, como se ha demostrado previamente para varios parásitos protozoarios. Como se esperaba, mientras que la secuencia de codificación fue muy conservada entre las especies, fue muy poco conservada entre los intrones de las distintas especies (38). En el caso de B. bovis, una comparación de la secuencia genómica parcial obtenida en este trabajo con un DNAc disponible en el GenBank (Q04709) mostró que el intrón supuesto no estaba presente en la secuencia del DNAc (38).
Gen de la proteína del cuerpo esférico (SBP) 1-2-3. Ruef et al (39) identificaron una proteína de 135-kDa que contiene una región conservada en las cepas de B. bovis de Texas, México y Australia. La proteína se localiza en los órganos del complejo apical de Babesia y fue designada proteína del cuerpo esférico 3 (SBP3). Los estudios de inmunofluorescencia mostraron la unión de anticuerpos monoclonales a la región de membrana de los eritrocitos infectados, pero no a sus homólogos no infectados, lo que demuestra una gran asociación con las proteínas de cuerpo esférico, SBP1 y SBP2, aisladas previamente en B. bovis. Con la identificación de proteínas un tercer cuerpo esférico, que se asocia con la cara citoplasmática de la membrana de los eritrocitos infectados, se ha identificado un complemento de distintas proteínas de B. bovis que pueden contribuir a la supervivencia intracelular, al crecimiento y desarrollo de este parásito. Esta proteína debe ser estudiada en futuras investigaciones por ser una región muy conservada con el objetivo de evaluar los procesos inmunológicos que genera esta infección en el hospedador (28).
Gen RAP. Proteínas asociadas a roptrias que proporcionan un buen nivel de detección de las infecciones tempranas y tardías de los bovinos debido a su grado de conservación entre diferentes regiones geográficas. Las comparaciones de secuencias entre genes rap-1 en B. bovis reveló entre el 98 y el 100% de identidad entre todos los aislados portugueses. (GenBank: FJ901342; FJ939723). Por lo tanto, de conformidad con las observaciones anteriores, las secuencias del gen B. bovis rap-1 de aislados portugueses son altamente conservados y similares a los publicados secuencia de B. bovis de la cepa Argentina (R1A, GenBank: AF030055), la cepa de Texas (T2Bo, GenBank AF030059) y la cepa de Australia (S2P, GenBank: AF030056) (40, 41). En cambio, aunque los amplicones de B. bigemina mostraron 100% de identidad de secuencia entre las cepas portuguesas (GenBank: FJ939724), estos tenían un 82% identidad con los publicados en otras regiones (GenBank: S45366) (42). Este estudio mostró la alta conservación del marcador molecular RAP 1 entre cepas de B. bovis pero no para B. bigemina lo que lo hace un marcador específico de especie (21)
En conclusión, los marcadores moleculares utilizados para la detección de las Babesias bovinas B. bovis y B. bigemina varían dependiendo de la región geográfica de estudio, su grado de conservación y los resultados de estudios previos que concluyen su utilidad diagnóstica. Según los datos obtenidos por la revisión de la literatura los marcadores moleculares más utilizados para la detección de especies de Babesia son la subunidad pequeña del RNAr, región muy conservada entre especies que permite, incluso con técnicas de detección múltiple, diferenciar, caracterizar y secuenciar Babesia bovis y Babesia bigemina, y tiene como ventaja que su nivel de conservación se encuentra distribuido en las cepas de diferentes regiones geográficas, aisladas entre sí. Por lo tanto, se presenta como un marcador molecular ideal para la amplificación del material genético con baja probabilidad de modificación. (22-24, 26)
El gen Citocromo B fue comparado con la subunidad pequeña del RNAr y, aunque presentó mayor número de copias, sigue predominando la conservación del marcador ribosomal; en este caso, de acuerdo con los objetivos planteados en estudios de amplificación de Babesia sp., se deben definir los criterios de selección para el marcador molecular, de acuerdo con los resultados esperados en la investigación, referidos al grado de conservación de la secuencia (16, 43).
En este sentido, uno de los principales criterios para la selección del marcador molecular a emplear para la detección de Babesia sp en bovinos se encuentra referido a su nivel de conservación. De acuerdo con la literatura revisada, tanto para B. bovis como B. bigemina, la amplificación de la subunidad pequeña del RNAr se presenta como el método más adecuado con alto nivel de sensibilidad, y reportes de identidad cercanos al 100%; igualmente el gen Citocromo B, aunque con menos estudios, ha sido reportado con porcentajes de identidad superiores al 90% para ambas especies. (Tabla 2).

Otro de los marcadores moleculares más utilizados es el gen Bv, específico de especie que corresponde a proteínas de las roptrias del merozoito, y presenta un polimorfismo limitado dentro de las especies de Babesia, lo cual sugiere que los miembros de esta familia tienen la posibilidad de adquirir y de tolerar polimorfismos sustanciales en la secuencia de aminoácidos; en consecuencia, sus posibles modificaciones puedan alterar potencialmente os resultados de las amplificaciones en diferentes regiones geográficas con cepas diferentes. Estudios previos realizados con este gen en cepas australianas, evidencian la posibilidad de explorar su uso como marcador molecular ideal específico de cepas circulantes en una región específica de Latinoamérica (44).
El gen RAP muestra ser muy conservado en la especie B. bovis pero no en B. bigemina; lo que lo hace un marcador específico en estudios que sólo busquen amplificar esta especie (3, 21)
Por otra parte, genes como la Beta tubulina, los msa, SBP, el factor de elongación 1alfa; requieren más estudios para hacer una comparación y selección de estas secuencias como marcadores, ya que aunque demuestran ser muy conservadas, no es posible inferir su comportamiento a lo largo del mundo, por falta de reportes que generen conclusiones para sugerir utilizarlos como marcadores moleculares. (5, 10, 18-20, 27, 28)
Es importante recalcar que la información acerca de los marcadores moleculares no sólo es de interés para estudios epizootiológicos en lo relacionado con el desarrollo de métodos de detección con altos niveles de sensibilidad; dos aspectos adicionales se hacen relevantes sobre los posibles usos de esta información.
En primer lugar, está el carácter inmunogénico de los marcadores descritos, de los cuales se concluye que mientras mayor sea la identidad presente entre los marcadores de los mismos aislados, y entre diferentes regiones geográficas, habrá mayor potencial inmunogénico; algunos ejemplos de esta utilidad los encontramos en el marcador msa-2c, sobre el cual algunos estudios evidencian la presencia de epítopes comunes que permiten suponer un alto potencial para el desarrollo de vacunas (19, 20), igual potencial se ha referido al marcador RAP-1 (21); en el caso de los genes que codifican las proteínas SBP1, SBP2, SBP3, estos sólo han sido detectados en B. bovis, lo cual evidencia que esta familia de genes posee un alto potencial para diagnóstico de esta especie, pero al mismo tiempo, dicha característica la convierte en un potencial candidato para el desarrollo de vacunas específicas de especie en diversas regiones geográficas (28); así como ocurre con la proteína SBP3, la cual se ha asociado con la supervivencia intracelular y el crecimiento y desarrollo del parásito, y por lo tanto, se convierte en un blanco potencial para investigaciones sobre la inmunidad del hospedador y su uso como vacuna. (27)
El segundo aspecto relevante se refiere a la posibilidad de discriminar cepas vacunales de cepas circulantes utilizando marcadores altamente conservados; estudios realizados con los genes Bv80 y BvVA1 se sugiere su utilización en pruebas de PCR para la certificación de la identidad de los parásitos en procesos de vacunación para B. bovis con parásitos vivos atenuados, de forma que permitan diferenciar cepas vacunales de cepas de campo. (1, 2). En este mismo sentido, los estudios sobre el marcador ssrRNA proponen que la sensibilidad de su amplificación facilita el análisis de vacunas y su capacidad para inducir o evitar el estado de portador; además, su detección puede ser utilizada para probar la eficacia de medicamentos contra el parásito y realizar estudios sobre la transmisión (25). Estudios sobre este mismo marcador como el de Salih et al (23) sugieren la necesidad de realizar futuras investigaciones que se centren en la caracterización de aislados de campo de diferentes áreas en una región geográfica determinada, utilizando cepas mantenidas tanto in vitro como in vivo para caracterizar su nivel de inmunogenicidad y determinar de esta manera su uso en la inmunización contra la babesiosis bovina, teniendo en cuenta el grado de conservación del marcador.
Agradecimientos
Universidad de Antioquia, Escuela de Microbiología y grupo de investigación en Microbiología Veterinaria por su apoyo logístico y académico.
Referencias
1. Lew AE, Dalrymple BP, Jeston PJ, Bock RE. PCR methods for the discrimination of Babesia bovis isolates. Vet Parasitol 1997; 71(4):223-37. [ Links ]
2. Lew AE, Bock RE, Croft JM, Minchin CM, Kingston TG, Dalgliesh RJ. Genotypic diversity in field isolates of Babesia bovis from cattle with babesiosis after vaccination. Aust Vet J 1997; 75(8):575-8. [ Links ]
3. Buening GM, Figueroa JV. Detection of Babesia bigemina infection: use of a DNA probe--a review. Mem Inst Oswaldo Cruz 1992;87(Suppl 3):207-11. [ Links ]
4. Ravindran R, Rao JR, Mishra AK. Detection of Babesia bigemina DNA in ticks by DNA hybridization using a nonradioactive probe generated by arbitrary PCR. Vet Parasitol 2006; 10;141(1-2):181-5. [ Links ]
5. Caccio S, Camma C, Onuma M, Severini C. The beta-tubulin gene of Babesia and Theileria parasites is an informative marker for species discrimination. Int J Parasitol 2000; 30(11):1181-5. [ Links ]
6. Oliveira MC, Oliveira-Sequeira TC, Araujo JP, Jr., Amarante AF, Oliveira HN. Babesia spp. infection in Boophilus microplus engorged females and eggs in Sao Paulo State, Brazil. Vet Parasitol 2005; 130(1-2):61-7. [ Links ]
7. Criado A, Martinez J, Buling A, Barba J, Merino S, Jefferies R, et al. New data on epizootiology and genetics of piroplasms based on sequences of small ribosomal subunit and cytochrome b genes. Vet Parasitol 2006; 142(3-4):238-47. [ Links ]
8. Altangerel K, Alhassan A, Iseki H, Sivakumar T, Boldbaatar D, Yokoyama N, et al. Evaluation of Babesia bigemina 200 kDa recombinant antigen in enzyme-linked immunosorbent assay. Parasitol Res 2009; 105(1):249-54. [ Links ]
9. Alhassan A, Pumidonming W, Okamura M, Hirata H, Battsetseg B, Fujisaki K, et al. Development of a single-round and multiplex PCR method for the simultaneous detection of Babesia caballi and Babesia equi in horse blood. Vet Parasitol 2005; 129(1-2):43-9. [ Links ]
10. Aktas M, Altay K, Dumanli N. Determination of prevalence and risk factors for infection with Babesia ovis in small ruminants from Turkey by polymerase chain reaction. Parasitol Res 2007; 100(4):797-802. [ Links ]
11. Pai M, McCulloch M, Gorman J, Pai N, Enanoria W, Kennedy G, et al. Systematic reviews and meta-analyses: an illustrated, step-by-step guide. Natl Med J India 2004; 17(2):86-95. [ Links ]
12. Silins GU, Blakeley RL, Riddles PW. Characterisation of genes encoding a nucleoside monophosphate kinase and a L35 ribosomal protein from Babesia bovis. Mol Biochem Parasitol 1996; 76(1-2):231-44. [ Links ]
13. Figueroa J, Alvarez J, Ramos J, Rojas E, Santiago C, Mosqueda J, et al. Bovine babesiosis and anaplasmosis follow-up on cattle relocated in an endemic area for hemoparasitic diseases. Ann N Y Acad Sci 1998; 849:1-10. [ Links ]
14. Quintao-Silva MG, Melo MN, Ribeiro MF. Comparison of duplex PCR and microscopic techniques for the identification of Babesia bigemina and Babesia bovis in engorged female ticks of Boophilus microplus. Zoonoses Public Health 2007; 54(3-4):147-51. [ Links ]
15. Bock R, Lew A, Minchin C, Jeston P, Jorgensen W. Application of PCR assays to determine the genotype of Babesia bovis parasites isolated from cattle with clinical babesiosis soon after vaccination against tick fever. Aust Vet J 2000; 78(3):179-81. [ Links ]
16. Buling A, Criado-Fornelio A, Asenzo G, Benitez D, Barba-Carretero JC, Florin-Christensen M. A quantitative PCR assay for the detection and quantification of Babesia bovis and B. bigemina. Vet Parasitol 2007; 147(1-2):16-25. [ Links ]
17. Salem G, Liu X, Johnsrude J, Dame J, Roman Reddy G. Development and evaluation of an extra chromosomal DNA-based PCR test for diagnosing bovine babesiosis. Mol Cell Probes 1999; 13(2):107-13. [ Links ]
18. Suarez CE, Norimine J, Lacy P, McElwain TF. Characterization and gene expression of Babesia bovis elongation factor-1alpha. Int J Parasitol 2006; 36(8):965-73. [ Links ]
19. Borgonio V, Mosqueda J, Genis AD, Falcon A, Alvarez JA, Camacho M, et al. msa-1 and msa-2c gene analysis and common epitopes assessment in Mexican Babesia bovis isolates. Ann N Y Acad Sci 2008; 1149:145-8. [ Links ]
20. Pérez-Llaneza A, Caballero M, Baravalle E, Mesplet M, Mosqueda J, Suarez CE, et al. Development of a tandem repeat-based multilocus typing system distinguishing Babesia bovis geographic isolates. Vet Parasitol 2010; 167(2-4):196-204. [ Links ]
21. Silva M, Henriques G, Sánchez C, Marques P, Suarez C, Oliva A. First survey for Babesia bovis and Babesia bigemina infection in cattle from Central and Southern regions of Portugal using serological and DNA detection methods. Vet Parasitol 2009; 166(1-2):66-72. [ Links ]
22. Luo J, Yin H, Guan G, Yang D, Liu A, Ma M, et al. A comparison of small-subunit ribosomal RNA gene sequences of bovine Babesia species transmitted by Haemaphysalis spp. in China. Parasitol Res 2005; 95(2):145-9. [ Links ]
23. Salih D, El Hussein A, Seitzer U, Ahmed J. Epidemiological studies on tick-borne diseases of cattle in Central Equatoria State, Southern Sudan. Parasitol Res 2007; 101(4):1035-44. [ Links ]
24. M'Ghirbi Y, Hurtado A, Barandika JF, Khlif K, Ketata Z, Bouattour A. A molecular survey of Theileria and Babesia parasites in cattle, with a note on the distribution of ticks in Tunisia. Parasitol Res 2008; 103(2):435-42. [ Links ]
25. Adham FK, Abd-el-Samie EM, Gabre RM, el-Hussein H. Detection of tick blood parasites in Egypt using PCR assay I. Babesia bovis and Babesia bigemina. Parasitol Res 2009; 105(3):721-30. [ Links ]
26. Durrani AZ, Kamal N. Identification of ticks and detection of blood protozoa in friesian cattle by polmerase chain reacton test and estimation of blood parameters in district Kasur, Pakistan. Trop Anim Health Prod 2008; 40(6):441-7. [ Links ]
27. Ruef B, Dowling S, Conley P, Perryman L, Brown W, Jasmer D, et al. A unique Babesia bovis spherical body protein is conserved among geographic isolates and localizes to the infected erythrocyte membrane. Mol Biochem Parasitol 2000; 105(1):1-12. [ Links ]
28. AbouLaila M, Yokoyama N, Igarashi I. Development and evaluation of two nested PCR assays for the detection of Babesia bovis from cattle blood. Vet Parasitol 2010; 172(1-2):65-70. [ Links ]
29. Zupanska A, Drummond P, Swetnam D, Al-Khedery B, Allred D. Universal primers suitable to assess population dynamics reveal apparent mutually exclusive transcription of the Babesia bovis ves1alpha gene. Mol Biochem Parasitol 2009; 166(1):47-53. [ Links ]
30. Cowman A, Bernard O, Stewart N, Kemp D. Genes of the protozoan parasite Babesia bovis that rearrange to produce RNA species with different sequences. Cell 1984; 37(2):653-60. [ Links ]
31. Dalrymple BP. Cloning and characterization of the rRNA genes and flanking regions from Babesia bovis: use of the genes as strain discriminating probes. Mol Biochem Parasitol 1990; 43(1):117-24. [ Links ]
32. Jasmer DP, Reduker DW, Goff WL, Stiller D, McGuire TC. DNA probes distinguish geographical isolates and identify a novel DNA molecule of Babesia bovis. J Parasitol 1990; 76(6):834-41. [ Links ]
33. Gill A, Cowman A, Stewart N, Kemp D, Timms P. Babesia bovis: molecular and biological characteristics of cloned parasite lines. Exp Parasitol 1987; 63(2):180-8. [ Links ]
34. Timms P, Barry D, Gill A, Sharp P, de Vos A. Failure of a recombinant Babesia bovis antigen to protect cattle against heterologous strain challenge. Res Vet Sci 1988; 45(2):267-9. [ Links ]
35. Carson CA, Timms P, Cowman AF, Stewart NP. Babesia bovis: Evidence for selection of subpopulations during attenuation. Exp Parasitol 1990; 70(4):404-10. [ Links ]
36. Dalrymple BP, Casu RE, Peters JM, Dimmock CM, Gale KR, Boese R, et al. Characterisation of a family of multi-copy genes encoding rhoptry protein homologues in Babesia bovis, Babesia ovis and Babesia canis. Mol Biochem Parasitol 1993; 57(2):181-92. [ Links ]
37. Jones SH, Lew AE, Jorgensen WK, Barker SC. Babesia bovis: Genome size, number of chromosomes and telomeric probe hybridisation. Int J Parasitol 1997; 27(12):1569-73. [ Links ]
38. Cacciò S, Cammà C, Onuma M, Severini C. The [beta]-tubulin gene of Babesia and Theileria parasites is an informative marker for species discrimination. Int J Parasitol 2000; 30(11):1181-5. [ Links ]
39. Ruef BJ, Dowling SC, Conley PG, Perryman LE, Brown WC, Jasmer DP, et al. A unique Babesia bovis spherical body protein is conserved among geographic isolates and localizes to the infected erythrocyte membrane. Mol Biochem Parasitol 2000; 105(1):1-12. [ Links ]
40. Suarez CE, Palmer GH, Jasmer DP, Hines SA, Perryman LE, McElwain TF. Characterization of the gene encoding a 60-kilodalton Babesia bovis merozoite protein with conserved and surface exposed epitopes. Mol Biochem Parasitol 1991; 46(1):45-52. [ Links ]
41. Hotzel I, Suarez CE, McElwain TF, Palmer GH. Genetic variation in the dimorphic regions of RAP-1 genes and rap-1 loci of Babesia bigemina. Mol Biochem Parasitol 1997; 15;90(2):479-89. [ Links ]
42. Figueroa JV, Chieves LP, Johnson GS, Buening GM. Detection of Babesia bigemina-infected carriers by polymerase chain reaction amplification. J Clin Microbiol 1992; 30(10):2576-82. [ Links ]
43. Salem GH, Liu XJ, Johnsrude JD, Dame JB, Roman Reddy G. Development and evaluation of an extra chromosomal DNA-based PCR test for diagnosing bovine babesiosis. Mol Cell Probes 1999; 13(2):107-13. [ Links ]
44. Lew A, Dalrymple B, Jeston P, Bock R. PCR methods for the discrimination of Babesia bovis isolates. Vet Parasitol 1997; 71(4):223-37. [ Links ]














