Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268
Rev.MVZ Cordoba vol.18 supl.1 Córdoba Dec. 2013
ORIGINAL
polimorfismos del gen BoLA-DRB3.2* en ganado criollo colombiano
Polymorphism of BoLA-DRB3.2* gen in creole colombian breeds
Darwin Hernández H,1 M.Sc, Andrés Posso T,1 M.Sc, Jaime Muñoz F,1 Ph.D, Guillermo Giovambattista,2 Ph.D, Luz Álvarez F,1* Ph.D.
1Universidad Nacional de Colombia, Facultad de Ciencias Agropecuarias, Laboratorio de Genética Animal, Palmira, Colombia.
2Universidad Nacional de la Plata, Facultad de Ciencias Veterinarias, Instituto de Genética Veterinaria IGEVET, La Plata, Provincia de Buenos Aires, República Argentina.
*Correspondencia: laalvarezf@unal.edu.co
Recibido: Agosto de 2012; Aceptado: Abril de 2013.
RESUMEN
Objetivo. Caracterizar el polimorfismo del gen BoLA-DRB3.2* en las razas bovinas criollas y colombianas. Materiales y métodos. En 360 muestras de ADN de ocho razas bovinas criollas (Blanco Orejinegro, Casanareño, Costeño con Cuernos, Chino Santandereano, Caqueteño, Hartón del Valle, Romosinuano y San Martinero), dos razas sintéticas Colombianas (Lucerna y Velásquez) y dos razas foráneas (Brahman y Holstein) se evaluó el polimorfismo del gen BoLA-DRB3.2 mediante técnicas moleculares (PCR-RFLP); se calculó el número promedio de alelos (NPA), las frecuencias, la heterocigocidad esperada (He) y observada (Ho), el equilibrio de Hardy-Weinberg, la estructura genética y los valores de FST y FIS. Resultados. El NPA fue 14.6 ± 3.8 siendo Caqueteño la raza con mayor NPA (25) y el menor el Chino Santandereano (10). Se encontraron 41 alelos BoLA-DRB3.2* los más frecuentes fueron *28, *37, *24, *23, *20, *27, *8, *16, *39 (0.17, 0.11, 0.10, 0.09, 0.09, 0.07, 0.07 y 0.06 respectivamente). Se encontró alta diversidad genética (He = 0.878) con mayor valor en Caqueteño (0.96) y menor en San Martinero (0.81). Todas las razas se encontraron en equilibrio de Hardy-Weinberg, se encontraron valores altamente significativos de diferenciación genética (FST= 0.044) y de coeficiente de endogamia (FIS = 0.249). Conclusiones. El ganado criollo colombiano posee alto polimorfismo del gen BoLA-DRB3.2* representado en los altos valores de NPA y diversidad génetica.
Palabras clave: Antígenos, biología molecular, diversidad genética (Fuente: CAB).
ABSTRACT
Objective. To characterize BoLA-DRB3.2*gen polymorphism in Colombian Creole breeds. Materials and methods. Using 360 DNA samples from eight Creole bovine breeds (Blanco Orejinegro, Casanareño, Costeño con Cuernos, Chino Santandereano, Caqueteño, Hartón del Valle, Romosinuano and San Martinero), two synthetic Colombian breeds (Lucerna and Velásquez) and two introduced breeds (Brahmán and Holstein), polymorphism of BoLA-DRB3.2* was evaluated using molecular techniques (PCR-RFLP). Allele average number (AAN), expected (He) and observed (Ho) allele frequencies, heterozygosity, Hardy-Weinberg equilibrium (HW), genetic structure and FST and FIS values were estimated. Results. AAN was 14.6 ± 3.8, Caqueteño breed displayed the highest AAN value (25) and Chino Santandereano the lowest (10). 41 alleles of BoLA-DRB3.2* were detected. The most frequent were *28, *37, *24, *23, *20, *27, *8, *16 and *39 (0.17, 0.11, 0.10, 0.09, 0.09, 0.07, 0.07 and 0.06 respectively). High genetic diversity was found (He=0.878) with the highest value for Caqueteño (0.96) and lowest for San Martinero (0.81). All breeds were in HW, and highly significant values of genetic differentiation (FST=0.044) and inbreeding coefficient (FIS=0.249) were found. Conclusions. The Colombian Creole breeds have a high BoLA-DRB3.2*gen polymorphism represented by the high AAN and genetic diversity values.
Key words: Antigens, molecular biology, genetic diversity (Source: CAB).
INTRODUCCIÓN
El Complejo Mayor de Histocompatibilidad (CMH) de los bovinos es conocido como Antígenos de los Leucocitos Bovinos (BoLA) y se localiza en el cromosoma 23. Los Antígenos son glicoproteínas que se ubican en la superficie celular y su función principal es la presentación de péptidos a las células B y T (1).
Existen tres clases de antígenos leucocitarios bovinos, los antígenos clase I que constan de dos cadenas polipeptídicas codificadas por locus diferentes. Una de ellas atraviesa la membrana celular, tiene un peso molecular aproximado de 45000 D y la cadena más corta llamada microglobulina β2 tiene un peso aproximado de 12000 D. Los genes BoLA clase I codifican proteínas implicadas en el reconocimiento de las células del hospedero que han sido infectadas y en la presentación de péptidos a las células citotóxicas T y T CD8+ (1,2).
Los antígenos clase II son dos cadenas de un tamaño similar. Los genes clase II están localizados en dos partes diferentes del cromosoma 23, posee dos regiones llamadas clase IIa y clase IIb separadas por 15cM. La clase IIa contiene los genes DR y DQ y la clase IIb los genes DYA, DYB, DMA, DMB, DOB, DOA, TAP1, TAP2, LAPM2 y LMP7. Estos genes producen proteínas implicadas en la comunicación entre las células B y T, procesos antígenos extracelulares con células T CD4+ y otras funciones inmunes (1).
Mientras que, los genes clase III codifican proteínas del sistema de complemento, moléculas relacionadas con la inflamación y proteínas de choque térmico (2).
Los genes y los productos de la región clase IIa son los más estudiados porque presentan altos niveles de polimorfismo. Han sido identificados 14 genes en esta región: DRA, DRB (DRB1, DRB2, DRB3), DQA (DQA1, DQA2, DQA3, DQA4, DQA5) y DQB (DQB1, DQB2, DQB3, DQB4, DQB5) (1), donde el gen DRB3 es el más estudiado, este tiene una longitud de 11,4 Kbp con cinco intrones y seis exones, de los cuales el más polimórfico es el exón 2 (BoLA-DRB3.2) de un tamaño aproximado de 270bp (3).
Los genes BoLA-DRB3.2 han sido asociados con caracteres productivos como producción de leche, proteína y grasa en leche (4-6) y con enfermedades como linfocitosis persistente, carga proviral, brucelosis, mastitis y parásitos como las garrapatas (5,7-13).
Algunos autores (9,10) señalan que los bovinos criollos expresan características adaptativas de gran importancia como tolerancia al calor y humedad y resistencia a ciertas enfermedades, lo cual incrementa su valor como recurso genético.
El objetivo del presente trabajo fue caracterizar el polimorfismo del gen BoLA-DRB3.2* en las razas criollas y sintéticas Colombianas mediante técnicas moleculares (PCR-Semianidado y RFLP).
MATERIALES Y MÉTODOS
Instalaciones y muestras de ADN. El estudio se realizó en el Laboratorio de Genética Animal de la Universidad Nacional de Colombia sede Palmira. Se utilizaron 30 muestras de ADN de cada raza de ganado criollo (Blanco Orejinegro - BON, Chino Santandereano - ChS, Costeño con cuernos - CCC, Caqueteño - CQT, Casanareño - CAS, San Martinero - SM, Romosinuano - RS, Hartón del Valle - HV), de cada raza sintética Colombiana (Lucerna - LUC y Velásquez - VEL) y razas foráneas (Brahman - B y Holstein - H) del banco de ADN del Laboratorio. La concentración se determinó utilizando ADN del bacteriófago lambda de concentraciones conocidas, visualizado mediante geles de agarosa al 0.8 % (p/v) teñidos con bromuro de etidio. El ADN fue diluido a 20 ng/µl.
Reacciones de amplificación por PCR. Se amplificó el segundo exón del gen BoLA-DRB.3, por un protocolo de PCR de dos pasos (semi-anidado) (14); con los cebadores: HL030 (5’-ATCCTC TCTCTGCAGCACATTTCC-3’), HL031 (5’-TTTAAATTCGCGCTCACCTCGCCGCT-3’), HL032 (5’-TCGC CGCTCAGTGAAACTCTC-3’); en la primer reacción de amplificación se utilizaron los oligonucleótidos HL030 y HL031 (1.25mM), en 25 µl de mezcla total, con 20 a 40 ng de ADN, 0.2 mM de cada dNTP, 1X de tampón PCR, 2.5 mM MgCl2 y 1U de Taq DNA Polimerasa (Fermentas®). El programa de amplificación constó de una desnaturalización inicial de 4 minutos a 94°C, 10 ciclos de 1 minuto a 94°C, 2 minutos a 60°C y 1 minuto a 72°C, con una extensión final de 5 minutos a 72°C (7). Para la segunda reacción se tomaron 4 µl de la primera reacción de PCR como molde y los oligonucleótidos HL030 y HL032 en un volumen total de 50 µl, con las mismas concentraciones de dNTPs, tampón PCR, MgCl2 y Taq DNA polimerasa. El programa de amplificación constó de una desnaturalización inicial de 4 minutos a 94°C y 25 ciclos de 1 minuto a 94°C, 30 segundos 65°C y 1 minuto a 72°C, con una extensión final de 5 minutos a 72°C (7). Todas las amplificaciones se realizaron en un termociclador modelo PTC-100® (Peltier Thermal Cycler BIO-RAD). El producto de ambas reacciones fue una banda de 282 pares de bases.
RFLP, Electroforesis e interpretación de los cortes. 10µl del producto de PCR de la segunda reacción se utilizaron como sustrato para la digestión con 5U de las enzimas RsaI, BstyI y HaeIII (Fermentas®) a 37°C durante 4 horas (7). La electroforesis se realizó en geles de agarosa SFRTM (Super Fine Resolution, AMRESCO®) al 3% (p/v) y teñidos con bromuro de etidio en una cámara SUB-CELL® GT (BIO-RAD). La lectura de los alelos se basó en la nomenclatura del 5th BoLA workshop (15).
Análisis estadístico. Se formaron los siguientes grupos: criollos (BON, CAS, CCC, ChS, CQT, HV, RS y SM), sintéticas (LUC y VEL) y foráneas (B y H). El ganado criollo colombiano (GCC) está formado por las razas criollos y las razas sintéticas. Se determinó el número promedio de alelos, las frecuencias alélicas, la heterocigocidad esperada (He) y observada (Ho) y el equilibrio de Hardy - Weinberg (EHW). La estructura genética se infirió a partir del AMOVA basado en las frecuencias alélicas de las poblaciones, se determinaron los coeficientes de endogamia (FIS) y de diferenciación genética (FST) usando el programa ALERQUIN versión 3.5 (16).
RESULTADOS
Se analizaron 360 animales, de los cuales 240 fueron criollos, 60 de razas sintéticas y 60 de razas foráneas. Se encontraron 41 alelos BoLA-DRB3.2*, de los cuales sólo 20 estuvieron presentes en las razas foráneas B y H. La frecuencia y distribución de los alelos de acuerdo con la raza se muestra en la tabla 1. Los alelos BoLA-DRB3.2 *23, *36 y *37 se encontraron en todas razas, mientras que los alelos *7, *9, *11, *19, *32, *33, *35, *39, *41, *45, *46, *48, *50 y *51 en sólo una raza, siendo estos alelos únicos para el GCC.
El NPA para toda la población fue de 14.6±3.8, para las razas criollas de 14.6±4.6, para las sintéticas de 12.5±0.71 y para las foráneas de 15±1.41. El CQT presentó el mayor número de alelos (25 alelos) y el ChS el menor (10 alelos), mientras que en las razas sintéticas el mayor número de alelos lo presentó LUC (13 alelos) y en las razas foráneas B (16 alelos).
La raza CQT presentó el mayor número de alelos únicos (8 alelos), de otro lado el BON, SM, RS, CAS, LUC y VEL presentaron sólo uno. Las razas foráneas no mostraron alelos únicos.
El 58% de los alelos encontrados en el ganado GCC y el 36% en las foráneas tuvieron frecuencias menores a 5%. Los alelos con frecuencias superiores a 5% en el GCC fueron: BoLA-DRB3.2 *28 (0.15), *37 (0.128), *24 (0.089), *23 (0.093) y *20 (0.078). Las frecuencias en cada grupo racial se detallan en la tabla 1. En las razas foráneas los alelos con frecuencia mayor al 5% fueron los BoLA-DRB3.2 *13 (0.167), *37 (0.15), *36 (0.09), *23 (0.079), *28 (0.061) y el *25 (0.052).
Las razas sintéticas compartieron en promedio el 33% de sus alelos con las razas criollas que las conforman.
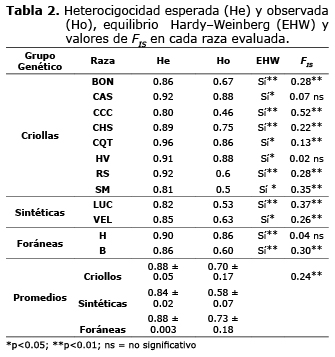
El promedio de He fue de 0.88±0.05 para los ganados criollos, para las sintéticas y las foráneas fue 0.84±0.02 y 0.88±0.003 respectivamente. En general la He fue mayor que la Ho para todas las razas. La raza con mayor diversidad genética (He) fue CQT seguido por CAS y RS, mientras que los valores más bajos de He los presentaron CCC y SM. El VEL mostró mayor valor de He que LUC. En las razas foráneas los valores de He fueron 0.86 y 0.90 para B y H respectivamente (Tabla 2).
Todas las razas de ganado criollo se encontraron en equilibrio de Hardy-Weinberg, aunque las razas criollas CAS y CQT y la VEL no mostraron diferencias altamente significativas (p<0.05).
El valor promedio de FIS fue 0.249 (Tabla 2). En el ganado criollo los valores más altos de endogamia lo presentaron CCC y el SM, en los sintéticos el LUC y en las foráneas el B (p<0.01). En las razas CAS y HV el FIS no fue significativamente diferente de cero (p<0.05) con bajos valores de fijación genética.
La estructura genética mostró un valor de FST de 0.044 (p<0.01) y un porcentaje de variación del 4.48% al comparar entre sí al GCC. El valor de FST fue menor al comparar los criollos con las razas sintéticas (0.043, p<0.01) y solo 0.41% de variación. Los mayores valores de FST se presentaron al comparar criollos y foráneas con 0.051 para B (4.46% de variación) y 0.052 para H (2.99% de variación) (p<0.01).
DISCUSIÓN
Los 41 alelos BoLA-DRB3.2* encontrados es el mayor número de alelos reportados para este gen en ganados criollos; sin embargo, el NPA encontrado por raza es menor (14.6±4.6) que el reportado en criollos de México 34 alelos en el Tamaulipas y 18 en el Chihuahua (17), en el criollo Saavedreño de Bolivia 22 (18), 21 en el criollo argentino (19) y 22 en criollo de Uruguay (20). El valor bajo de NPA por raza se explica porque sólo fueron utilizados 30 por cada raza criolla colombiana y los demás autores presentan al menos 50 animales por raza (5, 12, 18-21). En razas foráneas presentes en América se reportan en el Holstein de Argentina valores que van desde 17 (13) a 26 (12) alelos, en la raza Gyr de Brasil se reportan 37 alelos (5) y en Holstein (21) y Brahman (9) de Colombia 27 y 11 alelos respectivamente. Un valore similar de NPA al aquí encontrado se reporta en Holstein (9).
En el ganado criollo colombiano sólo la raza BON tiene reportes de estudios en este mismo gen donde muestran 31 alelos (n=140) (9) y en 27 alelos (n=25), (21), en el presente estudio se encontraron 15 alelos para este mismo grupo racial.
El GCC compartió al menos un alelo con frecuencia mayor al 5% con otras razas criollas genotipificadas, el alelo *37 lo compartió con el Saavedreño (18), el *20 y *24 con el criollo argentino (19), con los criollos mexicanos Chihuahua y Tamaulipas el *23 y el *24 (17). No se encontraron alelos de alta frecuencia compartidos con el criollo uruguayo (20). Un resultado similar se encontró al comparar el GCC y las razas foráneas presentes en América, así los alelos *23 y *24 de alta frecuencia en el Holstein de Argentina (12,13) también lo fueron en el GCC, el *27 en el Gyr brasilero (5) y el *24 en el Holstein de Colombia (21).
Los alelos *28 y *37 los de más alta frecuencia en el GCC con presencia en todas las razas pueden ser considerados alelos fijos en el GCC. Por otro lado, en la raza CQT que tuvo el mayor número de alelos la mayoría de ellos presentaron bajas frecuencias (≤5%), por lo que se pueden catalogar como alelos únicos.
En la raza BON se reporta (9) que los alelos más frecuentes son el *11, *34, *1 y *12. Sin embargo, en el presente estudio no se encontró ninguno de estos alelos para el mismo grupo racial.
De igual manera, varios de los alelos aquí reportados han sido asociados con resistencia a algunas enfermedades, los alelos *11, *23 y *28 con la ausencia de linfocitos persistente (13), *11 y *12 con baja carga proviral del virus de la leucosis bovina (12), *23, *26 y *31 asociados con resistencia a Brucelosis (9), con resistencia a garrapatas el *8, *16, *20 y *27 (10), con diferentes tipo de mastitis el *3, *11, *31 y *33 (5,6). Esta metodología de genotipaje puede ser utilizada en programas de mejoramiento animal con el fin de aumentar la frecuencia de los genes relacionados con resistencia a enfermedades.
El valor de He fue mayor que el de Ho en todas las razas, lo que indica un posible déficit de heterocigotos, lo que puede ser explicados por efectos de endogamia en la muestra evaluada. El promedio de He (0.88±0.05) aquí reportado para el ganado criollo fue mayor que para los criollos argentinos (19) y mexicano Chihuahua (17) (0.865 y 0.83 respectivamente) pero menor que el reportado para los criollos boliviano Saavedreño (18) y mexicano Tamaulipas (17) (0.91 y 0.92 respectivamente). Un valor similar de He se reporta en Holstein (7).
Los altos valores de FIS encontrados en algunas razas, pueden ser explicados por la procedencia de las muestras evaluadas, algunas razas sólo fueron muestreadas en una sola finca como es el caso de BON, CAS, CCC, RS y VEL de los cuales sólo el CAS presentó un valor bajo (0.073) y no fue significativamente diferente de cero, por el contrario, razas que fueron muestreadas en diferentes localidades presentaron bajo valor de FIS, como es el caso del HV (8 fincas) y H (6 fincas).
Los valores de FST encontrados al comparar los criollos indica poca diferenciación genética (FST <5%). De otro lado, al comparar los criollos con las razas foráneas el valor de FST indica una diferenciación genética moderada.
Esta investigación corrobora la gran diversidad genética del ganado criollo colombiano y afirma la necesidad de desarrollar planes estratégicos de conservación y utilización, ya que ellos pueden convertirse en reservorios de genes de interés.
Agradecimientos
A la Dirección Nacional de Investigación de la Universidad Nacional de Colombia-Palmira DIPAL, por la financiación (Proyecto 2030000).
REEFERENCIAS
1. Takeshima SN, Aida Y. Structure, function and disease susceptibility of the bovine major histocompatibility complex. Anim Sci J 2006; 77:138-150. [ Links ]
2. Lewin B. Genes IX, México, Mc Graw Hill; 2009. p. 570-604. [ Links ]
3. Russell GC, Smith JA, Oliver RA. Structure of the BoLA-DRB3 gene and promoter. Eur J Immunogenet 2004; 31:145-151. [ Links ]
4. Machado MA, Nascimento CS, Martinez ML, Silva MV, Campos AL, Teodoro RL, et al. Associação do loco BoLA-DRB3.2 com produção de leite em bovinos da raça Gir. Arq Bras Med Vet Zootec 2005; 5(3):380-389. [ Links ]
5. Do Nascimento CS, Machado MA, Martínez ML, Barbosa MVG, Martins MFG, Campos AL, et al. Association of the bovine major histocompatibility complex (BoLA) BoLA-DRB3 gene with fat and protein production and somatic cell score in Brazilian Gyr dairy cattle (Bos indicus). Genet Mol Biol 2006; 29(4):641-647. [ Links ]
6. Zambrano JC, Echeverri JZ, López AH. Asociación de los alelos del gen BoLA DRB3.2 con características productivas en vacas del hato Paysandú de la Universidad Nacional de Colombia. Rev Colom Cien Pecu 2009; 22(3):448-449. [ Links ]
7. Dietz AB, Detilleux JC, Freeman AE, Kelley DH, Stabel JR, Kehrli ME. Genetic association of bovine lymphocyte antigen DRB3 alleles with immunological traits of Holstein cattle. J Dairy Sci 1997; 80:400-4005. [ Links ]
8. Rupp R, Hernández A, Mallard BA. Association of bovine leukocyte antigen (BoLA) DRB3.2 with immune response, mastitis, and production and type traits in Canadian Holsteins. J Dairy Sci 2007; 90:1029-1038. [ Links ]
9. Martínez R, Toro R, Montoya F, Burbano M, Tobon J, Gallego J, et al. Caracterización del locus BoLA-DRB3 en ganado criollo colombiano y asociación con resistencia a enfermedades. Arch Zootec 2005; 54: 349-356. [ Links ]
10. Martínez ML, Machado MA, Nascimento CS, Silva MVGB, Teodoro RL, Furlong J, et al. Association of BoLA-DRB3.2 alleles with tick (Boophilus microplus) resistance in cattle. Genet Mol Res 2006; 5(3):513-524. [ Links ]
11. Castro GS, Trujillo EB, Duran CV. polimorfismos del gen BoLA-DRB3 en el bovino sintético colombiano Lucerna y asociación con conteo de células somáticas y mastitis. Rev Col Cienc Pec 2006; 19(3):270-279. [ Links ]
12. Juliarena MA, Poli M, Sala L, Ceriani C, Gutierrez S, Dolcini G, et al. Association of BLV infection profiles with alleles of the BoLA-DRB3.2 gen. Anim Genet 2008; 39:432-438. [ Links ]
13. Panei CJ, Suzuki K, Echeverria MG, Serena MS, Metz GE, Gonzales ET. Association of BoLA-DRB3.2 alleles with resistance and susceptibility to persistent lymphocytosis in BLV infected Cattle Argentina. Int Dairy J 2009; 4(3):123-128. [ Links ]
14. Van Eijk MJ, Russ I, Lewin HA. Order of bovine DRB3, DYA, and PRL determined by sperm typing. Mamm Genome 1992; 4:113-118. [ Links ]
15. Nomenclature. International Society for Animal Genetics. [en línea] [Fecha de acceso 20 de Julio de 2010]. URL disponible en http://www.projects.roslin.ac.uk/bola/bolahome.html. [ Links ]
16. Excoffier L, Lischer HEL. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Resour 2010; 10:564-567. [ Links ]
17. Fernández IG, Ríos JGR, Gayosso AV, Ulloa RA, Morales RAA. Polymorphism of locus DRB3.2 in populations of creole cattle from Northern Mexico. Genet Mol Biol 2008; 31(4):880-886. [ Links ]
18. Ripoli MV, Liron JP, De Luca JC, Rojas F, Dulout FN, Giovambattista G. Gene frequency distribution of the BoLA-DRB3 locus in Saavedreño creole dairy cattle. Biochem Genet 2004; 42:231-240. [ Links ]
19. Giovambattista G, Golijow CD, Dulout FN, Lojo MM. Gene frequencies of DRB3.2 locus of Argentine Creole cattle. Anim Genet 1996; 27:55-56. [ Links ]
20. Kelly L, Nicolini M, D'Angelo A, Nimo G, Rincon J, Piaggio J, et al. Polimorfismos del gen DBR3.2 en bovinos criollos del Uruguay. Arch Zootec 2003; 52:77-80. [ Links ]
21. Zambrano JC, Echeverri JZ, López AH. Análisis y frecuencias de los alelos del antígeno leucocitario bovino BoLA DRB3.2 en vacas del hato Paysandú de la Universidad Nacional de Colombia. Rev Colom Cien Pecu 2009; 22(3):448-449. [ Links ]