INTRODUCCIÓN
El mango (Mangifera indica L.) es una reconocida fruta tropical de origen en el Noreste de la India, la región Indo-Birmánica y las montañas de Bangladesh (Mukherjee, 1957). El mango se destaca por su gran diversidad, pulpa carnosa, particular sabor y aroma, amplia aceptación y creciente demanda en los mercados internacionales (Maldonado et al. 2016).
En la actualidad, el perfeccionamiento de herramientas genéticas para el mango son escazas, por lo que muy poco han contribuido al mejoramiento genético de estos frutos, a nivel mundial. La diversidad genética es esencial para la producción agrícola sostenible, puesto que reduce la vulnerabilidad ante plagas y enfermedades (Zhou et al. 2002; Lobo, 2006); sin embargo, el fitomejoramiento depende de la riqueza genética del material utilizado para este fin (Cooper et al. 2001).
Las técnicas moleculares son fundamentales para la identificación y la diferenciación de plantas, integrando taxonomía, genética y evolución (Riley et al. 2023); no obstante, la extracción de ADN a partir de tejidos vegetales presenta desafíos, ya que muchas plantas contienen altos niveles de metabolitos secundarios (Avalos & Pérez-Urria, 2009; Lawal & Agbator, 2021), que pueden interferir con la PCR. La eliminación de estos metabolitos, a menudo, requiere una purificación adicional del ADN, mediante el uso de disolventes orgánicos. Los métodos moleculares empleados en estudios de diversidad genética de especies dependen de la obtención de ADN íntegro, puro y de la más alta calidad (Roudsary et al. 2022).
Para poder desarrollar eficientemente las herramientas moleculares es fundamental contar con protocolos de obtención de ADN, de buena concentración, alta pureza, baratos, sencillos y que no produzcan desechos tóxicos (Pommer & Murakami, 2009; Healey et al. 2014; Sánchez et al. 2021).
Para el aislamiento y la purificación de ADN de buena calidad, se utilizan métodos basados en la técnica de bromuro de cetiltrimetilamonio (CTAB), debido a su bajo costo (Pardo-Pérez et al. 2021; Asha et al. 2023). Por otro lado, existen kits comerciales, que permiten una extracción rápida de ADN; aunque son reproducibles y proporcionan material de alta pureza, una limitación de estos kits es su alto costo y la baja cantidad de ADN recuperado (Ríos et al. 2016).
Las hojas de mango tienen presencia de fenoles (Carrillo-Tomalá et al. 2020), los cuales, son compuestos químicos inhibidores (Sánchez et al. 2021), que se unen y precipitan el ADN (Azmat et al. 2012;Pardo-Pérez et al. 2021). Para el caso de la extracción de ADN en hojas tiernas de mango, Saldaña & Salazar (2007) reportan que, de los cuatro métodos usados, el descrito por Doyle & Doyle (1990), como el mejor en obtención de cantidad de ADN intacto.
Dada la dificultad para el procesamiento de las muestras en fresco y teniendo en cuenta la ausencia de un protocolo de extracción de ADN en tejido seco de mango, se hace necesario implementar un método para aislar ADN en tejido seco, pues la mayoría de estas técnicas están estandarizadas para extraer ADN en tejido fresco (Kumari et al. 2018; Kumar et al. 2020). Por tal razón, el presente estudio tuvo como objetivo el análisis comparativo de dos protocolos de extracción de ADN en hojas secas de mango (Mangifera indica L.), para uso en técnicas moleculares.
MATERIALES Y MÉTODOS
Obtención de material biológico: Se colectaron hojas en fase madura de 15 árboles de mango, ubicados en la Vereda Km 12 de Montería (N 8°40'41.7", O 75°48'07.0"), las cuales, se colocaron en una bolsa resellable, que contenía gel de sílice y se almacenaron a temperatura ambiente.
Extracción de ADN: Se examinaron dos métodos de extracción de ADN: el kit comercial de extracción Wizard Genomic de Promega® (USA, Promega A1120), que se ejecutó atendiendo las indicaciones del fabricante y el método de Doyle & Doyle (1990), con las siguientes modificaciones: se reemplazó alcohol isoamilico 24:1, en vez de octanol, se centrifugó a 13.500rpm y se precipitó el ADN con una solución de etanol absoluto en reemplazo del isopropanol.
Concentración y pureza del ADN: La concentración (ng/µL) y la pureza del ADN obtenido se evaluaron espectrofotométricamente a 260 y 280 nm y la relación A260/A280, se utilizó para evaluar la contaminación con proteínas, empleando la espectrofotometría Colibri Microvolume Spectrometer (TitertekBerthold, Berthold Detection Systems GmbH, Bleichstrasse, Pforzheim, Germany), descrita por Brondmann (2008). Para verificar la integridad del ADN, se corrieron 5μL de ADN en una cámara de electroforesis (Clase II Owl separation Systems, Inc. Portsmount, USA modelo B1A), en un gel de agarosa al 1,5 de (p/v), con TBE 1X como buffer y a un voltaje constante de 120 V, durante 90 min. Se hizo el teñido del ADN con Gelred Nucleic Acid 10000X water Merk y se visualizaron las bandas en un transiluminador DyNA Light™ UV (Labnet International Inc.).
Amplificación del ADN: Se hizo en un termociclador Bioard T100 #1861096 (Los Ángeles, USA), de los marcadores MiIIHR12a, MiIIHR23a y MiIIHR34b (Ravishankar et al. 2011). El protocolo PCR se efectuó en un volumen final de 20μL, así: de 10μL de 2x DreamTaq HotStart PCR Master Mix (Thermo Scientific Lithuania); 2μL de cada primer; 7μL de agua estéril (Water Nuclease-free (Thermo Scientific Lithuania) y 1μL de ADN (100ng/µL). El programa empleado es 3 min a 95 °C; 30 s a 60 °C; 30 s a 56 °C; 2 min a 72 °C, repitiendo 23 ciclos del paso 2 al 4 y un paso final de extensión de 10 min a 72 °C.
Análisis estadístico: Los resultados fueron procesados con ayuda del paquete estadístico SPSS para Windows, versión 22.0 (SPSS Inc., 2018). En cada uno de los dos métodos de extracción utilizados, se determinaron medias y desviaciones estándar (s). En la determinación del método que muestra diferencias significativas en la concentración y calidad de ADN, se utilizaron las pruebas de Tukey y Dunnet (Miller, 1996). Para la estimación de la proporción de éxito de las amplificaciones de los marcadores utilizados, se determinó mediante la prueba de Chi-cuadrado (Halos et al. 2004).
RESULTADOS Y DISCUSIÓN
Al comparar las metodologías utilizadas para la extracción de ADN a partir de hojas secas de mango (Mangifera indica L.), la que mejor resultados evidenció, en términos de concentración y de pureza, fue Doyle & Doyle (1990) con modificaciones, en donde la relación entre la cantidad de absorción resultante en 260 y 280 nm fue 1,9, lo que indica que la mayor parte de la absorción la realizan los ácidos nucleicos y, por tanto, el ADN extraído está bien calificado y su pureza es adecuada, mientras la relación para el kit Promega fue de 1,42, valor sensiblemente inferior, lo cual, puede indicar la presencia de proteínas, fenol u otros contaminantes, que se absorben fuertemente a 280 nm o cerca de ellos (Russo et al. 2022).
Además, el ADN extraído con el Kit presentó una coloración turbia durante el proceso de incubación, lo que muestra que para los ácidos nucleicos la turbidez revela presencia de compuestos aromáticos (fenoles) y proteínas (Sahu et al. 2012; Sánchez et al. 2021), los cuales, en sus formas oxidadas, se unen al ADN covalentemente, lo que resulta en un ADN inadecuado para la mayoría de las aplicaciones moleculares (Pommer & Murakami, 2009 y Sánchez et al. 2021), mientras que para el protocolo Doyle y Doyle modificado, resultó un ADN traslúcido, que se ajusta a lo reportado por Sánchez et al. (2021), donde obtuvieron, por el método Doyle y Doyle, para hojas liofilizadas de guayaba (Psidium guajava L.), un ADN incoloro. Asimismo, el ADN traslúcido revela una extracción limpia y de calidad, evidenciado en la relación de absorbancia A260/A280.
Los resultados mostraron para el rendimiento de ADN y la pureza del ADN utilizando un espectrofotómetro UV, un mayor rendimiento de ADN con el método de Doyle y Doyle modificado (289,7±42,0 ng/μL), mientras que el menor se obtuvo con el método Kit Promega (95,9±31,8 ng/μL) (Tabla 1).
En la prueba de comparación entre medias de Tukey, el método Doyle y Doyle modificado fue el que obtuvo la mayor concentración de ADN, lo que indica que, en este caso, sí fue posible obtener buena cantidad de ADN. Al comparar los resultados mediante la prueba de Dunett, se encontraron diferencias significativas y mayores concentraciones de ADN para el método Doyle y Doyle modificado (Tabla 1)
Tabla 1 Comparación de medias para la eficiencia de dos métodos diferentes de extracción de ADN, mediante la prueba de rangos múltiples de Duncan (P ≤ 0,01), en muestras de hojas secas de Manguifera indica L.

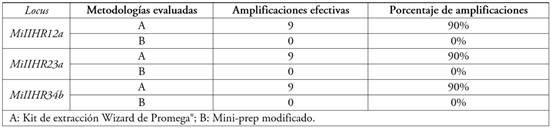
En la tabla 2, se observa que el mayor porcentaje de efectividad en la amplificación se obtuvo a partir del ADN extraído por el método de Doyle y Doyle modificado, siendo del 90 %, para cada uno de los tres marcadores. Este resultado se podría relacionar con la calidad del ADN que se obtuvo mediante el protocolo, debido a que las muestras extraídas con el método Doyle & Doyle modificado evidenciaron un valor promedio de 1,9, lo cual, se ajusta a lo reportado por Russo et al. (2022) y Pérez et al. (2011), quienes argumentaron que muestras de ADN con valores entre 1,8 y 2,0 son más estables en cuanto a calidad y pureza, por ser poco susceptibles a contaminantes, siendo este ADN óptimo para amplificar con la mayoría de marcadores. También, se puede observar que el porcentaje efectivo de amplificaciones para ADN extraído por el método de Kit de Promega evidenció 0 % en los tres marcadores, lo cual, se pudo deber a la contaminación por metabolitos secundarios, resultado que se ajusta a lo indicado por Pardo-Pérez et al. (2021) y Ferreira & Grattapaglia et al. (1998), quienes expresaron que la presencia de estos metabolitos en ácidos nucleicos inhibe la acción de la Taq polimerasa.