INTRODUCCIÓN
El uso de tecnologías de información se ha convertido en elemento fundamental en la investigación de la biodiversidad, especialmente, en el escenario actual, dada la necesidad de consolidar y estructurar los datos provenientes de una amplia gama de fuentes, tanto espaciales como temporales, de las características de dicha biodiversidad (Shin & Choi, 2015; Soltis et al. 2016; Chen & Hu, 2021; Alberti & Massone, 2022). La información que se produce de estos datos posibilita la comprensión del comportamiento de las especies, desde sus patrones evolutivos, ecológicos, hasta su respuesta al cambio climático (Davenport & Prusak, 1998); sin embargo, el almacenamiento creciente de registros de biodiversidad genera volúmenes masivos de información, lo que limita su manipulación eficaz con las herramientas actuales, requiriendo el uso de técnicas analíticas más eficientes, para el modelamiento y explotación de la información (Hampton et al. 2013).
Actualmente, existen referentes de infraestructuras de datos globales para el estudio de la biodiversidad y el monitoreo de carbono, que propenden la accesibilidad, el uso, la distribución y el procesamiento de los datos, tales como GBIF (Global Biodiversity Infomation Facility), ForestPlot, NEON (National Ecological Observatory Network) e ICOS (Integrated Carbon Observatory System) (Sierra et al. 2017; ForestPlots.NET, 2020; GBIF, 2020). Estas infraestructuras, además sirven como herramienta de apoyo en la toma de decisiones y planteamiento de políticas de desarrollo sostenible (GBIF, 2022), pues permiten ajustar modelos para la comprensión y la generalización de las causas, patrones, mecanismos y consecuencias de los fenómenos naturales, favoreciendo las evaluaciones implementadas por el Grupo de Observaciones de la Tierra GEO (GEO, 2015; Cooper & Noonan-Mooney, 2013) y, subsecuentemente, los objetivos del protocolo de Kyoto (ONU, 1998), la Organización de las Naciones Unidas ONU (2018), el Grupo Intergubernamental de Expertos sobre el Cambio Climático IPCC (2019) y la Organización de las Naciones Unidas para la Alimentación y la Agricultura FAO (2022). Colombia cuenta con el Sistema de Información de Biodiversidad (SiB), el cual, es parte del Sistema de Información Ambiental de Colombia (SIAC) y es, en sí mismo, un nodo de información articulado al GBIF (Muñoz et al. 2007; SiB, 2017).
Estas infraestructuras de datos, por sí mismas, no logran una integración automática y escalable entre los datos y el desarrollo de nuevo conocimiento en ambientes de trabajos científicos; por lo general, requieren replicación de datos, procedimientos de exportación, importación, tratamiento y análisis adicionales, generando entornos que consumen tiempo y son vulnerables a errores (Noreña-P. et al. 2018). Como resultado, el procesamiento de datos se debe repetir en cada tarea al momento de ser abordada, lo que resulta en un mayor gasto de energía (Grattarola et al. 2019). Así, pues, se deben aunar esfuerzos en prácticas científicas más competentes, donde los datos y los procesos se gestionan con mayor eficiencia, fiabilidad y reproducibilidad. Esto implica flexibilidad a la hora de integrar herramientas de almacenamiento, análisis y visualización de datos, entendidos aquí como el conjunto de enfoques metodológicos, algoritmos y herramientas de software (Hu & Che, 2019; Andjarwirawan et al. 2020).
En el caso particular de los grupos de investigación académica, permanentemente, se están generando conjuntos de datos que se suelen almacenar en diferentes temas, de manera que se distribuyen en innumerables documentos, hojas de cálculo y archivos, lo que conlleva a que los datos resultantes logren escasamente ser sintetizados en el corto plazo, incluso, si existe un individuo exclusivamente en esta labor (Devictor & Bensaude-Vincent 2016; Senterre & Wagner, 2016). En este contexto, es necesario crear un espacio que conecte diferentes infraestructuras de datos con tecnologías en la nube, como lo son Amazon Web Services (AWS), Google Cloud, Oracle entre otros. Esto permitiría mejorar la gestión y el procesamiento de datos de biodiversidad y promover la colaboración entre grupos de investigación.
Es aquí, donde el concepto de ambiente virtual hace referencia a un sistema que implementa, administra y controla múltiples instancias virtuales, permitiendo el intercambio de datos, a largo plazo, para usos más allá de su propósito inicial (Bart et al. 2018; Pimentel et al. 2019). En este sentido, posee características para explotar al máximo el potencial de las tecnologías encargadas del almacenamiento, análisis y modelización de la información tanto en el componente de software como de hardware (Bart et al. 2018).
En esta investigación se desarrolló un laboratorio virtual para el análisis, la gestión y la sistematización de los datos y procesos, teniendo por objeto desarrollar una arquitectura de referencia, que mejore la interactividad, la colaboración, la reproducibilidad, la latencia, el rendimiento y la persistencia en el manejo de datos de biodiversidad y sirviendo como núcleo de recolección y estandarización de la información, generada por el proyecto “Distribución de la diversidad genética de especies maderables amenazadas como base del manejo forestal sostenible en los bosques húmedos del pacífico colombiano”. Para lograr estos objetivos, se implementó una base de datos relacional, que permite la integración y la gestión eficiente de grandes volúmenes de datos de biodiversidad. Además, se desarrollaron entornos de trabajo colaborativos para facilitar la interacción y la cooperación entre investigadores en tiempo real. También, se establecieron procedimientos para la depuración y la estandarización de datos, asegurando la calidad y la consistencia de la información recolectada. Finalmente, se evaluó el rendimiento del laboratorio virtual.
Este trabajo destaca un enfoque integral en la gestión de datos ecológicos, combinando la integración de datos de diferentes fuentes, como inventarios forestales, estudios ecológicos y la experiencia práctica de los autores en la implementación de soluciones de laboratorio virtual. Esta aproximación permite la creación de un laboratorio virtual, que aplica técnicas de análisis y visualización de datos para la toma de decisiones en la gestión de la biodiversidad. La implementación de soluciones en la nube junto a herramientas de software libre permite una mayor escalabilidad y acceso a los datos y herramientas de análisis desde cualquier lugar, lo que facilita la colaboración y la toma de decisiones en tiempo real, permitiendo una mayor flexibilidad y portabilidad en la implementación de estas tecnologías en el campo forestal.
MATERIALES Y MÉTODOS
Área de estudio. Para este estudio se visitaron 13 localidades ubicadas en tres departamentos del pacífico colombiano, desde junio a diciembre del 2021, cuya cobertura geográfica se puede observar en la figura 1.
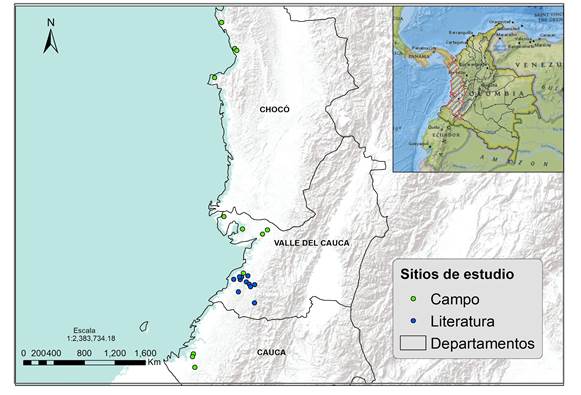
Figura 1 Mapa de sitios de estudio del proyecto principal, con información tomada en campo y literatura.
Se recolectaron datos dasométricos y geográficos en el campo, utilizando las metodologías descritas por Melo & Vargas (2003) y Chapman & Wieczorek (2022), respectivamente. Las muestras botánicas se clasificaron siguiendo la metodología de Gentry (1996) y se utilizaron bolsas Ziploc y gel de sílice para la preservación de las muestras genéticas, según las recomendaciones de Bocanegra-González & Guillemin (2018). Los datos se digitalizaron en hojas de cálculo en formato .xlsx, para minimizar los errores humanos y se generó un archivo geográfico, mediante el uso del GPS en formato .gpx, para cada archivo tabular.
Desarrollo de la base de datos. Se utilizó Python y Google Colaboratory junto con los módulos pandas y pygpx, para manejar, transformar y concatenar archivos .xlsx y .gpx. Se fusionaron, usando la llave relacional en un DataFrame y se exportaron a un archivo consolidado en Excel, para la inserción de nuevos datos. Los datos se integraron en una base de datos de modelo relacional y se desarrollaron varios procedimientos, para asegurar que los valores fueran comparables entre todas las fuentes de información. Se generó un código de identificación único para los proyectos recopilados y se estandarizaron los sistemas de georreferenciación (Van Rossum, 1995; Google, 2023; McKinney, 2010; Leslie, 2022; Svob et al. 2014; Nakamura et al. 2021; Python Software Foundation, 2022; Coordinate Systems Worldwide, 2022).
La información taxonómica fue actualizada y estandarizada utilizando la API del GBIF (Chamberlain et al. 2022). La base de datos fue diseñada para gestionar dos tipos de proyectos: inventarios forestales y estudios ecológicos, implementando técnicas de control de calidad y transformación de datos en cinco formatos de archivo (.xlsx, .gpx, .docx, .shp y .pdf). La base de datos relacional fue implementada utilizando PostgreSQL (PostgreSQL Global Development Group, 2022) y SQLAlchemy (Bayer, 2013), como se muestra en la figura 2.
La estructura final de la base de datos consta de 13 tablas relacionales divididas en dos grupos, siguiendo el modelo estrella, descrito por Svob et al. (2014), Giménez (2019) y Alberti & Massone (2022). El primer grupo está compuesto por las tablas que almacenan información general del proyecto y la localidad de muestreo, como projects, places e inventory_details. El segundo grupo de tablas se enfoca en almacenar información taxonómica y dasométrica, a nivel de individuo, así como información sobre experimentos ecológicos, genéticos y de propagación, incluyendo las tablas biodiversity_records, measurements, taxonomy_details, observations_details, collections, experiments, experiment_types y experiment_records. El diseño de las tablas se basó en llaves primarias y foráneas, para establecer relaciones entre las tablas, incluyendo las pautas de Chapman & Wieczorek (2022), para la correcta georreferenciación y Bayer (2013), para la gestión de la base de datos.
Arquitectura de referencia para el laboratorio virtual. En este estudio, se utilizó la plataforma de código abierto Docker versión 19.03.8, sobre la distribución Ubuntu 22.10, usando el kernel Linux 5.19.0-29-generic, para la implementación del laboratorio virtual, mientras que los entornos de trabajo para los investigadores se generaron mediante el uso del software JupyterHub (Hu & Che, 2019; Andjarwirawan et al. 2020, Jupyter project, 2022a).
Por su parte, los Jupyter Notebooks son entornos de desarrollo interactivo basado en la web para el manejo de código y datos. La interfaz flexible de los notebooks permitió a los usuarios configurar y organizar sus flujos de trabajo de ciencia de datos, estadística y computación científica, tanto en Python como en R (R Development Core Team, 1993; Jupyter project, 2022b), permitiendo un flujo de trabajo estructurado y reproducible en cada uno de los procesos y análisis realizados a lo largo del proyecto, siguiendo las recomendaciones de Carneiro et al. (2018), Pimentel et al. (2019) y Beg et al. (2021).
Adicionalmente, se implementaron los servicios de Amazon Web Services (AWS), los cuales, brindan una visión unificada en torno a la captura, ingesta, almacenamiento y análisis de datos desarrollados en el laboratorio virtual (Coker et al. 2019). Durante la implementación del laboratorio virtual en AWS se utilizó Internet Gateway y Route 53, para permitir la conexión con el navegador web. Se estableció una Virtual Private Cloud (VPC) y se implementaron dos instancias Elastic Compute Cloud (EC2), para responder a las peticiones realizadas por el intérprete de órdenes seguro y el software TablePlus (Raccoon & Pham, 2022). También, se virtualizaron los contenedores mediante Elastic Container Service (ECS) y Fargate, y se usó Relational Database Service (RDS), para la base de datos PostgreSQL. Para el almacenamiento y distribución de los Notebooks se usó Elastic File System (EFS) y Simple Storage Service (S3), como repositorio para imágenes, documentos y resultados. La infraestructura fue monitoreada por CloudWatch, Config y se estableció el registro de acceso y privacidad con Identity and Access Management (IAM) y Congnito. El laboratorio virtual ejecutó los servicios y aplicaciones, tal como se muestra en la figura 3.
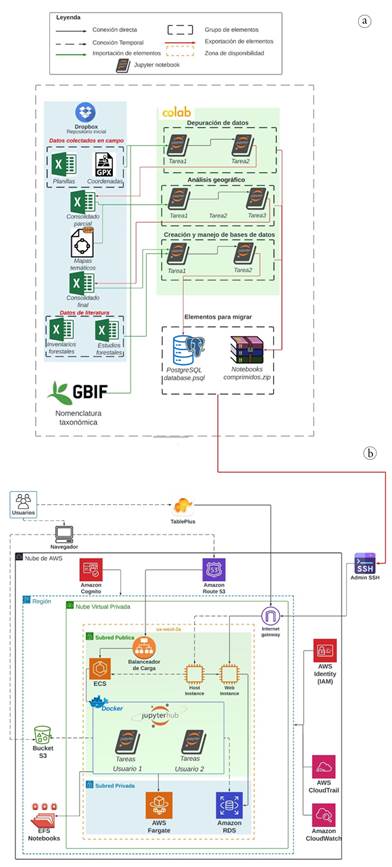
Figura 3 Infraestructura y procesos para el desarrollo del laboratorio virtual en la nube. a) procesos realizados fuera del laboratorio; b) procesos y servicios que forman el laboratorio virtual.
Se evaluó el rendimiento de los notebooks en el servidor JupyterHub después de implementar la infraestructura del laboratorio virtual. Los temas abordados incluyen la depuración de datos, la creación de una base de datos en PostgreSQL a partir de archivos tabulares y análisis geográfico, cada uno con dos notebooks en Python y el tema de diseño experimental con un solo notebook en R. Para facilitar la reproducibilidad y la colaboración se ha creado un repositorio en GitHub de libre acceso, que contiene todos los notebooks y recursos utilizados en este estudio https://github.com/juanpac96/virtual_laboratory_of_biodiversity.
RESULTADOS Y DISCUSIÓN
Análisis de la base de datos. Se logró poner en funcionamiento el laboratorio virtual para cinco usuarios, mediante la integración de una base de datos final en PosgreSQL, el motor de bases de datos TablePlus, la aplicación de JupyterHub virtualizada en un contenedor de Docker y los servicios de cómputo en la nube ofrecidos por AWS. Mientras tanto, la base de datos final contiene un total de 28.058 registros, compuestos por seis proyectos clasificados en dos inventarios forestales y cuatro estudios de investigación ecológica, realizados entre 2004 y 2022. El número de observaciones registradas por proyecto varía entre 130 a 18.575, siendo el proyecto cuatro el que posee mayor cantidad de registros, mientras que el proyecto seis posee la menor cantidad de observaciones. Un breve análisis de los registros colectados en los años en los que se realizaron los seis proyectos revela una alta concentración durante 2004 y 2008.
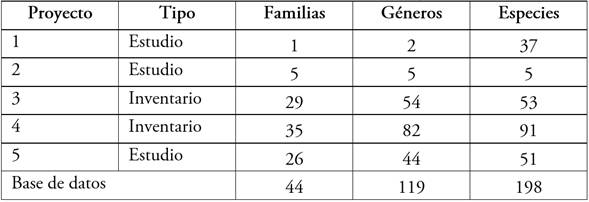
La base de datos cuenta con un total de 28.058 entradas, de las cuales, 26.117 corresponden a los inventarios forestales, mayormente documentando especímenes arbóreos, mientras que 1.941 entradas provienen de los estudios ecológicos. Se identificaron 128 especies pertenecientes a 104 géneros y 40 familias en los inventarios, mientras que en los estudios ecológicos se identificaron 91 especies, 49 géneros y 28 familias. En total, 21 especies fueron registradas, tanto en los inventarios como en los estudios ecológicos, mientras que 70 especies solo fueron registradas en los inventarios y 107 únicamente en los estudios ecológicos. Entre las familias con mayor número de especies registradas se encuentran Fabaceae, con 54 especies; Arecaceae, con 13 especies; Malvaceae, con 11 especies y Moraceae, con 10 especies. La base de datos contiene un total de 44 familias, 119 géneros y 198 especies registradas en cuanto a nomenclatura taxonómica. La descripción de los registros taxonómicos, a nivel de proyecto y base de datos, se puede visualizar en la tabla 1.
Tabla 1 Resumen de la información taxonómica forestal presente en la base de datos de tres departamentos del Pacífico colombiano.
Control de calidad y validación de los datos taxonómicos. En cuanto a la identificación taxonómica se puede apreciar un incremento en la identificación de los individuos cuando se transita del nivel de especie al de género, ya que se observó un aumento del 2,56 % en la cantidad de registros con géneros identificados, que abarcan un 63,25 % del total de registros, mientras que los individuos que alcanzaron a llegar al nivel de especies, representan el 60,69 % del total de registros en la base de datos, revelándose, también, que el mayor grado de identificación taxonómica fue realizado por los estudios ecológicos.
Para garantizar el control y la calidad de la base de datos, se implementaron medidas de restricción y validación en los datos ingresados en cada campo y fila de las tablas correspondientes. Dichas restricciones de fila incluyen: restricciones de valores únicos, condición del tipo de datos almacenados, controles de comparación de campos, codificación de valores de campo y encriptación de valores sensibles.
Con la ayuda de estos parámetros se reveló que la tabla con mayores presencias de errores fue la biodiversity_records, que detectó anomalías en las columnas de latitud, longitud, elevación, nombres comunes y fechas, debido a la presencia de caracteres especiales dentro del respectivo campo y la presencia de valores duplicados en la columna de los códigos de registros.
En menor medida, se detectaron errores en la columna de encargada del almacenamiento de los valores de las variables dasométricas de la tabla measurements, concentrándose en mayor media en las variables de altura total, altura comercial, diámetro a la altura del pecho (DAP) y se caracterizaban por el uso de caracteres especiales y errores de toma de valores en campo. Para el resto de las once tablas no se detectó ninguna violación a las restricciones implementadas. La información general de las tablas que forman la base de datos de este estudio se puede observar en la tabla 2.
De manera global, las restricciones y las condiciones en la tabla de tipo de datos, valores nulos y duplicación de datos ayudaron a detectar errores de digitación en planillas de campo e información suministradas por los inventarios forestales y estudios ecológicos. Adicionalmente, con el uso del software TablePlus y en paralelo con Python, se ejecutan consultas de lenguaje SQL en la búsqueda de incoherencias en las variables dasométricas, taxonómicas y espaciales, gastando un promedio de 0,641 segundos por consulta.
Para ilustrar la utilidad de la base de datos se realizó un análisis simple de la ocurrencia de especies, consultando la base de datos, donde se encontró que las tres especies de mayor ocurrencia registrados por los estudios ecológicos son: Carapa guianensis, con 17,48 %; Humiriastrum procerum, con 11,91 % y Magnolia calimaensis, con el 4,42 %, mientras que en los inventarios forestales, las tres especies más abundantes son: Euterpe cuatrecasana, con 15,69 %; Rhizophora harrisonii, con 2,66 % e Inga edulis, con el 2,46 %, de los 26.117 registros suministrados por los inventarios forestales. Por su parte, las variables dasométricas presentaron un comportamiento diferente en la distribución de las observaciones en la variables, puesto que no todas las variables contempladas en la base de datos están presentes en todos los proyectos, tal es el caso de la variable diámetro de copa, que presentan un bajo número de 431 observaciones, pues solo fueron contempladas por los estudios 1 y 2, mientras que la variable DAP se distribuye entre las magnitudes de 5 a 80 cm, en donde se puede observar que las tres variables poseen histogramas sesgados positivamente. En el mismo sentido, la distribución de las observaciones para la altura total y altura comercial se da entre el orden de los 0,5 a los 40 m, con tendencia a una distribución normal.
Evaluación de las capacidades del ambiente de desarrollo. Una vez realizada y depurada la base de datos se procedió a desplegar el servidor de JupyterHub del laboratorio virtual en fase de prueba, con un personal de cinco investigadores y un contenedor en Docker, mediante el uso de los servicios de orquestación de contenedores ECS y AWS Fargate. Con el servidor de JupyterHub en funcionamiento se procedió a ejecutar todos los notebooks migrados de Google Colaboratory dentro del entorno de JupyterHub, para realizar una comparativa entre el tiempo de ejecución y el porcentaje de memoria RAM usada por cada plataforma, tal como se ve en la tabla 3. El promedio del tiempo de los notebooks ejecutados en Google Colaboratory es de 06min:02seg, con una desviación estándar de ± 03min:58seg, mientras que en el servidor de JupyterHub se obtuvo un tiempo de ejecución promedio de 04min:18seg, con una desviación estándar de ± 03min:48seg, observándose un mejor rendimiento en los notebooks ejecutados en JupyterHub, debido a que las librerías usadas para el análisis geográfico y conexión a la infraestructura del GBIF de cada ambiente de trabajo vienen instaladas, por defecto, desde la configuración del servidor y no es tarea del usuario.
Tabla 3 Comparación del comportamiento de los notebooks entre las dos plataformas de desarrollo.
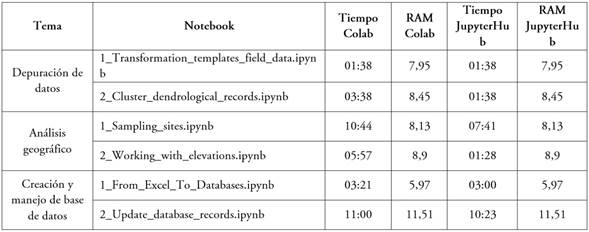
Nota: Tabla resumen entre las plataformas Google Colaboratory y JupyterHub de los tiempos en minutos y segundos junto con el porcentaje (%) de uso en memoria RAM.
Por el contrario, en la plataforma de Google Colaboratory se debe instalar dichas librerías, cada vez que se requiera ejecutar un notebook para realizar cualquier tarea relacionadas con estas librerías y deben ser instaladas por el usuario, lo que generó un aumento en el tiempo de ejecución considerablemente.
Para el componente de memoria RAM no se registró un cambio significativo, ya que el porcentaje de uso promedio de dicho componente fue de 8,48 %, de un total 12 GB disponibles en cada plataforma, al momento de la evaluación de cada notebook, lo que indicó que los códigos usados en cada tarea no son muy demandantes de recursos computacionales. El objetivo del Notebook relacionado con el tema del diseño experimental, ejecutado con el lenguaje de programación R, fue verificar su compatibilidad con el software JupyterHub. Los resultados obtenidos fueron tiempos de ejecución de 1 minuto y 10 segundos, con un consumo de RAM del 8,2 %, lo que indica que este software puede ser útil para futuros proyectos que requieran el uso simultáneo de más de un lenguaje de programación.
Análisis y discusión del rendimiento en el laboratorio virtual. A continuación, se llevó a cabo un análisis del rendimiento del laboratorio virtual, evaluado a través de dos métricas clave: tiempo de respuesta y uso de memoria RAM. Se compararon dos plataformas de desarrollo, Google Colaboratory y JupyterHub y se encontró que el tiempo de respuesta fue similar en ambas, pero JupyterHub requiere un poco más de memoria RAM. Los tiempos de ejecución de los notebooks variaron entre 1:30 minutos y 11:00 minutos, lo que proporciona un amplio margen de tiempo para llevar a cabo tareas y análisis, permitiendo una mayor flexibilidad y escalabilidad. Los resultados indican que la interactividad y la reproducibilidad son propiedades importantes del laboratorio virtual. Con relación a los servicios EC2 y RDS se registraron métricas, como tiempo de respuesta de 200 ms, uso de CPU del 30 % y RAM 50 %, junto a un tráfico de red alrededor de 0,1 GB/s. En el caso de ECS, el tiempo de inicio y detención del contenedor fue de 10 y 5 segundos, respectivamente. En AWS Fargate, se observó un uso promedio de CPU y RAM del 80 % y 75 %, respectivamente. Finalmente, en el servicio S3 se almacenaron 4 GB de objetos, con tiempos de respuesta inferiores a 100 ms.
El laboratorio virtual desarrollado en este estudio permitió la recopilación y el análisis de grandes conjuntos de datos sobre biodiversidad, apoyando la idea de que la combinación de datos a diferentes escalas permite un seguimiento más completo en el espacio y en el tiempo, identificando patrones y tendencias en la distribución de especies (Hernandez et al. 2022). Estudios recientes, como los realizados por Pöttker et al. (2023), han demostrado la eficacia de utilizar la librería Keras de TensorFlow con Python para entrenar redes neuronales convolucionales (CNNs) en la clasificación de comunidades vegetales, a partir de imágenes multiespectrales. De manera similar, nuestro estudio también utilizó Python, pero se enfocó en procesar y analizar datos de una base de datos en PostgreSQL con librerías, como Pandas y Sqlalchemy; mientras que Pöttker et al. (2023) aplicaron Python para el análisis espacial avanzado y la identificación de patrones fenológicos, nuestro enfoque se centró en la gestión y análisis de grandes conjuntos de datos taxonómicos, provenientes de exploraciones de campo. Ambos enfoques resaltan la versatilidad y potencia de Python en el análisis de datos ecológicos.
En este estudio se destaca la eficacia de las plataformas de computación en la nube para el procesamiento de grandes volúmenes de datos ecológicos. Kovács et al. (2023) utilizaron Google Earth Engine (GEE) para generar mapas globales de características de vegetación, logrando tiempos de reconstrucción temporal de 20-30 segundos. Con el empleo de AWS y JupyterHub se logró un tiempo promedio de ejecución de notebooks de 04:18 minutos, en comparación con 06:02 minutos en Google Colaboratory. La diferencia en los tiempos de procesamiento se debe a que GEE está optimizado para el análisis y la visualización de datos geoespaciales, permitiendo una integración eficiente con grandes conjuntos de datos satelitales; en cambio, Google Colaboratory es una plataforma general de notebooks basada en la nube, que requiere la instalación manual de bibliotecas para cada ejecución, lo que aumenta el tiempo de procesamiento. Aunque el enfoque no utilizó GEE, la combinación de JupyterHub y AWS proporcionó un entorno personalizado y optimizado para la gestión de datos específicos, destacando la flexibilidad y la eficiencia en la ejecución de análisis complejos y la gestión de grandes volúmenes de datos.
Asimismo, se destaca la importancia del monitoreo de bosques tropicales utilizando tecnologías avanzadas y análisis de datos. Tanto en este estudio como el de Roberts et al. (2022), se empleó Python para el procesamiento y análisis, integrando múltiples fuentes de información, lo que permite una respuesta rápida ante eventos de deforestación y degradación de bosques, facilitando la conservación en áreas críticas de Colombia.
En general, los estudios en la literatura enfatizan la importancia de adoptar un enfoque holístico para estudiar la biodiversidad, uno que incorpore datos de múltiples fuentes y disciplinas para proporcionar una comprensión más completa del mundo natural. Aprovechando tecnologías avanzadas, como el aprendizaje automático, la teledetección y el análisis de datos, los investigadores pueden desarrollar estrategias efectivas para conservar la biodiversidad y mitigar los impactos del cambio climático (Agrillo et al. 2021; Li et al. 2021; Musvuugwa et al. 2021). A pesar de los desafíos y debilidades, la computación en la nube y la ciencia de datos se están convirtiendo en herramientas comunes para el desarrollo de nuevo conocimiento (Borowiec et al. 2022).
Una de las limitaciones de este estudio es que no se probaron todos los servicios presentes en la AWS, ni se usaron otros proveedores de servicios en la nube, como Google, Oracle o Azure de Microsoft, para poder comparar cuál es el más eficiente o preciso. Además, la arquitectura aquí presente no representa una solución definitiva y cualquier otro investigador puede añadir o eliminar servicios y herramientas, según sus necesidades específicas. Proponer futuras investigaciones que exploren la eficacia de diferentes proveedores de servicios en la nube y ajusten la arquitectura del laboratorio virtual, según los requisitos específicos de cada estudio, sería beneficioso.
En el futuro, se espera la combinación de datos de diversas fuentes, como imágenes (Arechiga et al. 2022), datos moleculares (Triana-Vallejos et al. 2022), sensores de movimiento/ubicación (Wägele et al. 2022) y observaciones sobre servicios ecosistémicos (García-López et al. 2022).