Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biotecnología en el Sector Agropecuario y Agroindustrial
Print version ISSN 1692-3561
Rev.Bio.Agro vol.11 no.spe Popayán Dec. 2013
IDENTIFICACIÓN DE LAS LEVADURAS NATIVAS PRESENTES EN ZUMOS DE PIÑA, MORA Y UVA
IDENTIFICATION OF YEAST IN THE NATIVE PINEAPPLE JUICE, BLACKBERRY AND GRAPE
IDENTIFICAÇÃO DE LEVEDURAS NESTES SUCO DE ABACAXINATIVA, MORAEUVA
LUZ ADRIANA MAMBUSCAY M.1, WILLIAM ANDRÉS LÓPEZ A.2, RAÚL ALBERTO CUERVO M.3, FRANCISCO E. ARGOTE V.4, ESTEBAN OSORIO C.5
1Bióloga. Joven Investigador Universidad San Buenaventura Sede Cali
2Biólogo. Joven Investigador Universidad San Buenaventura Sede Cali
3Biólogo. Magíster en Ciencias Biológicas. Doctorando en Ciencias Biológicas. Docente Asociado Programa de Agroindustria, Universidad de San Buenaventura Sede Cali.
4Ingeniero Agroindustrial. Especialista en Docencia Universitaria y Gerencia de Mercadeo. Magíster en Mercadeo Agroindustrial. Docente Asociado Programa de Agroindustria, Universidad de San Buenaventura Sede Cali.
5Biólogo. Doctor en Bioquímica. Docente Titular Universidad del Valle
Correspondencia: feargote@usbcali.edu.co
Recibido para evaluación: 07-11-2012. Aprobado para publicación: 27-05-2013.
RESUMEN
Las frutas son microhábitats importantes para una variedad de especies de levaduras en la naturaleza debido a su alta concentración de azúcares simples, bajo pH y la constante visita por insectos vectores. Este trabajo tuvo como objetivo principal el aislamiento e identificación de las levaduras encontradas en los zumos de mora, piña y uva. La identificación de las cepas se realizó mediante el uso de técnicas moleculares. Primero se realizó el análisis RFLP del gen ribosomal 5,8s y sus espaciadores transcritos internos con las enzimas CfoI, HaeIII y HinfI, para establecer agrupaciones de acuerdo a los patrones de restricción. Posteriormente 28 cepas fueron seleccionadas como representantes de los grupos para secuenciar el dominio D1/D2 del gen ribosomal 26s y poder definir a que especie pertenecían los diferentes aislados. Adicionalmente se hizo una caracterización fisiológica parcial de cada una de las cepas. Finalmente se logró la identificación de 66 cepas levaduriformes, distribuidas en las siguientes especies: Hanseniaspora uvarum, Wickerhamomyces pijperi, Candida sp., Pichia kluyveri, Hansenia sporauvarum, Candida boidinii, Candida oleophila, Meyerozyma caribbica, Hanseniaspora pseudo guilliermondii, Pichia sp.1, Pichia sp.2, Candida pseudointermedia, Pichia membranifaciens, Issatchenkia terrícola y Candida azyma
PALABRAS CLAVES: Frutas,Técnica, Dominio D1/D2, RFLPs.
ABSTRACT
Fruits are important micro-habitats for a variety ofyeast speciesin the wild dueto its high concentration of simple sugars,lowpHand constant visits by insect vectors.This work had asmain objective the isolation and identification of yeasts found in blackberry juice, pineapple and grape. The identification of this trains was performed by using molecular techniques.First performed RFLP analysis ofribosomal gene internal transcribed spacers5, 8syherwith enzymes CfoI, HaeIII and HinfIto establish groups according to restriction patterns. Then 28 strainswere selected as representatives of groups to sequence the D1/D2 domain of 26S ribosomal gene and to define the specie of the different isolates. Additionally it was a partial physiological characterization of each of the strains. Finally achieving the identification of 66 yeast strains, divided into the following species:Hanseniasporauvarum, Wickerhamomyces pijperi, Candida sp., Pichiakluyveri, Hanseniasporauvarum, Candida boidinii, Candida oleophila, Meyerozyma caribbica, Hanseniaspora pseudo guilliermondii, Pichiasp.1, Pichiasp.2, Candida pseudointermedia , Pichiamembranifaciens, Issatchenkiaterrícola andCandida azyma
KEY WORDS:Fruit,Technical,Domain D1/D2, RFLPs.
RESUMO
As frutas são importantes microhabitats para uma variedade de espécies de leveduras na natureza devido à sua alta concentração de açúcares simples, baixo pH e visitas constantes por insetos vetores. Este trabalho teve como objetivo principal o isolamento e identificação das leveduras encontradas no suco de amora, abacaxi e uva. A identificação das estirpes foi realizada utilizando técnicas moleculares. Primeiro realizada uma análise de RFLP do gene ribossomal espaçadores internos transcritos 5,8syla com enzimas CFOI, HaeIII e HinfI para estabelecer grupos de acordo com os padrões de restrição. Em seguida, 28 foram selecionados como representantes de grupos a sequência do domínio D1/D2 do gene ribossomal 26S e para definir a espécie dos diferentes isolados. Além disso, ele era um caracterização parcial fisiológica de cada uma das estirpes. Finalmente, chegando à identificação de 66 cepas de leveduras, divididos nas seguintes espécies: Hanseniaspora uvarum, Wickerhamomyces pijperi, Candida sp, Pichia kluyveri, Wickerhamomyces anomalus, Candida boidinii, Candida oleophila, Meyerozyma caribbica, Hanseniaspora pseudoguilliermondii, Pichia sp.1, Pichia. sp.2, Candida pseudointermedia, Pichia membranifaciens, tIssatchenkia terrícola e Candida azyma
PALAVRAS-CHAVE:Frutas, Técnicas, Dominio D1/D2, RFLPs.
INTRODUCCIÓN
Las levaduras son hongos unicelulares que pueden ser clasificados en dos grupo filéticos: levaduras ascomicetosasanamórficas y teleomórficas y levaduras basidiomicetosasanamórficas [1].
Debido a su gran diversidad fisiológica pueden crecer en un amplio rango de hábitats.Al ser organismos heterotróficos requieren para su crecimiento nutrientes minerales y una cantidad significativa de carbono orgánico. La variedad de hábitats en los cuales se pueden encontrar levaduras incluyen: suelos, insectos, plantas, frutas, exudados de árboles, algas, ambientes marinos y la atmosfera. De igual manera, su presencia también es común en alimentos manufacturados, bebidas fermentadas, en intestinos de animales entre otros [2].
Las frutas son microhábitats importantes para una variedad de especies de levaduras en la naturaleza debido a su alta concentración de azúcares simples, bajo pH y la constante visita por insectos vectores [3]. En estos sustratos la sucesión de levaduras está involucrada en varios procesos bioquímicos y ecológicos, incluyendo el deterioro de las frutas. Esto ocurre debido a la habilidad de las levaduras para utilizar rápidamente azúcares simples presentes en los sustratos. La presencia de especies proteolíticas y pectinolíticas sobre estos sustratos puede cumplir un papel muy importante en el establecimiento y mantenimiento de la comunidad levaduriforme[4]. Durante la colonización de las frutas algunos de estos factores pueden conferirle ventajas adaptativas a algunas especies. La presencia de cepas productoras de b-glucosidasa puede contribuir a mejorar las características aromáticas de las frutas, además de ser de importancia biotecnológica para su aplicación en la industria alimenticia[5].
En Colombia son pocos los estudios enfocados a la diversidad de levaduras presente en diferentes sustratos. En este sentido se destaca el estudio de Osorio et al. (2008), en el cual se evaluó la diversidad de levaduras asociada al “champús”, reportando las siguientes especies: S. cerevisiae, Issatchenkiaorientalis, P. fermentans, Zygosaccharomycesfermentati, Torulosporadelbruekii, P. kluyverivar. kluyveri, y Galactomycesgeotrichum [6].
López et al. (2009) identificaron las especies más representativas en chichas de maíz, piña y arracacha: Candidatropicalis, Pichia kluyveri, Pichiaguilliermondii, Hanseniaporaguilliermondii, Pichiafermentans, Saccharomycescerevisiae, Candida maltosa, Rhodotorulaglutinis, Torulasporadelbrueckii, Hanseniaspora uvarum, Kazachstania exigua, Kluyveromycesmarxianus, Yarrowialypolitica, Candidaparapsilosis, Debaromyceshansenii, Cryptococcusarboriformis, Saccharomycesmartiniae, Dekkeraanomala, Aureobasidiumpullulans y Candida pseudointermedia[7].
El propósito de esta investigación fue identificar las especies de levaduras más representativas en tres frutas: uva, piña y mora. En este estudio se usaron métodos moleculares para la caracterización de las levaduras nativas presentes en las tres frutas. Para realizar esta caracterización se usaron las técnicas, análisis RFLP del gen ribosomal 5,8s y sus espaciadores transcritos internos, usando las enzimas de restricción Hhal, Haelll y Hinfl; y el secuenciamiento del dominio D1/D2 del gen ribosomal 26s.
MÉTODO
Preparación del zumo y aislamiento de las levaduras
El zumo de las frutas (piña, mora y uva) se preparó agregando 10 g de la fruta en 100 mL de agua destilada estéril. Se realizaron 5 preparaciones de cada zumo. Para cada preparación diferente se hizo un muestreo, en cada uno de estos, se realizaron diluciones seriadas (10-1-10-5) y aislamiento en medio de cultivo YPDA (1% extracto de levadura, 2% peptona micológica, 2% de glucosa, 2% agar) suplementado con 25mg/L de penicilina y cloranfenicol. Las placas se incubaron a 30°C por 3 días y se aislaron las colonias más representativas.
Caracterización fenotípica
Los aislados fueron analizados según criteriosmorfológicos y fisiológicos de acuerdoa las descripciones realizadas por Boekhoutet al. (2004). Se evaluó la morfología celular, modo de reproducción vegetativa y caracterizaciónfisiológica. La habilidad para fermentar (2% de fuente de carbono) y asimilar (0,5% defuente de carbono) glucosa, maltosa, sacarosa, y fructosa fue evaluada por la acumulación de dióxido de carbono en tubos durhamy turbidez en escala de Wickerham,respectivamente. Final-mente, se evaluó la halo-tolerancia(10, 15% p/v) en medio YNB, y la termotolerancia (28°C, 30°C y 35°C) en medio YPDA (Boekhoutet al., 2004). Todas las pruebas fisiológicas se realizarona 30°C.
Extracción de ADN genómico de levaduras
Para la extracción de ADN genómico de levaduras, se empleó el protocolo de Osorio-Cadavid et al. (2009): se tomó un cultivo fresco que se puso a crecer en medio YPD a 120rpm por 16 horas a 28°C. Posteriormente, se recuperaron las células por centrifugación. (MicrocentrífugaEppendorf) a 8000 rpm durante 5 min en un tubo de microcentrífuga. Luego se resuspendió el precipitado de células en 0,5mL de una solución de Sorbitol 1M, EDTA 0,1M, (pH 5), que contenía 50 Unidades (U) de la enzima Beta-glucoronidasa (Sigma-Aldrich). La suspensión se incubó en baño maría a 37°C por 60 minutos, agitando periódicamente. Seguidamente se centrifugó la suspensión a 8000 rpm por 10 minutos y se resuspendió el precipitado en 0,5mL de Tris-HCl 50mM, EDTA 20mM, (pH 7,4). Se añadieron 50μl de SDS al 10% y se incubó en baño maria a 65°C por 30 minutos. Inmediatamente se agregaron 0,2mL de acetato de potasio (KAc) 5M (pH 4,8), se resuspendió (mínimo durante 30 segundos) y se dejó en baño de hielo por 30 minutos.
Después, se centrifugó a 14 000 rpm por 5 minutos y se trasladó el sobrenadante a otro tubo, repitiendo este paso nuevamente para eliminar otras impurezas. Luego se adicionó 1 mL de isopropanol (conservado a -20°C) y se incubó a temperatura ambiente por 5 minutos agitando suavemente el tubo. Posteriormente, se centrifugó a 14 000 rpm por 10 minutos y se descartó el sobrenadante. A continuación, se añadieron 0,5 mL de etanol al 70% (conservado a -20°C), y se centrifugó a 14 000 rpm por 5 minutos descartandose el sobrenadante. Finalmente, se dejó secar el precipitado a temperatura ambiente, y se resuspendió en 50μl de TE (Tris-EDTA pH 7,4), se incubó por 5 minutos en baño maría a 100°C y se dejó enfriar a temperatura ambiente. La extracción se verificó añadiendo 3μl en gel de agarosa al 1% usando TBE 1X (Tris-Borato-EDTA) a 100-110v, durante 30min.
PCR-RFLP´S de la región 5.8s-ITS
Para la amplificación de la región que comprende el gen ribosomal 5,8S y sus dos espaciadores transcritos internos (ITS1 e ITS2), se utilizaron los primers ITS1 (5´ CCGTAGGTGAACCTGCGG-3´) e ITS4(5´-TCCTCCGCTTATTGATATGC-3´) (White 1990; Suárez 2007).
Se preparó un cóctel de reacción de 28μl que contenía: 0,5μM ITS1, 0,5μM ITS4, 10μM DNTPs, 1X NH4+, 1,5mM MgCl2 , y 1,2 unidades (U) de Taq Polimerasa, y se adiciono 7μl de ADN (600ng aproximadamente) para un volumen final de 35μl. La región del gen ribosomal 5,8S se amplificó en un termociclador (M.J. Research PTC-150, minicycler), bajo las siguientes condiciones: denaturación inicial a 94ºC por 5minutos, 30 ciclos de denaturación a 94ºC por 30s, apareamiento a 55ºC por 30s, y extensión a 72ºC por 1min, con una extensión final de 10min a 72ºC. Los amplificados se verificaron mediante electroforesis en geles de agarosa al 1% teñidos con bromuro de etidio.
La digestión de la región amplificada se realizó con las enzimas CfoI, HaeIIIy HinfI(Esteve-Zarzoso et. al 1999). Para la reacción con cada enzima se utilizaron 9μl del amplificado, 2 U de enzima y 1X del buffer de la enzima. La incubación se realizó por dos horas en un baño maría a 37°C. Los fragmentos de ADN generados por cada enzima fueron visualizados en geles de agarosa al 1,5% teñidos con bromuro de etidio. Los tamaños, tanto de los amplificados como de los fragmentos de restricción, se calcularon usando el software UvigelStartMw v11 ©, tomando como referencia el marcador de peso molecular Generuler 50pb.
Secuenciación del dominio D1/D2 gen rRNA 26S
Los aislados del cepario de la Universidad San Buenaventura - Cali fueron identificados usando la secuencia del dominio D1/D2 del gen ribosomal 26S. Los primers utilizados para amplificar esta esta región fueron: NL1 (5´-CATATCAATAAGCGGAGGAAAAG-3´)yNL4(5´-GTCCGTGTTTCAAGACGG-3´).
Las reacciones de amplificación (PCR) se realizaron en un termociclador (Multigene- Labnet, USA) bajo las siguientes condiciones: desnaturalización inicial a 94ºC por 5minutos, 30 ciclos de desnaturalización a 94ºC por 1min, apareamiento a 55ºC por 30seg, y extensión a 72ºC por 1min, con una extensión final de 10min a 72ºC. (Osorio-Cadavid et al. 2008). Para la amplificación, se tomarán 7uL (aproximadamente 1ng/μL) de ADN Genómico extraído y se resuspendió en 28µL de mezcla de PCR: 0,5μM ITS1, 0,5μM ITS4, 10μM dNTPs, 1X NH4+, 1,5mM MgCl2, y 1U de Taq Polimerasa. Los amplificados fueron visualizados por medio de electroforesis en geles de agarosa al 1,5% y visualizadas con Bromuro de Etidio a 240nm.
Los productos de PCR fueron purificados y secuenciados por la empresa Macrogen-USA. Posteriormente, las secuencias (Forward y Reverse) enviadas vía Internet fueron ensambladas y corregidas mediante el programa Chromas Pro v. 1,42 ® y comparadas con las secuencias reportadas en las bases de datos NCBI y CBS-KNAW usando el algoritmo “Basic Local AlignmentSearchTool (BLAST)”.
RESULTADOS
A partir de los muestreos realizados se aislaron 66 cepas levaduriformes, de las cuales 28 pertenecieron al zumo de mora (LM), 20 al zumo de piña (LP) y 18 al zumo de uva (LU). Cada uno de los aislados se caracterizó morfológicamente, las colonias presentaron variedad en su elevación y margen, el color predominante fue el blanco, seguido del crema y por último el rosado.La forma de todas las colonias fue circular. Entre las características observadas en las células están: la forma, que varió entre redonda, ovalada, cilíndrica y apiculada. Posterior a esta caracterización se realizó la extracción de ADN genómico de todos los aislados obtenidos, encontrando valores promedio de concentración y pureza (ABS 260/280) de ácidos nucléicos de 9677ng/ul y 2,1 respectivamente. En la figura 1 se muestra la corrida electroforética del ADN extraído a 18 cepas de levaduras.
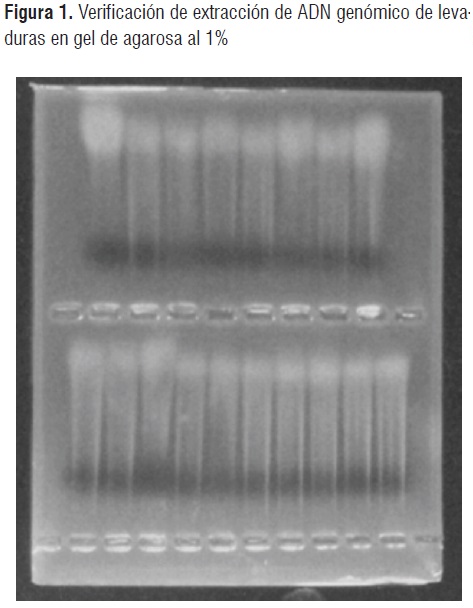
La caracterización molecular se inició con el análisis RFLP de la región que incluye el gen ribosomal 5,8s y sus espaciadores transcritos internos (5,8s-ITS). Los amplificados obtenidos fueron digeridos con las enzimas de restricción Cfol, Haelll y Hinfl, para obtener los fragmentos de restricción. Las amplificaciones y las digestiones fueron verificadas por electroforesis en gel de agarosa al 1,5% (figura 2 y 3).
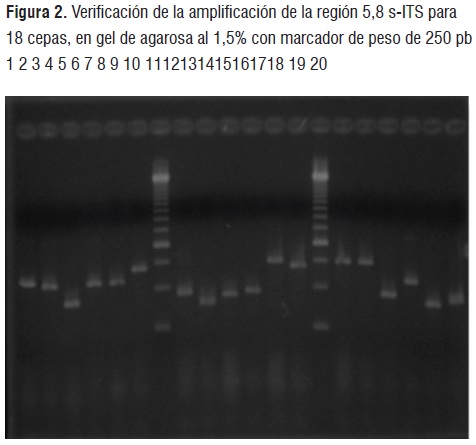
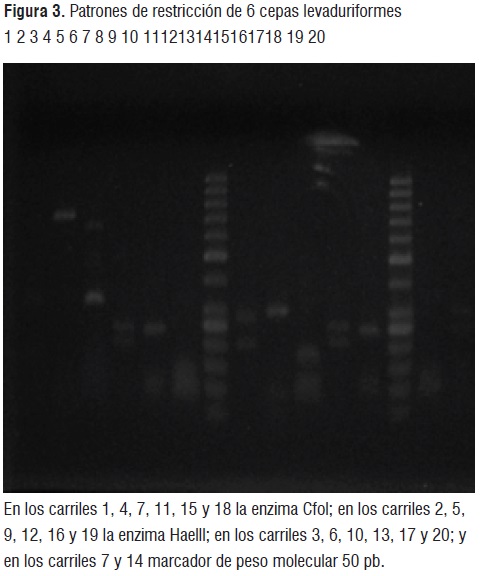
Los patrones de restricciónse usaron para establecer las agrupaciones consignadas en el cuadro 1. Con base en esta información se seleccionaron las cepas a las cuales se les secuenció el domino D1/D2 del gen ribosomal 26s. Se obtuvieron en total 28 secuencias que fueron usadas para la comparación mediante el algoritmo BLAST en las bases de datos NCBI y CBS-KNAW. Por medio de estas comparaciones se pudieron identificar hasta el nivel de especie los integrantes de los grupos 1, 2, 4, 5, 6, 7, 8, 9, 11, 12, 14 y 15, mientras que los integrantes de los grupos 3, 10 y 13 sólo pudieron ser asignados a nivel de género (Cuadro 2).
En cuanto a la distribución de las especies, sólo Wickerhamomyces pijperi y Candida boidinii estuvieron presentes en dos de las tres frutas (ambas en mora y piña), mientras que las demás fueron únicas en cada fruto (Cuadro 1).
En el zumo de mora, la mitad de las especies reportadas solo cuentan con un aislado, mientras que la especie Wickerhamomyces pijperi está representada por 13 de las 28 cepas aisladas. Las especies Hanseniaspora uvarumy Candida boidinii presentan 6 y 5 cepas respectivamente. De igual manera, de las 8 especies encontradas en el zumo de piña 3 estuvieron representadas por un único aislado; no bostante no hay una dominancia marcada por alguna de las especies. Por otro lado, en el zumo de uva sólo se reportaron 2 especies a partir de 18 aislados, dominando la especie Candida azyma con más del 83% de las cepas. En el caso de las especies compartidas, aunque Wickerhamomyces pijperi dominó ampliamente en los aislados del zumo de mora, sólo presentó un aislado en el zumo de piña. Algo similar pasó con la especie Candida boidinii, que tuvo 5 aislados en el zumo de mora y sólo uno el zumo de piña.
En el cuadro 3 se encuentran consignadas algunas características fenotípicas de los aislados de cada grupo conformado. El 71% de las cepas fermentaron glucosa, el 6% sacarosa, el 2% maltosa y ninguna fermentó lactosa. De las 15 especies reportadas 13 tuvieron al menos 1 cepa con la capacidad de asimilar la sacarosa, y 9 con la capacidad de asimilar la maltosa. La especie Candida pseudointermedia fue la única capaz de crecer en los cuatro azúcares, además de fermentar maltosa. En cuanto a la especie Candida azyma todas sus cepas crecieron en medio con 16% de NaCl, siendo igualadas únicamente por una cepa de pichia sp.1 y las dos cepas de Pichia membranifaciens.
Discusión
Las frutas son fuentes importantes de levaduras debido a su gran cantidad de azúcares simples y a la constante visita de vectores. De acuerdo a Skineret al. (1980) y a Phaff (1990), la microbiota natural de las futas está compuesta comúnmente de levaduras y hongos levaduriforfmes de los génerosAureobasidium, Rhodotorula, Sporobo-lomyces, Cryptococcus, Candida, Pichia,Aureobasidium, Rhodotorula, Sporobo-lomyces, Cryptococcus, Candida, Pichia, Hanseniasporay raramente Saccha-romycesy Schizosaccharomyces[8,9].
En este estudio se encontró una gran riqueza de especies principalmente en los zumos de mora y piña. Adicionalmente, varias de las especies reportadas tienen un gran potencial biotecnológico, especialmente en el campo del biocontrol.
En este sentido, la especie P. membranifaciens fue usada por Cao et al. (2010) para el control del moho azul (Penicilliumexpansum) en peras. El tratamiento de la levadura adicionado con molibdato de amonio inhibió la germinación de las esporas y la elongación del tubo germinal del hongo [10]. Además no encontraron cambios en los parámetros de calidad de las frutas, por lo que puede considerarse un tratamiento promisorio para el control de esta enfermedad postcosecha. Por otro lado, la secreción de toxinas killer por parte de esta especie también ha sido aprovecha para el control de levaduras contaminante en procesos fermentativos.
Santos et al. (2009) encontraron que la toxina PKTM2 inhibió el crecimiento de Brettanomycesbruxellensis, una levadura contaminante de procesos fermentativos, sin afectar a Saccharomycescerevisiae [11].Del total de especies encontradas en este estudio W. anomalus es la más reportada en estudios de biocontrol y biorremediación. Pan et al. (2004) probaron su capacidad para degradar hidrocarburos aromáticos policíclicos, pudiendo degradar naftaleno, dibenzotiofeno, fenantreno y criseno; tanto de manera individual como combinada [12]. Yang et al. (2011) reportaron valores de degradación de carbofurano del 95,2% en medios donde el insecticida era la única fuente de carbono, éste fue metabolizado por lacepa teniendo como intermediario el benzofuranol que después fue degradado [13]. En el campo del biocontrol se ha probado su efectividad en la reducción del crecimiento de mohos en granos de cereales durante su almacenamiento. Melinet al. (2007) optimizaron una formulación liofilizada de esta especie con viabilidades superiores al 80%; y al ser almacenados a 30% por un año no mostró reducción significativa de la viabilidad [14]. Estos estudios son de gran importancia porque evidencian la diversidad metabólica de esta levadura.
En estudios de producción de enzimas extracelulares se ha reportado la producción de b-glucosidadasa, peroxidasa y b-glucanasa, y en menor medida celulasa y poligalacturonasa; por parte de varias especies del género Pichiaespecialmente Pichiacaribbica (M. caribbica) aisladas de ecosistemas oléicos [15]. En este sentido H. uvarum, una especie común en la elaboración del vino, presenta producción de enzimas extracelulares como b-glucosidasa y b-xilosidasa, que por medio de su actividad liberan compuestos aromáticos importantes en el vino [16]. Además, al igual que la especie P. memebranifaciens ha sido usada como biocontrol, en el tratamiento contra el moho gris de la uva mejorado con la adición de molibdato de amonio.
Aunque los estudios de riqueza en zumos y mostos fermentados de uva presentan una gran variedad de especies, en este trabajo sólo se reportan dos especies C. azyma, con el 83% de los aislados en uva; e I. terrícola con el 17%. No obstante estas especies también fueron aisladas por Chavan et al. (2009) durante el estudio de la flora levaduriforme en diferentes variedades de uva de la India para la fabricación de vinos. Cabe resaltar que todos los aislados de C. azyma fermentaron glucosa y fueron halotolerantes y termotolerantes hasta 35°C. Por otro lado las especies P. kluyveri y C. pseudointermedia fueron reportadas en la fase inicial, durante la fermentación de la chicha de piña. La caracterización fenotípica parcial realizada en esta investigación mostró la gran variedad metabólica de las especies aisladas a partir de las tres frutas [17]. En cuanto a la identificación, 9 de los 64 aislados sólo pudieron ser asignados a nivel de género, lo que puede indicar la existencia
de especies nuevas en estos sustratos de acuerdo a los parámetros de identificación basados en el dominio D1/D2 del gen ribosomal 26s [16].AGRADECIMIENTOS
Este trabajo se pudo realizar gracias a la financiación por parte de la Universidad de San Buenaventura-Cali y al Programa Jóvenes investigadores e innovadores “Virginia Gómez de Pineda” año 2010.
REFERENCIAS
[1] BOEKHOUT, T., ROBERT, V., SMITH, M., STALPERS, J., YARROW,D., BOER, P., BUIS, R., GIJSWIJT, G., KURTZMAN, C.P., FELL,J.W., GUÉHO, E., GUILLOT, J., and ROBERTS, I. Yeasts ofthe World Version 2.0. Amsterdam (Netherlands);ETI Biodiversity Center, p. 520-525. [ Links ]
[2] KURTZMAN, C.P. and ROBNETT, C.J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences. Antonie van Leeuwenhoek, (73), 1998, p. 331-371 [ Links ]
[3] ROSI, I., VINELLA, M. and DOMIZIO, P. Characterization of b-glucosidase activity in yeasts of oenological origin. J. Appl. Bacteriol., (77), 1994, p. 519-527. [ Links ]
[4] TRINDADE, R.C., RESENDE, M.A., SILVA, C.M. and ROSA, C.A. Yeasts associated with fresh andfrozen pulps of Brazilian tropical fruits. System Appl. Microbiol.,(25), 2002, p. 294-300. [ Links ]
[5] ROMO-SÁNCHEZ, S., ALVES-BAFFI, M., ARÉVALO-VILLENA, M., ÚBEDA-IRANZO, J. and BRIONES-PÉREZ, A. Yeast biodiversity from oleic ecosystems: Study of their biotechnological properties. Food microbiology, (27), 2010, p. 487-492. [ Links ]
[6] OSORIO-CADAVID, E., RAMÍREZ, M., LOPEZ, W.A.yMAMBUSCAY, L.A. Estandarización de un protocolo sencillo para la extracción de ADN genómico de levaduras. Revista Colombiana de Biotecnología, (11), 2009, p.125-131 [ Links ]
[7] LOPEZ, W.A., RAMÍREZ, M., MAMBUSCAY, L.A. y OSORIO-CADAVID, E. Diversidad de levaduras asociadas a chichas tradicionales de Colombia. RevistaColombiana de Biotecnología,(12), 2009, p. 176-186 [ Links ]
[8] SKINNER, F.A., PASSMORE, S.M. and DAVENPORT,R.R. Biology and Activities of Yeasts. 2 ed. London (Inglaterra): Academic Press,1980, 125-128 p. [ Links ]
[9] PHAFF, H.J. Specific habitats of yeasts and their isolation. USFCC Newsletter, 18(4), 1990, p. 11-12. [ Links ]
[10] CAO, S., YUAN, Y., HU, Z. and ZHENG, Y. Combination of Pichiamembranifaciens and ammonium molybdate for controlling blue mould caused by Penicilliumexpansum in peach fruit. International Journal of Food Microbiology,141, 2010, p. 173-176 [ Links ]
[11] SANTOS, A., SAN MAURO, M., BRAVO, E. and MARQUINA, D. PMKT2, a new killer toxin from Pichiamembranifaciens, and its promising biotechnological properties for control of thespoilage yeast Brettanomycesbruxellensis. Journal of Microbiology 155, 2009, p. 624-634. [ Links ]
[12] PAN, F., YANG, Q., ZHANG, S. and YANG, M. Biodegradation of polycyclic aromatic hydrocarbons by Pichiaanomala. Biotechnology Letters, 26, 2004, p. 803-806 [ Links ]
[13] YANG, L., CHEN, S., HU, M., HAO, W., GENG, P. and ZHANG, Y. Biodegradation of carbofuran by Pichiaanomala strainHQ-C-01 and its application for bioremediation of contaminated soils. Biol. fertile soils, 47, 2011, p. 917-923. [ Links ]
[14] MELIN, P., HÅKANSSON, S., and SCHNÜRER, JOHAN. Optimisation and comparison of liquid and dry formulation sof the biocontrol yeast Pichiaanomala J121. Appl.Microbiol.Biotechnol., 73, 2007, p. 1008-1016 [ Links ]
[15] OSORIO-CADAVID, E., CHAVES-LOPEZ, C., TOFALO, R., PAPARELLA, A. and SUZZI, G. Detection and identification of wild yeasts in champús, a fermented Colombian maize beverage. Food Microbiology,25, 2008, p. 771-777 [ Links ]
[16] KURTZMAN, C.P. and FELL, J.W. The Yeasts, a Taxonomic Study.4 ed. Amsterdam (Holanda): Elsevier, 1998, 21-30 p. [ Links ]
[17] CHAVAN, P., MANE, S., KULKARNI, G., SHAMIM, S., GHORMADE, V., NERKAR, D., SHOUCHE, Y. and DESPANDE, M. Natural yeast flora of different varieties of grapes used for wine making in India. Food microbiology, 26, 2009, p. 801-808 [ Links ]
[18] JACQUES, N. y CASAREGOLA, S. 2008. Safety assessment of dairy microorganisms: The hemiascomycetous yeasts. International journal of food microbiology, 126, 2008, p. 321-326. [ Links ]
[19] ESTEVE-ZARZOSO, B., BELLOCH, C., URUBURU, F. and QUEROL, A. Identification of yeasts by RFLP analysis of the 5.8S rRNA gene and the two ribosomal internal transcribedspacers. International Journal of SystematicsBacteriology,(49),1999, p. 329-337 [ Links ]
[20]KURTZMAN, C.P. and FELL, J.W. The Yeasts, a Taxonomic Study.5 ed. Amsterdam (Holanda): Elsevier, 2010, 1082p. [ Links ]
[21] WHITE, T.J., BURNS, T., LEE, S. and TAYLER, J., Amplification and Direct Sequencing of Fungal RibosomalRNA Genes for Phylogenetics. Journal of Molecular Biology, 2 (4), 1990, p. 32-40. [ Links ]













