Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.24 no.3 Bogotá Sept. 2004
Epidemiología molecular de infección nosocomial por Klebsiella pneumoniae productora de beta-lactamasas de espectro extendido
Paula Andrea Espinal 1 , José Ramón Mantilla 1,2 , Carlos H. Saavedra 3,5 , Aura Lucía Leal 3 ,
Celia Alpuche 4 , Emilia María Valenzuela 2
1 Posgrado Interfacultades de Microbiología, Universidad Nacional de Colombia, Bogotá, D.C., Colombia.
2
Instituto de Biotecnología, Universidad Nacional de Colombia, Bogotá, D.C., Colombia.3
Facultad de Medicina, Universidad Nacional de Colombia, Bogotá, D.C., Colombia.4
Facultad de Medicina, Universidad Nacional Autónoma de México, México, D.F., México.5
Hospital Universitario Clínica San Rafael, Bogotá, D.C., Colombia.La epidemiología molecular aplicada al estudio de las infecciones nosocomiales ha sido fundamental para la formulación y la evaluación de las medidas de control; con este fin, se caracterizaron microbiológica y molecularmente aislamientos de Klebsiella pneumoniae productores de beta-lactamasas de espectro extendido (BLEE) obtenidos de pacientes en un hospital de tercer nivel de Bogotá, D.C., Colombia. Se tipificaron quince aislamientos por electroforesis en gel de campo pulsado (PFGE) y por amplificación de secuencias de AND repetidas (REP-PCR). La susceptibilidad antimicrobiana y la producción de BLEE se determinaron de acuerdo con las normas de NCCLS. Las beta-lactamasas se evaluaron por isoelectroenfoque y PCR. El 80% de estos aislamientos se asociaron con infección nosocomial y de éstos, el 91,7% provenía de unidades de cuidado intensivo. La susceptibilidad antibiótica mostró 13 patrones de resistencia; 87% de los aislamientos presentó corresistencia a amikacina, 53% a gentamicina, 33,3% a ciprofloxacina, 40% a cefepime, 66,7% a piperacilina/tazobactam, 60% trimetoprim/sulfametoxazol y 46,7% a cloranfenicol. Todos fueron sensibles a imipenem. En la mayoría de los aislamientos se detectó producción simultánea de beta-lactamasas del tipo TEM y SHV y el 93,3% produjo ceftazidimasa de pI 8.2 del tipo SHV-5. Los 15 aislamientos fueron agrupados por PFGE y REP-PCR en 11 y 12 patrones electroforéticos, respectivamente. Esta variabilidad genética está relacionada con infecciones nosocomiales de origen endógeno más que por infecciones cruzadas.
Palabras clave: Klebsiella pneumoniae, BLEE-SHV-5, resistencia bacteriana, infección nosocomial, genotipificación.
Molecular epidemiology of nosocomial infection by extended-spectrum beta-lactamases-producing Klebsiella pneumoniae
Molecular epidemiology applied to the study of nosocomial infection has been fundamental in formulating and evaluating control methods. From patients in a level 3 Bogota hospital, Klebsiella pneumoniae samples were isolated that produced extended-spectrum beta-lactamases (ESBL). Each of 15 isolates was characterized microbiologically and by molecular characters realized by pulsed field gel electrophoresis (PFGE) and by repetitive-DNA sequences amplification (REP-PCR). Antimicrobial susceptibility and ESBL production was determined in accordance with NCCLS guidelines. The beta-lactamases were evaluated by isoelectric-focusing and PCR. Twelve (80%) of the isolates were associated with nosocomial infection; 11 of them were from intensive care units. The antibiotic susceptibility displayed 13 resistance patterns - 87% presented co-resistance to amikacin, 53% to gentamicin, 33% to ciprofloxacin,40% to cefepime, 67% to piperacillin/tazobactam, 60% to trimethoprim/sulfamethoxazole and 47% to chloranphenicol. All were sensitive to imipenem. Production of TEM and SHV beta-lactamases was detected simultaneously in most isolates by isoelectric focusing and 93.3% produced a ceftazidimase of pI 8.2 of the SHV-5 type. The 15 isolates were grouped into 11 and 12 electrophoretic patterns by PFGE and REP-PCR, respectively. The degree of genetic variability indicated an endogenous origin of the nosocomial infections.
Key words: Klebsiella pneumoniae, ESBL, SHV-5, bacterial resistance, nosocomial infection, genotyping.
Klebsiella pneumoniae es un patógeno de importancia en el ámbito hospitalario, frecuentemente relacionado con infecciones nosocomiales en unidades de cuidado intensivo y salas de pediatría, desde donde se aíslan cada vez con más frecuencia cepas resistentes a cefalosporinas de tercera generación con resistencia combinada (corresistencia) a múltiples antibióticos (1-5). Con la proliferación de cepas multirresistentes, el problema de la infecciones intrahospitalarias causadas por K. pneumoniae es aún mayor, ya que al ser más difíciles de tratar dan lugar a incrementos de las tasas de mortalidad, de las estancias hospitalarias y de los costos de atención (4,6). Los estudios realizados en Colombia señalan a K. pneumoniae como el cuarto patógeno hospitalario causante de infección (7,8) y cuya resistencia a cefalosporinas de tercera generación se ha incrementado significativamente (Leal AL,GREBO. Resistencia antimicrobiana en hospitales de tercer nivel en Bogotá, 2001-2002. VI Congreso Nacional de Enfermedades Infecciosas, Cartagena, Colombia, junio de 2003. Infectio 2003;7:108); sin embargo, existe poco conocimiento de los factores que han favorecido el desarrollo de la infección nosocomial, de los mecanismos de resistencia y de las características genéticas de las enterobacterias productoras de beta-lactamasas causantes de infección intrahospitalaria en nuestros hospitales.
Con el fin de contribuir al conocimiento de estas características, en este trabajo se estudiaron aislamientos de K. pneumoniae asociados con infecciones nosocomiales. Mediante la combinación de procedimientos microbiológicos, bioquímicos y de biología molecular se detectaron e identificaron beta-lactamasas de espectro extendido (BLEE). Se estableció la relación genética de la población bacteriana mediante dos procedimientos de genotipificación: amplificación de secuencias de ADN repetidas (REP-PCR) y electroforesis en gel de campo pulsado (PFGE, del inglés pulsed field gel electrophoresis) (9-13).
Materiales y métodos
Aislamientos bacterianos
Para el presente estudio se consideraron 18 casos de pacientes con infección, hospitalizados durante el periodo abril 2001-2002 en el Hospital Universitario Clínica San Rafael de Bogotá. De estos pacientes, se obtuvieron 22 aislamientos de K. pneumoniae identificados con el sistema MicroScan y confirmados con el sistema API 20E (BioMérieux S.A.).
Para el estudio de epidemiología molecular, solamente se incluyeron los 15 aislamientos de K. pneumoniae resistentes a cefalosporinas de tercera generación.
Pruebas de susceptibilidad antimicrobiana
La susceptibilidad antimicrobiana se determinó por el método automatizado MicroScan (panel 13) y se confirmó mediante la técnica de difusión en agar Mueller-Hinton con discos de amikacina, gentamicina, ciprofloxacina, piperacilina/ tazobactam, cloranfenicol, trimetoprim/ sulfametoxazol, imipenem, cefepime, ceftazidima, ceftriaxona, cefotaxima y aztreonam (Oxoid). La prueba confirmatoria de producción de BLEE se realizó con base en las recomendaciones del National Committee for Clinical Laboratory Standards (NCCLS) (14); se emplearon discos de cefotaxima (30 µg) y ceftazidima (30 µg) solos y en combinación con ácido clavulánico (10 µg) (Oxoid).También se empleó la prueba de E-test (AB-Biodisk) utilizando tiras con gradientes de concentración de ceftazidima y de cefotaxima, solas y en combinación con ácido clavulánico. En las dos metodologías se empleó como control positivo la cepa ATCC 700603 de K. pneumoniae productora de BLEE y como control negativo, la cepa ATCC 25922 de Escherichia coli.
Determinación del punto isoeléctrico de las BLEE
Los extractos crudos de beta-lactamasas obtenidos por sonicación de cultivos bacterianos se analizaron mediante isoelectroenfoque (IEF) sobre geles de poliacrilamida con rangos de pH 3 a 9, en el equipo PhastSystem (Pharmacia). Las beta-lactamasas se detectaron por hidrólisis de nitrocefín (500 µg/ml). Los valores de los puntos isoeléctricos (pI) se determinaron de acuerdo con el método descrito por Matthew et al. (15), mediante comparación con beta-lactamasas de pI conocidos. Cuando la hidrólisis sobre el nitrocefín se hizo evidente (bandas anaranjadas), se continuó con la prueba biológica.
Detección de BLEE mediante bioensayo
sobre geles de IEF
Con el fin de detectar la actividad frente a ceftazidima o frente a cefotaxima de las beta-lactamasas separadas por isoelectroenfoque se realizó la prueba biológica (bioensayo) descrita por Silva et al. (16). El gel de acrilamida previamente impregnado con ceftazidima (8 µg/ ml) o con cefotaxima (8 µg/ml) se incubó en una cámara húmeda a 37 º C durante 30 minutos, para permitir la hidrólisis total de la cefalosporina por las beta-lactamasas separadas. Posteriormente, el gel se cubrió con células en crecimiento logarítmico de la cepa E. coli J53-2, sensible a ceftazidima y a cefotaxima, suspendidas en agar suave (0,7%). Después de incubar en cámara húmeda a 37 º C, se registraron los puntos de crecimiento bacteriano sobre el gel para establecer el valor de pI de las enzimas con capacidad de hidrolizar el antibiótico.
Transferencia de los genes codificantes de
las BLEE por conjugación y análisis de plásmidos
La transferencia de plásmidos por conjugación se realizó de acuerdo con el método descrito por Sirot et al. (17). Se promovió la conjugación entre una cepa representativa de grupos de K. pneumoniae resistentes a ceftazidima (CAZ r ) y E. coli J53-2 sensible a CAZ y resistente a rifampicina (Rif r ).
En los transconjugantes de E. coli Rif r CAZ r , portadoras de un solo plásmido, se confirmó la producción de BLEE por la prueba E-Test, se evaluó la susceptibilidad antimicrobiana y por IEF se determinaron los puntos isoeléctricos de la beta-lactamasa producida. El ADN plasmídico de los transconjugantes se obtuvo por el método de lisis alcalina y se analizó por electroforesis (18).
Identificación por PCR del gen bla SHV-5
El ADN plasmídico de los aislamientos de K.
pneumoniae y de los transconjugantes portadores de un solo plásmido se amplificó por reacción en cadena de la polimerasa (PCR, del inglés polimerase chain reaction) para la identificación del gen bla SHV-5 . Se utilizaron los iniciadores A (5'-ACTGAATGAGGCGCTTCC)
y B (5'-TCCCGCA GATAAATCACC) siguiendo las condiciones de amplificación previamente descritas (19).
Tipificación molecular por REP-PCR y electroforesis en gel de campos pulsados (PFGE)
La amplificación por REP-PCR se realizó de acuerdo con protocolos publicados (9-11) con algunas modificaciones. Se utilizaron como iniciadores: REP1R- Dt 5'- IIINCGNCGNCATCNGGC-3' y REP2- Dt, 5-NCGNCTTATCNGGGCTAC-3' sintetizados por Invitrogen. En el termociclador iCycler (Bio-Rad) se amplificaron 100 ng de ADN en un volumen de reacción de 25 µl que contenía: 200 µM de cada dNTP, 6 mM de cloruro de magnesio; 2 µM de cada iniciador; 0,2 mg/ml de albúmina sérica bovina (BSA); 5% de dimetilsulfóxido (DMSO); solución tampón para PCR 1X y 2 unidades de Taq ADN-polimerasa (Invitrogen). La amplificación se realizó con un paso inicial de 94 º C por 7 minutos y, luego, 30 ciclos de: desnaturalización (92 º C, 30 segundos), asociación (60 º C, 1 minuto) y extensión (70 º C, 3 minutos), seguidos por un paso de extensión final (70 º C, 8 minutos). Se evaluaron 10 ml de producto amplificado mediante electroforesis en geles de agarosa al 2% con solución tampón TBE 0,5 X durante 3 horas a 4,6 V/cm.
La tipificación por PFGE se realizó siguiendo la metodología descrita por Miranda (12). Se digirió con la enzima XbaI (10 U/µl) (Gibco BRL) durante 18 horas. Los fragmentos generados se separaron en gel de agarosa PFGE (Bio-Rad) al 1% en solución tampón TBE 1X, en el equipo para campos pulsados CHEF-DR III (Bio-Rad), con pulsos de 5 a 60 segundos por 20 horas a 200 voltios.
Los patrones electroforéticos registrados en el sistema Gel-Doc de Bio-Rad se analizaron usando el software NTSYSpc versión 2.0. Se generó una matriz de similaridad basado en la presencia y ausencia de bandas y, luego, se creó un dendrograma basado en el algoritmo UPGMA (del inglés, unweighted pair group method with arithmetic averages).
<>Investigación epidemiológica
Se elaboró un formato de información clínica para cada uno de los pacientes a quienes se les aisló el microorganismo en estudio. Se incluyeron las siguientes variables: edad y sexo del paciente, fecha de ingreso y egreso del hospital, antecedentes, terapia antibiótica, hospitalizaciones previas, clasificación de la infección, fuente del aislamiento, caracterización de la calidad de la muestra y estado del paciente al egreso. Para el manejo de la información se creó una base de datos utilizando el programa EpiInfo 2002 del CDC ( Centers for Disease Control and Prevention, Atlanta).
Resultados
Características clínicas
De acuerdo con los criterios para la clasificación de las infecciones intrahospitalarias del CDC (20) y del Comité de Infecciones del hospital, se encontró que 11 de 18 pacientes hospitalizados en la institución (61,1%) tuvieron infección intrahospitalaria, 3 (16,6%) infección extra-hospitalaria y 4 (22,2%) colonización. Los casos de infección se presentaron con mayor frecuencia en pacientes menores de 2 años (7/18) y adultos entre 60 y 80 años (9/18).
Para el análisis molecular se estudiaron 15 aislamientos obtenidos de 11 pacientes, resistentes a cefalosporinas de tercera generación en el tamizaje para detección de BLEE. De tres pacientes se obtuvo más de un aislamiento, los cuales se incluyeron en el estudio como episodios independientes debido a que se recogieron en fechas o desde sitios diferentes durante el tiempo de hospitalización. Los aislamientos se recuperaron de pacientes que se encontraron en las unidades de cuidado intensivo (12/15, 80%), medicina interna (2/15, 13,3%) y pediatría (1/15, 6,7%). La muestra más frecuente a partir de la cual se recuperó el microorganismo fue de hemocultivo (26,7%). Las características de los pacientes de los cuales se obtuvieron los aislamientos productores de BLEE se resumen en el
cuadro 1.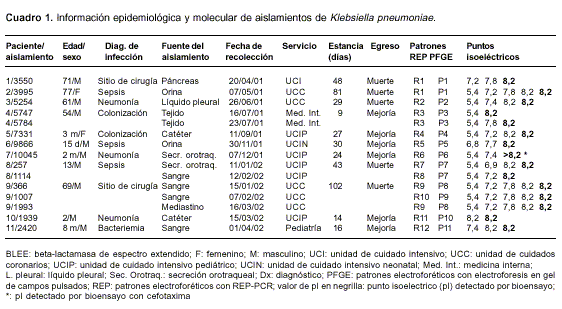
Las causas de infección intrahospitalaria por K. pneumoniae productora de BLEE estuvieron principalmente asociadas con infección del sitio quirúrgico, sepsis y neumonía. El 81,8% de estos pacientes estuvo en una unidad de cuidado intensivo en algún momento de su hospitalización y tuvo estancias prolongadas con rangos entre 14 y 102 días. En 5 pacientes la muerte fue atribuible a la infección.
Susceptibilidad antimicrobiana
Los aislamientos resistentes a cefalosporinas de tercera generación y aztreonam mostraron resistencia combinada con ampicilina (100%), gentamicina (53,3%), amikacina (86,7%), ciprofloxacina (33,3%), cefepime (40%), piperacilina/tazobactam (66,7%), trimetoprim/ sulfametoxazol (60%) y cloranfenicol (46,7%).
Todos los aislamientos fueron sensibles a imipenem. Con esta información se establecieron 13 patrones de resistencia o antibiotipos (Abiot) (
cuadro 2).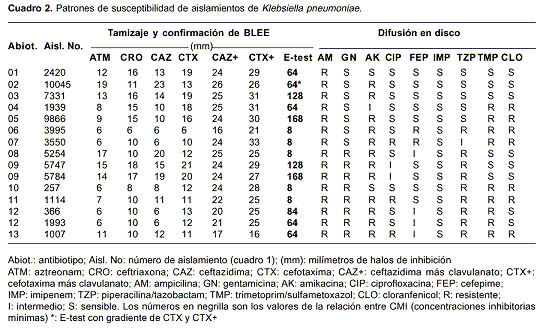
En la prueba de tamizaje con sensidiscos, 14 aislamientos fueron resistentes a los cuatro antibióticos (ceftazidima, cefotaxima, ceftriaxona y aztreonam) y un aislamiento (10045) fue sensible a ceftazidima (CAZ s ), aunque resistentea cefotaxima, ceftriazona y aztreonam. Todos los aislamientos se confirmaron como productores de BLEE. Con la prueba de E-test se obtuvieron los mismos resultados.
Caracterización de beta-lactamasas
Los aislamientos productores de BLEE presentaron entre 1 y 5 beta-lactamasas con pI de 5,4, 6,8, 6,9, 7,2, 7,4, 7,7, 7,8, 8,2 y 8,9 de manera combinada, valores que sugieren la presencia de beta-lactamasas tipo TEM y SHV (6). Los pI más frecuentes fueron 8,2 en 93,3% de los aislamientos y 5,4 en 73,3%, posiblemente asociados con SHV-5 y TEM-1, respectivamente (
cuadro 1) (20,21). El bioensayo para detectar beta-lactamasas separadas por isoelectroenfoque, mostró que 14 de los 15 aislamientos produjeron una enzima de pI 8,2 con actividad hidrolítica sobre ceftazidima y no se observó esta actividad por parte del aislamiento 10045 (CAZ s ). Con excepción del aislamiento 10045, en los demás aislamientos se detectó el gen bla SHV-5 por PCR.Dentro de la gran variedad de beta-lactamasas producidas por las cepas de este centro hospitalario, se detectaron algunas cuyos puntos isoeléctricos no se han informado como BLEE (21). Así, siete aislamientos produjeron beta-lactamasas con pI 7,2; tres con pI 7,4, uno con pI 6,9 y otro con dos beta-lactamasas con pI 6,8 y 7,7. Ninguna de las beta-lactamasas mencionadas mostró actividad frente a CAZ en el bioensayo.
Por conjugación se transfirieron plásmidos de 23 kb, aproximadamente, que confirieron a E. coli J53-2 resistencia a ceftazidima. El análisis por isoelectroenfoque de los transconjugantes mostró la producción de BLEE con pI 8,2 y por PCR se detectó el gen bla SHV-5 ; además, los trans-conjugantes adquirieron resistencia a cefotaxima, ampicilina, aztreonam y la respectiva resistencia a amikacina, gentamicina y cloranfenicol de la cepa de K. pneumoniae parental.
Tipificación molecular
En la tipificación molecular mediante electroforesis en gel por campos pulsados (PFGE) se obtuvieron patrones con 18-20 bandas entre 48 kb y 820 kb. Los 15 aislamientos se agruparon en 11 patrones electroforéticos (P1 a P11) (
cuadro 1, figura 1).
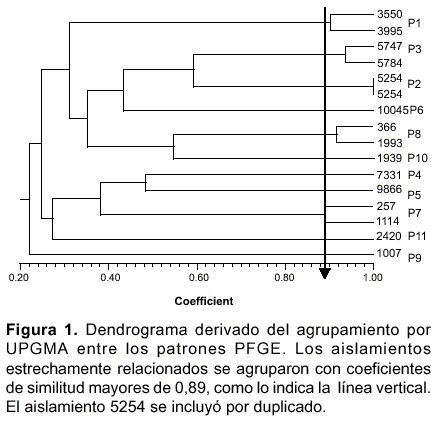
Cada uno de los patrones P1, P3, P7 y P8 agrupó dos aislamientos; los demás correspondieron cada uno a un aislamiento. Los aislamientos agrupados con un coeficiente de similitud superior a 0,89 se consideraron estrechamente relacionados de acuerdo con las indicaciones de Tenover et al. (13) y la información obtenida de los datos epidemiológicos.
En la tipificación por REP-PCR, los productos generados mostraron de 12 a 21 bandas con tamaños entre 0,3 kb y 2,6 kb. Los aislamientos se agruparon con un valor de coeficiente de similitud superior a 0,87 (
figura 2). Este valor se definió mediante comparación de las agrupaciones obtenidas con PFGE que fue la técnica de referencia y la información obtenida con los datos epidemiológicos y moleculares. Con REP-PCR se obtuvieron 12 patrones electroforéticos (R1 a R12) cuadro 1, figura 2), los patrones R1, R3 y R9 agruparon dos aislamientos y cada uno de los 9 restantes un aislamiento.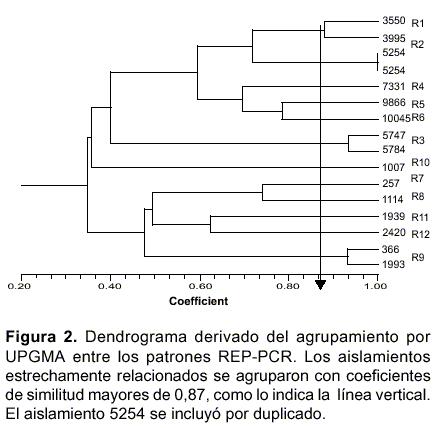
La comparación de las agrupaciones obtenidas con PFGE y con REP-PCR mostró lo siguiente: los patrones electroforéticos P1, P3 y P8 correspondieron a los patrones R1, R3 y R9 respectivamente; el patrón P7 agrupó 2 aislamientos que fueron discriminados como R7 y R8 mediante REP-PCR (
cuadro 1). La agrupaciónP1/R1 incluyó 2 aislamientos considerados estrechamente relacionados, con patrones de resistencia muy semejantes (antibiotipos 06 y 07), recuperados de 2 pacientes hospitalizados en diferentes unidades: uno en la Unidad de Cuidados Intensivos (UCI) (abril de 2001) y el otro en la Unidad de Cuidados Coronarios (UCC) (mayo de 2001). Durante su hospitalización, el paciente No. 2 de la UCC había estado previamente en la UCI, momento en el cual pudo adquirir el mismo tipo de cepa aislada del paciente No.1 (
cuadro 1).Dos aislamientos productores de BLEE que fueron recuperados del mismo paciente en meses consecutivos (enero y febrero de 2002), se agruparon en el patrón electroforético P7; sin embargo, estos aislamientos presentaron antibiotipos diferentes y fueron discriminados por REP-PCR en los patrones electroforéticos R7 (antibiotipo 10) y R8 (antibiotipo 11). La agrupación P3/R3 incluyó 2 aislamientos del mismo paciente hospitalizado en medicina interna (julio de 2001), presentaron el antibiotipo 09 y genéticamente fueron considerados estrechamente relacionados.
La agrupación P8/R9 incluyó dos aislamientos con iguales patrones de resistencia (antibiotipo 12) recuperados del paciente No. 9 en diferentes meses (
cuadros 1 y cuadro 2). De este paciente se obtuvo otro aislamiento que no se relacionó genéticamente con los otros dos aislamientos, ni por PFGE (patrón P9) ni por REP-PCR (patrón R10) (cuadro 1); además, mostró un patrón de resistencia diferente (antibiotipo 13) al de la agrupación P8/R9.Discusión
K. pneumoniae es un patógeno de importancia en el ambiente hospitalario, frecuentemente relacionado con infecciones nosocomiales en unidades de cuidado intensivo y salas de pediatría.
Su elevada resistencia a los antibióticos ß-lactámicos, aminoglucósidos y a quinolonas es un problema clínico en la actualidad. En los casos de infección por K. pneumoniae estudiados, prevalecieron los factores asociados con riesgo para infección nosocomial, como son las edades extremas de los pacientes, la hospitalización en una UCI, las estancias prolongadas y el uso previo de antibióticos, los cuales no difieren con respecto a lo informado en la literatura (22,23).
En hospitales y centros de salud de diferentes partes del mundo (1-4,23) se han descrito brotes debidos a la diseminación de cepas bacterianas productoras de BLEE y a la transferencia de plásmidos que portan los genes codificantes de las BLEE entre diferentes especies de la familia Enterobacteriaceae, lo cual ha conducido a un cambio significativo de la resistencia anti-microbiana en el ámbito hospitalario. Sin embargo, la magnitud de este problema no se ha establecido claramente debido a la limitada capacidad de detección de este tipo de resistencia de algunos de los sistemas manuales y automáticos utilizados para este fin (24,25). En este trabajo, para la detección de BLEE se obtuvieron idénticos resultados con el método de disco combinado recomendado por el NCCLS y con la prueba de E-test, por lo cual esta prueba podría ser una alternativa en los centros hospitalarios del país para los análisis confirmatorios de detección de BLEE. La detección microbiológica de BLEE por los análisis mencionados fue ratificada por isoelectroenfoque, el cual permitió detectar gran variedad de betalactamasas del tipo SHV y TEM; en la mayoría de los casos producida simultáneamente por la misma bacteria. La beta-lactamasa más frecuente fue la de pI 8,2, lo cual indica la posible diseminación en este centro hospitalario de la enzima BLEE- SHV-5 por transferencia horizontal de plásmidos conjugativos.
Con los bioensayos para determinar la actividad hidrolítica de enzimas separadas por isoelectroenfoque fue posible identificar beta-lactamasas que no fueron detectadas inicialmente, resultado que pudo deberse a la dificultad en la observación del cambio de color del nitrocefín cuando se hidroliza. El bioensayo permitió establecer que la actividad de BLEE en 14 aislamientos era exclusivamente de la betalacta-masa con pI 8,2. El aislamiento 10045 (CAZ s ) en un bioensayo con cefotaxima mostró actividad hidrolítica con pI superior a 8,2 (
cuadro 2); este resultado sugiere la producción de una cefotaximasa (CTX-M), caracterizada por una gran actividad hidrolítica frente a cefotaxima y poca o ninguna actividad frente a ceftazidima (26). Estos resultados permiten sugerir que el empleo simultáneo del gel de isoelectroenfoque y la prueba biológica mejora el nivel de detección y la especificidad de acción de alguna de las enzimas separadas electroforéticamente.Una beta-lactamasa con actividad hidrolítica sobre cefalosporinas de tercera generación (especialmente sobre ceftazidima) y pI 8,2 fue descrita inicialmente en aislamientos de Chile (23) y catalogada como BLEE-SHV-5 (2,27). En este estudio, en 14 de 15 aislamientos de K. pneumoniae se encontró BLEE tipo SHV-5 que también se hainformado con relativa frecuencia en México (4) y Argentina (28), lo cual sugiere que este tipo de BLEE codificada en plásmidos se ha diseminado en países de Latinoamérica.
La resistencia simultánea a diferentes clases de antibióticos (multirresistencia) es la consecuencia fenotípica de la diseminación de genes portados por elementos genéticos transferibles y de la selección de cepas por el uso inadecuado de antibióticos. En este estudio, muchas de las cepas productoras de BLEE presentaron resistencia simultánea a gentamicina, amikacina y cloranfenicol.
La resistencia simultánea a los aminoglucósidos y fluoroquinolonas se ha encontrado con frecuencia en enterobacterias; sin embargo, no debe tenerse como un marcador específico porque la resistencia a estos agentes se ha observado tanto en BLEE positivos como en BLEE negativos (23). Las quinolonas son potenciales opciones terapéuticas para el tratamiento de infecciones causadas por K. pneumoniae productoras de BLEE; sin embargo, la resistencia a estos antibióticos ha incrementado. Paterson et al. (29) describieron la epidemiología de la resistencia a ciprofloxacina en cepas de K. pneumoniae y su relación con la producción de BLEE y encontraron que 18% (15/83) de los aislamientos productores de BLEE eran resistentes a ciprofloxacina (29). En un estudio en Taiwán esta resistencia simultánea fue del 18,5% (39/21) (30). Aunque la resistencia simultánea de K. pneumoniae a ciprofloxacina (33,3%) encontrada en este estudio es similar para Estados Unidos (31), es un valor preocupante que supera lo encontrado en algunos países suramericanos (23,1%) y otras regiones del mundo según lo informado en estudios SENTRY (31). De igual manera, es preocupante la resistencia a cefepime y a otros antibióticos. Los resultados de este hospital podrían ser el reflejo de lo que ocurre en otros centros hospitalarios en Colombia de similar complejidad, por lo cual es conveniente iniciar un estudio amplio para conocer la prevalencia de BLEE en Enterobacteriaceae causantes de infección intrahospitalaria.
La multirresistencia de los microorganismos dificulta la efectividad de los tratamientos y genera limitaciones terapéuticas en muchas instituciones, pues cada día aumentan los niveles de resistencia a estos antibióticos. Aunque en este estudio no se pudo establecer causalidad entre la resistencia y la mortalidad, es importante considerar que la presencia de multirresistencia se puede asociar con el fracaso terapéutico.
La tipificación molecular realizada mediante REP-PCR mostró resultados concordantes con respecto a los encontrados con PFGE, considerado el estándar por excelencia para la tipificación bacteriana. Las agrupaciones observadas en el dendrograma de REP-PCR correspondieron a las mismas de PFGE, excepto en el caso de dos aislamientos que fueron agrupados por PFGE en el mismo patrón electroforético (P7) y separados por REP-PCR en los patrones R7 y R8. Estos aislamientos mostraron patrones de resistencia diferentes; por lo tanto, es posible que se trate de cepas con cromosomas muy parecidos que han adquirido elementos extracromosómicos que determinan patrones electroforéticos diferentes con el sistema REP-PCR. Esta discrepancia sugiere la necesidad de utilizar, por lo menos, dos marcadores diferentes para lograr establecer con mayor eficacia las posibles relaciones genéticas y epidemiológicas entre aislamientos. Los resultados de este trabajo indican la utilidad del ensayo de REP-PCR como una alternativa para tipificaciones rápidas. Aunque en algunos estudios es conveniente confirmar los resultados con procedimientos más discriminatorios.
En el presente estudio, la correlación de los datos clínicos y epidemiológicos con las características de la resistencia y la información molecular permitieron hacer inferencias que podrían ser importantes para tomar medidas en el control de la infección nosocomial por K. pneumoniae. El estudio de las características de la población permitió identificar tres patrones de infección.
1) infecciones de origen endógeno, las cuales se caracterizaron por la diversidad genética, probablemente asociada con la selección de organismos resistentes causada por la excesiva exposición a antibióticos y la estancia hospitalaria prolongada; 2) infecciones cruzadas, debido al hallazgo de cepas estrechamente relacionadas en espacios distantes del hospital, posiblemente transportados por personal hospitalario u objetos inertes relacionados con la atención del paciente; este caso se presentó en dos pacientes que coincidieron en tiempo en la UCI (agrupación P1/ R1), y 3) casos de recidiva infecciosa, corres-pondientes a la reaparición de manifestaciones clínicas causadas por una misma cepa bacteriana o por cepas pertenecientes al mismo grupo.
Ejemplo de este caso es la infección del paciente No. 8, quien inicialmente fue afectado por una cepa sensible a amikacina y a ciprofloxacina (aislamiento 257) y un mes después presentó infección por una cepa con resistencia a estos antibióticos (aislamiento 1114), aunque estrechamente relacionada con la cepa 257.
Probablemente, la persistencia de la cepa 257 en el ambiente hospitalario le permitió adquirir genes de resistencia, posiblemente transferidos a través de plásmidos.
En el presente trabajo cabe resaltar que la caracterización bioquímica de los mecanismos de resistencia y la tipificación molecular son herramientas útiles para estudios epidemiológicos y para conocer el comportamiento de las poblaciones bacterianas, de procesos específicos relacionados con la infección intrahospitalaria y las características de la resistencia a antibióticos en la institución, que permiten al comité de infecciones de las instituciones hospitalarias aplicar medidas tempranas y dirigidas a optimizar los esfuerzos en el control de infecciones y a evitar su progresión y propagación.
Agradecimientos
Agradecemos a la División de Investigaciones de Bogotá DIB de la Universidad Nacional de Colombia, al Posgrado Interfacultades de Microbiologìa de la Universidad Nacional de Colombia y a Colciencias por la financiación del proyecto (código No. 1101-04-12675). Al Hospital Universitario Clínica San Rafael; a la Unidad de Medicina Experimental, Universidad Nacional Autónoma de México; a Jesús Silva (Instituto Nacional de Salud Pública, México) y a Edgar Prieto (Facultad de Medicina, Universidad Nacional de Colombia) por la colaboración con este estudio.
Correspondencia:
José Ramón Mantilla, Laboratorio de Epidemiología Molecular Instituto de Biotecnología, Universidad Nacional de Colombia, Ciudad Universitaria, Bogotá, D.C., Colombia.
Teléfono: (571) 316 5000, extensión 16965
Recibido: 26/03/04; aceptado: 080/07/04
Referencias
1. Podschun R, Ullmann U. Klebsiella spp. as nosocomial pathogens: epidemiology, taxonomy, typing methods, and pathogenicity factors. Clin Microbiol Rev 1998;11: 589-603. [ Links ]
2. Bradford P. Extended-spectrum ß-lactamases in the 21 st century: characterization, epidemiology and detection of this important resistance threat. Clin Microbiol Rev 2001;14:933-51. [ Links ]
3. Li L, Lim C. A novel large plasmid carrying multiple ß-lactam resistance genes isolated from a Klebsiella pneumoniae strain. J Applied Microbiol 2000;88:1038-48. [ Links ]
4. Silva J, Gatica R, Aguilar C, Becerra Z, Garza U, Velásquez M et al. Outbreak of infection with extended spectrum ß-lactamase-producing Klebsiella pneumoniae in a Mexican hospital. J Clin Microbiol 2001; 39:3193-6. [ Links ]
5. Bingen E, Desjardins P, Arlet G, Bourgeois F, Kurkdjlan P, Lambert N et al. Molecular epidemiology of plasmid spread among extended broad-spectrum ß-lactamase-producing Klebsiella pneumoniae isolates in a pediatric hospital. J Clin Microbiol 1993;3:179-84. [ Links ]
6. Paterson D, Ko W, Gottberg A, Casellas J, Mulazimoglu L, Klugman K et al.Outcome of cephalosporin treatment for serious infections due to apparently susceptible organisms producing extended-spectrum ß-lactamases: implications for the clinical microbiology laboratory. J Clin Microbiol 2001;39:2206- 12. [ Links ]
7. Robledo C, Robledo J.Panorama de la resistencia a los antibióticos en Colombia. Rev Panam Infect 1999; (Suppl.1):26-32. [ Links ]
8. Crespo M, Vélez J, Castañeda C.Patrones de sensibilidad de las enterobacterias y P. aeruginosa causantes de bacteremia: un estudio de 4 años. Infectio 2001;5:116. [ Links ]
9. Malathum K, Singh K, Weinstock G, Murray B.Repetitive sequence-based PCR versus pulsed field gel electrophoresis for typing of Enterococcus faecalis at the subspecies level. J Clin Microbiol 1998;36:211-5. [ Links ]
10. Coenye T, Spilker T, Martin A, LiPuma J. Comparative assessment of genotyping methods for epidemiologic study of Burkholderia cepacia genovar III. J Clin Microbiol 2002;40:3300-7. [ Links ]
11. Versalovic J, Koeuth T, Lupski J. Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes. Nuc Ac Res 1991; 19:6823-31. [ Links ]
12. Miranda G, Kelly C, Solórzano F, Leanos B, CoriaR, Patterson JE. Use of pulsed field gel electrophoresis typing to study an outbreak of infection due to Serratia marcescens in a neonatal intensive care unit. J Clin Microbiol 1996;34:3138-41. [ Links ]
13. Tenover F, Arbeit R, Goering R, Mickelsen P, Murray B, Persing D et al. Interpreting chromosomal DNA restriction patterns by pulsed-field gel electrophoresis: criteria for bacterial strain typing. J Clin Microbiol 1995; 33:2233-9. [ Links ]
14. National Committee for Clinical Laboratory Standards. Performance standards for antinmicrobial susceptibility testing. Eleven informational supplement. NCCLS approved standard M100-S11. Wayne, PA: National Committee for Clinical Laboratory Standards; 2002. [ Links ]
15. Matthew M, Harris A, Marshall H, Ross G. The use of analytic isoelectric focusing for detection and identification of ß-lactamases. J Gen Microbiol 1975;88:169-78. [ Links ]
16. Silva J, Aguilar C. ß-lactamase bioassay: a simplified method to determine extended-spectrum ß-lactamases (ESBL) in enterobacteria. Arch Med Res 1997;28:285-7. [ Links ]
17. Sirot D, De Champs C, Chanal C, Labia R, Darfeville-Michaund A, Perroux P et al. Translocation of antibiotic resistance determinants including an extended-spectrum ß-lactamase between conjugative plasmids of Klebsiella pneumoniae and Escherichia coli. Antimicrob Agents Chemother 1991;35:1576-81. [ Links ]
18. Sambrook J, Fritsch EM, Maniatis T. Molecular cloning: a laboratory manual. Second edition. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press; 1989. [ Links ]
19. Siu L, Hsueh P, Lin F, Chang S, Luh K, Ho M, Lee C. Bacteremia due to extended-spectrum ß-lactamase-producing Escherichia coli and Klebsiella pneumoniae in a pediatric oncology ward: clinical features and identification of different plasmids carrying both SHV-5 and TEM-1 genes. J Clin Microbiol 1999;37:4020-7. [ Links ]
20. Centers for Disease Control and Prevention. Hospital infection program. National nosocomial infections surveillance (NNIS) report, data summary from october 1986-april 1996, issued may 1996: a report from the NNIS system. Am J Infect Control 1996;24: 380-8. [ Links ]
21. Bush K, Jacoby JA. Amino acid sequences for TEM, SHV and OXA extended spectrum and inhibitor resistant ß-lactamases. Published by the Lahey Clinic. Disponible en:
http://www.lahey.org/studies/webt.htm. [ Links ]22. Livermore D. ß-lactamases in laboratory and clinical resistance. Clin Microbiol Rev 1995;8:557-84. [ Links ]
23. Essack S, Hall L, Pillay D, McFadyen M, Livermore D. Complexity and diversity of Klebsiella pneumoniae strains with extended-spectrum ß-lactamases isolated in 1994 and 1996 at a teaching hospital in Durban, South Africa. Antimicrob Agents Chemother 2001;45:88-95. [ Links ]
24. Cormican M, Marshall S, Jones R. Detection of extended-spectrum ß-lactamase (ESBL)-producing strains by the E-test ESBL screen. J Clin Microbiol 1996; 34:1880-4. [ Links ]
25. Carter M, Oakton K, Warner M, Livermore D. Detection of extended-spectrum ß-lactamases in Klebsiellae with the Oxoid combination disk method. J Clin Microbiol 2000;38:4228-32. [ Links ]
26. Dutour C, Marchandin H, Boyer M, Chanal C, Sirot D, Sirot J. CTX-M-1, CTX-M-3, and CTX-M-14 ß-lactamases from Enterobacteriaceae Isolated in France. Antimicrob Agents Chemother 2002;46:534-7. [ Links ]
27. Gutmann L, Ferre B, Goldstein F, Rizk N, Pinto-Schuster E, Acar J, et al. SHV-5, a novel SHV-type ß-lactamase that hydrolyzes broad-spectrum cephalosporins and monobactams. Antimicrob Agents Chemother 1989;33:951-6. [ Links ]
28. Quinteros M, Radice M, Gardella N, Rodriguez MM, Costa N, Korbenfeld D et al. Extended-spectrum ß-lactamases in Enterobacteriaceae in Buenos Aires, Argentina, public hospitals. Antimicrob Agents Chemother 2003;47:2864-7. [ Links ]
29. Paterson D, Mulazimoglu L, Casellas JM, Ko W, Goossens H, Gottberg A et al. Epidemiology of ciprofloxacin resistance and its relationship to extended-spectrum ß-lactamase production in Klebsiella pneumoniae isolates causing bacteremia. Clin Infect Dis 2002;30:473-8. [ Links ]
30. Yu W, Jones R, Hollis R, Messer S, Biedenbach D, Deshpande L, et al. Molecular epidemiology of extended-spectrum ß-lactamase-producing, fluoro-quinolone- resistant isolates of Klebsiella pneumoniae in Taiwan. J Clin Microbiol 2002;40:4666-9. [ Links ]
31. Winokur PL, Canton R, Casellas JM, Legakis N.Variations in the prevalence of strains expressing extended-spectrum ß-Lactamases phenotype and characterization of isolates from Europe, the Americas and the Western Pacific region. Clin Infec Dis 2001;32: 94-103. [ Links ]














