Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Antropología
Print version ISSN 0486-6525
Rev. colomb. antropol. vol.48 no.1 Bogotá Jan./June 2012
COMPOSICIÓN GENÉTICA DE UNA POBLACIÓN DEL SUROCCIDENTE DE COLOMBIA
GENETIC STRUCTURE OF A SOUTHWESTERN COLOMBIAN POPULATION
LILIANA CÓRDOBAA,
JHARLEY JAIR GARCÍAA, LUZ STELLA HOYOSB, CONSTANZA DUQUEA,
WINSTON ROJASA, SILVIO CARVAJALB, LUISA F. ESCOBARB,C, INGRID REYESB,
NOHELIA CAJASB, ADALBERTO SÁNCHEZD, FELIPE GARCÍAD, GABRIEL BEDOYAA,
ANDRÉS RUIZ-LINARESA,E
AGRUPO DE GENÉTICA MOLECULAR, INSTITUTO DE BIOLOGÍA, UNIVERSIDAD DE ANTIOQUIA, MEDELLÍN, COLOMBIA. (lilianac27@yahoo.com)
BGRUPO DE INVESTIGACIÓN EN TOXICOLOGÍA GENÉTICA Y CITOGENÉTICA, DEPARTAMENTO DE BIOLOGÍA, UNIVERSIDAD DEL CAUCA, POPAYÁN, COLOMBIA.
CDEPARTMENT OF PHARMACOLOGICAL SCIENCES AND PATHOLOGY, STONY BROOK UNIVERSITY, STONY BROOK, NUEVA YORK, ESTADOS UNIDOS.
DDEPARTAMENTO DE CIENCIAS FISIOLÓGICAS, FACULTAD DE SALUD, UNIVERSIDAD DEL VALLE, CALI, COLOMBIA.
EDEPARTAMENT OF GENETICS, EVOLUTION AND ENVIRONMENT, UNIVERSITY COLLEGE LONDON, LONDRES, REINO UNIDO.
Recibido: 29 de agosto de 2011 Aprobado: 10 de octubre de 2011
Resumen
La población actual del departamento del Cauca es el resultado de la mezcla de tres poblaciones parentales (europea, amerindia y africana). En este estudio se determinó la composición genética de 306 residentes del departamento mediante la utilización de 34 variantes autosómicas, 9 variantes en el cromosoma X, 6 en el ADNmt y 8 en el cromosoma Y. Los análisis de las variantes autosómicas y del cromosoma X revelaron que la población europea y la amerindia han contribuido en mayor proporción al actual acervo genético de la población estudiada. Los resultados de las variantes en el ADNmt y del cromosoma Y sugieren un fuerte sesgo sexual (flujo génico asimétrico) en el proceso de mezcla, en el que los cruces interétnicos fueron principalmente entre los colonizadores europeos y las mujeres nativas.
Palabras clave: departamento del Cauca, mezcla, variantes genéticas.
Abstract
The contemporary population of the Province of Cauca is the result of the admixture between three parental populations (European, Amerindian and African). Genetic composition was determined in 306 residents of the Province with the use of 34 autosomal markers, 9 markers in chromosome X, 6 and 8 mtDNA in chromosome Y. Autosomal and X chromosome data indicate that both European and Amerindian populations have contributed to a greater extent to the current genetic pool of the population. The mtDNA and the Y chromosome data, indicate a strong sexual bias (asymmetrical gene flow) in the admixing process, where interethnic crosses were mainly between European men and native women.
Key words: Province of Cauca, admixture,genetic markers.
INTRODUCCIÓN1
La ciudad de Popayán fue fundada en 1537 por el español Sebastián de Belalcázar; posteriormente, en 1540, se conformó la gobernación de Popayán, que comprendía los actuales departamentos de Cauca, Valle del Cauca, Nariño, parte de Chocó y parte de Antioquia. El establecimiento del dominio hispánico en la gobernación, en las primeras décadas del siglo XVI, tuvo grandes repercusiones demográficas. La conquista en el altiplano, aunque violenta, no fue tan destructiva como en el norte (departamento del Valle); los indígenas lograron sobrevivir en mayor número, se ubicaron en los "pueblos de indios" y fueron los principales proveedores de productos alimenticios de los hacendados españoles (Díaz 1983, 1994).
En el siglo XVIII el incremento de la minería estimuló el crecimiento de la esclavitud (Díaz 1983). Así, en 1716 llegaron a Popayán los primeros esclavos de diferentes regiones de África, principalmente de la costa occidental y regiones vecinas (Senegal, Gambia, Sierra Leona, Costa de Marfil, Costa de Oro, Berrión, Golfo de Biafra, Angola y Mozambique) (Díaz 1994). Según las estadísticas, del total de esclavos desembarcados en Cartagena, el 15% llegaron a Popayán (Castellanos 1980).
Para finales del siglo XVIII la composición étnica de la población de la ciudad de Popayán era en un 20,5% blanca, en un 27,7% indígena, el 32,7% eran libres (negros libres, mestizos y mulatos) y eran esclavos el 18,7%. A comienzos del siglo XX los blancos constituían el 25,3%, los indígenas el 34%, los mestizos el 20,4% y el 19,8% eran negros (Díaz 1983, 1994). El departamento del Cauca, según el censo realizado en 2005 por el Departamento Administrativo Nacional de Estadística (DANE), cuenta con 1.182.022 habitantes de gran diversidad étnica. El 56,25% de su población es blanca y mestiza, y está ubicada principalmente en el centro del departamento. En la zona de Tierradentro, hacia el oriente, están las comunidades indígenas que constituyen el 21,55% de la población, de las etnias yanacona, inga, coconuco, totoró, paez, guambiana, eperara-siapidara y embera, que lo hacen uno de los departamentos con más comunidades indígenas del país. Por otro lado, en la costa del Pacífico viven los afrodescendientes que conforman el 22,2% de la población (Colombia, DANE 2005).
La composición genética de la población estudiada ha sido estimada principalmente con el uso de variantes en el ADN mitocondrial (Salas et ál. 2008). La importante información filogeográfica de las variantes presente en el ADN mitocondrial (ADNmt), nuclear y del cromosoma Y ha permitido la reconstrucción de los eventos demográficos y evolutivos pasados, como las migraciones humanas y los procesos de mezcla (Mendizábal et ál. 2008). Las variaciones en el ADNmt y el cromosoma Y muestran una fuerte estructura filogeográfica, al punto que todos los haplogrupos están confinados a un único continente y pueden ser usados para trazar las migraciones fuera de este (Jobling et ál. 2004).
La estimación de la composición genética de las poblaciones resultantes de mezclas ha sido importante en los análisis genéticos de muchas poblaciones humanas (Chakraborty y Weiss 1986); tales estimativos son útiles tanto en las investigaciones de epidemiología genética como en los estudios de asociación genética (Chakraborty y Weiss 1988; Long 1991), en los que la existencia de subgrupos o de una subestructura poblacional puede llevar a falsas asociaciones en un rasgo (Choudhry et ál. 2006; Hoggart et ál. 2003; Li 1969).
Para estimar la composición genética individual se dispone de polimorfismos en los autosomas y en el cromosoma X que exhiben diferencias en las frecuencias alélicas entre poblaciones parentales, polimorfismos que son denominados marcadores informativos de ascendencia (ancestry informative markers, [AIM]) (Bonilla et ál. 2004; Parra et ál. 1998). Estos polimorfismos son considerados una herramienta eficiente para obtener estimativos de mezcla precisos y detectar estructuras poblacionales (Choudhry et ál. 2006; Pfaff et ál. 2001).
El objetivo principal del presente estudio fue evaluar la composición genética a través de variantes en el ADN gonosómico, autosómico y mitocondrial en una muestra de la población del departamento del Cauca, así como su relación con otras poblaciones latinoamericanas.
MATERIALES Y MÉTODOS
Muestra de estudio
Se analizaron 306 muestras de hombres voluntarios residentes en los municipios de Popayán, Santander de Quilichao, Piendamó, Timbío, Puerto Tejada, El Tambo y El Bordo (departamento del Cauca) y originarios de diferentes zonas del departamento. El ADN se extrajo de sangre periférica por el método de salting-out en el laboratorio de Genética Toxicológica y Citogenética de la Universidad del Cauca, previa firma de consentimiento informado (con la aprobación del Comité de Ética de la Universidad del Cauca). La concentración y calidad de las muestras fueron determinadas utilizando un NanoDrop2000.
Genotipificación
LINAJES MATERNOS: ADN MITOCONDRIAL
Cada muestra fue genotipificada para las mutaciones en la región codificante usando polimorfismos de longitud de fragmentos de restricción PCR-RFLP y PCR-AFLP2, cinco polimorfismos nucleotídicos simples (SNP) y una inserción/ deleción (In/Del), de acuerdo con las condiciones experimentales descritas previamente (Bailliet et ál. 1994; Hertzberg et ál. 1989; Macaulay et ál. 1999). Estas variantes discriminan seis haplogrupos mitocondriales; los principales haplogrupos amerindios se definieron como A (+HaeIII 663), B (deleción de nueve pares de bases en la región intergénica COII/tRNAlys), C (−HincII 13259) y D (−Alu 5176). Además, se determinaron el paragrupo africano L (+HpaI 3592) y el haplogrupo europeo H (−AluI 7025).
LINAJES PATERNOS: CROMOSOMA Y
Nueve variantes bialélicas y seis repeticiones cortas en tándem (short tandem repeat [STR]) fueron genotipificadas. Las variantes bialélicas definen algunos de los haplogrupos de acuerdo con las recomendaciones del consorcio del cromosoma Y (Karafet et ál. 2008), y se resolvieron por PCR-RFLP. En resumen, el clado DE*(xE1b1a) se define por el estado derivado en YAP y ancestral en M2; el estado derivado en M2 es diagnóstico para definir el clado E1b1a; el paragrupo F* se define por el estado ancestral en M9; el clado A se define por los estados ancestral en M2 y derivado en P97; mientras que el estado ancestral en P97 define al clado B. Los estados ancestral en M9 y derivado en 12f2a son característicos para el clado J; los estados derivado y ancestral en M9 y 92R7 respectivamente son diagnósticos para el paragrupo K*; los estados derivado en 92R7 y ancestral en SRY-2627 son propios del clado P*; el estado derivado en SRY-2627 es diagnóstico para R1b1b2d; el estado derivado en M242 define el haplogrupo Q*. Para globalizar, se consideró que la frecuencia del haplogrupo E1b1a representa la contribución africana, el haplogrupo Q, la contribución amerindia y los restantes haplogrupos y paragrupos hallados, la contribución europea.
Los STR DYS19, DYS388, DYS390, DYS391, DYS392 y DYS393 fueron genotipificados de acuerdo con lo descrito por Thomas et ál. (1999). Se analizaron por electroforesis capilar utilizando un analizador genético ABI-310®, y la determinación de genotipos se realizó con el programa GeneMapper versión 4.0 (AppliedBiosystems).
VARIANTES INFORMATIVAS DE COMPOSICIÓN GENÉTICA AUTOSÓMICA
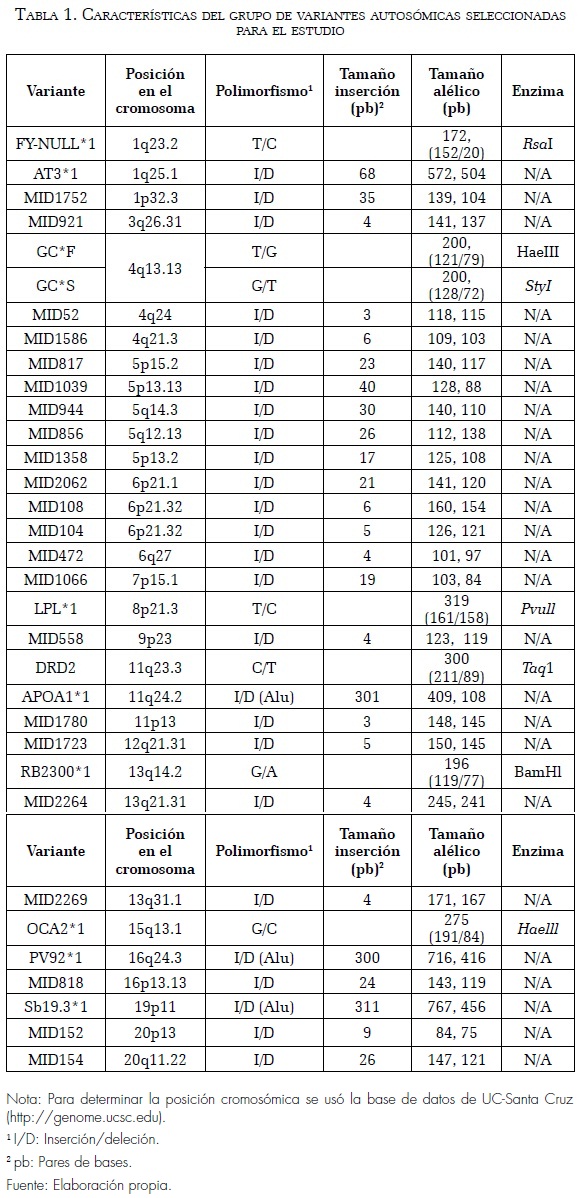
Se seleccionaron 34 variantes, incluidas SNP e In/Del, con base en la diferencia de las frecuencias alélicas (denotada por δ , donde δ = frecuencia del alelo A en la población 1 – frecuencia de A en la población 2) entre las subpoblaciones parentales europea, africana y amerindia, de acuerdo con la bibliografía (Carvajal- Carmona et ál. 2000; Carvajal-Carmona et ál. 2003; Cintado et ál. 2009; Parra et ál. 1998; Vergara et ál. 2009), incluidas bases de datos en línea (http://www.marshfieldclinic.org/mgs/). Los SNP fueron analizados por PCR-RFLP, y se resolvieron los fragmentos de digestión en geles de agarosa al 2,5% teñido con bromuro de etidio. Para identificar los In/Del, los segmentos amplificados se analizaron directamente en geles de agarosa o de poliacrilamida teñidos con bromuro de etidio, o por electroforesis capilar en un analizador genético ABI-310®, en cuyo caso la determinación de los genotipos se realizó con el programa GeneMapper v. 4.0. La tabla 1 muestra las variantes utilizadas en el análisis, con los cebadores y enzimas de restricción necesarias para su caracterización. Una variante se consideró lo suficientemente informativa si la diferencia en las frecuencias alélicas era mayor a 0,30 entre cualquiera de los grupos parentales.
VARIANTES DE COMPOSICIÓN GENÉTICA EN EL CROMOSOMA X
Se tipificaron nueve variantes, según las condiciones descritas en Hoggart et ál. (2003), por electroforesis en geles de agarosa al 2%, teñidos con bromuro de etidio o por electroforesis capilar utilizando un analizador genético ABI-310 (AppliedBiosystems). Además, la asignación de genotipos se realizó con el programa GeneMapper v. 4.0. En la tabla 2 se muestran las características de las variantes tipificadas.
ANÁLISIS ESTADÍSTICOS
Las frecuencias de cada haplogrupo mitocondrial y del cromosoma Y, junto con la inferencia de haplotipos y sus frecuencias, se calcularon utilizando el programa Arlequin v. 3.11 (Excoffier, Laval y Schneider 2005). La diversidad génica para cada locus se calculó así: D = (1- Σpi2), donde pi equivale a las frecuencias alélicas.
Las relaciones filogenéticas entre los haplotipos dentro de los haplogrupos y entre los haplogrupos se determinaron utilizando el algoritmo median joining (MJ) propuesto por Bandelt, Forster y Rohl (1999), implementado en el programa Network v. 4.5.16. Además, se realizó la búsqueda del origen geográfico de los haplotipos de mayor frecuencia usando la base de datos de referencia de haplotipos del cromosoma Y (Y Chromosome Haplotype Reference Database, YHRD) en http://www.yhrd.org.
Para las variantes autosómicas y del cromosoma X se calcularon las frecuencias genotípicas y alélicas y se realizó la prueba de equilibrio de Hardy-Weinberg con el programa Arlequin v. 3.11 (Excoffier, Laval y Schneider 2005); para obtener la significancia del análisis controlado por el número de pruebas se realizó una prueba de Bonferroni (Bland y Altman 1995). Se estimaron los porcentajes de mezcla grupal e individual, y el número promedio de generaciones desde la finalización del evento de mezcla (τ -suma de intensidades), mediante un método híbrido que combina la estadística clásica y bayesiana, implementado en el programa Admixmap v. 3.2 (Hoggart et ál. 2004).
RESULTADOS
Linajes maternos: ADNmt
El haplogrupo C fue el de mayor frecuencia en la población (0,3), seguido por A (0,28), B (0,21) y D (0,1), y en menor frecuencia por los haplogrupos de origen no amerindio, africano (0,0695) y europeo (0,0065). Un 0,034 de las muestras, de probable origen africano o europeo, no pudieron ser asignadas a los clados mayoritarios aquí analizados y se denominaron como otros. Así, el 89% de la muestra tiene ascendencia de origen amerindio; el 6,95%, descendencia africana y la europea constituye el 0,065% de la muestra.
Linajes paternos: cromosoma Y
Para la población en general se observó que el haplogrupo de mayor frecuencia fue el P* (0,39), de origen europeo; seguido del haplogrupo amerindio Q (0,17); DE(xE1b1a) con una frecuencia de 0,108; J, con una frecuencia de 0,065; los paragrupos F*, con 0,104, y K*, con 0,069; y los clados E1b1a, con 0,055, y R1b1b2d, con 0,039. En total, la contribución paterna de origen europeo fue de 77,5%, mientras que la amerindia fue de 17% y la africana, de 5,5%.
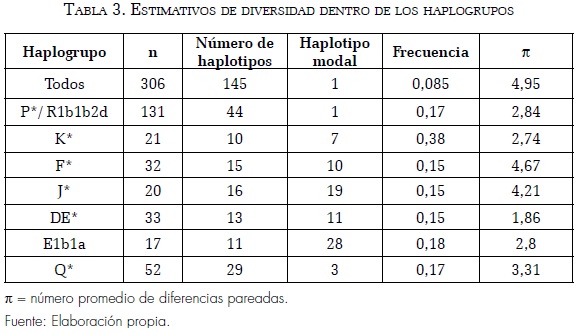
Se determinaron 145 haplotipos en los 306 cromosomas Y analizados (tabla anexa 1), de los cuales el más frecuente fue el haplotipo 1 (14/12/24/11/13/13). En la tabla 3 se muestran el número de haplotipos por haplogrupo, los haplotipos modales, sus frecuencias y el número promedio de diferencias por pares. Se observa que los clados menos diversos son DE*, K* y P*. Se destaca la poca diversidad de este último haplogrupo, a pesar de ser el más frecuente en la población.
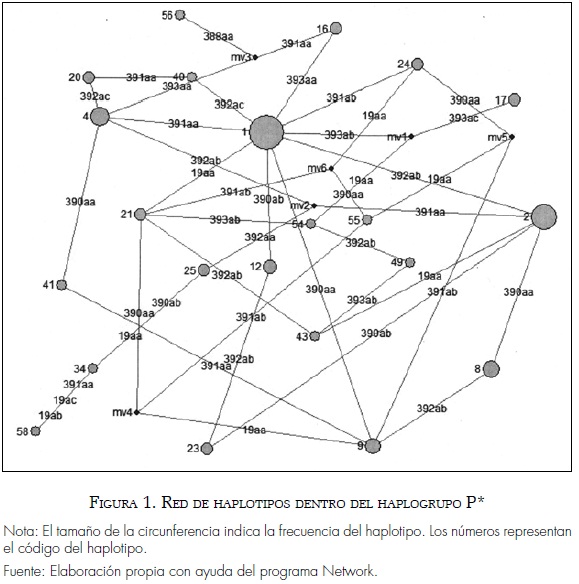
Para cada haplogrupo se construyeron redes utilizando el algoritmo median joining con los haplotipos de los microsatélites, en las que solo se incluyeron los haplotipos compartidos por dos individuos o más (figura 2 y figuras anexas 1, 2 y 3). Por ejemplo, en el caso del haplogrupo P*, el haplotipo 1 es el modal; por esto se considera el ancestral y de este se derivan la mayoría de los haplotipos (2, 4, 9, 12, 16, 21, 24 y 40) que se encuentran a un paso mutacional (figura 1).
Variantes informativas de la composición genética en el cromosoma X
Las frecuencias alélicas de cada una de las variantes analizadas se muestran en la tabla 4. Se determinó que la contribución promedio del componente europeo fue de 57,3%, la de la fracción amerindia fue 32%, mientras que el componente africano aparece en menor frecuencia con 10,7%. En cuanto a la mezcla individual, se observa que el componente africano es muy homogéneo en todos los individuos, con mayor variación en los aportes amerindio y europeo. Es de resaltar que todos los individuos tienen más del 40% de ascendencia amerindia y que, además, el 16,3% de ellos tienen más del 70%.
Variantes informativas de composición genética autosómica
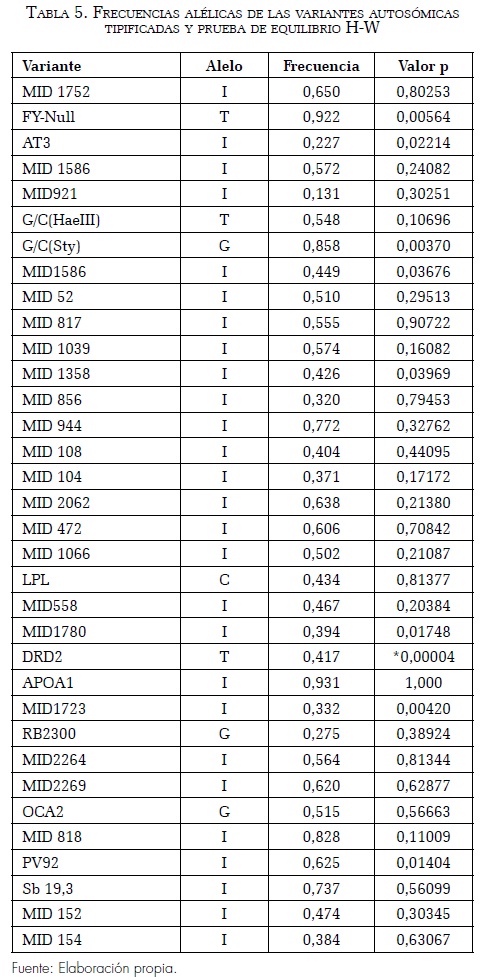
Las frecuencias alélicas y los valores p de la prueba de equilibrio de Hardy-Weinberg se muestran en la tabla 5. Los resultados de la mezcla genética promedio muestran que la contribución europea en la muestra fue del 0,48 (± 0,033); la amerindia, de 0,411 (± 0,03) y la africana, de 0,107 (± 0,0169). En los estimativos de la mezcla individual se observan rangos muy amplios de distribución: en el componente africano, por ejemplo, se observa una variación de 0,015 hasta 0,6; en el componente amerindio, de 0,14 a 0,72 y en el europeo, de 0,24 a 0,75.
DISCUSIÓN
Teniendo en cuenta los datos del ADNmt y del cromosoma Y se puede inferir que el actual acervo genético de la muestra de la población del Cauca está compuesto por la mezcla de tres poblaciones parentales, europea, amerindia y africana, patrón similar al encontrado en otras poblaciones mestizas de Colombia (Carvajal-Carmona et ál. 2003; Carvajal-Carmona et ál. 2000; Rojas et ál. 2010) y en otros países de Latinoamérica (Martínez- Marignac et ál. 2007; Martínez et ál. 2007).
La composición de linajes del ADNmt y del cromosoma Y encontrada en este estudio amplía esa idea, ya que se hallaron linajes amerindios, africanos y europeos (incluido el Medio Oriente). Sin embargo, la distribución de los linajes en ambos sistemas muestra que esas poblaciones ancestrales no contribuyeron de igual manera al proceso de mezcla, debido a que hasta un 72,8% de los cromosomas Y son de origen europeo, pero solo un 0,65% del ADN mitocondrial es del mismo origen. En el componente amerindio se observa un aporte de los hombres del 17%, frente a un 89% de contribución materna; y en los africanos se observa el fenómeno contrario, es decir un mayor aporte de los hombres.
Todos estos resultados sugieren un fuerte sesgo sexual (flujo génico asimétrico) en el proceso de mezcla de la población estudiada, en el que los cruces interétnicos fueron principalmente entre los colonizadores europeos y las mujeres nativas. Una mezcla sesgada de hombres europeos de esta clase ha sido reportada previamente en otras poblaciones colombianas (Carvajal-Carmona et ál. 2003; Carvajal-Carmona et ál. 2000; Rojas et ál. 2010) y en otros países de Latinoamérica y del Caribe (Alves-Silva et ál. 2000; Carvalho-Silva et ál. 2001; Mendizábal et ál. 2008).
En la población estudiada el haplogrupo mitocondrial de mayor frecuencia fue el C (0,3), que se ha encontrado en gran frecuencia en las poblaciones amerindias de Colombia, ubicadas al sur del país (guambianos, coreguajes, inganos y ticunas) (Keyeux et ál. 2002). El haplogrupo A fue el segundo más frecuente (0,28) en la muestra de la población caucana, y es el más frecuente en las comunidades indígenas de la zona occidental de la cordillera de los Andes y en el norte del país (Keyeux et ál. 2002), y también es el más frecuente en las poblaciones mestizas de Colombia (Rojas et ál. 2010).
En cuanto a la contribución paterna de la muestra de la población del Cauca, se determinó el gran aporte de origen europeo (0,728); particularmente, los haplogrupos más frecuentes son el P*, encontrado en un 39%, y el R1b1b2d, en un 3,9% de los cromosomas de la población observada. El haplogrupo P* es más frecuente en el occidente de Europa, con un gradiente en el que la frecuencia disminuye hacia el oriente de Europa (Semino et ál. 1996). El haplotipo 1 (14/12/24/11/13/13) es el más común en las poblaciones del occidente de Europa (Brion et ál. 2003; Pérez-Lezaun et ál. 1997). Este haplotipo también es el más frecuente entre las poblaciones mestizas de Colombia (Rojas et ál. 2010). Tanto la red tipo estrella (figura 1) como la distribución unimodal de las diferencias por pares (figura anexa 1) son características de poblaciones de crecimiento rápido a partir de un haplotipo ancestral (modal).
El haplogrupo J (6,5%), en Europa, exhibe un gradiente en el que la frecuencia disminuye del suroriente hacia el noroccidente (Semino et ál. 1996). El mismo haplogrupo se ha reportado en altas frecuencias en el Medio Oriente, el centro y occidente del Mediterráneo y el norte de África (Di Giacomo et ál. 2004; Semino et ál. 2004). Dentro del haplogrupo se encontró el haplotipo modal Cohen (cmh) (haplotipo 19), que es el principal haplotipo de algunas poblaciones del Medio Oriente (Thomas et ál. 1998), particularmente de los judíos asquenazis y sefardíes derivados de la casta Cohen (Di Giacomo et ál. 2004; Thomas et ál. 1998; Thomas et ál. 2000). La diversidad relativa encontrada en este haplogrupo es alta y la ausencia de un haplotipo modal en la red puede indicar que estos cromosomas fueron introducidos a la población por varios fundadores y/o en migraciones recientes (figura anexa 2).
Dentro del haplogrupo E1b1a es de resaltar la alta frecuencia de la repetición 12 en el locus DYS388 (0,96), típica de los grupos bantú-hablantes (Thomas et ál. 2000), así como los alelos 15 y 21 en los locus DYS19 y DYS390, típicos de las poblaciones del África subsahariana (Willuweit y Roewer 2007). La frecuencia de estos alelos es similar a la reportada por Rojas et ál. (2010) respecto de la población afrodescendiente del palenque de San Basilio. Además, la búsqueda del origen geográfico de los haplotipos indica que todos se encuentran reportados en el África subsahariana, en las poblaciones de Camerún, Guinea Bissau y Angola, entre otras (Willuweit y Roewer 2007).
La frecuencia del haplogrupo Q en el Cauca es similar a la reportada en la población mestiza de Nariño (21%), y es similar también al promedio de las poblaciones mestizas de Colombia (16%) (Rojas et ál. 2010). Los haplotipos más frecuentes se corresponden con los más frecuentes en las poblaciones amerindias (Bortolini et ál. 2003; Lell et ál. 2002; Ruiz-Linares et ál. 1999). El haplotipo 3 (13/12/24/10/14/13) y los haplotipos a un paso mutacional conforman el 46% del total de los haplotipos y la distribución de las diferencias por pares (figura anexa 3), y se constata que son característicos de poblaciones en expansión.
Al estimar los componentes de mezcla promedio con las variantes en el cromosoma X, el componente amerindio se encuentra en un 57,3%, cifra menor a la hallada en las poblaciones mestizas de Colombia (Rojas et ál. 2010). La mezcla europea se encontró en un 32%, cifra similar a la reportada para algunas poblaciones colombianas (p. e., Valle del Cauca y Norte de Santander). Por último, el componente africano del 10% aquí registrado es similar al encontrado en población antioqueña (Rojas et ál. 2010). Como las mujeres aportan dos tercios de los cromosomas X a una población, se espera que las variantes en este cromosoma tengan mayor contribución amerindia que la encontrada en los autosomas. Al comparar los componentes promedio de la población del Cauca con otras poblaciones mestizas de Colombia, se observa que para este sistema esta población se asemeja más a las poblaciones de afro-Chocó y Antioquia (Rojas et ál. 2010).
Para estimar la mezcla genética autosómica se utilizó un panel de 34 variantes con δ ≥ 0,30, valor recomendado para realizar estimativos confiables a partir de ellas (Bonilla et ál. 2004; Martínez-Marignac et ál. 2007). Con estos polimorfismos se determinó que la muestra de estudio tiene actualmente un 48,2% de aporte europeo, un 41,1% de amerindio y un 10,7% de africano.
El parámetro de suma de intensidades para la población se estimó en 10,9 (IC 95%: 6,63-18,6), alrededor de 11 generaciones; asumiendo que una generación corresponde a 25 años, el final de la mezcla ocurrió hace 275 años (en la primera mitad del siglo XVIII). Este parámetro se ha estimado en algunas poblaciones de América Latina y el Caribe: en Cartagena se calculó en 7,2 generaciones (95% IC: 5,9-8,76) (Vergara et ál. 2009); en la ciudad de México se estimó en 6,7 generaciones (IC 95%: 5,7-8,0) (Martínez-Marignac et ál. 2007); y de manera similar en los cubanos este parámetro se estimó en 6,1 generaciones (IC 95% 1,24-14,7) (Díaz-Horta et ál. 2010). Contrastan estos resultados con los de la población de Trinidad y Tobago, con un promedio de 2,4 generaciones desde que finalizó la mezcla (IC 95% de 0,7-7,4) (Molokhia et ál. 2003).
CONCLUSIONES
1. Los linajes maternos, en un gran porcentaje, son de origen amerindio (89%), y se desataca el haplogrupo C como el más frecuente en la población (0,3).
2. Los linajes paternos europeos fueron los predominantes, seguidos por los nativos americanos y en más baja frecuencia, los africanos.
3. El haplogrupo P*, a pesar de ser el más frecuente, es poco diverso, lo cual indica que fueron menos los individuos fundadores y que de estos pocos individuos se derivó el resto de la población, al contrario de lo encontrado en los otros haplogrupos de menor frecuencia.
4. El patrón de mezcla de la población se ajusta con lo reportado para las otras poblaciones de América Latina, en las que los cruces fueron principalmente entre hombres europeos y mujeres amerindias.
AGRADECIMIENTOS
Los autores agradecen a las instituciones Universidad de Antioquia, Universidad del Cauca y Universidad del Valle. Al Departamento Administrativo de Ciencia, Tecnología e Innovacion (Colciencias) (proyecto "Efecto de la mezcla ancestral y los polimorfismos en genes del metabolismo y reparación en el riesgo de cáncer por exposición ocupacional a solventes orgánicos", código 459/1103459214-29-2008) y al Comité para el Desarrollo de la Investigación (CODI) de la Universidad de Antioquia (estrategia de sostenibilidad 2009-2011) por la financiación. Finalmente, a los evaluadores y editores de la Revista Colombiana de Antropología por sus correcciones y sugerencias.
Notas
1 Este artículo es producto de una tesis de maestría, basada en un trabajo de investigación.
2 PCR: polymerase chain reaction; RFLP: restriction fragment length polymorphim; AFLP: amplified fragment length polymorphism.
REFERENCIAS
1. ALVES-SILVA, JULIANA, MAGDA DA SILVA SANTOS, PEDRO GUIMARAES, ALESSANDRO FERREIRA Y HANS BANDELT. 2000. "The Ancestry of Brazilian mtDNA lineages". American Journal of Human Genetics 67 (2): 444-461. [ Links ]
2. BAILLIET, GRACIELA, FRANCISCO ROTHHAMMER, FRANCISCO CARNESE, CLAUDIO BRAVI Y NÉSTOR BIANCHI. 1994. "Founder Mitochondrial Haplotypes in Amerindian Populations". American Journal of Human Genetics 55 (1): 27-33. [ Links ]
3. BANDELT, HANS, PETER FORSTER Y ARNE ROHL. 1999. "Median-joining Networks for Inferring Intraspecific Phylogenies". Molecular Biology Evolution 16 (1): 37-48. [ Links ]
4. BLAND, J. MARTIN Y DOUGLAS ALTMAN. 1995. "Multiple Significance Tests: The Bonferroni Method". British Medical Journal 310 (6973): 170. [ Links ]
5. BONILLA, CAROLINA, MARK SHRIVER, ESTEBAN PARRA, ALFREDO JONES Y JOSÉ FERNÁNDEZ. 2004. "Ancestral Proportions and Their Association with Skin Pigmentation and Bone Mineral Density in Puerto Rican Women from New York City". Human Genetics 115 (1): 57-68. [ Links ]
6. BORTOLINI, MARÍA CATIRA, FRANCISCO SALZANO, MARK THOMAS, STEVEN STUART, SELJA NASANEN, CLAITON BAU Y ANDRÉS RUIZ-LINARES. 2003. "Y-Chromosome Evidence for Differing Ancient Demographic Histories in the Americas". American Journal of Human Genetics 73 (3): 524-539. [ Links ]
7. BRION, MARÍA, ANTONIO SALAS, A. GONZÁLEZ-NEIRA, MARÍA VICTORIA LAREU Y ÁNGEL CARRACEDO. 2003. "Insights into Iberian Population Origins through the Construction of Highly Informative Y-Chromosome Haplotypes Using Biallelic Markers, STRs, and the MSY1 Minisatellite". American Journal of Physical Anthropology 122 (2): 147-161. [ Links ]
8. CARVAJAL-CARMONA, LUIS G., ROEL OPHOFF, SUSAN SERVICE, JAANA HARTIALA, JULIO MOLINA, PEDRO LEÓN, JORGE OSPINA, GABRIEL BEDOYA, NELSON FREIMER Y ANDRÉS RUIZ-LINARES. 2003. "Genetic Demography of Antioquia (Colombia) and the Central Valley of Costa Rica". Human Genetics 112 (5-6): 534-541. [ Links ]
9. CARVAJAL-CARMONA, LUIS, IVÁN D. SOTO, NICOLÁS PINEDA, DANIEL ORTIZ-BARRIENTOS, CONSTANZA DUQUE, JORGE OSPINA-DUQUE, MARK MCCARTHY, PATRICIA MONTOYA, VÍCTOR M. ÁLVAREZ, GABRIEL BEDOYA Y ANDRÉS RUIZ-LINARES. 2000. "Strong Amerind/White Sex Bias and a Possible Sephardic Contribution among the Founders of a Population in Northwest Colombia". American Journal of Human Genetics 67 (5): 1287-1295. [ Links ]
10. CARVALHO-SILVA, DENISE, FABRICIO SANTOS, JORGE ROCHA Y SERGIO PENA. 2001. "The Phylogeography of Brazilian Y-Chromosome Lineages". American Journal of Human Genetics 68 (1): 281-286. [ Links ]
11. CASTELLANOS, JORGE. 1980. La abolición de la esclavitud en Popayán, 1832-1852. Cali: Universidad del Valle. [ Links ]
12. CHAKRABORTY, RANAJIT Y KENNETH WEISS. 1986. "Frequencies of Complex Diseases in Hybrid Populations". American Journal of Physical Anthropology 70 (4): 489-503. [ Links ]
13. CHAKRABORTY, RANAJIT Y KENNETH WEISS. 1988. "Admixture as a Tool for Finding Linked Genes and Detecting that Difference from Allelic Association between Loci". Proceedings of the National Academy of Science of the United States of America 85 (23): 9119-9123. [ Links ]
14. CHOUDHRY, SHWETA, NATASHA COYLE, HUA TANG, KEYAN SALARI, DENISE LIND, SUZANNE CLARK Y ESTEBAN BURCHARD. 2006. "Population Stratification Confounds Genetic Association Studies among Latinos". Human Genetics 118 (5): 652-664. [ Links ]
15. CINTADO, ALBERTO, OSMEL COMPANIONI, MARCELO NAZABAL, HANLET CAMACHO, ANNIA FERRER, MARÍA DE COSSIO Y MARTA DUENAS. 2009. "Admixture Estimates for the Population of Havana City". Annals of Human Biology 36 (3): 350-360. [ Links ]
16. COLOMBIA, DEPARTAMENTO ADMINISTRATIVO NACIONAL DE ESTADÍSTICA (DANE). 2005. Censo General 2005, http://www.dane.gov.co [ Links ]
17. DI GIACOMO, FABIO, FRANCESCA LUCA, LUIS POPA, NEJAT AKAR, NICHOLAS ANAGNOU, JURAJ BANYKO Y ANDREA NOVELLETTO. 2004. "Y Chromosomal Haplogroup J as a Signature of the Post-neolithic Colonization of Europe". Human Genetics 115 (5): 357-371. [ Links ]
18. DÍAZ-HORTA, ÓSCAR, ALBERTO CINTADO, MARÍA FERNÁNDEZ-DE-COSSIO, MARCELO NAZABAL Y ANNIA FERRER. 2010. "Relationship of Type 1 Diabetes to Ancestral Proportions and HLA DR/DQ Alleles in a Sample of the Admixed Cuban Population". Annals of Human Biology 37 (6): 778-788. [ Links ]
19. DÍAZ, ZAMIRA. 1983. Guerra y economía en las haciendas: Popayán, 1780-1830. Bogotá: Banco Popular. [ Links ]
20. DÍAZ, ZAMIRA. 1994. Oro, sociedad y economía. Bogotá: Banco de la República. [ Links ]
21. EXCOFFIER, LAURENT, GUILLAUME LAVAL Y STEFAN SCHNEIDER. 2005. "Arlequin (Version 3.0): An Integrated Software Package for Population Genetics Data Analysis". Evolutionary Bioinformatics Online 1: 47-50. [ Links ]
22. HERTZBERG, MARK, KATHERINE MICKLESON, SUSAN SERJEANTSON Y RON TRENT. 1989. "An Asian-specific 9-bp Deletion of Mitochondrial DNA is Frequently Found in Polynesians". American Journal of Human Genetics 44 (4): 504-510. [ Links ]
23. HOGGART, CLIVE J., ESTEBAN J. PARRA, MARK D. SHRIVER, CAROLINA BONILLA, RICK A. KITTLES, DAVID G. CLAYTON Y PAUL M. MCKEIGUE. 2003. "Control of Confounding of Genetic Associations in Stratified Populations". American Journal of Human Genetics 72 (6): 1492-1504. [ Links ]
24. HOGGART, CLIVE J., MARK D. SHRIVER, RICK A. KITTLES, DAVID G. CLAYTON Y PAUL M. MCKEIGUE. 2004. "Design and Analysis of Admixture Mapping Studies". American Journal of Human Genetics 74 (5): 965-978. [ Links ]
25. JOBLING, MARK, CHRIS TYLER SMITH Y MATTHEW HURLES. 2004. Human Evolutionary Genetics: Origins, Peoples, and Disease, vol. 1. Abingdon: Garland Science. [ Links ]
26. KARAFET, TATIANA. M., FERNANDO. L. MÉNDEZ, MONICA B. MEILERMAN, PETER. A. UNDERHILL, STEPHEN L. ZEGURA Y MICHAEL F. HAMMER. 2008. "New Binary Polymorphisms Reshape and Increase Resolution of the Human Y Chromosomal Haplogroup Tree". Genome Research 18 (5): 830-838. [ Links ]
27. KEYEUX, GENOVEVA, CLEMENCIA RODAS, NANCY GÉLVEZ Y DEE CARTER. 2002. "Possible Migration Routes into South America Deduced from Mitochondrial DNA Studies in Colombian Amerindian Populations". Human Biology 74 (2): 211-233. [ Links ]
28. LELL, JEFFREY, REM SUKERNIK, YELENA B. STARIKOVSKAYA, BING SU, LI JIN, THEODORE G. SCHURR, PETER A. UNDERHILL Y DOUGLAS C. WALLACE. 2002. "The Dual Origin and Siberian Affinities of Native American Y Chromosomes". American Journal of Human Genetics 70 (1): 192-206. [ Links ]
29. LI, CHING. 1969. "Population Subdivision with Respect to Multiple Alleles". Annals of Human Genetics 33 (1): 23-29. [ Links ]
30. LONG, JEFFREY. 1991. "The Genetic Structure of Admixed Populations". Genetics 127 (2): 417-428. [ Links ]
31. MACAULAY, VINCENT, MARTIN RICHARDS, EILEEN HICKEY, EMILCE VEGA, FULVIO CRUCIANI, VALENTINA GUIDA, ROSARIA SCOZZARI, BATSHEVA BONNÉ-TAMIR, BRYAN SYKES Y ANTONIO TORRONI. 1999. "The Emerging Tree of West Eurasian mtDNAs: A Synthesis of Control-region Sequences and RFLPs". American Journal of Human Genetics 64 (1): 232-249. [ Links ]
32. MARTÍNEZ, HELIOS, ÁLVARO RODRÍGUEZ-LARRALDE, MARY HELEN IZAGUIRRE Y DINORAH CASTRO DE GUERRA. 2007. "Admixture Estimates for Caracas, Venezuela, Based on Autosomal, Y-chromosome, and mtDNA Markers". Human Biology 79 (2): 201-213. [ Links ]
33. MARTÍNEZ-MARIGNAC, VERÓNICA L., ADÁN VALLADARES, EMILY CAMERON, ANDREA CHAN, ARJUNA PERERA, RACHEL GLOBUS-GOLDBERG, NIELS WACHER, JESÚS KUMATE, PAUL MCKEIGUE, DAVID O'DONNELL, MARK D. SHRIVER, MIGUEL CRUZ Y ESTEBAN J. PARRA. 2007. "Admixture in Mexico City: Implications for Admixture Mapping of Type 2 Diabetes Genetic Risk Factors". Human Genetics 120 (6): 807-819. [ Links ]
34. MENDIZÁBAL, ISABEL, KARLA SANDOVAL, GEMMA BERNIELL-LEE, FRANCESC CALAFELL, ANTONIO SALAS, ANTONIO MARTÍNEZ-FUENTES Y DAVID COMAS. 2008. "Genetic Origin, Admixture, and Asymmetry in Maternal and Paternal Human Lineages in Cuba". BioMedCentral Evolutionary Biology (8): 213-223. [ Links ]
35. MOLOKHIA, MARIAM, CLIVE HOGGART, ALAN PATRICK, MARK SHRIVER, ESTEBAN PARRA, J. YE Y PAUL MCKEIGUE. 2003. "Relation of Risk of Systemic Lupus Erythematosus to West African Admixture in a Caribbean Population". Human Genetics 112 (3): 310-318. [ Links ]
36. PARRA, ESTEBAN J., AMY MARCINI, JOSHUA AKEY, JEREMY MARTINSON, MARK A. BATZER, RICHARD COOPER, TERRENCE FORRESTER, DAVID B. ALLISON, RANJAN DEKA, ROBERT E. FERRELL Y MARK D. SHRIVER. 1998. "Estimating African American Admixture Proportions by Use of Population-specific Alleles". American Journal of Human Genetics 63 (6): 1839-1851. [ Links ]
37. PÉREZ-LEZAUN, ANNA, FRANCESC CALAFELL, EVA MATEU, DAVID COMAS, R. RUIZ-PACHECO Y JAUME BERTRANPETIT. 1997. "Microsatellite Variation and the Differentiation of Modern Humans". Human Genetics 99 (1): 1-7. [ Links ]
38. PFAFF, CARRIE, ESTEBAN PARRA, CAROLINA BONILLA, K. HIESTER, PAUL MCKEIGUE, M. KAMBOH et ál. 2001. "Population Structure in Admixed Populations: Effect of Admixture Dynamics on the Pattern of Linkage Disequilibrium". American Journal of Human Genetics 68 (1): 198-207. [ Links ]
39. ROJAS, WINSTON, MARÍA V. PARRA, OMER CAMPO, MARÍA A. CARO, JUAN G. LOPERA, WILLIAM ARIAS, CONSTANZA DUQUE, ANDRÉS NARANJO, JAIR GARCÍA, CANDELARIA VERGARA, JAIME LOPERA, ERICK HERNÁNDEZ, ANA VALENCIA, YURI CAICEDO, MAURICIO CUARTAS, JAVIER GUTIÉRREZ, SERGIO LÓPEZ, ANDRÉS RUIZ-LINARES Y GABRIEL BEDOYA. 2010. "Genetic Make Up and Structure of Colombian Populations by Means of Uniparental and Biparental DNA Markers". American Journal of Physical Anthropology 143 (1): 13-20. [ Links ]
40. RUIZ-LINARES, ANDRÉS, DANIEL ORTIZ BARRIENTOS, MAURICIO FIGUEROA LOZANO, NATALIA REGINA MESA HERRERA, JUAN G. MÚNERA, GABRIEL DE JESÚS BEDOYA BERRÍO, IVÁN DARÍO VÉLEZ, LUIS FERNANDO GARCÍA, ANNA PÉREZ LEZAUN, JAUME BERTRANPETIT, MARCUS W. FELDMAN, DAVID GOLDSTEIN. 1999. "Microsatellites Provide Evidence for Y Chromosome Diversity among the Founders of the New World". Proceedings of the National Academy of Science of the United States of America 96 (11): 6312-6317. [ Links ]
41. SALAS, ANTONIO, MARÍA AMPARO ACOSTA, V. ÁLVAREZ-IGLESIAS, MARÍA CEREZO, CHRIS PHILLIPS, MARÍA VICTORIA LAREU Y ÁNGEL CARRACEDO. 2008. "The mtDNA Ancestry of Admixed Colombian Populations". American Journal of Human Biology 20 (5): 584-591. [ Links ]
42. SEMINO, ORNELLA, CHIARA MAGRI, GIORGIA BENUZZI, ALICE A. LIN, NADIA AL-ZAHERY, VINCENZA BATTAGLIA, LILIANA MACCIONI, COSTAS TRIANTAPHYLLIDIS, PEIDONG SHEN, PETER J. OEFNER, LEV A. ZHIVOTOVSKY, ROY KING, ANTONIO TORRONI, L. LUCA CAVALLI-SFORZA, PETER A. UNDERHILL Y A. SILVANA SANTACHIARA- BENERECETTI. 2004. "Origin, Diffusion, and Differentiation of Y-chromosome Haplogroups E and J: Inferences on the Neolithization of Europe and Later Migratory Events in the Mediterranean Area". American Journal of Human Genetics 74 (5): 1023-1034. [ Links ]
43. SEMINO, ORNELLA, GUISSEPPE PASSARINO, AGNESSE BREGA, MARC FELLOUS Y A. SILVANA SANTACHIARA-BENERECETTI. 1996. "A View of the Neolithic Demic Diffusion in Europe through Two Y Chromosome-specific Markers". American Journal of Human Genetics 59 (4): 964-968. [ Links ]
44. THOMAS, MARK, TUDOR PARFITT, DEBORAH A. WEISS, KARL SKORECKI, JAMES F. WILSON, MAGDEL LE ROUX, NEIL BRADMAN Y DAVID B. GOLDSTEIN. 2000. "Y Chromosomes Traveling South: The Cohen Modal Haplotype and the Origins of the Lemba-the "Black Jews of Southern Africa". American Journal of Human Genetics 66 (2): 674-686. [ Links ]
45. THOMAS, MARK, KARL SKORECKI, H. BEN-AMI, TUDOR PARFITT, NEIL BRADMAN Y DAVID GOLDSTEIN. 1998. "Origins of Old Testament Priests". Nature 394 (6689): 138-140. [ Links ]
46. VERGARA, CANDELARIA, LUIS CARABALLO, DILIA MERCADO, SILVIA JIMÉNEZ, WINSTON ROJAS, NICHOLAS RAFAELS, TRACEY HAND, MÓNICA CAMPBELL, YUHJUNG J. TSAI, LI GAO, CONSTANZA DUQUE, SERGIO LÓPEZ, GABRIEL BEDOYA, ANDRÉS RUIZ-LINARES Y KATHLEEN C. BARNES. 2009. "African Ancestry is Associated with Risk of Asthma and High Total Serum IgE in a Population from the Caribbean Coast of Colombia". Human Genetics 125 (5-6): 565-579. [ Links ]
47. WILLUWEIT, SASCHA Y LUTZ ROEWER. "Y Chromosome Haplotype Reference Database (YHRD): Update". 2007. Forensic Science International Genetics 1 (2): 83-87. [ Links ]
ANEXOS
TABLA ANEXA 1. HAPLOTIPOS INFERIDOS DE LOS MICROSATÉLITES
FIGURA ANEXA 1. DISTRIBUCIÓN DE LAS DIFERENCIAS POR PARES, HAPLOGRUPO P*
FIGURA ANEXA 2. RED DE HAPLOTIPOS DENTRO DEL HAPLOGRUPO J*
FIGURA ANEXA 3. RED DE HAPLOTIPOS DENTRO DEL HAPLOGRUPO Q*