Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
DYNA
versão impressa ISSN 0012-7353
Dyna rev.fac.nac.minas vol.80 no.177 Medellín jan./fev. 2013
USO DE PATRONES DE DIFRACCIÓN DE LUZ LÁSER Y ANÁLISIS DE IMAGENES PARA DIFERENCIACIÓN DE TRES TIPOS DE COLONIAS BACTERIANAS
LASSER DIFFRACTION PATTERNS AND IMAGE ANALYSIS TO DIFFERENTIATE THREE TYPES OF BACTERIAL COLONY
GLORIA ARANGO
Química MSc, Universidad Nacional de Colombia - Sede Medellín, garango@unal.edu.co
CARLOS RAMÍREZ
Físico MSc, Universidad Nacional de Colombia - Sede Medellín, caramire@unal.edu.co
LUIS GONZALO VARGAS
Físico, Universidad Nacional de Colombia - Sede Medellín, gvargas@unal.edu.co
LAURA MARÍA MUÑOZ
Ing. Biológica. Est. MSc. Universidad Nacional de Colombia - Sede Medellín, lmmunoze@unal.edu.co
JUAN CARLOS PÉREZ
Ing. Agrónomo Ph.D. Universidad Nacional de Colombia - Sede Medellín, jcperez@unal.edu.co
Recibido para revisar Julio 17 de 2012, aceptado Diciembre 21 de 2012, versión final Enero 15 de 2013
RESUMEN: La diferenciación rápida de colonias bacterianas es esencial en muchos campos industriales, científicos y de salud humana y animal. Generalmente esa diferenciación se logra por técnicas microscópicas, bioquímicas o genéticas muy elaboradas que requieren tiempo y entrenamiento. En este estudio se utilizaron técnicas de análisis de imágenes sobre patrones de difracción de luz láser para evaluar su sensibilidad en la diferenciación de colonias bacterianas. Adicionalmente se estudió el efecto del tiempo de crecimiento de las colonias en la generación de estos patrones y se determinó la influencia de la cantidad de medio de cultivo en la difracción de luz láser. Los patrones de difracción del láser se obtuvieron sobre cultivos puros de cinco aislamientos bacterianos B1, B2, B3, B2Ca y B2Cb. En la primera prueba de diferenciación se utilizaron colonias de B2, B3, B2Ca. En el estudio del efecto del tiempo de crecimiento sobre patrones de difracción, las bacterias B1 y B2Cb con uno o dos días de crecimiento. Finalmente, el efecto de la cantidad de medio de cultivo en la difracción de la luz, se evaluó en una prueba en cajas petri con 10, 15 o 20 ml de medio de cultivo estéril sin colonias de bacterias. Mediante un análisis de escalado multidimensional realizado con los parámetros de textura extraídos de las imágenes de los patrones, se obtuvo un agrupamiento adecuado de los patrones asociados a las colonias de cada bacteria evaluada, lo cual indicó que las colonias bacterianas se pueden diferenciar por sus patrones de difracción. Adicionalmente se encontró que no hay variaciones significativas en los patrones de difracción obtenidos de dos aislamientos bacterianos en dos tiempos de crecimiento. El espesor del medio de cultivo afectó la difracción del haz de luz pues patrones obtenidos de cajas petri con 10 ml se separan completamente de los patrones obtenidos con 15 y 20 ml, aunque no hay diferencias entre los dos últimos volúmenes. Los resultados indican que esta técnica sencilla parece ser altamente sensible para diferenciar colonias bacterianas en medios de cultivo, y su refinamiento e implementación para investigación o uso industrial podría lograrse a partir de materiales fácilmente disponibles a nivel local.
PALABRAS CLAVE: Difracción láser; biodiversidad; ImageJ; microorganismos del suelo.
ABSTRACT: Fast and accurate bacterial colony identification is a common requirement in many fields, and usually is supported by the use microscopic, biochemical and molecular tools. But procedures involving these tools might be expensive or time consuming. This study uses image analysis of laser diffraction patterns, obtained from intact bacterial colonies in order to asses its suitability to differentiate bacterial colonies. We also tested the effect of the colony incubation period on these patterns, and the role of the volume of culture media on the laser light diffraction patterns. Laser diffraction patterns were obtained from the five bacterial isolates B1, B2, B3, B2Ca y B2Cb. Bacterial colony differentiation by its diffraction patterns was assessed using isolates B2, B3, B2Ca. The role of colony incubation period on obtained diffraction patterns was tested by using isolates B1 and B2Cb, whereas agar plates without colonies were used to assess the role of different volumes of culture media on its laser diffraction patterns. It was found that the volume of culture media may reduce the quality of the laser diffraction patterns. A multivariate dimensional analysis on laser diffraction patterns also indicated that these can be used to differentiate the bacterial colonies included in the test. Finally, the length of the incubation period on the differences among diffraction patterns of the same isolate appears to be negligible. It is concluded that this simple technique could be used to support the design of sensible equipments based mainly on cheap, locally available supplies, which in turn might support industry and research needs requiring fast and accurate bacterial colony detection.
KEYWORDS: laser diffraction, biodiversity, ImageJ, soil microorganisms
1. INTRODUCCIÓN
La clasificación de colonias bacterianas es rutinaria en muchas áreas de investigación microbiológica. Para diferenciar colonias bacterianas, tradicionalmente se emplean caracteres morfológicos como circularidad, forma de los bordes, brillo y elevación de las colonias. Pero cada característica tiene a su vez muchas categorías y debido al alto número de combinaciones posibles entre ellas, se describen patrones muy complejos que son difíciles de clasificar en grupos morfológicos o fenotipos en forma segura.
Recientemente se presentó una tecnología basadas en difracción de luz láser para diferenciar colonias bacterianas [1]. Esa tecnología aprovecha el hecho de que la forma, tamaño, estructura interna e índice de refracción de las células individuales o colonias de células generan en conjunto variaciones angulares en la trayectoria de los haces de luz que las atraviesan [2], generando patrones de difracción únicos a cada tipo de colonia. Esta tecnología es promisoria debido a que las colonias son estructuras tridimensionales cuya arquitectura interior y exterior no puede ser diferenciada fácilmente, pero cambios pequeños que no se detectan por observación directa, si interferirían enormemente en la difracción de un rayo de luz que las atraviese, lo que permitiría su diferenciación con base en análisis de las imágenes del patrón de difracción. Antecedentes de este tipo de investigaciones han sido presentados anteriormente [1] [2] [3] [4] [5]. Estos estudios diferenciaron colonias bacterianas de especies de Listeria, serotipos de E. Coli, y otros géneros de bacterias como Salmonella, Bacillus y Vibrio mediante difracción de un rayo de luz láser que las atravesaba mientras crecían en cajas de Petri. La estrategia general consiste en cuantificar parámetros que describen las imágenes de los patrones de difracción y a partir de esos valores obtener un análisis dimensional simple que permita agrupar patrones similares entre sí y que se representan como distancias en un plano geométrico [2]. Este tipo de análisis requiere un uso intensivo de algoritmos y memoria que no está disponible en computadores de uso común.
Los sistemas semiautomáticos para reconocimiento de colonias bacterianas son costosos y no están disponibles en todas partes, y su desarrollo a nivel local está limitado por la falta de investigación básica que los soporte. Sin embargo, actualmente se pueden obtener fácilmente imágenes digitales, fuentes de luz láser y software libre que en conjunto permitan obtener rápidamente los patrones de difracción y su análisis estadístico. Si se acoplan apropiadamente, esos elementos permitirían desarrollar localmente sistemas sensibles para uso en industria e investigación. El objetivo general de esta investigación fue evaluar un sistema de diferenciación de colonias bacterianas basado en la incorporación de láseres de bajo costo y paquetes de software libre para análisis estadísticos y de imágenes. Los resultados indican que es posible diferenciar colonias mediante un montaje relativamente simple combinado con condiciones de cultivo apropiadas, lo que permitiría el desarrollo posterior de sistemas sensibles y semiautomáticos para diferenciar colonias bacterianas.
2. MATERIALES Y MÉTODOS
2.1 Material biológico
Se eligieron 5 colonias bacterianas cuyas características morfológicas se presentan en la tabla 1. Estas fueron aisladas de suelo y raíz en estudios anteriores y preservadas en solución salina al 0.85%. Para los experimentos descritos abajo se tomó 10 ml de esta solución y se sembraron por dispersión en al menos 3 cajas de Petri con 10 ml de agar nutritivo estéril, se incubaron a 37oC durante 2 y 3 días, tiempo en el cual se observaron colonias individuales de cada bacteria.
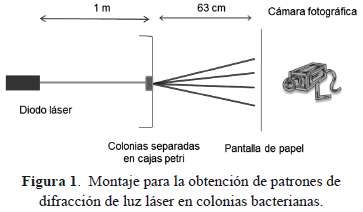
2.2. Montaje para la obtención de patrones de difracción
La figura 1 presenta las principales características del montaje utilizado. Como fuente de luz se empleó un láser Apinex™ con haz de luz de color verde (532 nm). Las imágenes fueron capturadas con una cámara Canon PS SX 30IS con resolución de 14 megapíxeles. Para alinear apropiadamente las colonias y el láser, se construyó a partir de un carro de microscopio, un dispositivo simple que además de sujetar la caja de Petri permitió su desplazamiento vertical y horizontal mediante tornillos de ajuste micrométrico. Este dispositivo se colocó a una distancia de 100 cm desde el láser. Las imágenes se capturaron en una pantalla localizada a 63 cm de la caja con la colonia de bacterias. Esa pantalla se construyó sujetando lateralmente un pliego de papel albanene blanco a dos soportes universales.
2.3. Diferenciación de colonias por sus patrones de difracción
Para ello se realizaron dos estudios. En el primero se obtuvieron en conjunto 18 patrones de difracción usando cuatro colonias del aislamiento B2, seis colonias del aislamiento B3 y nueve colonias del aislamiento B2Ca. Las colonias se crecieron en agar nutritivo de acuerdo al procedimiento descrito anteriormente. En el segundo estudio se determinó el efecto de dos tiempos de crecimiento (2 y 3 días) utilizando patrones de difracción láser de 29 colonias del aislamiento B1 y 45 colonias del aislamiento B2Cb. El número de colonias utilizado se definió con base en la obtención de colonias bien separadas en una misma caja de Petri. La decisión de utilizar colonias sacadas de una misma caja de Petri se tomó debido a las variaciones incontrolables que podrían encontrarse en el vidrio de las cajas de diferentes fabricantes.
2.4. Efecto del volumen de medio de cultivo en la difracción de luz láser.
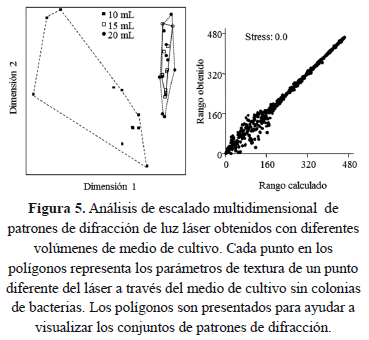
Para este estudio no se emplearon colonias de bacterias. Los patrones de difracción se obtuvieron al pasar aleatoriamente el chorro de luz por 11 puntos diferentes sobre la superficie del medio de cultivo en una misma caja de Petri, en cajas que contenían 10, 15 o 20 ml de agar nutritivo. Este número de patrones de difracción así como los mencionados anteriormente se consideró adecuados para el análisis no paramétrico de escalado multidimensional que se menciona más adelante y que se usó en forma exploratoria en esta investigación.
2.5. Cuantificación de parámetros en imágenes de patrones de difracción y análisis estadístico
Para el análisis cuantitativo de los patrones de difracción, las imágenes se cargaron en el software libre ImageJ [6] y se determinaron en ellas parámetros de textura como: segundo momento angular, contraste, correlación, entropía y diferencia inversa del momento. Esos parámetros fueron definidos previamente [7] y usados en una imagen digital en el contexto de esta investigación y describen la complejidad del patrón de difracción mediante el análisis de distribución espacial de pixeles grises, que varían en intensidad de color desde negro hasta blanco. Dichos parámetros se cuantificaron mediante un "plugin" para análisis de texturas en ImageJ, que está disponible libremente como GLMC-texture.java [8]. Para reducir la complejidad del conjunto de datos y su representación espacial, los valores de textura se analizaron mediante escalado multidimensional usando distancias. euclidianas en el software libre PAST [9]. (http://www.nhm.uio.no/norlex/past/download.html). En ese análisis la fidelidad de la representación gráfica del conjunto de datos de textura en dos dimensiones está indicada por valores más bajos de "stress" y por una baja dispersión de puntos en un diagrama de Sheppard [2].
3. RESULTADOS
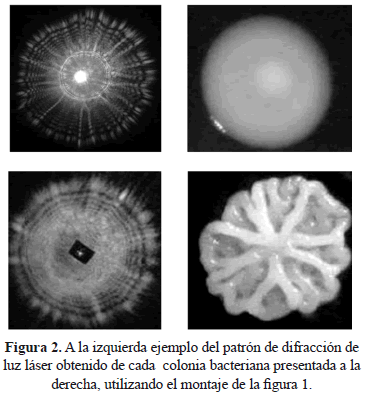
Los patrones de difracción generados por las colonias bacterianas permitieron diferenciarlas claramente. La figura 2 presenta a manera de ilustración los patrones de difracción obtenidos de dos colonias bacterianas usadas para los análisis dimensionales que se presentan mas adelante.
3.1. Diferenciación de colonias por sus patrones de difracción.
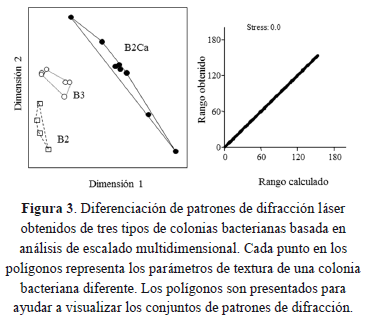
El análisis dimensional de los patrones generados por las colonias de los aislamientos B2, B3 y B2Ca presentaron buena separación de los tres morfotipos (figura 3). En esa misma figura cada grupo de colonias se diferenció del otro, y las colonias de un mismo grupo aparecen más similares entre sí. Esas distancias entre colonias están bien soportadas por el valor bajo de stress y el diagrama de Shepard, que indican una representación adecuada de la similaridad entre colonias a través de los valores de textura usados para el análisis
3.2. Efecto del tiempo crecimiento de dos tipos de colonias bacterianas en la generación de patrones de difracción láser.
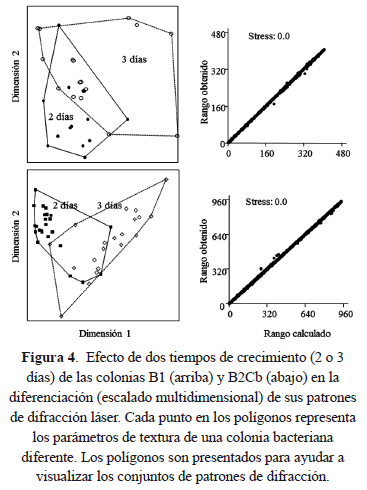
En el estudio para determinar el efecto del tiempo de crecimiento de dos tipos de colonias bacterianas en la generación de patrones de difracción láser, se observa una superposición de los patrones de difracción obtenidos en cada tiempo de crecimiento, lo que sugiere que hay poco efecto de esta condición en las características del patrón de difracción, sin que éstos se separen como grupos diferentes. La mayor dispersión de patrones de difracción observada en la figura 3, se debe posiblemente a que las colonias más pequeñas presentan también patrones de menor tamaño, lo que afectaría ligeramente el análisis de textura (figura 4).
3.3. Efecto del volumen de medio de cultivo en la difracción de luz láser.
El medio de cultivo presentó un efecto significativo en la difracción de luz láser. La caja con 10 ml de medio presentó menor interferencia al paso de luz, y los patrones obtenidos del chorro de luz capturados sobre la pantalla fueron más brillantes y completamente diferenciados de los obtenidos con otros volúmenes de medio (figura 5). Con 15 y 20 ml de medio se presentó mayor interferencia al paso de luz, y los patrones obtenidos no se separaron entre sí. En conjunto, el análisis multidimensional y el diagrama de Shepard (stress: 0.0006453) indicaron mayor dispersión de los valores de textura en los patrones obtenidos con 10 ml, aunque los patrones son más intensos a simple vista, comparado con los obtenidos con otros volúmenes de medio de cultivo.
4. DISCUSIÓN
El conteo y la diferenciación de colonias bacterianas en cajas de Petri han sido empleados rutinariamente en estudios ambientales para determinar la efectividad de prácticas de descontaminación de suelos [10], así como en otras áreas ambientales o de la salud [4]. El análisis de patrones de difracción en este estudio permitió diferenciar preliminarmente colonias de tres tipos diferentes de bacterias creciendo en medio de cultivo sólido. Sin embargo, el efecto del tiempo de crecimiento de las colonias en la clasificación de los patrones de difracción parece depender del aislamiento bacteriano analizado. Adicionalmente, el volumen de medio de cultivo afecta la difracción del chorro de luz que genera los patrones. Por consiguiente la diferenciación de colonias bacterianas por sus patrones de difracción láser parece ser un método confiable, rápido y económico, si se determinan adecuadamente las condiciones que afectan la generación de patrones de difracción.
Los sistemas tradicionales de detección de microorganismos consumen mucho tiempo y se basan en tecnologías costosas que no están disponibles en cualquier lugar. Investigaciones anteriores presentaron prototipos análogos a los de este estudio para generar y capturar imágenes de difracción de luz láser por colonias de bacterias creciendo en medio sólido. Sin embargo, el análisis de imágenes se basó en algoritmos complejos y con demandas computacionales muy altas, lo que posiblemente limitó el desarrollo y la adopción de estos sistemas a nivel comercial. Por otra parte, estudios anteriores se concentraron en la detección rápida de microorganismos patógenos de importancia en la industria de alimentos [4].
En el presente estudio, para el análisis de imágenes o estadístico se utilizó software disponible libremente en internet y otros elementos disponibles localmente. Estas condiciones permitirían el diseño de equipos sensibles a muy bajo costo, para usar en investigación con otros tipos de microorganismos, como los empleados aquí, que fueron aislados del suelo o raíces de plantas. Esto es especialmente importante en suelos tropicales que concentran una gran diversidad de microorganismos, pero para cuyo estudio no siempre están disponibles herramientas sensibles, rápidas, fáciles de usar y de bajo costo. La construcción de equipos basados en láseres y cámaras fotográficas ofrecería muchas ventajas para la investigación, debido a que los resultados obtenidos con un mismo equipo deben ser altamente reproducibles aún en diferentes ambientes. Esto se debe a las propiedades intrínsecas de estos elementos. Por ejemplo, un patrón de difracción resulta de la interacción entre la estructura interna de las colonias, el tamaño de las mismas y la longitud de onda de la luz láser que incide sobre ellas, a diferencia de otros métodos más complejos en los cuales las condiciones ambientales podrían afectar considerablemente su desempeño. Por ejemplo, los resultados de pruebas moleculares que son ampliamente usadas en microbiología pueden ser afectados por el tipo de polimerasa [11], las condiciones de extracción de ADN [12] [13], o el equipo mismo en el que se realiza la prueba. [14]. Aunque la identificación de estos aislamientos no se realizó debido a restricciones actuales para el acceso a material genético, investigaciones futuras permitirán la identificación taxonómica de estos microorganismos.
5. CONCLUSIONES
El uso de software libre y elementos disponibles localmente permitió obtener un sistema de muy bajo costo para diferenciar preliminarmente colonias bacterianas por sus patrones de difracción de luz láser.
El uso combinado de técnicas microbiologicas estándar con otros tipos de láseres u otros sistemas de captura y análisis de imágenes podría contribuir al desarrollo de sistemas semi-automáticos, altamente sensibles y de bajo costo para detectar microorganismos de interés ambiental. Para ello será necesario extender estos resultados a un mayor número de aislamientos bacterianos y condiciones de crecimiento en el laboratorio.
AGRADECIMIENTOS
Esta investigación fue realizada gracias al apoyo de las Escuelas de Física y Geociencias de la Facultad de Ciencias de la Universidad Nacional de Colombia, Sede Medellín.
REFERENCIAS
[1] Hirleman, E.D., Bae, E.W., Huff, K., Banada, P. and Bhunia. A., Light scattering endows bacterial colonies with unique fingerprints. The International Society for Optical Engineering. 2007. [ Links ]
[2] Banada, P.P., Guo, S., Bayraktar, B., Bae, E., Rajwa, B., Robinson, J.P., Hirleman, E.D. and Bhunia,.A.K., Optical forward-scattering for detection of Listeria monocytogenes and other Listeria species. Bios. Bioelectr. 22, pp. 1664-1671, 2007. [ Links ]
[3] Guo, S., Optical scattering for bacterial colony detection and characterization, Purdue University, West Lafayette, IN, 2004. [ Links ]
[4] Leavesley, S., Bayraktar, B., Venkatapathi, M., Hirleman, E.D., Bhunia, A.K., Robinson, J.P., Hassler, R., Smith, L. and Rajwa, B., Quantification of morphology of bacterial colonies using laser scatter measurements and solid element optical modeling, pp. 64460E, In A. Wax and V. Backman, (eds.) Biomedical Applications of Light Scattering. Vol. 6446. Int. Soc. Opt. Eng.; Bellingham, WA, 2007. [ Links ]
[5] Rajwa, B., Bayraktar, B., Banada, P.P., Huff, K., Bae, E., Hirleman, E.D., Bhunia, A.K. and Robinson. J.P., Phenotypic analysis of bacterial colonies using laser light scatter and pattern-recognition techniques [6864-27], pp. 1-7 Biomedical Applications of Light Scattering II, Vol. 6864. Int. Soc. Opt. Eng.; Bellingham, WA, 2008. [ Links ]
[6] Rasband, W.S., ImageJ [Online]. Available by National Institutes of Health, (http://imagej.nih.gov/ij/ ) (verificado Diciembre 1, 2011). [ Links ]
[7] Haralick, R.M., Shanmugam, K. and Dinstein, I.H., Textural features for image classification. Systems, Man and Cybernetics, IEEE Transactions on 3:610-621, 1973. [ Links ]
[8] Cabrera, J., Texture Analyzer [Online]. Available from: National Institutes of Health, http://rsbweb.nih.gov/ij/plugins/download/GLCM_Texture.java (verified December 1), 2010. [ Links ]
[9] Hammer, Ø., Harper, D.A.T. and Ryan, P.D., PAST: Paleontological Statistics Software Package for Education and Data Analysis. 2001 Palaeontol Electronica, Vol. 4. (http://palaeo-electronica.org/2001_1/past/issue1_01.htm) 2001. [ Links ]
[10] Gómez, W., Gaviria, J. and Cardona, S., Evaluación de la bioestimulación frente a la atenuación natural y la bioaumentación en un suelo contaminado con una mezcla de gasolina - diesel. DYNA 76, pp. 83-93, 2009. [ Links ]
[11] Al-Soud, W.A. and Radstrom, P., Capacity of nine thermostable DNA polymerases to mediate DNA amplification in the presence of PCR-inhibiting samples. Appl. Env. Microb. 64, pp. 3748-3753, 1998. [ Links ]
[12] Rossen, L., Norskov, P., Holmstrom, K., and Rasmussen, O.F., Inhibition of PCR by components of food samples, microbial diagnostic assays and DNA-extraction solutions. Int. J. Food Microb. 17, pp. 37-45, 1992. [ Links ]
[13] Schrader, C., Schielke, A., Ellerbroek, L. and Johne, R., PCR inhibitors-occurrence, properties and removal. J. of Applied Microb. 113, pp. 1014-1026, 2012 [ Links ]
[14] Meunier, J.R. and Grimont, P.A.D., Factors affecting reproducibility of random amplified polymorphic DNA fingerprinting. Res. Microb. 144, pp. 373-379, 1993. [ Links ]