Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Colombiana de Ciencias Químico - Farmacéuticas
versão impressa ISSN 0034-7418versão On-line ISSN 1909-6356
Rev. colomb. cienc. quim. farm. v.40 n.2 Bogotá jul./dez. 2011
Artículo de investigación clínica
Comparación de cuatro métodos de restauración del ADN en muestras de plasma y láminas de citología cérvico-uterina como una herramienta para mejorar la calidad de la muestra
Comparison of four methods of DNA restoration in samples from plasma and pap smears as a tool for better the samples
Pilar Caterin Márquez1, Dabeiba Adriana García-Robayo2, Marcos Castillo3, Ignacio Briceño3, Jairo Amaya4, Fabio Ancízar Aristizábal-Gutiérrez*5
1Facultad de Ciencias Básicas, Pontificia Universidad Javeriana, Bogotá, D. C., Colombia.
2Centro de Investigaciones Odontológicas, Pontificia Universidad Javeriana, Bogotá, D. C., Colombia.
3Facultad de Medicina, Universidad de La Sabana, Chía, Cundinamarca, Colombia.
4Unidad de Colposcopia, Hospital de Engativá, Bogotá, D. C., Colombia.
5 Departamento de Farmacia, Facultad de Ciencias, Universidad Nacional de Colombia, Bogotá, D. C., Colombia.*Correo electrónico: faaristizabalg@unal.edu.co.
Recibido para evaluación: 3 de junio de 2011.
Aceptado para publicación: 5 de septiembre de 2011.
RESUMEN
Objetivos: comparar cuatro métodos de restauración del ADN en plasma y láminas cérvico-uterinas como una herramienta para mejorar la calidad de la muestra. Métodos: a 20 muestras de plasma sanguíneo y 20 muestras de láminas citológicas, se les realizó aislamiento de ADN mediante kit comercial y fenol-cloroformo. A todas las muestras se les realizó un tratamiento pre-PCR con cuatro diferentes tipos de actividad de ADN polimerasa: 1. Exonucleasa y endonucleasa 5'-3'. 2. Exonucleasa 5'-3'. 3. Klenow, y 4. Klenow más ligasa. Los diferentes métodos se evaluaron mediante PCR en tiempo real con el gen ALU. Resultados: todos los métodos de restauración mejoran la calidad del ADN en los dos tipos de muestras. El método 3 mostró mejores resultados en plasma y en lámina, incrementando la concentración del ADN de 0,0022 ng/µL a 0,6474 ng/µL en láminas de citología y de 0,0039 ng/µL a 0,435 ng/µL en plasma sanguíneo. Conclusiones: ADN de las muestras de plasma y lámina al ser tratadas con un proceso de restauración aumenta la calidad del ADN en comparación a las muestras no tratadas.
Palabras clave: frotis de muestra exocervical, plasma, restauración, reacción en cadena de la polimerasa, polimerasa Taq
SUMMARY
Objetives: To compare four methods of restoration of DNA in plasma and PAP smears as a tool to improve the quality of the samples. Methods: 20 blood samples and 20 PAP smears samples, we performed DNA isolation by commercial kit and phenol-chloroform respectively. Then all samples underwent a pre-PCR treatment with four different types of activity DNA polymerase: 1. Exonuclease and endonuclease 5'-3'. 2. Exonuclease 5'-3'. 3. Klenow, and 4. Klenow more ligase. Different restoration methods were evaluated quantitatively by real-time PCR with gene ALU. Results: All restoration methods improve the quality of DNA in both types of samples. However, the 3th method showed better results in both plasma and PAP smears, increasing the concentration of DNA from 0.0022 ng/mL to 0.6474 ng/mL in PAP smears and 0.0039 ng/mL to 0.435 ng/mL in blood plasma. Conclusions: DNA from plasma samples and PAP smears to be treated with a restoration process increases the quality of DNA compared to untreated samples.
Key words: PAP smears, plasma, restoration, polymerase chain reaction, Taq polymerase.
INTRODUCCIÓN
El cáncer cervical es una de las principales causas de morbilidad y mortalidad en mujeres (1, 2). La tasa de incidencia anual del cáncer cervical en el mundo es de 529.409 nuevos casos; es la primera causa de muerte en mujeres en países en desarrollo y la segunda en el mundo, aun cuando se trata de una patología altamente curable al ser detectada a tiempo (3, 4). La detección temprana es efectiva, porque las lesiones precursoras se desarrollan algunas veces lentamente en el cáncer invasivo, típicamente durante un período mayor a diez años (5); estas lesiones precursoras (displasias o neoplasias intraepiteliales cervicales) son detectables con la citología cervical.
En los países donde la citología ha sido introducida, los índices de cáncer cervical se han reducido sustancialmente (5). Las muestras de láminas de citología cérvico-uterinas son una fuente importante para el estudio de VPH en pacientes con cáncer. Con todo esto, los datos no son precisos debido a que los procesos en sí mismos producen contaminaciones y alteraciones que pueden afectar el ADN, siendo contraproducente para los resultados de los estudios (6). Por ejemplo, el ADN en láminas de citología puede ser afectado por el uso de colorantes, resinas, etanol, entre otros, que son reconocidos como contaminantes que inhiben la amplificación mediada por la PCR (7, 8), y el ADN de plasma sanguíneo puede ser degradado por la influencia de ADNsas, mecanismos o acciones físicas como la hidrólisis, oxidación o destrucción de enzimas (8). El plasma es una fuente valiosa de un gran número de indicadores moleculares que describen las condiciones generales de pacientes con cáncer, ya que existe mayor cantidad de ADN circular en plasma o suero de pacientes con cáncer que en pacientes sanos (9, 10). El plasma sanguíneo es útil para realizar seguimiento de los pacientes en tratamiento para la detección temprana de recurrencias, metástasis e integración viral en cáncer de cuello uterino (6). Sin embargo, las muestras de sangre y su procesamiento pueden afectar de manera significativa el ADN en cuanto a su estructura, cantidad y calidad, impidiendo realizar estudios moleculares. Por ende, se justifica explorar nuevos procedimientos para obtener ADN de un tamaño adecuado, y es aquí donde un tratamiento de restauración toma vida como una alternativa para tratar las alternaciones del ADN dañado (7, 8, 10, 11). Esta técnica se ha aplicado con muy buenos resultados en la recuperación de ADN proveniente de diferentes tipos de muestras (8, 11), y en ADN de tejidos posmortem para análisis forenses (7).
De aquí la importancia de este estudio de investigación, en el cual se comparan cuatro métodos de restauración del ADN en muestras de plasma y láminas de citologías cérvico-uterinas como una estrategia para mejorar la calidad de la muestra.
MATERIALES Y MÉTODOS
Recolección de muestras
El estudio se realizó bajo las normas éticas y legales para investigación con humanos, según lo dispuesto en la resolución 8430/1993 del Ministerio de Salud. Las pacientes incluidas en el estudio, firmaron un consentimiento informado aprobado por un comité de ética. Se incluyeron 20 mujeres, mayores de edad, que asistieron a la toma de muestra citológica en un hospital de Bogotá, que presentaron o no lesiones intraepiteliales de cuello uterino, durante un período de un mes. No se incluyeron pacientes que no deseaban participar en el estudio, en estado de embarazo, que hayan recibido tratamiento para la neoplasia cervical o infección por VPH, con antecedentes de terapia antineoplásica o inmunomoduladora. A cada paciente se sometió a un examen ginecológico de rutina; se le tomaron muestra de sangre con anticoagulante (EDTA) y dos láminas de citología cérvico-uterina: una para visualización con tinción de Papanicolaou y otra para la extracción del ADN, igualmente teñidas.
Extracción de ADN
Las muestras de sangre se procesaron con el Mini Kit de ADN de QIAamp (QUIAGEN; Germany), siguiendo el protocolo de instrucción de muestras de sangre. Las láminas después de la tinción, se embebieron en xileno para luego cubrirlas con medio líquido plástico (resina) (Shandon Consul – Mount, Histology Fomulation, Xylene Base, Thermo Electron Corporation) y se calentaron a 50 ºC hasta que se endureciera el medio (40 a 60 minutos). Rápidamente, las láminas se colocaron en agua caliente por 60 minutos, en seguida se removió el medio con ayuda de unas pinzas y se colocaron en tubos de microcentrífuga de 1,5 mL con 500 µL de PBS 1X (12). Antes de realizar las pruebas moleculares, las muestras de lámina se centrifugaron a 8.000 rpm durante 5 minutos a 4 ºC. El ADN se extrajo por digestión del tejido con proteinasa K (2 mg/mL), extracción por fenol-cloroformo-isoamílico y precipitación con etanol según las especificaciones de Sambrook y Russel (13). Se evaluó la calidad del ADN obtenido de las muestras por PCR convencional, amplificando un fragmento de 110 pb del gen constitutivo de β-globina por electroforesis en geles de agarosa al 2% (10).
Evaluación de la calidad del ADN
La calidad del ADN se confirmó por PCR con primers PC03/PC04, los cuales amplificaron un fragmento de 110 pb del gen constitutivo de β-globina (10). La mezcla de la reacción contenía buffer de PCR 1X, MgCI2 3,5 mM, mezcla de dNTP 800 µM (200 µM de cada uno), PC03 1 µM, PC04 1 µM, 1,25 U de Taq polimerasa (Promega M7502) y agua destilada estéril hasta completar un volumen de reacción de 25 µL. Las reacciones se corrieron en el equipo My CyclerTM Termal Cycler (Biorad) con las siguientes condiciones: 4 minutos a 94 ºC, seguidos por 40 ciclos de amplificación de 94 ºC por 1 minuto, 55 ºC por 1 minuto y 72 ºC por 1 minuto, y una extensión final a 72 ºC por 4 minutos. Como control positivo se usó ADN linfocitario y como control negativo agua destilada. El producto de la PCR se visualizó mediante una electroforesis en gel de agarosa al 1% teñido con bromuro de etidio, identificando la banda amplificada de 110 pb. Esta electroforesis se corrió durante 1 hora a 100 voltios en buffer TBE1x utilizando el equipo Biorad Power PAC 3.000 y visualizado con un digitalizador de geles con el software Quantity One de Biorad.
Restauración del ADN
Método 1: se utilizó Taq polimerasa con actividad exonucleasa y endonucleasa 5'-3' (Promega M7502) (10). Método 2: se utilizó Taq polimerasa con actividad exonucleasa 5'-3' (Bioline BIO-21071) (7). Los ensayos de restauración que contenían Taq polimerasa con actividad exonucleasa y endonucleasa 5'-3' (Promega M7502) y Taq polimerasa con actividad exonucleasa 5'-3' (Bioline BIO-21071); las muestras de ADN se incubaron durante 1 hora a 55 ºC en 25 µL, en una solución que contenía 10 mM Tris/HCL (pH 8,3), 1,5 mM MgCl, 2% Triton X-100 y 200 µM de cada dNTP. Posteriormente, 1 U Taq polimerasa se adicionó y la polimerización se realizó a 72 ºC durante 20 minutos. Al terminar el proceso, las muestras restauradas se denaturaron a 95 ºC por 5 minutos e inmediatamente enfriadas en hielo y almacenadas a -20 ºC hasta su amplificación. Método 3: se utilizó Klenow (Epicentre® Biotechnologies Exo- Minus Klenow KL0810250) (8). Método 4: se utilizó Klenow + Ligasa (Epicentre® Biotechnologies T4 Ligase LH810H). En la restauración que se utilizó la Klenow (Epicentre® Biotechnologies), se realizó una solución que contenía 50 mM Tris-HCL (pH 7,5), 100 mM NaCl, 1 mM dithiothreitol (DTT), 0,1 mM EDTA y 0,1% Triton® X-100, dNTP y buffer: 0,2 M Tris-HCL (pH 7,5), 50 mM MgCl2 y 5 mM DTT. Posteriormente se adicionó 5 µL de la muestra de ADN para un volumen final de 25 µL y la mezcla se incubó a 37 ºC durante 10 minutos. Transcurrido el tiempo de preincubación, se adiciona 1 µL de Klenow, se incubó a 37 ºC por 1 hora y se almacenó a -20 ºC. Finalmente, para los ensayos que utilizaron Klenow + Ligasa (Epicentre® Biotechnologies), se preparó la misma solución del método de restauración de la Klenow, con la diferencia que después de la incubación se le adicionó 1 µL de Klenow (Epicentre® Biotechnologies Exo- Minus Klenow KL0810250), 1 µL de Ligasa (Epicentre® Biotechnologies T4 Ligase LH810H), 1,3 µL de ATP 10X e incubándolas a 22 ºC toda la noche.PCR en tiempo real
Para determinar cuánto mejora la calidad del ADN y para comparar los métodos de restauración, se realizó una cuantificación del ADN de las muestras restauradas y no restauradas de plasma y lámina. Para esto, el ADN se midió usando el sistema de PCR en tiempo real (PCR-TR) con SYBR® Green (Master Mix, Applied Biosystems), usando secuencias repetitivas del gen ALU (secuencias altamente conservadas en el genoma humano) de 115 pb y 247 pb (14, 15). Las PCR en tiempo real se llevaron a cabo en un volumen de reacción final de 25 µL que contenía 1X Power SYBR® Green PCR Master Mix (Applied Biosystems), 0,2 µM de cada primer y 2 µL de ADN. Las amplificaciones del ADN se corrieron por duplicado, en placas de reacción de 96 pozos en el equipo 7500 de PCR de tiempo real (Applied Biosystems). Una curva estándar de diferentes concentraciones de ADN linfocitario patrón (15 ng/µL) se corrió en paralelo con cada análisis como referencia cuantitativa equivalente a las muestras desconocidas. La amplificación se inició a 95 ºC por 10 minutos y complementada por 50 ciclos de amplificación (denaturación a 95 ºC por 20 segundos, anillamiento a 64 ºC por 30 segundos y una extensión de 72 ºC por 32 segundos). Durante el ciclaje, las emisiones de cada una de las muestras se registraron y los datos de fluorescencia se procesaron por el software 7500 versión 2.0.1, para producir los valores del CT (por su sigla en inglés Threshold Cycle) de cada una de las muestras.
RESULTADOS Y DISCUSIÓN
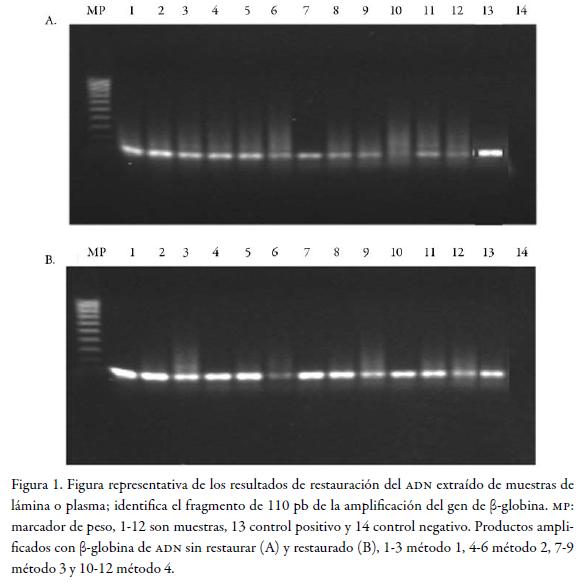
La calidad del ADN obtenido después del proceso de restauración se evaluó amplificando por PCR el gen de β–globina, identificando el fragmento de amplificación de 110 pb en el gel de agarosa al 1%. Las muestras sin restaurar se muestran en la figura 1A. En la figura 1B se exponen las mismas muestras restauradas por los distintos métodos de restauración.
En general, todos los métodos de restauración mejoran la intensidad de banda, mostrando un incremento en el ADN tratado frente al ADN no tratado. Al calcular la densidad óptica relativa (DOR) de las bandas mediante el software Image J versión 1.40g, mostró que la DOR de las bandas del ADN tanto de las muestras de plasma y lámina que no tuvieron tratamiento era menor y que la DOR de las bandas del ADN con tratamiento fue mayor.
Debido a que se observó una mejora en la calidad del ADN al ser restaurado, se decidió realizar un análisis cuantitativo por medio de PCR en tiempo real, usando las secuencias repetitivas ALU de un fragmento de 115 pb. Para lo cual se hizo una cuantificación absoluta tomando como referencia una curva estándar de concentraciones conocidas la cual mostró un R2 = 0,992.
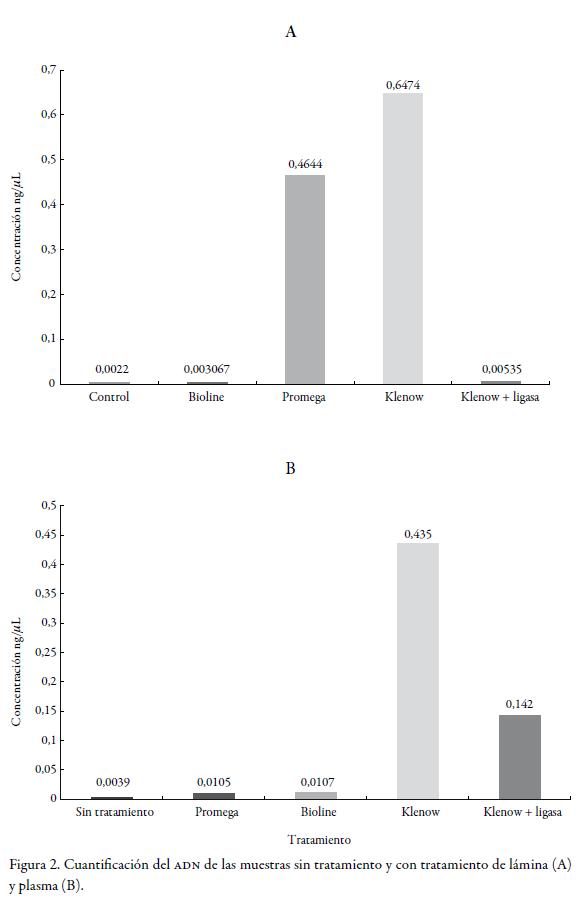
La media de las concentraciones de ADN (ng/µL) extraído de las muestras de lámina sin tratamiento fue de 0,0022, mientras que en las muestras sometidas al tratamiento de restauración con Bioline, Promega, Klenow y Klenow + Ligasa fue de 0,0030, 0,0464, 0,6474 y 0,0053, respectivamente (figura 2A).
La media de las concentraciones de ADN (ng/µL) extraído de las muestras de plasma sin tratamiento fue de 0,0039, mientras que en las muestras sometidas al tratamiento de restauración con Bioline, Promega, Klenow y Klenow + Ligasa fue de 0,0107, 0,0105, 0,435 y 0,142, respectivamente (figura 2B). Estos resultados permiten constatar que hay un incremento significativo en la concentración relativa del ADN en las muestras de plasma al ser tratadas con un proceso de restauración. Dado que hay diferentes concentraciones de ADN en las muestras tanto de plasma como en las extraídas de láminas, sometidas a los cuatro métodos de restauración, se realizaron análisis de varianzas (ANOVA) para determinar si hay diferencias significativas entre estos cuatro métodos para los dos tipos de muestras (plasma y lámina). Aplicando esta prueba, el valor crítico de F fue de 3,92; así se pudo reiterar que hay diferencias significativas entre los métodos de restauración con un p ≤ 0,05.
En cuanto a cuál es el mejor método de restauración, los resultados revelaron que el método que utilizó la Klenow presentó mayores ganancias de concentración de ADN, que los métodos que utilizaron Taq polimerasa con actividad exonucleasa 5'-3' (Bioline), o Taq polimerasa con actividad exonucleasa y endonucleasa 5'-3' (Promega) y Klenow + Ligasa.
Los resultados de este estudio revelaron que pese a que las muestras de plasma o lámina de citología cérvico-uterina contienen información valiosa, al ser sometidas a pruebas moleculares como la PCR puede presentar datos erróneos debido a que el ADN presente en estas muestras no es de buena calidad. Estudios retrospectivos han utilizado un proceso de restauración para mejorar la calidad del ADN (7, 8, 10, 11); este proceso permite que el ADN que ha sufrido roturas (nicks) en una de las cadenas perdiendo algunas de las bases nitrogenadas, como lo que se cree que sucede con el ADN de plasma y de lámina, puedan ser reparadas al ser sometidas a un tratamiento de restauración (7). El ADN en láminas de citología puede ser afectado por el uso de colorantes, resinas, etanol, entre otros, que son reconocidos como contaminantes que inhiben la amplificación mediada por la PCR, eliminando toda evidencia (7, 8).
El método de restauración consiste en un tratamiento de prePCR, en el cual el ADN es incubado en una solución con dNTP, buffer de PCR, MgCl2 y ADN polimerasa. El método es basado sobre el hecho de que la degradación del ADN se relaciona con el rompimiento al azar de hebras sencillas y la pérdida de algunos nucleótidos aislados formando los denominados nicks. Se ha planteado que la reacción de la polimerasa restaura los espacios (nicks) usando la hebra íntegra como molde. Gracias a la posición aleatoria de los nicks, la reconstrucción de los fragmentos da un tiempo suficiente para que sea un éxito la amplificación (7, 8). El proceso de restauración no es un tratamiento que incremente el número de copias molde al no incluir ciclos repetitivos de síntesis y denaturación característicos de la PCR, sino que repara las cadenas de modo y, por tanto, los primers podrán encontrar con mayor eficacia su secuencia blanco (7, 10).
Nuestros resultados revelaron que hay un incremento del ADN molde en las muestras restauradas frente a las muestras sin restaurar, indicando que sí hay una mayor eficiencia en la PCR cuando el ADN es restaurado. En contraste, estudios recientes obtuvieron resultados similares; los cuales afirman que hay una mejora en la calidad del ADN al ser sometido a un proceso de restauración e incluso sostienen que no se necesitan grandes cantidades de ADN para incrementar la calidad del mismo (7, 8, 10).
El análisis cuantitativo por medio de PCR en tiempo real, permitió identificar con una muy alta probabilidad cuánto mejora la calidad del ADN de las muestras sometidas al proceso de restauración. Este sería el primer reporte cuantitativo y comparativo; porque en lo reportado hasta el momento en la literatura especializada, los trabajos de restauración describen sólo los resultados de manera cualitativa luego de haber determinado el efecto de restauración en el ADN degradado.
Otro hallazgo relevante de esta investigación, fue la evaluación del efecto que tiene la actividad de la polimerasa en dicho proceso, que al igual que el análisis cuantitativo, no existe reporte alguno sobre el efecto de las distintas actividades de los diferentes tipos de polimerasa en el proceso de la restauración. Gracias a los resultados obtenidos en esta prueba, se pudo determinar que sí hay diferencia significativa entre los cuatro métodos en los dos tipos de muestra; en donde el método de restauración que utilizó sólo la Klenow muestra importantes y mejores incrementos de calidad de ADN, tanto en plasma como en lámina. Probablemente lo que favoreció este resultado, es que la Klenow carece de la actividad degradadora de exonucleasas 5'-3' que la hace ineficaz para muchas aplicaciones, pero podría favorecer el proceso de restauración (16). Sin embargo, la enzima que sólo tenía actividad exo, presentó mayores resultados que la enzima que tenía actividad endo y exo. Este resultado pudo estar ligado al tipo de degradación que presentaron las muestras, ya que a menudo el daño que sufre el ADN se da en los extremos de los fragmentos, favoreciendo así el método de restauración que utilizó la polimerasa con actividad exonucleasa; ya que por su capacidad reparadora en forma progresiva en los extremos de la cadena, recorta los fragmentos sueltos o defectuosos de uno de los filamentos y los sustituye por los complementarios, permitiendo así la restauración de la doble cadena. Por ende, la acción de la Taq polimerasa está limitada a las copias que tienen rompimientos en cadena sencilla (17).
Otro factor que pudo favorecer la restauración que utilizó la enzima con actividad exo, es que la Taq polimerasa con actividad endo provoca cambios rápidos en las propiedades físicas del ADN; mientras que las exonucleasas tienen efectos menos drásticos sobre las propiedades físicas de los ácidos nucleicos. Además, las endonucleasas al actuar en posiciones internas de la cadena, van reduciendo los fragmentos cada vez más pequeños, lo que conlleva a que posiblemente estos fragmentos no sean detectados en la PCR, obteniendo datos erróneos (18).
Comentario aparte se puede hacer sobre el resultado del uso de la combinación de Klenow + Ligasa, dado que arrojan incrementos importantes en la cantidad de molde detectado, pero los niveles son mucho menores de cuando sólo se empleó Klenow; este hecho podría estar más relacionado a la forma como se aplicó el método, en el cual se colocaron las dos enzimas simultáneamente a la temperatura óptima de la ligasa, condición en la que posiblemente la actividad de la polimerasa fue limitada.
En conclusión, el ADN de las muestras de plasma y lámina al ser tratadas con un proceso de restauración aumenta la calidad del ADN, en comparación a las muestras no tratadas; lo que concuerda con los análisis cualitativos, los cuales expresan que hay una mejora de calidad en el ADN en las muestras restauradas, y entre menos sea agresiva la actividad de las polimerasas es más efectivo el proceso de restauración como una estrategia para reforzar pruebas diagnósticas moleculares, además podrían llegar a ser de utilidad para el estudio de ADN/ARN de muestras parafinadas.
AGRADECIMIENTOS
A la Universidad de La Sabana por su financiación para la realización de este proyecto, al Departamento de Farmacia y laboratorio de equipos comunes de la Universidad Nacional de Colombia por facilitarnos las instalaciones y equipos para desarrollar el proyecto y, en especial, a todas las pacientes que aceptaron formar parte del estudio.
REFERENCIAS
1. C.B. Woodman, S.I. Collins, L.S. Young, The natural history of cervical HPV infection: Unresolved issues, Nat. Rev. Cancer, 7, 11 (2007). [ Links ]
2. K.A. Ault, Epidemiology and natural history of human papillomavirus infections in the female genital tract, Infect. Dis. Obstet. Gynecol., 2006, 40470 (2006). [ Links ]
3. J. Ferlay, H.R. Shin, F. Bray, D. Forman, C. Mathers, D.M. Parkin, Estimates of worldwide burden of cancer in 2008: GLOBOCAN 2008, Int. J. Cancer, 127, 2893 (2010). [ Links ]
4. J. Thompson, L.K. Thomas, K.R. Shroyer, Human papillomavirus: Molecular and cytologic/histologic aspects related to cervical intraepithelial neoplasia and carcinoma, Hum. Pathol., 39, 154 (2008). [ Links ]
5. E.R. Unger, E Barrt, Human papillomavirus and cervical cancer, Emerg. Infect. Dis., 10, 2031 (2004). [ Links ]
6. W. Pornthanakesam, K. Shotelersul, W. Termrungruanglert, N. Voravud, S. Niruthisard, A. Mutirangura, Human papillomavirus ADN in plasma of patients with cervical cancer, BMC Cancer, 1, 2 (2001). [ Links ]
7. S. Bonin, F. Petrera, B. Niccolini, G. Stanta, PCR analysis in archival post-mortem tissue, Mol. Pathol., 56, 84 (2003). [ Links ]
8. C.M. Pusch, I. Giddings, M. Scholz, Repair of degrades duplex ADN from prehistoric samples using Escherichia coli ADN polymerase I and T4 ADN ligase, Nucleic Acids Res., 26, 857 (1998). [ Links ]
9. G. Sozzi, L. Roz, D. Conte, L. Mariani, F. Andriani, P. Verderio, U. Pastorino, Effects of prolonged storage of whole plasma or isolated plasma ADN on the results of circulating ADN quantification assays, J. Natl. Cancer Inst., 97, 1848 (2005). [ Links ]
10. Y. Arias, E. Carrillo, F. Aristizábal, Plasma ADN restoration for PCR applications, J. Clin. Pathol., 60, 952 (2007). [ Links ]
11. D. Mitchell, E. Willerslev, A. Hansen, Damage and repair of ancient DNA, Mutat. Res., 571, 265 (2005). [ Links ]
12. R.T. Miller, P.E. Swanson, M.R. Wick, Fixation and epitope retrieval in diagnostic immunohistochemistry: A concise review with practical considerations, Appl. Immonohistochem. Mol. Morphol., 8, 228 (2000). [ Links ]
13. J. Sambrook, D.W. Russell, "Molecular cloning: A laboratory manual", 3rd ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 2001. [ Links ]
14. M.A. Batzer, P.L. Deininger, Alu repeats and human genomic diversity, Nat. Rev. Genet., 3, 370 (2002). [ Links ]
15. K. Umetani, J. Kim, S. Hiramatsu, H. Reber, O. Hines, A. Bilchik, D.S.B. Hoons, Increased integrity of free circulating DNA in sera of patients with colorectal or periampullary cancer. Direct quantitative PCR for alu repeats, Clin. Chem., 52, 1062 (2006). [ Links ]
16. H. Klenow, I. Henningsen, Selective elimination of the exonuclease activity of the deoxyribonucleic acid polymerase from Escherichia coli B by limited proteolysis, Proc. Natl. Acad. Sci. USA, 65, 168 (1970). [ Links ]
17. S. Mourgues, J. Trzcionka, J.J. Vasseur, G. Pratviel, B. Meunier, Incorporation of oxidized guanine nucleoside 5'-triphosphates in DNA with DNA polymerases and preparation of single-lesion carrying DNA , Biochemistry, 47, 4788 (2008). [ Links ]
18. W. Yang, Nucleases: Diversity of structure, function and mechanism, Q. Rev. Biophys., 44, 1 (2010). [ Links ]