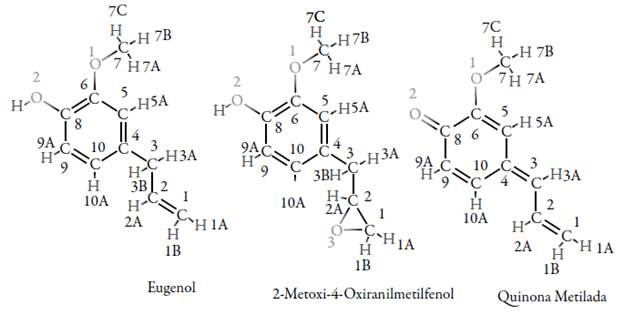
INTRODUCCIÓN
A lo largo de la evolución, las plantas han desarrollado una serie de vías metabólicas para la producción de sustancias químicas, muchas de ellas ofrecen ventajas evolutivas. En este proceso, varias plantas desarrollaron la capacidad de biotransformar la fenilalanina en fenilpropanoides [1-2]. En este sentido, diversos estudios han demostrado la capacidad antinflamatoria, antimicrobiana y citotóxica de diversos fenilpropanoides, como el cinamaldehído y el eugenol. Por lo que es imperativo que futuras investigaciones profundicen en estos aspectos, ya que esto permitiría encontrar nuevos enfoques terapéuticos [3].
Asimismo, diversos estudios demuestran que el eugenol posee actividad anti-inflamatoria, analgésica, antioxidante, antimicrobiana, antiparasitaria, anticancerígena y anti-fúngica. Por lo tanto, es de suma importancia estudiar el potencial farmacológico del eugenol [4-5].
Por otro lado, desde hace algunas décadas, se ha venido desarrollando la metodología de relaciones cuantitativas estructura-actividad (QSAR). La cual posee muchas aplicaciones en química médica, ya que a partir de la determinación teórica de determinadas propiedades fisicoquímicas es posible predecir el comportamiento de moléculas y con ello determinar si cumplen con la regla de cinco de Lipinski [6]. Consecuentemente, QSAR permitiría evaluar el potencial farmacológico del eugenol y derivados como quinona metilada (2-metoxi-4-alilideno-2,5-hexadieno-1-ona) [7] y 2-metoxi-4-oxiranil-metilfenol [8-9].
MATERIALES Y MÉTODOS
Este estudio determinó las propiedades fisicoquímicas y reactividad del eugenol, quinona metilada (2-metoxi-4-alilideno-2,5-hexadieno-1-ona) y 2-metoxi-4-oxiranilme-tilfenol (2M4F). Por lo que se realizó el siguiente procedimiento:
Propiedades fisicoquímicas
Se utilizó el software ChemAxon [10-11] para la determinación de las siguientes propiedades fisicoquímicas: área de la superficie topológica polarizada (TPSA), refractividad molar, constante de disociación acida (pKa), punto isoeléctrico, coeficiente de reparto (Log P), número de donantes y aceptores de hidrógeno, fracción de carbonos saturados en sp3 (FSP3), volumen de van der Waals y área superficial de van der Waals.
Se determinó el desplazamiento químico del 1H RMN, utilizando el software predictor 1D 1H NMR spectra [12].
Además, se utilizó el método de Joback [13-14] para la determinación de la energía libre de Gibbs de formación en condiciones estándar de reacción (AfG°), los cálculos fueron realizados utilizando la plataforma virtual Cheméo.
Reactividad
Se utilizó el software SMARTCyp versión 4.2.2 [15] para la determinación de la energía de activación y área de superficie accesible por solvente (SASA), de átomos de carbono frente al CYP2C (isoenzima del citocromo P450).
Para el estudio de la reactividad del eugenol y derivados se utilizó el software EAWAG-BBD, el cual puede predecir reacciones catabólicas en microorganismos aeróbicos. Dicho software reconoce grupos funcionales y predice las reacciones metabólicas más probables [16].
Análisis estadístico
Se determinó el coeficiente de correlación de Spearman de las siguientes variables:
- El desplazamiento químico del 1H RMN (variable independiente) y la energía de activación de átomos de carbono (variable dependiente).
- El desplazamiento químico del 1H RMN (variable independiente) y el SASA de átomos de carbono (variable dependiente).
Si dos o más átomos de hidrógeno unidos a un mismo carbono presentan valores distintos, se calculará una media aritmética y este resultado se usará para calcular el coeficiente de correlación.
Para el cálculo del coeficiente de correlación se usará el software STATA versión 15.
RESULTADOS
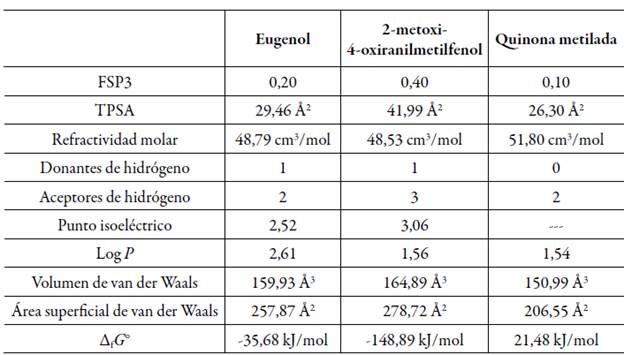
En la tabla 1 se puede observar los resultados obtenidos de las propiedades fisicoquímicas del eugenol y sus derivados; mientras en la tabla 2, el pKa del grupo alcohol del eugenol y 2-metoxi-4-oxiranilmetilfenol.
Tabla 2 pKa de los grupos químicos ionizables.
| Grupos químicos | Eugenol | 2-metoxi4-oxiranilmetilfenol |
|---|---|---|
| -OH (2) | 9,94 | 10,16 |
En la tabla 3 puede observarse el desplazamiento químico del 1H RMN del eugenol y de los derivados.
Tabla 3 Desplazamiento químico del 1H RMN.
| Átomo de hidrógeno | Eugenol (ppm) | 2-metoxi-4-oxiranilmetilfenol (ppm) | Quinona metilada (ppm) |
|---|---|---|---|
| 1A | 4,85 | 3,06 | 5,12 |
| 1B | 4,85 | 3,11 | 4,99 |
| 2A | 5,69 | 3,23 | 6,20 |
| 3A | 3,13 | 2,86 | 6,21 |
| 3B | 3,13 | 2,86 | |
| 5A | 6,59 | 6,58 | 6,86 |
| 7A | 3,78 | 3,78 | 3,91 |
| 7B | 3,78 | 3,78 | 3,91 |
| 7C | 3,78 | 3,78 | 3,91 |
| 9A | 6,63 | 6,63 | 6,85 |
| 10A | 6,53 | 6,58 | 7,53 |
De otro lado, en la tabla 4 se muestra la energía de activación de cada átomo de carbono frente al CYP2C, del eugenol y derivados. De la misma manera, en las tablas 5 puede observarse el SASA.
Tabla 4 Energía de activación de átomos de carbono frente a CYP2C.
| Átomo de carbono | Eugenol (kJ/mol) | 2-metoxi 4-oxiranilmetilfenol (kJ/mol) | Quinona metilada (kJ/mol) |
|---|---|---|---|
| 1 | 65,6 | 75,9 | 40,1 |
| 2 | 65,6 | 75,9 | 80,8 |
| 3 | 59,9 | 66,4 | 80,8 |
| 4 | 999 | 999 | 999 |
| 5 | 77,2 | 77,2 | 80,8 |
| 6 | 999 | 999 | 999 |
| 7 | 62,2 | 62,2 | 62,2 |
| 8 | 999 | 999 | 999 |
| 9 | 77,2 | 77,2 | 80,8 |
| 10 | 74,1 | 74,1 | 80,8 |
Tabla 5 Área de superficie accesible por solvente (SASA) de átomos de carbono frente al CYP2C.
| Átomo de carbono | Eugenol (Å2) | 2-metoxi4-oxiranilmetilfenol (Å2) | Quinona metilada (Å2) |
|---|---|---|---|
| 1 | 62,46 | 52,42 | 61,01 |
| 2 | 29,01 | 17,76 | 28,21 |
| 3 | 28,14 | 28,04 | 22,92 |
| 4 | 4,06 | 3,67 | 5,82 |
| 5 | 23,39 | 22,57 | 22,90 |
| 6 | 5,19 | 5,19 | 7,09 |
| 7 | 64,33 | 64,33 | 65,80 |
| 8 | 6,22 | 6,22 | 7,49 |
| 9 | 28,6 | 28,6 | 28,09 |
| 10 | 27,46 | 26,64 | 26,97 |
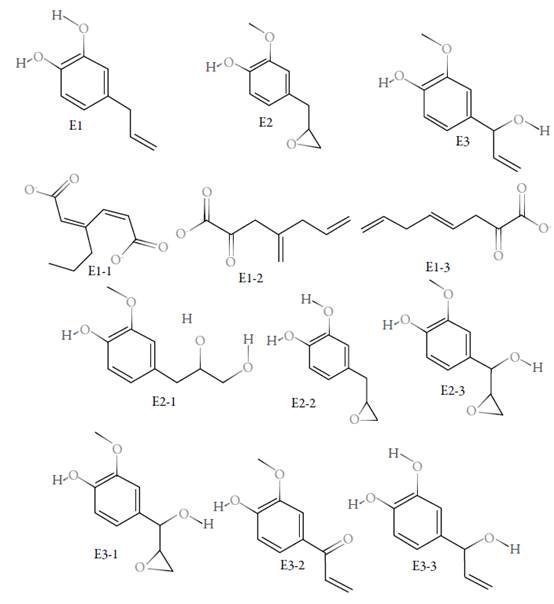
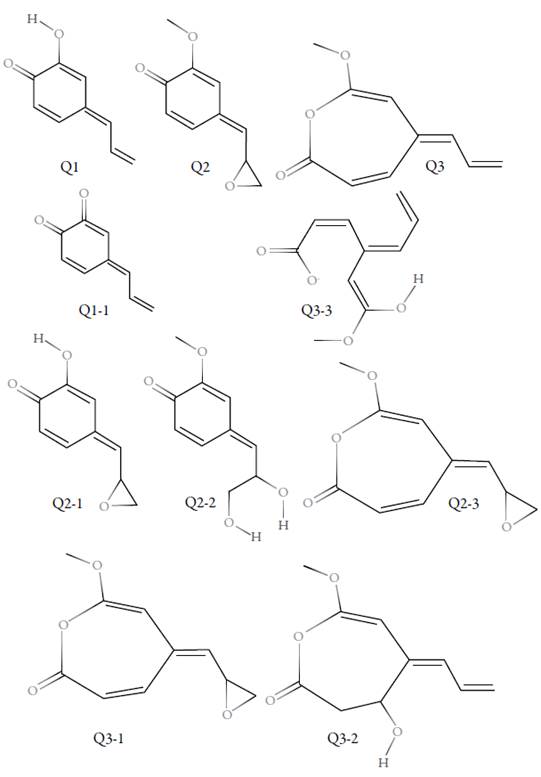
Por otra parte, en la figura 2 se puede apreciar los compuestos producto del catabolismo del eugenol, predicho por el software EWAG-BBD. Los compuestos E1, E2 y E3 son los productos de la primera etapa del catabolismo. Los compuestos E1-1, E1-2 y E1-3 son los productos del catabolismo del compuesto E1; igualmente, los compuestos E2-1, E2-2 y E2-3 son derivados del compuesto E2, y los compuestos E3-1, E3-2 y E3-3 derivan del compuesto E3.
En la tabla 6 están consignadas las reacciones predichas por el software EWAG-BBD para eugenol y derivados.
Tabla 6 Reacciones catabólicas del eugenol, 2-metoxi-4-oxiranolmetilfenol y quinona metilada.
| Eugenol | 2-metoxi4-oxiranilmetilfenol | Quinona metilada |
|---|---|---|
| O-desalquilación en el carbono 7 (E1) | O-desalquilación en el carbono 7 (P2) | O-desalquilación en el carbono 7 (Q1) |
| Epoxidación del grupo alqueno (E2) | Apertura del anillo epóxido (P1) | Epoxidación del grupo alqueno de los carbonos 1 y 2 (Q2) |
| Hidroxilación en el carbono 3 (E3) | Hidroxilación en el carbono 3 (P3) | Transferencia de un átomo de oxígeno, formándose un grupo éter entre los carbonos 6 y 8 (Q3) |
Entre paréntesis, el símbolo asignado por este estudio para cada compuesto.
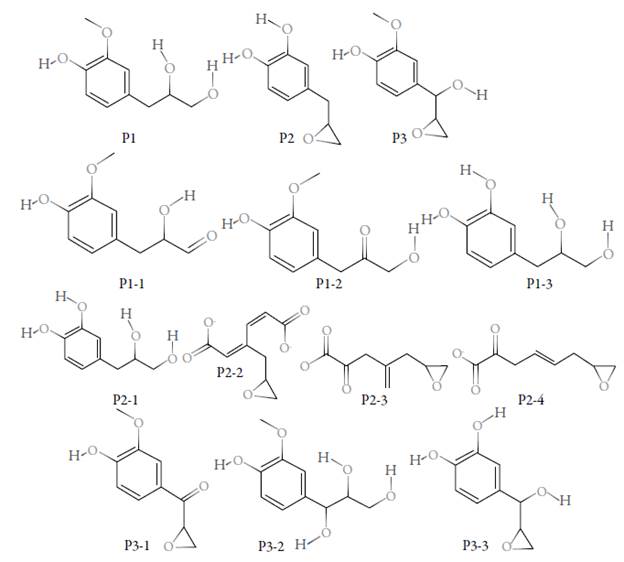
En la figura 3 se puede apreciar los compuestos producto del catabolismo del 2M4F, predicho por el software EWAG-BBD. Los compuestos P1, P2 y P3 son los productos de la primera etapa del catabolismo. De la misma manera, los compuestos P1-1, P1-2 y P1-3 son los productos del catabolismo del compuesto P1; los compuestos P2-1, P2-2 y P2-3 son derivados del compuesto P2, y los compuestos P3-1, P3-2 y P3-3 derivan del compuesto P3.
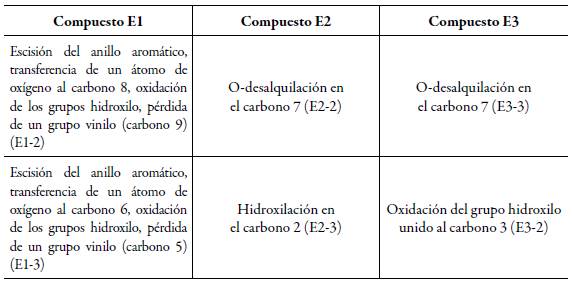
En la tabla 7 están las reacciones predichas por el software EWAG-BBD, de los compuestos E1, E2 y E3. Mientras en la tabla 8, las reacciones predichas por el software EWAG-BBD, de 2M4F.
Tabla 7 Reacciones catabólicas de compuestos E1, E2 y E3 (puede observarse entre paréntesis el producto de cada reacción).
Entre paréntesis, el símbolo asignado por este estudio para cada compuesto
Tabla 8 Reacciones catabólicas de compuestos derivados de 2-metoxi-4-oxiranilmetilfenol
| Compuesto P1 | Compuesto P2 | Compuesto P3 |
|---|---|---|
| Oxidación del grupo hidroxilo unido al carbono 1 (P1-1) | Apertura del anillo epóxido (P2-1) | Apertura del anillo epóxido (P3-2) |
| Oxidación del grupo hidroxilo unido al carbono 2 (P1-2) | Escisión del anillo aromático, transferencia de dos átomos de oxígeno a los carbonos 6 y 8, oxidación de los grupos hidroxilo (P2-2) | Oxidación del grupo hidroxilo unido al carbono 3 (P3-1) |
| O-desalquilación en el carbono 7 (P1-3) | Escisión del anillo aromático, transferencia de un átomo de oxígeno al carbono 8, oxidación de los grupos hidroxilo, pérdida de un grupo vinilo (carbono 9) (P2-3) | O-desalquilación en el carbono 7 (P3-3) |
| Escisión del anillo aromático, transferencia de un átomo de oxígeno al carbono 6, oxidación de los grupos hidroxilo, pérdida de un grupo vinilo (carbono 5) (P2-4) |
Entre paréntesis, el símbolo asignado por este estudio para cada compuesto.
De otro lado, en la figura 4 se pueden apreciar los compuestos productos del catabolismo de quinona metilada, predichos por el software EWAG-BBD. Los compuestos Q1, Q2 y Q3, son los productos de la primera etapa del catabolismo. El compuesto Q1-1 es producto del catabolismo del compuesto Q1; en ese sentido, los compuestos Q2-1, Q2-2 y Q2-3 derivan del compuesto Q2, y los compuestos Q3-1, Q3-2 y Q3-3, del compuesto Q3.
En la tabla 9 se observan las reacciones predichas por el software EWAG-BBD de los compuestos Q1, Q2 y Q3.
Tabla 9 Reacciones catabólicas de compuestos derivados de quinona metilada.
| Compuesto Q1 | Compuesto Q2 | Compuesto Q3 |
|---|---|---|
| Oxidación del grupo hidroxilo (Q1-1) | O-desalquilación en el carbono 7 (Q2-1) | Epoxidación del grupo alqueno de los carbonos 1 y 2 (Q3-1) |
| Apertura del anillo epóxido (Q2-2). | Hidroxilación en el carbono 10 (Q3-2). | |
| Transferencia de un átomo de oxígeno, formándose un grupo éter entre los carbonos 6 y 8 (Q2-3) | Escisión del anillo aromático, transferencia de un átomo de oxígeno al carbono 8 (Q3-3) |
Entre paréntesis, el símbolo asignado por este estudio para cada compuesto.
Finalmente, en la tabla 10 se presentan los resultados del coeficiente de correlación de Spearman. Podemos observar que existe una correlación lineal positiva entre el desplazamiento químico del 1H RMN y la energía de activación de átomos de carbono; asimismo, esta correlación es estadísticamente significativa (p<0,05) con un nivel de confianza del 95%. Además, se halló una correlación lineal negativa entre el desplazamiento químico del 1H RMN y el SASA de átomos de carbono; sin embargo, esta correlación no es estadísticamente significativa (p>0,05) con un nivel de confianza del 95%.
DISCUSIÓN
Los resultados presentados en la tabla 1 y 2 indican que el eugenol y derivados cumplen con la regla de cinco de Lipinski, esto guarda relación con lo esperado. Ya que un estudio previo encontró resultados similares utilizando otros programas [7]. Por tanto, estos valores posicionan al eugenol y derivados como posibles fármacos. Esto debido a que poseen menos de 5 donantes de hidrógeno, sus masas molares son menores a 500 Daltons, Log P menor a 5, tienen más de 10 enlaces rotativos y no poseen más de 10 átomos de nitrógeno y oxígeno. Por tanto tienen una buena bioabsorción y la capacidad para penetrar membranas biológicas.
Además, los resultados de ∆f G° indican que el eugenol y 2-metoxi-4-oxiranilmetilfenol (2M4F) poseen valores negativos, lo que indica que las reacciones que los generan son termodinámicamente favorables [17]. Sin embargo, el método de Joback presenta una serie de limitaciones, pues este método simplifica la estructura molecular; los cálculos también se realizan considerando que las reacciones se llevan a cabo a 25°C, 1 atm y 1 molar de concentración de cada reactivo. Por lo que estos resultados deben comprobarse experimentalmente [13-14].
En la tabla 3 puede verse el desplazamiento químico del 1H RMN del eugenol y derivados. Se encontraron valores más altos en átomos de hidrógeno unidos a enlaces doble carbono-carbono, esto debido a los enlaces pi (π) [17]. De otro lado, los resultados presentados en la tabla 4 indican que los carbonos 1, 2, 3 y 7 del eugenol y 2M4F presentan menor energía de activación, de la misma forma, los carbonos 1 y 7 de quinona metilada presentan menor energía de activación. Esto indica que dichos carbonos podrían ser blanco de reacciones químicas catalizadas por el CYP2C [15].
Asimismo, los resultados de la tabla 5 indican que los carbonos 1 y 7 del eugenol y derivados, presentan un SASA más alto, lo que permitiría que estos carbonos participen en reacciones químicas catalizadas por el CYP2C [15]. Los resultados de las tablas 4 y 5 también indican que en los carbonos 4, 6 y 8 es menos probable que se dé una reacción catalizada por el CYP2C [15].
En una investigación previa se estudió el metabolismo del eugenol mediado por el cito-cromo P450, encontrando que los carbonos 1 y 2 son susceptibles a la epoxidación, el carbono 3 a la hidroxilación y el carbono 7 a la o-desalquilación [7].
Como podemos observar el catabolismo del eugenol y 2M4F en microorganismos aeróbicos, se da principalmente en los carbonos 1, 2, 3 y 7; estos resultados son similares a los encontrados en el metabolismo catalizado por CYP2C. Además, se encontró que en quinona metilada las reacciones catabólicas se dan en los carbonos 1, 2 y 7.
Este estudio propone que la correlación entre el desplazamiento químico del 1H RMN y la energía de activación de los átomos de carbono podría deberse a la manera en que los aminoácidos y el grupo prostético del centro activo del citocromo P450 interactúan con los átomos de carbono del eugenol y derivados. Esta interacción sería similar a la forma en como los núcleos atómicos del hidrógeno interactúan con un campo magnético externo (como el RMN).
CONCLUSIONES
El eugenol y derivados (2-metoxi-4-oxiranilmetilfenol y quinona metilada) poseen propiedades fisicoquímicas que los posicionan como candidatos a nuevos fármacos. Asimismo, pueden ser metabolizados por el citocromo P450, siendo este metabolismo similar al observado en bacterias aeróbicas.
Por último, existe una correlación lineal positiva entre el desplazamiento químico del 1H RMN y la energía de activación de átomos de carbono.