INTRODUCCIÓN
La COVID-19 es una enfermedad infecciosa viral, reportada a partir del brote en Wuhan (China) en diciembre de 2019, es considerada como la pandemia más importante del siglo XXI [1]. Hasta el 17 de diciembre de 2020 se reportaron 39 560 casos de defunción en Colombia [2] y 1 658 645 a nivel mundial [3]. El agente causal fue confirmado por la Organización Mundial de la Salud (OMS) como el virus 2019-nCoV, mientras que el Comité Internacional de Taxonomía de Virus (ICTV) le asignó el nombre de SARS-CoV-2 [3, 4].
Los coronavirus son virus de ácido ribonucleico (ARN), pertenecientes a la familia Coronaviridae, que pueden causar enfermedades tanto en animales como en humanos [5]. Generalmente, atacan las vías respiratorias superiores desencadenando sintoma-tologías desde el resfriado común hasta enfermedades más graves como el síndrome respiratorio de Oriente Medio (Mers) y el síndrome respiratorio agudo severo (SARS) [6]. Los principales afectados son personas con alguna comorbilidad como hipertensión, diabetes, cardiopatías, asma, adultos mayores y personas inmunosuprimidas. Los casos severos, generalmente, manifiestan una neumonía grave, que conduce a un estado crítico y posteriormente la muerte. Se estima que, aproximadamente el 80 % de los infectados con SARS-CoV-2 presentan pocos síntomas (fiebre, fatiga, tos y dificultad para respirar) o son asintomáticos; el 15 % desarrolla un cuadro severo de la enfermedad y el 5 % llega a estadios graves y críticos [4, 7].
En atención al estado de emergencia que supone el brote por SARS-CoV-2, muchos países han implementado medidas de contención y mitigación para retrasar los picos de infección y disminuir la demanda en los sistemas de salud. Así, el diagnóstico temprano es vital para controlar la propagación del virus dentro de la población [8-10]. La OMS y el Centro para el control y la prevención de enfermedades de EE.UU. (CDC) aprobaron la técnica RT-qPCR (por sus siglas en inglés Quantitative reverse transcrip-tion PCR) como prueba estándar para la detección del virus SARS-CoV-2 [11,12]. Este análisis detecta la presencia de ácidos nucleicos virales con alta sensibilidad y especificidad, en muestras de esputo, hisopados de garganta y secreciones del tracto respiratorio inferior, sin embargo, entre sus limitantes se cuentan la necesidad de equipos complejos, sondas fotosensibles y personal capacitado o especializado en biología molecular. Además, el tiempo transcurrido entre la toma de la muestra y la obtención del resultado puede ser entre 24 y 72 h o incluso días según el lugar donde se encuentre ubicado el paciente [8].
Existen pruebas rápidas como las de detección de antígeno y anticuerpos asociados con la infección por el virus, estas pruebas se consideran fáciles de usar y menos costosas que las pruebas de RT-qPCR en tiempo real. Un resultado positivo en una prueba rápida se considera muy exacto, sin embargo, la principal desventaja de los ensayos serológicos es su baja sensibilidad, ya que suelen presentarse casos de falsos negativos, por ende, es necesario una prueba confirmatoria por RT-qPCR, repercutiendo en un incremento de los costos asociados, así, su utilidad puede ser limitada para el diagnóstico, ya que requiere que el paciente desarrolle un nivel detectable de anticuerpos y esto puede tardar varios días e incluso semanas [4, 8].
Las estrategias para la detección temprana de SARS-CoV-2 suelen ser la principal limitante para controlar el contagio, esto ocurre con frecuencia en países en desarrollo como Colombia, Perú y Ecuador. La identificación eficaz de los casos de infección es un reto que evidencia la necesidad de implementar métodos de diagnósticos rápidos y precisos en detección temprana, que puedan complementar las estrategias propuestas por las autoridades sanitarias para hacer frente a la rápida propagación del virus [13].
Para dar respuesta a esa problemática, países latinoamericanos como Uruguay y Argentina han avanzado en la implementación del método LAMP (por sus siglas en inglés loop mediated isothermal amplification) como una nueva alternativa diagnóstica para la enfermedad, ya que es económica, accesible y puede ser fácilmente utilizada en todo el país empleando infraestructura de baja complejidad [13].
El método LAMP, desarrollado por Notomi et al. [14], es un método de amplificación isotérmica de ácidos nucleicos (ADN o ARN) que ha sido aplicado a la detección de patógenos tales como virus y bacterias [15], presenta sensibilidad diagnóstica y especificidad similar al método estándar (RT-qPCR), debido a que emplea cebadores específicamente diseñados y una ADN polimerasa (Bst) aislada de Bacillus stearother-mophilus, con actividad de desplazamiento de cadena [10, 16, 17].
A partir de la secuencia a amplificar, previamente, las 6 regiones de reconocimiento de los cebadores deben ser diseñadas, la etapa de amplificación lineal comienza con la hibridación del cebador doble FIB o BIP (por sus siglas en inglés Forward Inner Primer y Backward Inner Primer) en el extremo 5' o 3', respectivamente, los cuales están diseñados para facilitar el acoplamiento y copia de la región de interés con sus regiones complementarias, formando estructuras tipo bucle que funcionan como moldes para la etapa cíclica de amplificación. Los cebadores externos (F3 y B3, por sus siglas en inglés Forward outer Primer y Backward outer Primer) abarcan la región completa de amplificación y permiten generar copias de la cadena original. Además, se emplean dos cebadores denominados FLB y BLP (por sus siglas en inglés Forward LOOP Primer y Backward LOOP Primer) que aceleran el proceso de amplificación y ayudan a mantener la estabilidad de las cadenas [14, 16, 18].
La lectura de los resultados puede realizarse de dos formas. I) Cuantitativa: empleando un turbidímetro para lectura en tiempo real. II) Cualitativa: detectando la turbidez, ya sea mediante el uso de agentes fluorescente como SYBR Green [19], el uso de indicadores de iones metálicos fluorescentes [16, 20] como el azul de hidroxinaftol y la calceína o el uso de indicadores de cambio de pH, entre otros [19, 21], tales como el rojo fenol que varia de color rosado a pH 8,8 con viraje a color amarillo cuando el pH disminuye por debajo de 6,2 [17, 22], este último es el método de detección predilecto, pues evita el uso de equipos adicionales o especializados.
Durante la síntesis de nuevas cadenas de ADN, la Bst polimerasa cataliza el crecimiento de la cadena en dirección 5'- 3', creando enlaces fosfodiéster entre los dNTPs de la cadena naciente, generando la liberación de pirofosfatos de magnesio (Mg2P2O7), esta acumulación ocasiona un cambio en el pH de la reacción, debido a la disminución en la concentración de los iones de magnesio [16, 19, 21, 22].
Entre las ventajas del método LAMP se encuentran el procesamiento de las muestras a una temperatura constante entre 60 y 65 °C, la amplificación del ADN blanco en menos de 30 min [16, 22-24], la exclusión de un termociclador como equipo robusto y un resultado de prueba rápido y confiable, ya que tiene una sensibilidad y especificidad similar e incluso superior a la qPCR, por lo que puede ser una técnica que ofrece una mayor capacidad diagnóstica para responder a las necesidades surgidas en una pandemia [24].
Una de las variantes del método LAMP es el método RT-LAMP (por sus siglas en inglés Reverse transcription loop-mediated isothermalamplification), en el cual se agrega una enzima transcriptasa a la mezcla de reacción. Esta enzima permite la conversión del ARN viral en ADN complementario, el cual será utilizado como molde para la amplificación. Este método tiene un gran potencial en la detección del virus, como lo demostraron Huang etal. [17], Buck etal. [24] y Lamb etal. [25], quienes reportaron una prueba rápida de LAMP para la detección de SARS-CoV-2 en un tiempo de reacción de 30 min para la amplificación del gen N, evidenciando una alta especificidad de los cebadores diseñados, ya que al evaluar virus como MERS, MHV y BtCoV (por sus siglas en inglés Middle East respiratory syndrome, Mouse Hepatitis Virus and Bat coro-navirus respectivamente) no se obtuvieron señales de amplificación [24, 25].
En el presente estudio se evaluó y comparó el límite de detección del método LAMP con respecto al método estándar RT-qPCR y el uso de la RT-LAMP como alternativa para la detección del gen N del SARS-CoV-2. La detección de la cantidad mínima de copias del gen viral en la prueba LAMP fue evaluada en un plásmido sintético denominado plásmido Covid+. Se evaluó la aplicabilidad del método RT-LAMP en el diagnóstico de muestras clínicas de SARS-CoV-2 en 25 muestras de pacientes colombianos, explorando su uso potencial en el diagnóstico molecular en Colombia.
METODOLOGÍA
Recolección y procesamiento de muestras clínicas
Se realizó un estudio descriptivo transversal. En el marco del proyecto "Generación de la capacidad técnica para fabricación mediante procesos biotecnológicos de reactivos y versiones in house de kits diagnóstico" desarrollado por el Laboratorio de Caracterización Molecular del Instituto de Biotecnología de la Universidad Nacional de Colombia (IBUN), Sede Bogotá; se realizó, previo consentimiento informado, la toma de 25 hisopados nasofaríngeos a adultos mayores de edad, con y sin síntomas de infección respiratoria aguda, que autorizaron la inclusión de sus muestras para el desarrollo del presente estudio. Las muestras fueron colectadas en medio de transporte viral universal y fueron debidamente transportadas y almacenadas hasta su posterior procesamiento.
La extracción de ARN viral se realizó utilizando el MGIEasy Magnetic Beads Virus DNA/RNA Extraction Kit® según las indicaciones del fabricante. Alícuotas de 20jxL de ARN viral eluído fueron usadas en el protocolo estándar para el diagnóstico de SARS-CoV-2, por RT-qPCR y almacenadas a -80 °C hasta la realización de los ensayos posteriores [12].
El estudio se ajustó a las buenas prácticas de laboratorio según los estándares internacionales y el uso de muestras clínicas fue aprobado por el comité de ética de la Facultad de Ciencias de la misma universidad. Todas las muestras fueron manipuladas e inactiva-das siguiendo las prácticas, técnicas de laboratorio, equipos de seguridad e instalaciones adecuadas para el manejo de muestras biológicas de nivel 3.
LAMP para SARS-CoV-2
Las reacciones de RT-LAMP para la amplificación del gen N, que codifica para la proteína de la nucleocápside del SARS-CoV-2, se llevaron a cabo empleando un grupo de cebadores específicos reportados por Buck et al. [24] y Zhang et al. [26] y el kit comercial WarmStart Colorimetric LAMP 2X Master Mix (ADN & ARN M1800) (NEB) (tabla 1), el cual contiene una polimerasa Bst 2.0 con actividad de desplazamiento de cadena y una solución de reacción especial optimizada, que incluye rojo fenol como indicador de pH.
Tabla 1 Secuencias de cebadores del gen N.
| Método | Cebador | Secuencia 5' - 3' |
|---|---|---|
| RT-LAMP | FIP | TCTGGCCCAGTTCCTAGGTAGTCCAGACGAATTC-GTGGTGG |
| BIP | AGACGGCATCATATGGGTTGCACGGGTGCCAAT-GTGATCT | |
| LOOP F | GGACTGAGATCTTTCATTTTACCGT | |
| LOOP B | ACTGAGGGAGCCTTGAATACA | |
| F3 | TGGCTACTACCGAAGAGCT | |
| B3 | TGCAG CATTGTTAG CAG GAT | |
| RT-qPCR | N_Sarbeco_F1 | CACATTGGCACCCGCAATC |
| N_Sarbeco_R1 | GAGGAACGAGAAGAGGCTTG | |
| N_Sarbeco_P1 | FAM-ACTTCCTCAAGGAACAACATTGCCA-BBQ |
El volumen de la mezcla de reacción fue de 12.5 μL (2X tampón de amplificación isotérmica del kit WarmStart Colorimetric LAMP); 10X de mezcla de cebadores (FIP, 16 μM; BIP, 16 μM; F3, 2 μM; B3, 2 μM; LF, 4 μM; LB, 4 μM) previamente preparados y 2,5 μL de ARN molde (cuantificado con el kit QubitTM RNA H S Assay), concentración de 7 ng/ μL; agua destilada estéril libre de DNAsas y RNAsas hasta completar volumen [24-26].
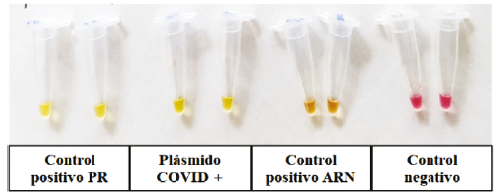
Las reacciones se prepararon sobre hielo y, posteriormente, se incubaron a 65 °C durante 30 min. Los ensayos fueron realizados por triplicado, como control negativo se empleó agua destilada estéril libre de DNAsas y RNAsas y como controles positivos, un plásmido previamente caracterizado denominado PR y una muestra clínica de ARN denominada (IB351), positiva para el virus SARS-CoV-2. La prueba fue evaluada por detección visual y considerada positiva al comparar la diferencia en el color con respecto al control negativo. Con estas condiciones de ensayo, se realizaron pruebas preliminares para evaluar la presencia del gen N en el plásmido Covid+.
RT-qPCR para SARS-CoV-2
Las condiciones de amplificación para este método se modificaron a partir del protocolo descrito por Corman et al. [12], para el diagnóstico y detección de SARS-CoV-2 por RT-qPCR. Se utilizó el kit comercial KiCqStartTM One-Step Probe RT-qPCR ReadyMixTM Sigma Aldrich®, se emplearon los cebadores y la sonda reportados para la amplificación del gen N del SARS-CoV-2 (tabla 1).
El volumen final de la mezcla de reacción fue de 10 μL: 2,5 μL de ARN molde, 5 μL del tampón de reacción 2X, 0,75 μL de primer F1 (10 μM), 1 μL de primer R1 (10 μM) y 0,25 μL de sonda (10 μM). El plásmido denominado Covid+ fue empleado como control positivo de amplificación y como control negativo se empleó agua destilada estéril libre de DNAsas y RNAsas. Los ensayos fueron realizados por triplicado.
La transcripción reversa se realizó a 55 °C durante 5 min., la amplificación a 94 °C durante 1 min para la activación inicial de la PCR, seguido de 45 ciclos de 94 °C por 10 segundos y 58 °C por 10 s. Con un ciclo adicional de 40 °C durante 30 s (total 50 min.). El ensayo se desarrolló en el equipo de tiempo real LightCycler® 480 (Roche).
Evaluación del límite de detección
El límite de detección del método LAMP y la qPCR fueron determinados usando diluciones seriadas del plásmido sintético COVID+ [15], una vez establecida la concentración del plásmido en la alícuota de trabajo, se calculó el número de copias y se evaluaron diluciones seriadas en un rango desde 182 millones de copias hasta ~1 copia por reacción (tabla 2). Estas diluciones fueron dispuestas por triplicado en tubos cónicos de 500 μL. Se realizó la lectura de los resultados según las especificaciones del método evaluado.
Tabla 2 Concentración de plásmido por factor de dilución.
| Dilución | Factor de dilución | Concentración (ng/μL) | Número de copias de plásmido |
|---|---|---|---|
| 1 | 1/10 | 7 | 182 424 242 |
| 2 | 1/100 | 7 x 10-1 | 18 242 424 |
| 3 | 1/1000 | 7 x 10-2 | 1 824 242 |
| 4 | 1/10 000 | 7 x 10-3 | 182 424 |
| 5 | 1/100 000 | 7 x 10-4 | 18 242 |
| 6 | 1/1 000 000 | 7 x 10-5 | 1 824 |
| 7 | 1/10 000 000 | 7 x 10-6 | 182 |
| 8 | 1/100 000 000 | 7 x 10-7 | 18 |
| 9 | 1/1 000 000 000 | 7 x 10-8 | 1,8 |
Ensayos RT-LAMP en muestras clínicas de ARN de pacientes para diagnóstico de SARS-CoV-2
La correlación del método RT-LAMP para el diagnóstico de SARS-CoV-2 se calculó comparando el resultado de la amplificación del gen N en 25 muestras clínicas de pacientes colombianos por el método RT-qPCR. Las muestras fueron previamente diagnosticadas por RT-qPCR así: 17 muestras positivas y 8 muestras negativas para el gen E del virus SARS-CoV-2 y empleado el gen RNAseP como control interno. Este último fue positivo para todas las muestras (datos no mostrados).
El ensayo de RT-LAMP se realizó en una mezcla de reacción de 12,5 μL que contenía 2X tampón de amplificación isotérmica del kit WarmStart Colorimetric LAMP; 10X de mezcla de cebadores (FIP, 16 μM; BIP, 16 μM; F3, 2 μM; B3, 2 μM; LF, 4 μM; LB, 4 μM) previamente preparados y 2,5 μL de ARN extraído de los hisopados de los pacientes, agua destilada estéril libre de DNAsas y RNAsas hasta completar volumen. La mezcla se incubó a 65 °C durante 30 min. en un baño seco.
RESULTADOS Y DISCUSIÓN
Presencia del gen N en el plásmido COVID+ con el método LAMP
Se confirmó la presencia del gen N en el plásmido sintético COVID+, desarrollando una prueba piloto con los reactivos de LAMP como se describe en la metodología. El cambio de color en la mezcla de reacción después de la incubación evidenció la presencia del gen N en el plásmido COVID+.
El resultado obtenido en la muestra ARN IB351 fue considerado positivo, lo que es consistente con lo reportado por Keller et al. (2020) [22], quienes evidenciaron variaciones en las tonalidades del indicador de color asociadas al cambio de pH en la solución de reacción en presencia de la hebra molde y la enzima. Así, los controles tanto positivo (PR y ARN IB351) como negativo (agua) se encuentran dentro de las especificaciones del ensayo (figura 1).
Evaluación del límite de detección
Los resultados de la evaluación del límite de detección del gen Npor el método LAMP, con la curva realizada a partir de las diluciones seriadas del plásmido COVID+, fueron determinados de manera visual, demostrando que 7x10-7 ng/μL de plásmido, equivalente a ~18 copias del gen N, pueden ser amplificados en un nivel detectable en 30 min (figura 2), esta es la concentración más baja donde un positivo pudo ser detectado por el cambio de color en la reacción. Los controles negativos se encuentran dentro de las especificaciones del ensayo.
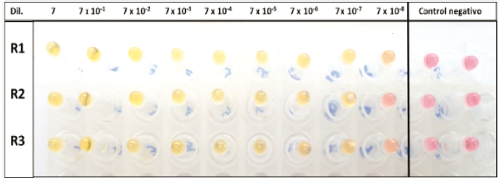
Figura 2 Evaluación del gen N-LAMP en el plásmido COVID+. R1: réplica 1; R2: réplica 2; R3: réplica 3.
Los resultados de la evaluación del límite de detección del gen N por el método de qPCR fueron establecidos mediante los valores CP calculados en el ensayo de detección de este gen a partir de las diluciones del plásmido COVID+ (tabla 3). Según el método lineal de la curva estándar, la concentración de las muestras detectadas se encontraba en un rango entre 9,66 hasta 5,55x 0-7 ng/μL aproximadamente. Los datos obtenidos sugieren reproducibilidad en cada uno de los puntos evaluados.
Tabla 3 Valores CP calculados en el ensayo gen N-qPCR de las diluciones del plásmido COVID+
| Concentración ng/μL plásmido | Réplica 1 CP | Réplica 2 CP | Réplica 3 CP | Concentración |
|---|---|---|---|---|
| 7 | 10,19 | 10,10 | 10,08 | 9,66 |
| 7 x 10-1 | 13,85 | 14,25 | 14,12 | 5,99 x 10-1 |
| 7 x 10-2 | 17,83 | 17,75 | 17,89 | 5,74 x 10-2 |
| 7 x 10-3 | 21,07 | 21,12 | 21,33 | 5,97 x 10-3 |
| 7 x 10-4 | 24,63 | 24,69 | 24,82 | 5,45 x 10-4 |
| 7 x 10-5 | 27,1 | 27,49 | 27,39 | 8,31 x 10-5 |
| 7 x 10-6 | 30,83 | 30,94 | 30,97 | 8,25 x 10-6 |
| 7 x 10-7 | 32,96 | 32,89 | 32,2 | 5,55 x 10-7 |
| 7 x 10-8 | N/D | N/D | N/D | N/A |
| Control negativo | N/D | N/D | N/D | N/A |
N/D: no detectado. N/A: no aplica
Los resultados obtenidos de la comparación del límite de detección de ambos métodos para la amplificación del gen N, realizados a partir de las diluciones seriadas del plás-mido COVID+ se muestran en la tabla 4.
Tabla 4 Comparación del límite de detección del método LAMP y qPCR.
| Concentración ng/μL plásmido | LAMP | qPCR | Número de copias de plásmido |
|---|---|---|---|
| 7 | + | + | 182 424 242 |
| 7 x 10-1 | + | + | 18 242 424 |
| 7 x 10-2 | + | + | 1 824 242 |
| 7 x 10-3 | + | + | 182 424 |
| 7 x 10-4 | + | + | 18 242 |
| 7 x 10-5 | + | + | 1824 |
| 7 x 10-6 | + | + | 182 |
| 7 x 10-7 | + | + | 18 |
| 7 x 10-8 | - | - | 1,8 |
T2 RT-LAMP y RT-qPCR para el diagnóstico de SARS-CoV-2
Una vez realizada la RT-LAMP en las 25 muestras de hisopado nasofaríngeo, el indicador de pH (rojo fenol) permitió evidenciar a simple vista, que 17 de las muestras evaluadas mostraron cambio de color rosa a amarillo, resultado que indicó la amplificación del gen N con el método RT-LAMP, mientras el control negativo y las muestras restantes mantuvieron el color rosa (figura 3). Estos resultados fueron comparados con el método estándar RT-qPCR (tabla 5), se obtuvo una concordancia del 100 % entre los dos métodos para el gen N, lo cual demuestra que la sensibilidad diagnóstica de RT-LAMP fue del 100% con una tasa de falsos negativos y falsos positivos de 0 % de acuerdo con el diagnóstico previamente realizado por RT-qPCR para el gen E (datos no mostrados).
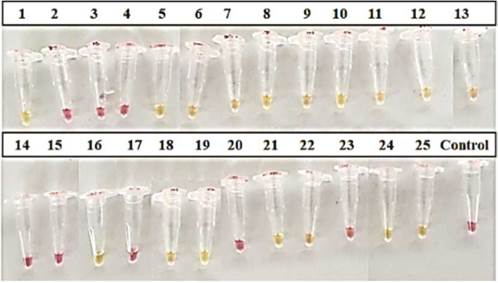
Figura 3 Detección del gen N de SARS-CoV-2 por RT-LAMP en muestras clínicas de pacientes colombianos.
Tabla 5, Aplicación del método RT-LAMP para la detección del SARS-CoV-2 en muestras clínicas.
| Tubo N.° | Muestras | qPCR CP | RT-LAMP | Tubo N.° | Muestras | qPCR CP | RT-LAMP | ||
|---|---|---|---|---|---|---|---|---|---|
| 1 | IB202 | 25,75 | + | A | 14 | IC915 | N/D | - | R |
| 2 | IB276 | N/D | - | R | 15 | IC581 | N/D | - | R |
| 3 | IB266 | N/D | - | R | 16 | IC432 | 33,24 | + | A |
| 4 | IC185 | N/D | - | R | 17 | ID450 | N/D | - | R |
| 5 | IB344 | 33,40 | + | A | 18 | ID548 | 25,52 | + | A |
| 6 | IB350 | 31,83 | + | A | 19 | ID684 | 32,99 | + | A |
| 7 | IB478 | 22,00 | + | A | 20 | ID792 | N/D | - | R |
| 8 | IB690 | 22,85 | + | A | 21 | ID888 | 19,90 | + | A |
| 9 | IB858 | 24,79 | + | A | 22 | ID913 | 33,44 | + | A |
| 10 | IB999 | 31,95 | + | A | 23 | IE078 | N/D | - | R |
| 11 | IB896 | 32,63 | + | A | 24 | IE171 | 33,12 | + | A |
| 12 | IC163 | 33,48 | + | A | 25 | IE278 | 33,18 | + | A |
| 13 | IC173 | 32,97 | + | A | Control | C- | N/D | - | R |
A: amarillo. R: rosa. N/D: no detectado
La mayoría de las pruebas de diagnóstico a nivel mundial se realizan con el método de RT-qPCR, se amplifica el ARN viral obtenido a partir de muestras nasofaríngeas. Sin embargo, frente a la continua demanda global y la competencia por reactivos y recursos, con el objeto de minimizar la dependencia de cualquier metodología, en este estudio hemos descrito el procedimiento para utilizar RT-LAMP como un método rentable, sensible, alternativo, de alto rendimiento y con resultados similares a los obtenidos por RT-qPCR para detectar SARS-CoV-2 en muestras clínicas.
Fueron empleados los cebadores diseñados para la amplificación del gen N de la nucleo-cápside del SARS-CoV-2, reportados por Zhang et al. [26] y previamente evaluados por Buck et al. [24] quienes reportaron correlación total entre los resultados obtenidos en RT-qPCR y RT-LAMP; confirmaron la especificidad y posible reactividad cruzada con ARN humano del juego de cebadores, además de evaluar la especificidad, en muestras clínicas negativas para SARS-CoV-2 previamente inoculadas con 14 tipos de virus como Mers-CoV, influenza A y B, algunas cepas de coronavirus estacionales, metap-neumovirus (MPV), virus sincitial respiratorio (RSV) y virus parainfluenza tipo 3 y 4 (PIV3, PIV4). En ninguno de los ensayos de RT-LAMP con el gen Nse obtuvo señal de amplificación, confirmando la especificidad de los cebadores para SARS-CoV-2.
El límite de detección del método LAMP fue de ~18 copias del gen Npor 12,5 μL de reacción, igual al reportado para la RT-qPCR, sugiriendo que este método es igual de sensible al método estándar para la detección de SARS-CoV-2. Estos resultados son equiparables a los descritos por Ganguli et al. [27], quienes obtuvieron un límite de detección de RT-LAMP equivalente a los obtenidos con RT-qPCR (50 copias por jxL de reacción). Renfei et al. [28] señalaron que la RT-LAMP detectó una cantidad de 30 copias en 25 μL de reacción, ligeramente más bajo que la RT-qPCR para el gen RdRp (gen de la ARN polimerasa dependiente de ARN del virus SARS-CoV-2).
El desempeño del método RT-LAMP, usando muestras clínicas de ARN positivas para SARS-CoV-2, determinó una concordancia del 100 % con respecto a la RT-qPCR (CP: 19,9-34,95), con la misma sensibilidad diagnóstica que el método estándar para el gen N de SARS-CoV-2 (p<0,01, test de chi cuadrado). Estos resultados son comparables a los obtenidos por Ganguli et al. [27], quienes obtuvieron una sensibilidad del 100 % del método RT-LAMP para el gen N en 10 muestras clínicas provenientes de pacientes positivos para el virus. De igual forma Referi et al. [28] reportaron una consistencia del 100 % en los datos entre RT-LAMP y RT-qPCR para el gen RdRp en 24 muestras clínicas de pacientes diagnosticados con la enfermedad.
El tiempo empleado en este estudio para el procesamiento de las muestras clínicas para la detección del gen N del SARS-CoV-2 por el método RT-LAMP fue de 30 min., lo cual corresponde a lo reportado en la literatura [21, 22, 24], constituyendo la principal ventaja frente al tiempo de procesamiento por la técnica estándar RT-qPCR que implica en promedio 120 min.
A partir de los resultados obtenidos y para el número de muestras clínicas evaluadas, puede sugerirse que el ensayo colorimétrico de RT-LAMP permite realizar una detección rápida y confiable del gen N del SARS-CoV-2 a partir de hisopados nasofaríngeos de pacientes colombianos, con un límite de detección de aproximadamente 18 copias del gen viral N, similar al establecido con la prueba estándar RT-qPCR, aplicada rutinariamente en el Instituto Nacional de Salud de Colombia y que en este estudio se usó como prueba de referencia. Este resultado es comparable con el reportado por Huang et al. [17], quienes evaluaron por este método, 8 muestras positivas y 8 muestras negativas registrando como límite detección para RT-LAMP aproximadamente 80 copias del gen. Los hallazgos encontrados por Tanner et al. [21] demostraron que la sensibilidad de la técnica en <10 copias y la rapidez en <30 min. para la detección, resultados que son consistentes con los obtenidos en este estudio.
Implementar esta técnica como prueba de diagnóstico diario, permitiría una disminución en los costos operativos, la rápida lectura de los resultados y el incremento del número de pruebas aplicadas a la población ya que, además de las ventajas mencionadas, al no requerir equipos de alto costo es aplicable en lugares con infraestructura limitada. Es probable que esta metodología dé paso a la aplicación de pruebas en puntos de atención fuera del ámbito del laboratorio de diagnóstico clínico, ampliando la expectativa de ser validado por las autoridades sanitarias competentes, tal como está ocurriendo en países de Latinoamérica.