Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Obstetricia y Ginecología
Print version ISSN 0034-7434On-line version ISSN 2463-0225
Rev Colomb Obstet Ginecol vol.61 no.4 Bogotá Oct./Dec. 2010
Dabeiba A. García, M.Sc.*, Markus Schmitt, Ph.D.**, Ángel Cid-Arregui, Ph.D.***, Marcos Castillo, M.D.****, Ignacio Briceño, Ph.D.*****, Fabio A. Aristizábal, Ph.D.******
Recibido: abril 30/10 - Aceptado: noviembre 12/10
* Centro de Investigaciones Odontológicas, Pontificia Universidad Javeriana. Bogotá (Colombia).
** Research Program Infection and Cancer, German Cancer Research Center (DKFZ). Heidelberg (Alemania).
*** Translationale Immunologie, German Cancer Research Center (DKFZ). Heidelberg (Alemania).
**** Facultad de Medicina, Universidad de La Sabana. Dinámica-IPS. Bogotá (Colombia).
***** Dinámica-IPS. Bogotá (Colombia).
****** Departamento de Farmacia, Facultad de Ciencias, Universidad Nacional de Colombia. Bogotá (Colombia). Correo electrónico: faaristizabalg@unal.edu.co
RESUMEN
Objetivo: genotipificar el VPH en muestras de pacientes con diferentes grados histopatológicos de lesión cervical.
Materiales y métodos: estudio de corte transversal en muestras de cepillado cervical de mujeres con diferentes grados de lesiones cervicales histopatológicas en la ciudad de Bogotá en el año 2009. Se utilizó Luminex® xMAP® una técnica altamente sensible, reproducible y automatizada, y Reverse Line Blot (RLB).
Resultados: las infecciones por los tipos de VPH de 16 y 18 fueron las más frecuentes en todos los tipos de patología del cérvix. Las infecciones mixtas predominaron con un 78,04% de los casos con patología negativa, 85,7% NIC I, 81,25% NIC II, 78,78% NIC III y 71,42% cáncer cervical. La concordancia entre los dos métodos fue del 67%.
Conclusión: las infecciones por los tipos de VPH 16 y 18 predominan en la población estudiada.
Palabras clave: papilomavirus, displasia cervical, patología, biología molecular.
SUMMARY
Objective: genotyping HPV in samples taken from patients having different histopathological degrees of cervical injury.
Materials and methods: this was a cross-sectional study of cervical scrape samples taken from females having different degrees of histopathological cervical injury in Bogotá during 2009. Luminex® xMAP® was used, which is a highly sensitive, reproducible and automated technique, as well as reverse line blot (RLB).
Results: infections caused by HPV types 16 and 18 were the most frequently found types of pathology of the cervix. Mixed infections predominated (78.04%); amongst cases having negative pathology, 85.7% was found for cervical intraepithelial neoplasia (CIN) I, 81.25% for CIN II, 78.78% for CIN III and 71.42% for cervical cancer. Agreement between both methods was 67%.
Conclusion: infection by VPH types 16 and 18 predominated in the population being studied.
Key words: papilloma virus, cervical dysplasia, pathology, molecular biology.
INTRODUCCIÓN
El cáncer cervical es un problema de salud pública a nivel mundial, especialmente en países en vías de desarrollo, y es una de las principales causas de morbilidad y mortalidad en mujeres.1,2 Hay 500.000 casos nuevos de cáncer cervical por año en el mundo y es la primera causa de muerte en mujeres en países en vías de desarrollo y la segunda en el mundo. No obstante, se trata de una patología altamente curable si se detecta a tiempo.3,4 El cáncer de cuello está precedido por una serie de lesiones precursoras que generalmente se desarrollan de modo lento precediendo al cáncer invasivo, típicamente durante un período mayor a 10 años.5 Estas lesiones precursoras son las anteriormente llamadas displasias o neoplasias intraepiteliales cervicales (NIC) y la actualmente denominada lesión escamosa intraepitelial (LEI).6 Estas lesiones son detectables mediante métodos de tamizaje, tales como la citología cervical o por medio de la detección de agentes asociados al desarrollo de estas lesiones precancerosas como el Virus del Papiloma Humano (VPH).7
El VPH es un virus ADN que desempeña un papel importante en el desarrollo del cáncer cervical.8,9 Hoy en día, se considera que la infección por VPH es una causa necesaria, pero no suficiente, para el desarrollo de esta neoplasia.10 Alrededor del 99% de mujeres con cáncer cervical presentan esta infección.10,11 Igualmente, se ha descrito su presencia en las lesiones precancerosas antes descritas. El virus actuaría conjuntamente con otros factores, no totalmente conocidos para el desarrollo final de la lesión invasora.4,12
La transmisión sexual es el mecanismo dominante para adquirir VPH, aunque la mayoría de las infecciones son transitorias y se eliminan a través del sistema inmune.5 Alrededor de 118 tipos del Virus del Papiloma han sido definidos completamente, 40 de estos han sido identificados en el tracto genital y se subdividen en: grupos de alto riesgo, riesgo indeterminado y bajo riesgo. Existen 15 tipos de VPH de alto riesgo (16, 18, 31, 33, 35, 39, 45, 51, 52, 56, 58, 59, 68, 73, 82)13 que son frecuentemente asociados con el cáncer cervical invasivo.13 El descubrimiento de que los VPH están etiológicamente ligados con el cáncer cervical ha estimulado los esfuerzos para aplicar este conocimiento con el fin de prevenir su infección mediante la vacunación y el desarrollo de técnicas que faciliten su detección.5
La detección temprana del virus podría revelar pacientes con lesiones precursoras. En este sentido, la PCR es el método más sensible para la detección de secuencias del ADN del VPH en muestras clínicas.14-16 Una de las técnicas basadas en la PCR más utilizadas para genotipificar el VPH es el Reverse Line Blot (RLB). Esta técnica utiliza los iniciadores denominados GP5+/GP6+, que amplifican la región L1 de 37 tipos del VPH. La técnica se basa en la hibridación de sondas para cada tipo viral sobre una membrana con los productos de PCR marcados con biotina que, a su vez, son conjugados con estreptavidina-peroxidasa y, finalmente, revelados con ECL en una película de autoradiografía.17 En los últimos años, se ha descrito una técnica automatizada basada en arreglos con perlas denominada xMAP® de Luminex®, la cual tiene un principio similar al RLB. Con respecto a los iniciadores, estos fueron mejorados incluyendo 8 iniciadores sentido adicionales y 2 antisentido, denominados BSGP5+/BIO-BSGP6+, además de 1 juego de iniciadores para β-globina denominados MS3/MS10 con el fin de detectar 11 tipos virales más, además de incluir un control de calidad de la muestra. La hibridación no se lleva a cabo en fase sólida, sino en fase líquida mediante el uso de perlas carboxiladas. Finalmente, la detección se lleva a cabo mediante un sistema automatizado, evitando de esta forma errores en la interpretación de los resultados.13
A la fecha, son pocos los estudios que genotipifican el VPH en muestras de mujeres en nuestra población por técnicas altamente sensibles como el Luminex® xMAP®, método que detecta 48 tipos virales de una manera automatizada, con lo cual se evitan errores en su interpretación desde un diagnóstico histopatológico negativo hasta diferentes grados de NIC y cáncer cervical.
Por lo tanto, este estudio busca determinar la prevalencia del VPH mediante genotipificación en muestras de pacientes con diferentes grados histopatológicos de lesión escamosa intraepitelial y evaluar la concordancia de los métodos RLB y xMAP® en la población objeto de estudio.
MATERIALES Y MÉTODOS
Diseño
Se hizo un estudio de corte transversal en mujeres que asistieron a consulta de colposcopia durante un período de ocho meses a las siguientes instituciones: Hospital San Ignacio, Fundación Santafé o Clínica Teletón (hospitales universitarios privados de referencia que atienden población con aseguramiento contributivo), el Hospital de Engativá (hospital público de mediana complejidad que atiende pacientes del aseguramiento subsidiado) y Dinámica-IPS (centro privado de diagnóstico), todos ubicados en la ciudad de Bogotá. Las pacientes aceptaron participar voluntariamente en el estudio por medio de la firma de un consentimiento informado. No se incluyeron pacientes en estado de embarazo, que hayan recibido tratamiento para la neoplasia cervical y/o infección por VPH, con antecedentes de terapia antineoplásica o inmunomoduladora. El ingreso fue independiente de que presentaran o no lesiones intraepiteliales del cuello uterino. El tamaño muestral fue por el número de mujeres estudiadas durante un período de 12 meses. Asimismo, se realizó un muestreo secuencial consecutivo por conveniencia.
Adquisición y procesamiento de las muestras
A cada paciente se le tomó una muestra de cepillado cervical para las pruebas moleculares y biopsia para el diagnóstico histopatológico. Los citocepillos se colocaron inmediatamente en buffer fosfato salino 1X y timerosal al 0,05%, posteriormente fueron digeridas con una solución de 20 mg/ml de proteinasa K (Bioline®) e incubadas a 37ºC durante toda la noche. Transcurrido el tiempo de incubación, se inactivaron las muestras a 95ºC por 5 minutos. Subsiguientemente, fueron centrifugadas a 10.000 rpm por 5 minutos y almacenadas a -20°C.
La calidad del ADN fue confirmada por PCR con los iniciadores PC03/PC04, los cuales amplifican un fragmento de 110 pb del gen constitutivo de β-globina.18 La mezcla de la reacción contenía buffer de PCR 1X, MgCI2 3,5mM,mezcladedNTPs200µMdecadauno, PC03 1 µM, PC04 1 µM, 1,25 U de Taq polimerasa (Promega®) y agua destilada estéril hasta completar un volumen de reacción de 25 µl. Las reacciones fueron corridas en el equipo My CyclerTM Termal Cycler (Biorad®) con las siguientes condiciones: 4 minutos a 94ºC, seguidos por 40 ciclos de amplificación de 94ºC por 1 minuto, 55ºC por 1 minuto y 72ºC por 1 minuto, y una extensión final a 72ºC por 4 minutos. Como control positivo, se usó ADN linfocitario y como control negativo agua destilada. El producto de la PCR se visualizó mediante una electroforesis en gel de agarosa al 1% teñido con bromuro de etidio identificando la banda amplificada de 110 pb. Esta electroforesis se corrió durante una hora a 100 voltios en buffer TBE1X utilizando el equipo Biorad® Power PAC 3000 y visualizado con un digitalizador de geles con el software Quantity One de Biorad®.
Genotipificación de VPH mediante RLB
A todas las muestras positivas para el gen de ß-globina, se les realizó una PCR, utilizando los iniciadores de la región L1 del VPH GP5+/BioGP6+.17 La mezcla de la reacción contenía 10 µl de muestra, buffer de PCR 1X, MgCI2 3,5 mM, mezcla de dNTPs 800 mM (200 mM de cada uno), GP5+ 1 µM, Bio-GP6+ 1 µM, 1,25 U de Taq polimerasa (Promega®) y agua destilada estéril hasta completar un volumen de reacción de 50 µL. Cada reacción se llevó a cabo en un termociclador My CyclerTM (Biorad®) y las condiciones de PCR fueron: un paso de denaturación a 94ºC por 4 minutos, seguido de 40 ciclos de amplificación y cada ciclo incluía un paso de denaturación a 94ºC por 1 minuto, un paso de anillamiento a 40ºC por 2 minutos y un paso de elongación a 72ºC por 1½ minuto. La elongación final fue prolongada por 4 minutos. Como control positivo se utilizaron los ADN extraídos de las líneas celulares de cáncer cervical, HeLa y SiHa, pertenecientes al Banco de Líneas Celulares, del grupo de farmacogenética de la Universidad Nacional de Colombia.
En un sistema Minibloter® 45 de Immunetics®, se unen covalentemente oligosondas que presentan grupos amino a una membrana que presenta grupos carboxilo, la cual es activada con EDAC 16% (P/V) (1-etil-3-(3-dimetilaminopropil) carbodiimida), las oligosondas acopladas detectan 37 tipos virales de VPH, las cuales fueron las reportadas por Van den Brule.17 Posteriormente, la membrana se gira 90 grados para realizar la hibridización de los productos denaturados de la PCR a 42ºC por 45 minutos y seguido por dos lavados por 10 minutos a 52ºC con 2X SSPE (1X SSPE contiene 0,18M NaCl, 10 mM NaH2PO4 y 1 mM EDTA pH 7,4; Invitrogen™) y 0,5% de SDS. Luego se incuba la membrana con el conjugado estreptividina peroxidasa (Roche®) por 45 minutos a 42ºC (dilución 1:4.000), seguido por dos lavados en 2X SSPE y 0,5% SDS por 10 minutos a 42ºC y dos lavados con 2X SSPE por 5 minutos a temperatura ambiente. La detección fue realizada usando detección con ECLTM (GE® Healthcare UK, AmershamTM) seguido por la exposición de la película toda la noche (Amersham Hyperfilm™ ECL™). La película fue revelada en el equipo Medical Film Procesador Modelo SRX-101 A Konica Minolta Medical & Graphic, Inc.
Genotipificación mediante Luminex® xMAP®
Inicialmente, se realizó una PCR multiplex con los iniciadores reportados por Markus y sus colegas en el 2008,15 denominados BSGP5+/GP6+, que constan de 9 iniciadores sentido 200nM BSGP5+ y 3 iniciadores antisentido 400nM BIO-BSGP6+, además de un par de iniciadores adicionales que amplifican el gen de β-globina 300 nM (MS3/ MS10).13 Las sondas con un grupo amino en el extremo 5’ para cada uno de los 48 tipos virales que se detectan son acopladas a las perlas carboxiladas mediante el procedimiento de carbodiimida, descrito por Dunbar y colaboradores en el 2003.19 Para cada combinación de sonda y perla, se tomaron 2,5 millones de perlas carboxiladas en 25 µL de 0,1 M ácido 2-(N-morfolino)-etanosulfónico, pH 4,5 (MES), con 400 pmol de sonda y 200 µg de N-(3-dimetilaminopropil)-N-etilcarbodiimida (EDC), se realizó la mezcla y se mantuvo en oscuridad bajo agitación por 30 minutos, se repitió el paso de la adición del EDC y la incubación. Posteriormente, se lavó con 0,5 ml de 0,2 g/l de Tween-20 y una vez con 0,5 ml de 1,0 g/l de SDS. Finalmente, se almacenó a 4°C en solución de TE. La hibridación se realizó en placas de 96 pozos con 10 µl del producto de la PCR con 33 µl de solución de hibridación 0,15 M TMAC (75 mM Tris-HCl, 6 mM EDTA, 1,5 g/l Sarkosyl, pH 8,0) y 2000 perlas acopladas a las sondas. Esta mezcla fue incubada a 95°C durante 10 minutos e inmediatamente colocada en hielo por 2 minutos, posteriormente se colocó a 41°C durante 30 minutos en agitación; luego se transfirieron las muestras a una placa de lavado (Millipore®, Bedford, MA) preequilibradas con solución de bloqueo (PBS–0,2 g/l Tween 20-2,0 M cloruro de tetrametilamonio), y se realizó un lavado con solución de bloqueo. Luego se adicionaron 75 µL de estreptavidina-R-ficoeritrina (sondas moleculares) 1:1.600 en 2.0 M TMAC y se incubó 20 minutos a temperatura ambiente en agitación, se lavó 3 veces con solución de bloqueo y, finalmente, se resuspendió en 100 µL de esta misma solución. La lectura se realizó en Luminex® 100 Analyzer, en donde se detecta el color interno de cadaperla ylafluorescenciadel reportero que en este caso fue la ficoeritrina, la media de la intensidad de fluorescencia (MIF) fue calculada para 100 perlas en cada muestra.13,20
De cada una de las pacientes incluidas en el estudio, se indagaron datos sociodemográficos tales como edad, estrato socioeconómico y factores de riesgo tales como edad de la primera relación sexual, número de compañeros sexuales, número de embarazos, edad del primer parto, uso de anticonceptivos, infecciones vaginales, enfermedades de transmisión sexual y antecedentes familiares de cáncer. Además, se determinó el tipo de VPH en los diferentes tipos de lesión escamosa intraepitelial o cáncer invasor.
Análisis estadístico
Los datos sociodemográficos de la población se analizaron mediante el software Statistix 9, con el cual se hallaron porcentajes, correlación de Pearson y chi-cuadrado. La significancia se determinó a un valor de p menor a 0,05. Los resultados de genotipificación fueron analizados hallando porcentajes mediante Excel®. Finalmente, para comparar los dos métodos de genotipificación de VPH y ver su concordancia se utilizó un estadístico kappa.21
RESULTADOS
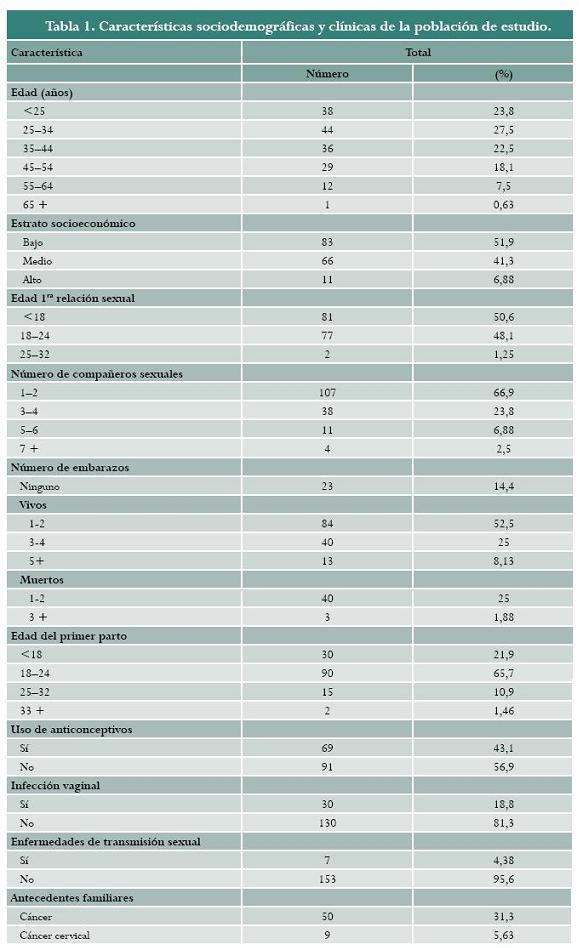
Durante el período de estudio, se incluyeron 160 mujeres con una edad promedio de 35±12 años. La media de la edad a la que se presentó la primera relación sexual fue de 17±2 años y un primer parto a una edad del promedio de 20±3 años. Los otros resultados sociodemográficos se muestran en la tabla 1.
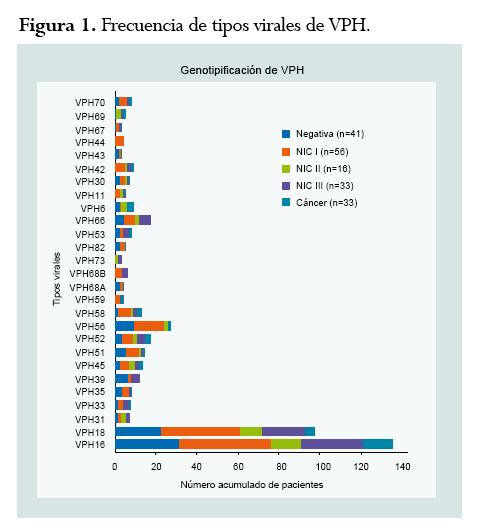
Los diagnósticos histopatológicos encontrados fueron: negativa para lesiones intraepiteliales cervicales en 41 mujeres (25,6%); 56 pacientes (35%) tenían NIC I, 16 pacientes (10%) NIC II, 33 (20,6%) NIC III y 14 pacientes (8,8%) tenían cáncer cervical. En todos los casos estudiados se detectó VPH. La frecuencia de los diferentes tipos de VPH por tipo de diagnóstico histológico se presenta en la Figura 1. Se observa un predominio de los tipos virales 16 y 18 en todos los grados histopatológicos. Los siguientes tipos virales más frecuentes fueron: VPH 56 (con una frecuencia del 16,8%) y VPH 52 y 66 (con una frecuencia del 10,6%). Por otra parte, las infecciones múltiples fueron más frecuentes que las infecciones simples. En los casos de histopatología negativa, las infecciones múltiples se observaron en el 78,04%, así como en el 85,7% de NIC I, 81,25% de NIC II, 78,78% de NIC III y 71,42% de cáncer cervical.
Se genotipificó el VPH por xMAP® en las 160 pacientes incluidas en el estudio. Sin embargo, 35 pacientes no se incluyeron para genotipificación paralela por Reverse Line Blot, ya que estas muestras no revelaron una banda clara y bien definida en el gel de agarosa para el gen de β-globina y no cumplieron con los requerimientos para su procesamiento por dicha técnica. La concordancia de las dos metodologías xMAP® y RLB mostró un valor del estadístico Kappa de 0,672. Además de las muestras positivas por las dos metodologías, el 67,2% coincidieron en el tipo viral detectado.
DISCUSIÓN
El presente trabajo muestra que los tipos virales más frecuentes en la población de estudio fueron VPH 16, seguido por VPH 18. Estos resultados son similares a lo reportado por Muñoz y colaboradores en el 2003.22 Las frecuencias de aislamiento de los tipos VPH 56, VPH 52 y VPH 66 son superiores a las reportadas por Clifford y colaboradores en el 2005, que fueron del 7,1%, 5,3% y 4,1%, respectivamente.23 Con respecto a la detección de infecciones simples y múltiples, en este estudio se obtuvieron frecuencias de 19,4% para infecciones simples y de 80,6% para infecciones múltiples, mientras que Muñoz y colaboradores encontraron frecuencias de 91,9% para infecciones simples y 8,1% para infecciones múltiples.22 Esta diferencia puede deberse a la distinta sensibilidad de las técnicas empleadas y a que la técnica utilizada en este estudio evaluaba 48 tipos virales, en comparación con 37 tipos virales detectados por los estudios citados.
La detección del VPH se ha convertido en una ayuda práctica para el diagnóstico en el cribado, la detección y el seguimiento de neoplasias cervicales, tal como en estudios epidemiológicos de la infección.24,25 No obstante, el VPH se ha divido en grupos de alto riesgo (AR), posiblemente de alto riesgo (pAR) y de bajo riesgo (BR), por lo que se hace necesario no sólo detectar el virus sino también genotipificarlo, con lo cual el pronóstico de la paciente es más cercano.22
Con respecto a la calidad, todas las muestras cumplieron los requerimientos por Luminex® xMAP®, la cual muestra una alta sensibilidad con los iniciadores MS3/MS10 del gen de β-globina, ya que detecta menos de 10 ng de ADN genómico.15 Sin embargo, para el RLB fue necesario evaluar las muestras por PCR convencional con geles de agarosa, la cual presenta una sensibilidad de más de 10 ng.26 Por esta razón, sugerimos que la exclusión que se debió hacer de las 35 muestras para el análisis por RLB se explica por la baja sensibilidad de PCR visualizada con electroforesis en agarosa para β-globina.
Finalmente, nuestro estudio mostró que la concordancia fue buena entre los dos métodos evaluados, con un índice kappa de 0,672. No obstante, son pocos los estudios en lo que se comparan estas dos técnicas. En uno de ellos, hecho por Schmitt y colaboradores,20 en 94 muestras se observó una concordancia muy buena entre estas dos técnicas, con un índice kappa de 0,92. En otro estudio, llevado a cabo por Geraets y colaboradores en 2009,27 el índice kappa para dicha concordancia fue de 0,88. Por su parte, en 2009 Feng y sus colegas28 encontraron un índice kappa de 0,85, aunque el xMAP® se basaba en otro conjunto de iniciadores.
CONCLUSIÓN
Los tipos virales 16 y 18 son los más frecuentemente detectados en pacientes que acuden a las clínicas de patología cervical y colposcopia. La concordancia entre las dos metodologías evaluadas es buena. Sin embargo, la técnica de Luminex® xMAP® surge como una nueva alternativa, aunque se necesitan nuevos estudios para corroborar estos hallazgos.
AGRADECIMIENTOS
A los departamentos de Ginecología y Obstetricia y de Patología de Dinámica-IPS, Hospital San Ignacio, Fundación Santafé, Hospital de Engativá y Clínica Teletón. Finalmente, a la Facultad de Medicina de la Universidad de La Sabana por su apoyo en la financiación.
REFERENCIAS
1. American Cancer Society. Cancer facts and figures. Washington, DC.: American Cancer Society; 1997. [ Links ]
2. Saslow D, Castle PE, Cox JT, Davey DD, Einstein MH, Ferris DG, et al. American Cancer Society Guideline for human papillomavirus (HPV) vaccine use to prevent cervical cancer and its precursors. CA Cancer J Clin 2007;57:7-28. [ Links ]
3. World Health Organization. State of the art of new vaccines research and development. Initiative for Vaccine Research. Geneva, Switzerland: World Health Organization; 2003. [ Links ]
4. Thompson J, Thomas LK, Shroyer KR. Human papillomavirus: molecular and cytologic/histologic aspects related to cervical intraepithelial neoplasia and carcinoma. Hum Pathol 2008;9:154-66. [ Links ]
5. Unger ER, Barr E. Human papillomavirus and cervical cancer. Emerg Infect Dis 2004;10:2031-2. [ Links ]
6. Crum CP. Symposium part 1: Should the Bethesda System terminology be used in diagnostic surgical pathology?: Point. Int J Gynecol Pathol 2003;22:5-12. [ Links ]
7. Leinonen M, Nieminen P, Kotaniemi-Talonen L, Malila N, Tarkkanen J, Laurila P, et al. Age-specific evaluation of primary human papillomavirus screening vs. conventional cytology in a randomized setting. J Natl Cancer Inst 2009;101:1612-23. [ Links ]
8. Kisseljov FL, Kisseljova NP, Kobzeva VK, Gritsko TM, Semenova LA, Pavlova LS, et al. State of Human Papilloma Virus ADN in Cervical Carcinomas. Molecular Biology 2001;35:399-404. [ Links ]
9. Wolf JK, Ramirez PT. The molecular biology of cervical cancer. Cancer Invest 2001;19:621-9. [ Links ]
10. Bosch FX, de Sanjosé S. Chapter 1: Human papillomavirus and cervical cancer: burden and assessment of causality. J Natl Cancer Inst Monogr 2003;31:3-13. [ Links ]
11. Walboomers JM, Jacobs MV, Manos MM, Bosch FX, Kummer JA, Shah KV, et al. Human papillomavirus is a necessary cause of invasive cervical cancer worldwide. J Pathol 1999;189:12-9. [ Links ]
12. Reeves WC, Brinton LA, García M, Brenes MM, Herrero R, Gaitan E, et al. Human papillomavirus infection and cervical cancer in Latin America. N Engl J Med 1989;320:1437-41. [ Links ]
13. Schmitt M, Dondog B, Waterboer T, Pawlita M. Homogeneous amplification of genital human alpha papillomaviruses by PCR using novel broadspectrum Gp5+ and Gp6+ primers. J Clin Microbiol 2008;46:1050-9. [ Links ]
14. Gravitt P, Hakenewerth A, Stoerker J. A direct comparison of methods proposed for use in widespread screening of human papillomavirus infection. Mol Cell Probes 1991;5:65-72. [ Links ]
15. Schiffman MH, Bauer HM, Lorincz AT, Manos MM, Byrne JC, Glass AG, et al. Comparison of Southern blot hybridization and polymerase chain reaction methods for the detection of human papillomavirus DNA. J Clin Microbiol 1991;29:573-7. [ Links ]
16. Cope JU, Hildesheim A, Schiffman MH, Manos MM, Lorincz AT, Burk RD, et al. Comparison of the hybrid capture tube test and PCR for detection of human papillomavirus DNA in cervical specimens. J Clin Microbiol 1997;35:2262-5. [ Links ]
17. van den Brule AJ, Pol R, Fransen-Daalmeijer N, Schouls L, Meijer C, Snijders PJ. GP5+/6+ PCR followed by reverse line blot analysis enables rapid and high-throughput identification of human papillomavirus genotypes. J Clin Microbiol 2002;40:779-87. [ Links ]
18. Arias YR, Carrillo EF, Aristizábal FA. Plasma DNA restoration for PCR applications. J Clin Pathol 2007;60:952-4. [ Links ]
19. Dunbar SA, Vander Zee CA, Oliver KG, Karem KL, Jacobson JW. Quantitative, multiplexed detection of bacterial pathogens: DNA and protein applications of the Luminex LabMAP system. J Microbiol Methods 2003;53:245-52. [ Links ]
20. Schmitt M, Bravo IG, Snijders PJ, Ghissann L, Pawlita M, Waterboer T. Bead-based multiplex genotyping of human papillomaviruses. J Clin Microbiology 2006;44:504-12. [ Links ]
21. Cortés-Reyes E, Rubio-Romero JA, Gaitán-Duarte H. Métodos estadísticos de evaluación de la concordancia y la reproducibilidad de pruebas diagnósticas. Rev Colomb Obstet Ginecol 2010:61:247-55. [ Links ]
22. Muñoz N, Bosch FX, de Sanjosé S, Herrero R, Castellsague X, Shah KV, et al. Epidemiologic classification of human papillomavirus types associated with cervical cancer. N Engl J Med 2003;348:518-27. [ Links ]
23. Clifford GM, Gallus S, Herrero R, Muñoz N, Snijders PJF, Vaccarella S, et al. The IARC HPV Prevalence Surveys Study Group. Worldwide distribution of human papillomavirus types in cytologically normal women in the International Agency for Research on Cancer HPV prevalence surveys: a pooled analysis. Lancet 2005;366:991-8 [ Links ]
24. Wei YC, Chou YS, Chu TY. Detection and typing of minimal human papillomavirus DNA in plasma. Int J Gynaecol obstet 2007;96:112-6. [ Links ]
25. Burd EM. Human Papillomavirus Detection and Utility of Testing. Clinical Microbiology Newsletter 2007;29:159-67. [ Links ]
26. Ahn SJ, Costa J, Emanuel JR. PicoGreen quantitation of DNA: effective evaluation of samples pre- or post-PCR. Nucleic Acids Res 1996;24:2623-5. [ Links ]
27. Geraetsa DT, Heidemanb DA, de Koninga MN, Snijdersb PJ, van Alewijka DC, Meijerb CJ, et al. High-throughput genotyping of high-risk HPV by the digene HPV Genotyping LQ Test using. J Clin Virol 2009;46:S21-6 [ Links ]
28. Feng Q, Cherne S, Winer RL, Balasubramanian A, Lee SK, Hawes SE, et al. Development and evaluation of a liquid bead microarray assay for genotyping genital human papillomaviruses. J Clin Microbiol 2009;47:547-53. [ Links ]
Conflicto de intereses: ninguno declarado.














