Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Colombiana de Obstetricia y Ginecología
versión impresa ISSN 0034-7434
Rev Colomb Obstet Ginecol vol.64 no.3 Bogotá jul./set. 2013
Wilmar Saldarriaga-Gil, MD, MSc1
Recibido: enero 17/13 - Aceptado: septiembre 18/13
1Ginecólogo obstetra. Ciencias Básicas Medicas. Embriología-Genética. Profesor Asociado, Departamentos de Morfología y Ginecoobstetricia. Grupo de Investigación MACOS. Director del Programa de Medicina, Universidad del Valle, Cali, Colombia. wsaldarriaga0608@yahoo.com
RESUMEN
Introducción: el cariotipo ha sido la prueba de oro para el análisis cromosómico en el diagnóstico prenatal en los últimos cuarenta años; sin embargo, a partir del 2011 se publican múltiples artículos y los primeros meta-análisis que muestran posibles ventajas de los array-CGH (del inglés array-comparative genomic hibridization) sobre el cariotipo en el diagnóstico prenatal.
Objetivo: hacer una reflexión acerca del uso de array-CGH en el diagnóstico prenatal y mostrar algunas de las potenciales ventajas y desventajas de esta prueba molecular con relación al cariotipo, así como su aplicación por obstetras, perinatólogos y especialistas en medicina materno-fetal.
Conclusión: los a-CGH son una nueva alternativa en el análisis cromosómico en el diagnóstico prenatal en fetos con anomalías anatómicas; pueden usarse en todos los casos en que se justifique una intervención invasiva en el diagnóstico prenatal de alteraciones cromosómicas, y es probable que los array-CGH reemplacen al cariotipo en el diagnóstico prenatal en esta década.
Palabras clave: diagnóstico prenatal, array-CGH, cariotipo.
ABSTRACT
Introduction: Karyotyping has been the gold standard for chromosomal analysis in prenatal diagnosis over the past 40 years. However, many articles and the first meta-analysis on the potential advantages of array-CGH (array comparative genomic hybridization) compared to karyotyping in prenatal diagnosis began to appear in 2011.
Objective: The objective of this article is to examine the use of array-CGH in prenatal diagnosis, and to show certain potential advantages and disadvantages of this molecular test over karyotyping, as well as its application by obstetricians, perinatologists and specialists in maternal-fetal medicine.
Conclusion: Array-CGH is a new option for chromosomal analysis in prenatal diagnosis of fetuses with anatomical abnormalities; it maybe used in all instances where an invasive intervention is warranted in the prenatal diagnosis of chromosomal abnormalities, and it may eventually replace karyotyping in prenatal diagnosis before this decade is out.
Key words: Prenatal diagnosis, array-CGH, karyotyping.
INTRODUCCIÓN
El diagnóstico prenatal es el conjunto de acciones orientadas a establecer el estado de salud del embrión o feto en la vida intrauterina. Este acercamiento se puede hacer a través de un abordaje no invasivo como la ecografía y pruebas en sangre materna, o invasivo que implica la toma de una muestra derivada de la gestación mediante biopsia de vellosidad corial (BVC), amniocentesis o cordocentesis (1); estos métodos invasivos están indicados cuando la probabilidad de encontrar un diagnóstico anormal es mayor que el riesgo que corre el embarazo de tener una complicación por el procedimiento (2). En el diagnóstico prenatal no invasivo, la intervención más usada es la ecografía, que desde su aparición en 1958 rápidamente llegó a la obstetricia, pasando de la observación en 2 dimensiones, 3 dimensiones multiplanar, cortes ortogonales y Doppler pulsado, lo que ha permitido una aproximación a detalles anatómicos mínimos (3). De la mano del desarrollo de la ultrasonografía obstétrica se disminuyeron los riesgos en los procedimientos invasivos, es así como en este momento se estima que la BVC, aumenta el riesgo basal de aborto en 1 a 2%, la amniocentesis en 0,8% y la cordocentesis en 2% (4).
En los casos en que estos procedimientos se realizan con la indicación de descartar o confirmar alteraciones cromosómicas numéricas o estructurales se procesa el cariotipo, método usado en humanos desde 1956, con él se hizo la relación etiológica de la alteración cromosómica en los síndromes de Down, Edwards, Patau, Turner, Klinefelter, entre otros; los primeros estudios en diagnóstico prenatal son de finales de los años sesenta (5, 6) e inicios de los setenta (7). En Colombia es en los años ochenta cuando llega la ecografía obstétrica, y su uso se hace rutinario en los años noventa; además, los laboratorios de citogenética estandarizaron el cariotipo con la tinción con giemnsa –conocido como bandas G–, e implementaron su uso en líquido amniótico y vellosidad corial también en los años noventa.
En los años noventa pruebas moleculares como FISH (fluorescent in situ hybridization), QF-PCR (quantitative fluorescent polymerase chain reaction) y MLPA (múltiplex ligation-dependent probe amplification), fueron desarrolladas en el análisis cromosómico, pero el cariotipo continuó siendo el estándar de oro en el diagnóstico prenatal (8, 9).
En el año 2000, los array-CGH (array comparative genomic hibridization) empiezan a ser utilizados en el diagnóstico prenatal como una prueba molecular para el análisis cromosómico, que al igual que el cariotipo detecta alteraciones cromosómicas numéricas y estructurales (10); hoy existen publicaciones que muestran ventajas de los array-CGH sobre el cariotipo y las otras técnicas moleculares, la cuales sugieren que son una prueba de primera línea en el análisis cromosómico en el diagnóstico prenatal (8-16).
Es importante que los ginecoobstetras, perinatólogos y especialistas en medicina materno fetal se documenten sobre la utilidad de esta prueba en cuanto a sus indicaciones, sus características operativas, ventajas y desventajas en relación con el cariotipo. Por tanto, el objetivo de este artículo es hacer una reflexión acerca del uso de array-CGH en el diagnóstico prenatal de alteraciones cromosómicas numéricas y estructurales.
ANÁLISIS CROMOSÓMICO EN EL DIAGNÓSTICO PRENATAL INVASIVO
Entre las indicaciones más frecuentes para el análisis cromosómico prenatal usadas en diferentes protocolos están: el antecedente de embarazo con alteración cromosómica; uno de los padres con alteración cromosómica; edad materna mayor a 38 o 40 años; tamizaje positivo de marcadores bioquímicos, más ecográficos, más edad materna; defectos anatómicos fetales detectados en la ecografía, que producen una probabilidad igual o mayor de un 1% de encontrar un defecto cromosómico (4, 12, 14). Como se mencionó, estas muestras se toman por métodos invasivos como la biopsia de vellosidad corial, la amniocentesis o la cordocentesis.
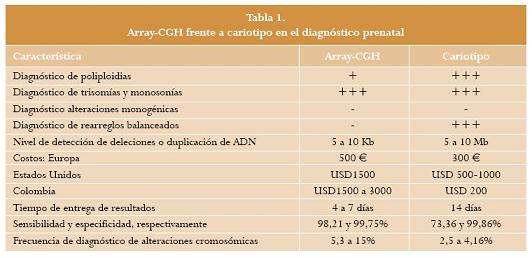
Cariotipo. Con el cariotipo se pueden diagnosticar alteraciones cromosómicas numéricas como: trisomías de cromosomas somáticos y sexuales (13, 18, 21, XXX, XXY, entre otras), monosomías (la del X); poliploidias (69 o 92 cromosomas en las molas hidatidiformes). Además, utilizando técnicas de bandeo de alta resolución se logran detectar defectos estructurales como pérdidas o exceso de material genético (inserciones o deleciones), entre 5 y 10 millones de pares de bases (Mb), como el síndrome Cri du Chat, originado por una deleción en el brazo corto del cromosma 5. También detecta translocaciones balanceadas, en las cuales un fragmento de un cromosoma se ubica en otro cromosoma (17). El cariotipo ha sido la prueba de oro en el análisis cromosómico en el diagnóstico prenatal en los últimos cuarenta años (14, 15, 18).
Esta prueba tiene varios determinantes, como que el proceso de cultivo celular y la observación microscópica de metafases requiere personal muy bien entrenado; lo anterior significa que es una prueba que depende del operador y el resultado se tiene en promedio en 14 días, tiempo que podría ser muy largo dada la ansiedad de la pareja por obtener un resultado, y en los países en que es permitida la interrupción voluntaria del embarazo sin condicionamientos, para tomar decisiones. En Colombia esta prueba tiene un costo de 200 dólares, que podría ser económico al compararlo con el de otros países, como por ejemplo Estados Unidos, donde cuesta entre 500 y 1000 dólares, o Europa donde tiene un costo de alrededor de 300 euros.
Otras pruebas usadas en el análisis cromosómico en el diagnóstico prenatal. Técnicas moleculares como FISH, QF-PCR y MLPA, usadas en el diagnóstico prenatal desde los años noventa, no lograron reemplazar el cariotipo, a pesar de la rápida entrega de resultados: 72 horas; el costo es similar o ligeramente superior, detectan alteraciones numéricas que involucran los cromosomas 13, 18, 21, X y Y, siendo locus específicos para esos cromosomas; sin embargo, no diagnostican alteraciones estructurales y no evalúan el resto de los cromosomas (19-21).
Array-CGH. Los array-CGH surgen como una prueba molecular para el análisis cromosómico, que al igual que el cariotipo detecta alteraciones cromosómicas numéricas y estructurales evaluando todos los cromosomas. Se utilizaron en principio en cáncer y en pacientes pediátricos (22, 23); posteriormente, en el 2000 se publican artículos que muestran el uso de esta prueba en el diagnóstico prenatal (10). En el 2009, el Colegio Americano de Obstetricia y Ginecología afirmó que el cariotipo es la principal herramienta en el diagnóstico prenatal, no obstante señaló que array-CGH puede ofrecerse como una prueba conjunta en el concierto de un adecuado asesoramiento genético para aquellos casos prenatales con defectos anatómicos y un cariotipo normal, y también en los casos de muerte fetal con anomalías congénitas, proyectando a los array-CGH como una herramienta de gran utilidad (24). En el 2010 se publicó un documento de consenso (25) y un artículo de análisis económico (26) los cuales sugieren que se debe considerar los a-CGH como primera prueba diagnóstica, reemplazando el cariotipo en pacientes con problemas neurológicos, autismo, déficit cognitivo o recién nacidos (RN) con anomalías congénitas de etiología desconocida. En el 2011 se empiezan a reportar publicaciones con más de 900 pacientes y metaanálisis que comparan los a-CGH, el cariotipo, MLPA y QF-PCR, encontrando ventajas de los array-HGC sobre las otras pruebas, específicamente sobre el cariotipo en el diagnóstico prenatal, al disminuir el tamaño de exceso o déficit de ADN que podría ser detectado y así aumentar la frecuencia de diagnóstico de alteraciones cromosómicas estructurales (8-16, 27, 28). Existen algunos autores que sugieren esta prueba como tamizaje en pacientes de bajo riesgo (9, 29).
Actualmente, en el diagnóstico prenatal los array-CGH emplean plataformas de segunda generación que usan oligonucleótidos que pueden encontrar exceso o déficit de ADN entre 500 Kb (miles de pares de bases) y 1 Mb (9, 18), y usualmente utilizan plataformas que contienen pozos (algunos los denominan pocillos), con un número de sondas de hibridación que detectan alteraciones entre 60.000 y 180.000 fragmentos de ADN, que logran examinar todo el genoma como en el cariotipo, pero mejoran el nivel de detección de pérdidas o exceso de material genético en fragmentos hasta 10 veces más pequeños; también existen plataformas construidas a conveniencia que detectan alteraciones que han demostrado relevancia clínica, suelen tener entre 7000 y 15.000 oligonucleótidos (9).
Sin embargo, los a-CGH son poco eficientes o no detectan rearreglos balanceados, ni poliploidias (sí detectados por el cariotipo), no detectan mutaciones puntuales (tampoco detectadas en el cariotipo), y pueden encontrar variantes de significancia clínica incierta (9, 15). Aunque el resultado está disponible a los 4 o 7 días, tienen como importante problema los costos: en Europa cuesta 500 euros, en Estados Unidos 1500 dólares, en Colombia entre 1500 a 3000 dólares, el proceso es sistematizado y el resultado no depende del operador.
EXACTITUD DEL CARIOTIPO EN COMPARACIÓN CON LOS ARRAY-CGH EN EL DIAGNÓSITCO PRENATAL
Los estudios comparativos de las diferentes pruebas de análisis cromosómico en diagnóstico prenatal en pacientes de alto riesgo, bajo las indicaciones ya descritas de someter a las pacientes a procedimientos invasivos, muestran frecuencias de diagnóstico con cariotipo entre el 2,55 y 4,16%9, y para array-CGH de 5,3% (13), 6,2% (8), 7,6% (15), e incluso hasta 13,3 y 15% (9); estos porcentajes de diagnóstico aumentan significativamente cuando la indicación son defectos anatómicos fetales, 9,3% (15), 17% (11), 39% (8). En general, en el diagnóstico prenatal el cariotipo tiene una sensibilidad de 73,36% y una especificidad de 99,86%, y los a-CGH una sensibilidad de 98,21% con especificidad de 99,75% (8). Puntualmente, en casos de defectos anatómicos la sensibilidad del cariotipo sigue siendo baja (76,6%) y la especificidad alta (99,86%), mientras que los a-CGH la sensibilidad es 96% y la especificidad 99,3% (8). Las características de ambas pruebas se resumen en la tabla 1.
El uso de esta prueba molecular en pacientes con anomalías anatómicas congénitas de etiología desconocida debe ir acompañado de un manejo por grupos multidisciplinarios que incluyen el obstetra, el perinatólogo, un genetista, un psicólogo. El trabajo en equipo debe ir desde el asesoramiento previo a la realización de la prueba hasta la interpretación de los resultados, que podrían no mostrar alteración, mostrar déficit o exceso de material genético con correlación fenotípica establecida, variantes de significado benigno y las variantes de significancia incierta (7, 8).
CONCLUSIÓN
Los array-CGH, al compararlos con el cariotipo, muestran un aumento significativo en la frecuencia de diagnóstico, manteniendo la sensibilidad y especificidad, sin someter al paciente a mayor riesgo; también con los array-CGH se obtienen resultados en menor tiempo, pero incrementan los costos y la probabilidad de encontrar variantes de naturaleza incierta que podrían aumentar la ansiedad de la pareja. Es probable que los array-CGH reemplacen al cariotipo en el diagnóstico prenatal en esta década.
REFERENCIAS
1. Saldarriaga W, Ballesteros A. Diagnóstico prenatal de anomalías genéticas. En: Galvis C, Baquero H, Garcia G. Texto de Neonatología. Bogotá: Distribuna; 2012. p. 497-502. [ Links ]
2. Gratacos E, Delgado J. Procedimientos invasivos ecoguiados en medicina fetal. En: Gratacos E, Gómez R, Nicolaides K, Romero R, Cabero L. Medicina fetal. Buenos Aires, Madrid: Medica Panamericana; 2007. p. 129-35. [ Links ]
3. Saldarriaga W, Artuz M. Ayudas diagnósticas en obstetricia. En: Saldarriaga W, Artuz M. Fundamentos de ginecología y obstetricia. Cali: Programa editorial Universidad del Valle; 2010. p. 265-77. [ Links ]
4. Stembalska A, Slezak R, Pesz K, Gil J, Sasiadek M. Prenatal diagnosis - principles of diagnostic procedures and genetic counseling. Folia Histochem Cytobiol. 2007;45:S11-6. [ Links ]
5. Jacobson CB, Barter RH. Intrauterine diagnosis and management of genetic defects. Am J Obstet Gynecol. 1967;99:796-807. [ Links ]
6. Steele MW, Breg WR Jr. Chromosome analysis of human amniotic-fluid cells. Lancet. 1966;1:383-5. [ Links ]
7. Wapner RJ, Driscoll DA, Simpson JL. Integration of microarray technology into prenatal diagnosis: counselling issues generated during the NICHD clinical trial. Prenat Diagn. 2012;32:396-400. [ Links ]
8. Armengol L, Nevado J, Serra-Juhé C, Plaja A, Mediano C, García-Santiago FA, et al. Clinical utility of chromosomal microarray analysis in invasive prenatal diagnosis. Hum Genet. 2012;131:513-23. [ Links ]
9. Mori M, Mansilla E, García-Santiago F, Vallespín E, Palomares M, Martín R, et al. Diagnóstico prenatal y array-hibridación genómica comparada (CGH) (I). Gestaciones de elevado riesgo. Diagn Prenat. 2012;23:34-48. [ Links ]
10. Lapierre JM, Cacheux V, Luton D, Collot N, Oury JF, Aurias A, et al. Analysis of uncultured amniocytes by comparative genomic hybridization: a prospective prenatal study. Prenat Diagn. 2000;20:123-31. [ Links ]
11. Lee CN, Lin SY, Lin CH, Shih JC, Lin TH, Su YN. Clinical utility of array comparative genomic hybridisation for prenatal diagnosis: a cohort study of 3171 pregnancies. BJOG 2012;119:614-25. [ Links ]
12. Strassberg M, Fruhman G, van den Veyver IB. Copynumber changes in prenatal diagnosis. Expert Rev Mol Diagn. 2011;11:579-92. [ Links ]
13. Shaffer LG, Dabell MP, Fisher AJ, Coppinger J, Bandholz AM, Ellison JW, et al. Experience with microarray-based comparative genomic hybridization for prenatal diagnosis in over 5000 pregnancies. Prenat Diagn 2012;2:976-85. [ Links ]
14. Hillman SC, Pretlove S, Coomarasamy A, McMullan DJ, Davison EV, Maher ER, et al. Additional information from array comparative genomic hybridization technology over conventional karyotyping in prenatal diagnosis: a systematic review and meta-analysis. Ultrasound Obstet Gynecol. 2011;37:6-14. [ Links ]
15. Breman A, Pursley AN, Hixson P, Bi W, Ward P, Bacino CA, et al. Prenatal chromosomal microarray analysis in a diagnostic laboratory; experience with >1000 cases and review of the literature. Prenat Diagn. 2012;32:351-61. [ Links ]
16. Fiorentino F, Caiazzo F, Napolitano S, Spizzichino L, Bono S, Sessa M, et al. Introducing array comparative genomic hybridization into routine prenatal diagnosis practice: a prospective study on over 1000 consecutive clinical cases. Prenat Diagn. 2011;31:1270-82. [ Links ]
17. Gardner M, Southerland G, Shaffer L. Autosomal reciprocal traslocations. En: Gardner RJM. Chromosome anormalities and genetic counseling. Oxford University Press; 2012. p. 67-111. [ Links ]
18. Brady PD, Devriendt K, Deprest J, Vermeesch JR. Array-Based Approaches in Prenatal Diagnosis. Methods Mol Biol. 2012;838:151-71. [ Links ]
19. Mansfield ES. Diagnosis of Down syndrome and other aneuploidies using quantitative polymerase chain reaction and small tandem repeat polymorphisms. Hum Mol Genet. 1993;2:43-50. [ Links ]
20. Pertl B, Yau SC, Sherlock J, Davies AF, Mathew CG, Adinolfi M. Rapid molecular method for prenatal detection of Down's syndrome. Lancet. 1994; 343:1197-8. [ Links ]
21. Schouten JP, McElgunn CJ, Waaijer R, Zwijnenburg D, Diepvens F, Pals G. Relative quantification of 40 nucleic acid sequences by multiplex ligationdependent probe amplification. Nucleic Acids Res. 2002;15;30:e57. [ Links ]
22. Bayani J, Thorner P, Zielenska M, Pandita A, Beatty B, Squire JA. Application of a simplified comparative genomic hybridization technique to screen for gene amplification in pediatric solid tumors. Pediatr Pathol Lab Med. 1995;15:831-44. [ Links ]
23. Erdel M, Duba HC, Verdorfer I, Lingenhel A, Geiger R, Gutenberger KH, et al. Comparative genomic hybridization reveals a partial de novo trisomy 6q23-qter in an infant with congenital malformations: delineation of the phenotype. Hum Genet. 1997;99:596-601. [ Links ]
24. ACOG. Comittee Opinion No. 446: array comparative genomic hybridization in prenatal diagnosis. Obstet Gynecol. 2009;114:1161-3. [ Links ]
25. Miller DT, Adam MP, Aradhya S, Biesecker LG, Brothman AR, Carter NP, et al. Consensus statement: chromosomal microarray is a first-tier clinical diagnostic test for individuals with developmental disabilities or congenital anomalies. Am J Hum Genet. 2010;86:749-64. [ Links ]
26. Regier DA, Friedman JM, Marra CA. Value for money? Array genomic hybridization for diagnostic testing for genetic causes of intellectual disability. Am J Hum Genet. 2010;86:765-72. [ Links ]
27. Shaffer LG, Rosenfeld JA, Dabell MP, Coppinger J, Bandholz AM, Ellison JW, et al. Detection rates of clinically significant genomic alterations by microarray analysis for specific anomalies detected by ultrasound. Prenat Diagn. 2012;32:986-95 [ Links ]
28. Lee CN, Lin SY, Shih JC, Su YN. Authors' response to: The clinical utility and indications of chromosomal microarray analysis in prenatal diagnosis. BJOG. 2013;120:120. [ Links ]
29. Querejeta ME, Nieva B, Navajas J, Cigudosa JC, Suela J. Diagnóstico prenatal y array-CGH II: gestaciones de bajo riesgo. Diagn Prenat. 2012;23:49-55. [ Links ]
Conflicto de intereses: ninguno declarado.













