Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de la Facultad de Medicina
Print version ISSN 0120-0011
rev.fac.med. vol.53 no.3 Bogotá July 2005
INVESTIGACIÓN ORIGINAL
Ibonne Aydee García Romero1, Emilia María Valenzuela de Silva2 Carlos Humberto Saavedra3, Aura Lucía Leal Castro4, Javier Eslava Schmalbac5, José Ramón Mantilla Anaya6
1.Química Farmacéutica, MSc en Microbiología, Instituto de Biotecnología, Universidad Nacional de Colombia. Bogotá.
2.Química Farmacéutica, MSc en Microbiología, Instituto de Biotecnología, Universidad Nacional de Colombia. Bogotá.
3.Médico Infectólogo, Facultad de Medicina, Universidad Nacional de Colombia. Bogotá. Hospital Universitario Clínica San Rafael.
4.Médico. Especialista en Microbiología y Parasitología Médica. MSc en control de Enfermedades Infecciosas, Facultad de Medicina. Universidad Nacional de Colombia, Bogotá.
5.Médico Cirujano, MSc en Epidemiología, Facultad de Medicina, Universidad Nacional de Colombia. Bogotá.
6.Químico Farmacéutico, MSc en Bioquímica, Instituto de Biotecnología. Universidad Nacional de Colombia, Bogotá.
Correspondencia:jrmantillaa@unal.edu.co
Resumen
Antecedentes. Las enterobacterias, antaño flora normal del tracto gastrointestinal, han cambiado su biología y emergido como agentes patógenos nosocomiales que se tornan resistentes los antibióticos conocidos.
Objetivo. Realizar la caracterización epidemiológico-molecular de 20 aislamientos de Enterobacter cloacae resistentes a cefalosporinas de tercera generación; provenientes de un hospital de tercer nivel de Bogotá-Colombia.
Material y métodos. Los aislamientos fueron identificados mediante sistemas automatizados Microscan y VITEK, se utilizó el Enterobacter asbureae como control externo inter-especie.
La confirmación de resistencia se hizo por técnica de difusión en agar, y una vez establecida se realizó BLEE para comprobación. La determinación de puntos isoeléctricos se hizo, mediante lisis por ultrasonido y la genotipificación mediante la metodología para bacterias Gramnegativas propuesta por Versalovic.
Resultados: Los aislamientos colectados durante un año fueron causantes de 15 casos de infección Intrahospitalaria y dos colonizaciones.
Todos los aislamientos presentaron resistencia a cefotaxima, ceftazidima, ceftriaxona, aztreonam y ciprofloxacina, 95% a amikacina, gentamicina y cloranfenicol, 75% a trimetoprim/sulfametoxazol, 20% a cefepime y todos fueron sensibles a imipenem.
Dos aislamientos fueron confirmados como productores de â-lactamasas de espectro extendido (BLEE) por la técnica microbiológica de disco combinado.
Por isoelectroenfoque presentaron dos â-lactamasas con puntos isoeléctricos (pI) de 5,4 y 8,2. En los 18 aislamientos no inhibidos por ácido clavulánico, se detectaron entre 2 y 4 â-lactamasas con pI de 5,4; 6,0; 7,0; 8,2 y mayor que 8,2; la resistencia a cefalosporinas de tercera generación podría ser atribuida a la hiperproducción de AmpC; los valores de pI sugieren la producción simultánea de â-lactamasas tipo SHV y TEM. La genotipificación mediante tres metodologías de rep-PCR (ERIC; REP y BOX) agrupó la población estudiada en siete clones: seis constituidos por un solo aislamiento y el clon predominante E1/B1/R1 agrupó 14 aislamientos causantes de infección en diez pacientes.
Conclusión. Se identificó un clon de Enterobacter cloacae multirresistente, endémico en una institución de tercer nivel en Bogotá, causante de infección nosocomial y quirúrgica en particular.
Palabras clave: enterobacter cloacae, cefalosporinas, â-lactamasas, isoeléctricos Mezclas anfolíticas), clones (células clonares), infección nosocomial (infección hospitalaria).
Summary
Background. Enterobacter species were normal in gastrointestinal tract, but nowadays, its biology has changed and there are nosocomial agents with antibiotics resistance.
Objective. To make an epidemiological and molecular characterization of 20 isolates of Enterobacter cloacae with third generation cephalosporin resistance, from a hospital of third level in Bogotá-Colombia.
Material and methods. Isolates were identified with Microscan and VITEK, Enterobacter asbureae was utilized as an inter-specie control.
Resistance was confirmed by agar diffusion and by BLEE techniques. Isoelectric points were determined by ultrasound lyses and genotypication by Versalovic´s system for gram negative bacteria.
Results. The isolates collected over the course of a year caused 15 cases of intra-hospital infection and two colonisations. All isolates presented resistance to cefotaxime, ceftazidime, ceftriaxone, aztreonam and ciprofloxacin, 95% to amikacin, gentamicin and chloramphenicol, 75% to trimethoprim/ sulphamethoxazole, 20% to cefepime and all were sensitive to imipenem. Two isolates were confirmed as extended spectre â-Iactamase (ESBL) producers by microbiologic al combined disktechnique; two â-Iactamases having 5.4 and 8.2 isoelectric points (pI) were presented by isoelectric focusing. Between 2 and 4 â-Iactamases having 5.4, 6.0, 7.0, 8.2 and >8.2 pl were detected in the 18 isolates which were not inhibited by clavulanic acid. Third-generation cephalosporin-resistance was attributed to AmpC hyper-production; pl values suggested simultaneous SHV and TEM â lactamase production. Genotyping by three rep- PCR methodologies (ERIC, REP and BOX) grouped the population studied into 7clones; 6 were constitute d by a single isolate and the predominant E1/B1/R1 clone grouped 14 isolates causing infection in 10 patients. This work led to a multiresistant Enterobacter cloacae clone being detected, considered endemic for the institution, in the studied surgical patients it was una predominantly cause of intrahospital infection.
Conclusion. We detected a clone of Enterobacter cloacae with multi-cephalosporin resistance, has an endemic strain in a hospital of third level in Bogotá, who caused nosocomial infection, in special of surgical patients.
Key words: enterobacter cloacae, cephalosporins, beta-lactamases, ampholyte mixtures, clone cells, infection, cross infection.
Introducción
Enterobacter cloacae hace parte de la flora normal del tracto gastrointestinal; sin embargo, en los últimos años ha emergido como un patógeno nosocomial importante, causante de infección en salas de cirugía, unidades de quemados y de sepsis neonatal (1-3). Adicionalmente, la aparición rápida de resistencia a determinados antibióticos durante la terapia antimicrobiana puede llegar a ser un problema serio (3,4). En general se acepta que la hiperproducción de una cefalosporinasa codificada por el gen AmpC es el mecanismo más frecuente de resistencia a 7µ-metoxi-cefalosporinas y monobactámicos, no solo de Enterobacter cloacae sino de otras especies de Enterobacter y enterobacterias como Citrobacter y Serratia (5,6). Esta â-lactamasa generalmente está codificada en el cromosoma y usualmente no se transfiere a otras bacterias (7). Otro mecanismo frecuente de resistencia a antibióticos â-lactámicos en enterobacterias es la producción de â-lactamasas de espectro extendido (BLEE). Estas enzimas inicialmente se id entificaron en Klebsiella spp.y en Escherichia coli, se derivan en su mayoría de las â-lactamasas tipo TEM y SHV y generalmente están codificadas por plásmidos (7,8). La identificación de enterobacterias productoras de BLEE que adicionalmente producen AmpC inducible o constitutiva, ha aumentado en todo el mundo; en estas especies la detección de BLEE mediante el efecto inhibitorio de ácido clavulánico es difícil y depende del nivel de producción de la enzima cromosomal (9-11).
Para la tipificación de Enterobacter cloacae con fines epidemiológicos se han usado métodos basados en características fenotípicas, tales como biotipificación, análisis de antibiograma, serotipificación y fagotipificación.
Algunas de estas técnicas no son lo suficientemente sensibles para distinguir entre diferentes cepas y otras no son fáciles de realizar en el laboratorio (12,13).
Se han utilizado la ribotipificación y la electroforesis en gel por campo pulsado para la tipificación de Enterobacter cloacae por su buen poder de discriminación (2,13); sin embargo estas técnicas son laboriosas y onsumen mucho tiempo. La introducción de técnicas como rep- PCR, basadas en la amplificación por PCR de secuencias repetidas, han facilitado la tipificación de diversos microorganismos ya que reducen los costos y el tiempo de los análisis (14-16).
En este trabajo se tuvo como objetivo caracterizar epidemiológica y molecularmente, mediante rep-PCR, aislamientos de Enterobacter cloacae asociados con infección preferiblemente intrahospitalaria, provenientes de un centro hospitalario de tercer nivel de complejidad y sugerir el posible mecanismo involucrado en la resistencia a â-lactámicos en estos aislamientos.
Material y métodos
Aislamientos bacterianos. Se estudiaron 20 aislamientos de Enterobacter cloacae resistentes a cefalosporinas de tercera generación, recolectados desde abril de 2001 hasta abril de 2002, provenientes de 16 pacientes del Hospital Universitario Clínica San Rafael de Bogotá.
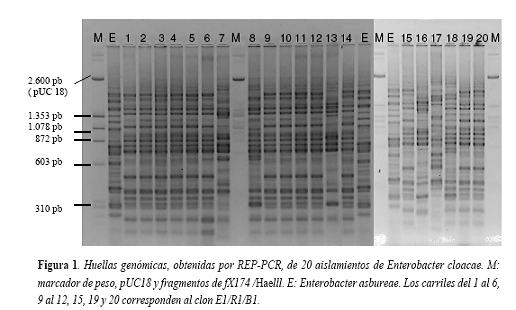
Los aislamientos fueron identificados por los sistemas automatizados MicroScan y VITEK. Se incluyó un aislamiento de Enterobacter asbureae como control externo interespecie para la validación de la caracterización molecular por rep-PCR (17) (Figura 1) .
Pruebas de susceptibilidad. El antibiograma se realizó por el sistema MicroScan (panel 13) del hospital, posteriormente se repitió en el laboratorio de epidemiología molecular del Instituto de Biotecnología de la Universidad Nacional de Colombia (IBUN) mediante la técnica de difusión en agar con discos (Oxoid®) de gentamicina, amikacina, ciprofloxacina, trimetoprim/sulfametoxazol, cloranfenicol, imipenem y cefepime. En el antibiograma se incluyeron tres cefalosporinas de tercera generación: cefotaxima, ceftazidima y ceftriaxona, y el monobactámico aztreonam. A los aislamientos que presentaron resistencia por lo menos a una cefalosporina de tercera generación o al aztreonam, se les realizó la prueba de confirmación para BLEE.
Como no se han establecido metodologías microbiológicas estándar para la confirmación de BLEE en esta especie bacteriana, se utilizó la técnica recomendada por la NCCLS (18) para Klebsiella pneumoniae, oxitoca y E.coli como han sugerido otros autores (11). Se utilizaron discos (Oxoid®) de ceftazidima y cefotaxima, solos y en combinación con ácido clavulánico. Como controles para evaluar la calidad de los sensidiscos se utilizó la cepa de Klebsiella pneumoniae ATCC 700603 y la cepa de Escherichia coli ATCC 25922.
Determinación de puntos isoeléctricos. Los extractos crudos de las â-lactamasas se obtuvieron mediante lisis por ultrasonido de las células bacterianas cultivadas en 10 ml de caldo tripticasa soya con cefotaxima (10 µg/ml); se utilizó un sonicador de vástago Vibra Cell (Sonic®) a una amplitud de 35% con tres ciclos de pulsos durante 30 segundos, con intervalos de dos minutos.
Los extractos se analizaron por isolectroenfoque en geles de poliacrilamida con un rango de pH entre 3,5 y 10 (ampholite-Bio-Rad ®) (19) usando el equipo manual mini-IEF (Bio-Rad®).
Las â-lactamasas se detectaron colocando sobre los geles una hoja de papel Whatman impregnada con solución de nitrocefín (100 µg/ml) (Oxoid®). Los valores de pI se determinaron por comparación con â-lactamasas de pI conocidos 5,4 (TEM-1), 7,6 (SHV-1), 7,8 (SHV-7), 8,2 (SHV-5) y el marcador preteñido con pI 4,75; 7,0 y 9,6 (IEF-standard-Bio-Rad®) (19).
Genotipificación por rep-PCR. La obtención del ADN total de los aislamientos se realizó siguiendo la metodología propuesta por Versalovic para bacterias gram-negativas (16). La cuantificación del ADN obtenido se realizó por comparación de la intensidad fluorescente de diluciones de la muestra frente a cantidades conocidas de ADN de fago lambda, teñidas con bromuro de etidio y separados por electroforesis en un minigel de agarosa (20). Posteriormente el ADN se almacenó a-20°C hasta la realización de la rep-PCR.
Para la obtención de las huellas genómicas se utilizaron tres metodologías de rep-PCR con tres conjuntos diferentes de iniciadores correspondientes a secuencias repetidas bacterianas (15,16). Para rep- PCR (secuencias repetidas palindrómicas extragénicas), los iniciadores: REP1R-dt 5'- IIINCGNCGNCATCNGGC-3' y REP2 5'- NCGNCTTATCNGGCCTAC-3' (15,16).
Para ERIC-PCR (secuencias consenso intergénicas repetidas enterobacteriales), los iniciadores: ERIC1 5'- ATGTAAGCTCCTGGGGATTCAC-3' y ERIC2 5'-AAGTAAGTGACTGGGGTGAGCG-3' (16) y para BOX-PCR (secuencias en mosaico del elemento BOX) el iniciador: BOXA1R 5'- CTACGGCAAGGCGACGCTGACG-3' (15). Todos los iniciadores fueron sintetizados por Invitrogen.
Las condiciones de amplificación fueron modificadas de protocolos publicados (14,15,17), las cuales incluyeron disminución de la concentración de dNTPs y adición de BSA en las tres metodologías; adicionalmente se aumentó el valor de la temperatura de asociación tanto para rep-PCR como para BOX-PCR. En el termociclador iCycler (Bio-Rad ®), se amplificaron 100 ng de ADN en un volumen final de reacción de 25 µl, que contenía 200 µM de cada dNTP, 6 mM de cloruro de magnesio, 2 mM de cada iniciador, 2 mg/ml de BSA, dimetilsulfóxido (5% para rep-PCR y 10% para ERIC-PCR y BOXPCR ®), buffer para PCR 1X, dos unidades de Taq-DNA polimerasa (Invitrogen®). La amplificación para rep-PCR se realizó con un paso inicial de desnaturalización de (94°C, 7 minutos) y luego 30 ciclos de: desnaturalización (92°C, 30 segundos), asociación (60°C, 1 minuto) y extensión (70°C, 3 minutos) seguidos por un paso de extensión final (70°C, 8 minutos). Para ERIC-PCR: desnaturalización inicial (94°C, 7 minutos), luego 30 ciclos de: desnat uralización (92°C, 30 segundos), asociación (52°C, 1 minuto) y extensión (70°C, 3 minutos) seguidos por extensión final (70°C, 8 minutos). Para BOXPCR desnaturalización inicial (95°C, 2 minutos), luego 30 ciclos de: desnaturalización (92°C, 30 segundos), asociación (60°C, 1 minuto) y extensión (65°C, 8 minutos) seguidos por extensión final (65°C, 8 minutos).
La evaluación de la PCR se realizó por electroforesis en geles de agarosa al 2% con Buffer TBE 0,5X a 4,6 v/cm, posteriormente los geles fueron teñidos en solución de bromuro de etidio 1 mg/ml. Los patrones de bandas fueron registrados en el sistema Gel-Doc System (Bio- Rad®). El análisis de las huellas genómicas obtenidas con las tres metodologías se realizó con el software NTSYSpc versión 2.0 mediante una matriz de presencia ausencia utilizando el algoritmo de agrupamiento UPGMA (unweighted pair group meted with arithmetic averages).
Información clínica. La recolección de la información clínica se llevó a cabo mediante una ficha elaborada para este estudio, fecha de ingreso, género, edad, hospitalización previa, fecha de colección del aislamiento, fuente, tipo de infección, servicio en los que se aisló el micro- organismo y condiciones de egreso.
Para el análisis de la información obtenida se creó una base de datos en Excel, con datos clínicos, bioquímicos y moleculares. Se hizo una presentación descriptiva de datos, y en los casos que se obtuvo relevancia clínica se aplicó la prueba exacta de Fisher. Se realizó el análisis mediante el programa estadístico Stata versión 6.0.
Resultados
Características clínicas. El estudio incluyó 20 aislamientos de Enterobacter cloacae provenientes de 11 hombres y cinco mujeres con una edad promedio de 63 años, las características clínicas de estos pacientes infectados o colonizados se encuentran resumidas en la tabla 1. Diecisiete de los 20 (85%) aislamientos de Enterobacter cloacae se consideraron causantes de infección nosocomial en concordancia con lo descrito por el Center for Disease Control and Prevention, Atlanta (21). Seis fueron aislados de líquido peritoneal, cinco de herida quirúrgica, cuatro de urocultivo, dos de hemocultivo, uno de humor vítreo, uno de cultivo de trayecto catéter y otro de cultivo de biopsia.
Tres aislamientos fueron considerados colonizaciones, uno se aisló de un paciente hospitalizado en UCC, el segundo de un paciente de nefrología y el tercero fue considerado colonización de origen extrahospitalario.
Nueve pacientes habían estado previamente hospitalizados desde uno hasta 90 días. Adicionalmente nueve pacientes fueron sometidos a cirugía y de estos siete fueron dados de alta y reingresaron con diagnóstico de infección nosocomial. Seis de los 16 pacientes murieron en el hospital, cuatro por causas atribuibles a la infección intrahospitalaria. Se encontró asociación entre la variable de herida quirúrgica con la variable rep-Tipo (E1/B1/R1).
Pruebas de susceptibilidad. Los resultados del antibiograma se muestran en la Tabla 1. Todos los aislamientos presentaron resistencia a cefotaxima, ceftazidima, ceftriaxona, aztreonam y ciprofloxacina, 95% a amikacina, gentamicina y cloranfenicol, 75% a trimetoprim/sulfametoxazol, 20% a cefepime y todos fueron sensibles a imipenem. En la prueba confirmatoria para BLEE solamente dos de los 20 aislamientos produjeron â-lactamasas inhibidas por ácido clavulánico.
Determinación de puntos isoeléctricos. Los perfiles de puntos isoeléctricos (pI) obtenidos para cada uno de los aislamientos se muestran en la Tabla 1. No fue posible precisar los valores superiores a 8,2 debido al estrecho margen de separación entre 8,2 y 9,6 de los marcadores utilizados.
Los 18 aislamientos no inhibidos por ácido clavulánico, produjeron una â-lactamasa con Pi mayor que 8,2 que correspondería a la cefalosporinasa AmpC. Los dos aislamientos confirmados, por la prueba de disco combinado, como productores de BLEE presentaron dos valores de puntos isoeléctricos: 5,4 y 8,2 (perfil de pIs: I); el aislamiento correspondiente al paciente remitido de otro centro hospitalario presentó dos pI: uno de 5,4 y otro mayor que 8,2 (perfil de pIs: II); un aislamiento presentó tres pI: 6,0; 8,2 y mayor que 8,2 (perfil de pIs: III) y otro presentó tres pIs con valores de 5.4, 6.0 y mayor que 8.2 (perfil de pIs: IV). Catorce de los 15 aislamientos estrechamente relacionados presentaron el perfil de puntos isoeléctricos V (pIs: 5,4; 6,0; 7,0 y mayor que 8,2) y el restante presentó el perfil VI (pIs: 6,0; 7,0; 8,2 y mayor que 8,2.
Genotipificación por rep-PCR. Las relaciones genéticas entre los 20 aislamientos de Enterobacter cloacae fueron investigadas por rep-PCR utilizando tres conjuntos diferentes de iniciadores. Los resultados se muestran en la tabla 1. REP y BOX-PCR agruparon los aisla-
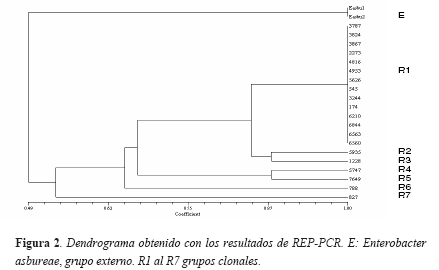
mientos en siete diferentes grupos clonales, para rep-PCR (R1 al R7) con un promedio de 20 bandas y para BOX-PCR (B1 al B7) con un promedio de 17 bandas. ERIC-PCR los clasificó en ocho grupos clonales (E1 al E8) con un promedio de 13 bandas. Catorce aislamientos provenientes de 10 pacientes presentaron patrones electroforéticos idénticos y se consideraron con rep-PCR y con BOX-PCR correspondientes al mismo grupo clonal R1 y B1 respectivamente; con ERIC-PCR, 13 de los 14 aislamientos presentaron patrones electroforéticos idénticos (E1) el aislamiento 545 (E1a) difirió en dos bandas por lo que se consideró con este marcador como estrechamente relacionado con E1. Nueve de los 10 pacientes que presentaron los aislamientos agrupados en el clon R1/B1/E1 habían sido intervenidos quirúrgicamente y el otro paciente presentó infección urinaria, este paciente había sido tratado por el servicio de nefrología antes de producirse la infección. Dos de los nueve pacientes intervenidos quirúrgicamente, también habían sido tratados por este servicio. Los seis aislamientos restantes tuvieron perfiles electroforéticos únicos por lo que cada uno se consideró un clon diferente. El análisis de agrupamiento realizado para los patrones de bandas obtenidas con los tres conjuntos de iniciadores a partir de una matriz de presencia-ausencia, mediante el algoritmo UPGMA permitió establecer que el aislamiento de Enterobacter cloacae más alejado genéticamente del clon predominante (E1/B1/R1) fue el aislamiento 827. El aislamiento de Enterobacter asbureae utilizado como control externo, el cual se analizó por duplicado con los tres conjuntos de iniciadores siempre, se agrupó a una gran distancia genética (Figura 2).
Discusión
La interpretación de los resultados del antibiograma permite dar una idea aproximada de los mecanismos causantes de la resistencia a las cefalosporinas de tercera generación. Así, los 20 aislamientos de Enterobacter cloacae fueron resistentes a cefalosporinas de tercera generación y al aztreonam, pero sensibles a imipenem; por lo tanto se puede presumir que la resistencia a estos â-lactámicos es debida a la acción de la cefalosporinasa AmpC o por BLEE del tipo serin-proteasas. De otro lado, 15 de los 20 aislamientos (80%) fueron sensibles a cefepime lo cual permite suponer que el mecanismo de resistencia más probable en estos aislamientos es la hiperproducción de AmpC ya que esta enzima generalmente tiene baja capacidad hidrolítica sobre cefalosporinas de cuarta generación.
Mediante isoelectroenfoque, en 18 aislamientos se observó la producción de âlactamasas con puntos isoeléctricos mayores a 8.2 valores que se han informado como característicos para diferentes AmpC (9,11). De esta manera, con las pruebas microbiológicas y moleculares se puede sugerir que el mecanismo más probable de resistencia a antibióticos âlactámicos en los aislamientos estudiados fue la hiperproducción de AmpC. Sin embargo, es factible que en todos los aislamientos estén presentes otros mecanismos de resistencia. Así, uno los cinco aislamientos resistentes a cefepime, pudo confirmarse como productor de BLEE; en los otros aislamientos es posible la producción simultánea de BLEE, con actividad hidrolítica sobre cefepime y de la cefalosporinasa AmpC, que al sobreexpresarse enmascara la detección BLEE; adicionalmente los aislamientos que presentaron resistencia a amikacina y a gentamicina, podrían producir concomitantemente algún tipo de BLEE, ya que los genes de resistencia a aminoglucósidos se han encon trado en plásmidos que contienen simultáneamente genes codificadores de BLEE (23). Todos los aislamientos fueron productores de â-lactamasas con puntos isoeléctricos que podrían corresponder a BLEE del tipo al tipo SHV y TEM, que son la â-lactamasas mas frecuentemente informadas en enterobacterias; sin embargo la posible hiperproducción de AmpC por estas cepas estaría enmascarando la acción de estas e impidiendo su detección por la técnica de disco combinado.
La detección de BLEE en Enterobacter sp. hiperproductor de AmpC; es importante por que permitiría controlar o descartar el uso de cefepime como antibiótico de elección, ya que algunas BLEE podrían hidrolizar a esta cefalosporina de cuarta generación (24) Adicionalmente, es importante conocer si los aislamientos que sobreexpresan AmpC también son productores de BLEE, puesto que estas enzimas generalmente están codificadas en plásmidos, lo cual implicaría la posible transferencia horizontal, entre cepas y aun entre diferente especies, de la resistencia a â-lactámicos de espectro extendido y la corresistencia a otros antibióticos codificada en estos plásmidos. La corresistencia a aminoglucósidos y fluoroquinolonas en aislamientos productores de BLEE y de sus enzimas parentales, codificadas en plásmidos, se ha documentado ampliamente para otras enterobacterias como K lebsiella pneumoniaey Escherichia coli; adicionalmente se ha descrito la transferencia de este tipo de plásmidos entre especies (25,26).
La expresión de BLEE en dos de los aislamientos, confirmada mediante la prueba de disco combinado, descarta la hiperproducción de AmpC como mecanismo de resistencia a âlactámicos.
Estas cepas pudieron haber adquirido plásmidos con genes codificantes de BLEE, transferidos desde el ambiente hospitalario o tener esta información como parte de su haber genético antes de iniciar la terapia antimicrobiana y por consiguiente su resistencia a â-lactámicos pudo deberse a la expresión de BLEE que implica un menor "costo biológico" para la bacteria que la hiperproducción de AmpC (9).
Los resultados de la tipificación por las tres metodologías de rep-PCR fueron concordantes. Sin embargo los patrones electroforéticos obtenidos con ERIC-PCR fueron más difíciles de interpretar debido a la intensidad variable de las bandas y a que la reproducibilidad estuvo más condicionada a la variación de la concentración del ADN molde, limitaciones probablemente asociadas con la naturaleza de los iniciadores; Wong y colaboradores (27) informan las mismas dificultades con esta técnica para Vibrio parahemolyticus, en este estudio los autores concluyen que ERIC-PCR tiene un buen poder discriminatorio pero tiene menor reproducibilidad con relación a la variante rep-PCR. Sin embargo, otro estudio llevado a cabo en Grecia por Tzelepi (11) sugiere el uso de ERIC-PCR en la tipificación de Enterobacter cloacae y Enterobacter aerogenes de origen hospitalario productores de BLEE.
El hecho de que el aislamiento 5747 obtenido de un paciente remitido de otra institución presentara un patrón electroforético diferente al clon endémico, permite afirmar que las tres metodologías aplicadas en este trabajo discriminan aislamientos de la misma especie no relacionados epidemiológicamente.
La tipificación de Enterobacter cloacae con BOX-PCR no se había informado hasta el momento, aunque esta metodología se ha aplicado otros microorganismos como Streptococcus pneumoniae, Burkholderia cepacia y Enterococcus faecalis, los autores de estos trabajos concuerdan en afirmar que esta metodología tiene un adecuado poder de discriminación buena reproducibilidad (14,27,28). Coenye y colaboradores (14) señalan que esta metodología puede ser una alternativa de tipificación rápida y menos costosa con respecto a técnicas como la electroforesis en gel por campos pulsados.
Los resultados obtenidos de este trabajo permiten recomendar esta metodología junto con rep-PCR como sistemas adecuados de tipificación de Enterobacter cloacae, con buena reproducibilidad y obtención de resultados en corto tiempo.
De otro lado, Cantón (9) informó prevalencia de clones endémicos de Enterobacter cloacae multirresistentes, productores de BLEE, aisladosdurante 12 años en diferentes áreas de un hospital de España. En contraste, Tzelepi en el Hospital Central de Grecia (11), encontró diversos clones de Enterobacter cloacae los cuales tenían plásmidos comunes que codificaban para BLEE, en su mayoría del tipo SHV-5, y también describió aislamientos de Enterobacter cloacae hiperproductores de â-lactamasa cromosomal AmpC que simultáneamente producen BLEE y concluye que hay una diseminación de plásmidos y no de clones particulares. En nuestro estudio la integración de la información epidemiológica con los resultados de la tipificación molecular permitió detectar un clon endémico multirresistente el cual tuvo una mayor asociación con pacientesintervenidos quirúrgicamente. Este clon podría estar en diferentes unidades del hospitalpor transmisión cruzada, puesto que componentes de este fueron aislados de pacientes atendidos pre viamente por otro servicio. Los resultados obtenidos no permiten establecer la fuente de contaminación cruzada por lo cual se sugiere la realización de un estudio prospectivo aleatorizado en la institución hospitalaria, que permita ubicarla. Los componentes de este clon fueron resistentes a cefalosporinas de tercera generación muy posiblemente por hiperproducción de AmpC y en ellos también se sugiere la producción simultánea de BLEE.
Este trabajo resalta que la utilización de la biología molecular, como herramienta de la epidemiología clínica, contribuye al conocimiento de la dinámica de las poblaciones bacterianas en el ambiente hospitalario, lo cual es fundamental para la implementación de estrategias que conduzcan al control de la infección hospitalaria y a la contención de la resistencia bacteriana.
Referencias
1. Gaston MA. Enterobacter: an emerging nosocomial pathogens. J Hosp Infect 1988;11:197-208. [ Links ]
2. Nierop WH, Duse AG, Stewaert RG, Bilgeri YR, Koornhof HJ. Molecular epidemiology of an outbreak of Enterobacter cloacae in the neonatal Intensive Care Unit of a Provincial Hospital in Gauteng, South Africa. J Clin Microbiol 1998;36:3085-8. [ Links ]
3. Sanders WE, Sanders CC. Enterobacter spp: Pathogens poised to flourish at the turn of the century. J Clin Microbiol 1997;10:220-41. [ Links ]
4. Dietz H, Pfeifle D, Widemann B.. The signal molecule for b-lactamase induction in Enterobacter cloacae is the anhidromuramyl-pentapeptide. Antimicrob Agents Chemother 1997;41:2113-20. [ Links ]
5. Livermore DM. Clinical significance of beta-lactamase induction and stable derepression on gram-negative rods. Eur J Clin Microbiol 1987;6:439-45. [ Links ]
6. Sanders CC. Chromosomal cephalosporinases responsible for multiple resistance to newer b-lactam antibiotics. Annu Rew Microbiol. 1987;41:573-93. [ Links ]
7. Bush K, Jacoby GA, Medeiros AA. A Functional classification scheme for b-lactamases and its correlation with molecular structure. Antimicrob Agents Chemother 1995;19:1211-33. [ Links ]
8. Bradford P. Extended-Spectrum b-lactamase in the 21st Century: Characterization epidemiology, and detection of this important resistance threat. Clin Microbiol Rev 2001;14:933-51. [ Links ]
9. Canton R, Oliver A, Coque T, Varela MC, Pérez Baquero JF. Epidemiology of extended-spectrum blacatamase- producing Enterobacter isolates in a Spanish hospital during a 12-year period. J Clin Microbiol 2002;40:1232-43. [ Links ]
10. De Champs C, Sirot D, Chanal C, Bonnet R, J. Sirot and the French study group. A 1998 Survey of extended-spectrum of b-lactamases among enterobacteriaceae in France. Antimicrob Agents Chemother. 2000;44:3177-9. [ Links ]
11. Tzelepi E, Giakkoupi P, Sofianou D, Loukova V, Kermeroglou A, Tsakris A. Detection of extendedspectrum b-lactamases in clinical isolates of Enterobacter cloacae and Enterobacter aerogenes. J Clin Microbiol 2000;38:542-6. [ Links ]
12. Arbeit RD. Laboratory Procedures for the Epidemiologic Analysis of Microorganisms. En: Murray PR, Baron EJ, Pfaller MA, Tenover FC, Yolken RH (ed) Manual of Clinical Microbiology. 6th ed. Washington D.C. ASM Press.1995:190-208. [ Links ]
13. Weischer M, Kolmos HJ. Ribotyping of selected isolates of Enterobacter cloacae and clinical data related to biotype, phage type, 0-serotype, and ribotype. APMIS 1993;101:879-86. [ Links ]
14. Coenye T, Spilker T, Martin A, Lipuma J. Comparative assessment of genotyping methods for epidemiologic study of Burkhorlderia cepacia Genomovar III. J Clin Microbiol 2002;40:3300-07. [ Links ]
15. Malathum K, Singh K, Weinstock G, Murray B. Repetitive sequence-based PCR versus Pulsed-field gel electrophoresis for typing of Enterococcus faecalis at the subspecies level. J Clin Microbiol 1998;36:211-15. [ Links ]
16. Versalovic J, Koeuth T, Lupski R. Distribution ofrepetitive DNA sequences in eubacteria and application to finger printing of bacterial genomes. Nucleic Acids Research 1991;19:6823-31. [ Links ]
17. Rademaker JL, Brujin FJ. Characterization and classification of microbes by rep_PCR genomic fringerprinting and computer assisted pattern analysis in: Caetano-Anollés G, and Gresshoff PM. DNA Markers. New York: Wiley-VCH; 1998:151-70. [ Links ]
18. National Committee for Clinical Laboratory Standards. Performance standards for antimicrobial susceptibility testing. Eleven Informational Supplement. NCCLS approved standard M100-S11. National Committee for Clinical Laboratory Standards, Wayne, PA. 2001. [ Links ]
19. Matthew M, Harris AM, Marshall H, Ross GW. The use of analytic isoelectric focusing for detection and identification of b-lactamases. J Gen Microbiol 1975;88:169-78. [ Links ]
20. Sambroock J, Fritsh EF. Maniatis T. Molecular cloning. A laboratory Manual, 2nd Ed. 3 vols. Cold Spring Harbor laboratory Press, Cold Spring Harbor,NY. 1989. [ Links ]
21. Center for Disease Control and Prevention, Hospital infection Program. National Nosocomial Infections Surveillance (NNIS) report, data summary from October 1986-April 1996, issued May 1996. A report from the NNIS System. Am J Infect Control 1996;24:380-38. [ Links ]
22. Shi Z, Liu PY, Lau Y, Lin Y, Hu B. Epidemiological typing of isolates from an outbreak of infection with Multidrug-resistant Enterobacter cloacae by repetitive extragenic palindromisc Unit b1-Primed PCR and Pulsed-field gel electrophoresis. J Clin Microbiol 1996;34:2784-90. [ Links ]
23. Pitout JD, Thomson KS, Hanson ND, Ehrhardt AF, Coudron P, Sanders CC. Plasmid-mediated resistance to extended-spectrum cephalosporins among Enterobacter strains. Antimicrob Agents Chemother 1998;42:596-600. [ Links ]
24. Gottlieb T. Wolfson C. Comparison of the MICs of cefepime for extended-spectrum -lactamase JAC 2000;46:323-42. [ Links ]
25. Aibinu IE, Ohaegbulam VC, Adenipekun EA, Ogunsola FT, Odugbemi TO, Mee BJ. Extended spectrum b-lactamase enzymes in clinical isolates of Enterobacter species from Lagos, Nigeria. J Clin Microbiol 2003;41:2197-2200. [ Links ]
26. Neuwirth C, Siebor E, López J, Pechinot A, Kazmierzak A. Outbreak of TEM-24-producing Enterobacter aerogenes in ha intensive care init and dissemination of the extended-spectrum â-lactamase to other members of the family Enterobacteriaceae. J. Clin. Microbiol. 1996;34: 76-79. [ Links ]
27. Wong H, Lin C. Evauation of typing of Vibrio paraemolyticus by three PCR methods using specific primers. J Clin Microbiol 2001;39 :4233-40. [ Links ]
28. Van-Belkum A, Sluijter M, Groot R, Verbrugh H, Hermans P. Novel BOX repeat PCR assay for highresolution typing of Streptococcus pneumoniae strains. J Clin Microbiol 1996;34:1176-79. [ Links ]













