Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de la Facultad de Medicina
Print version ISSN 0120-0011
rev.fac.med. vol.57 no.4 Bogotá Oct./Dec. 2009
INVESTIGACIÓN ORIGINAL
DETERMINACIÓN DEL GEN aac(6')-aph(2") ASOCIADO CON RESISTENCIA A AMINOGLUCÓSIDOS EN CEPAS DE STAPHYLOCOCCUS COAGULASA NEGATIVA EN UNA UNIDAD NEONATAL EN BOGOTÁ
Detection of gene aac(6')-aph(2") in associated with aminoglucosides resistance in Staphylococcus coagulase-negative strains isolates from one neonatal unit in Bogota.
Liliana Muñoz M.1, Gladys Pinilla B1,Ariel I. Ruiz-Parra2,Yolanda Cifuentes C3,Eva A. Gallego1
1 Profesora, Facultad de Ciencias de la Salud, programa de Bacteriología y Laboratorio Clínico, Universidad Colegio Mayor de Cundinamarca. 2 Profesor Titular, Departamento de Obstetricia y Ginecología e Instituto de Investigaciones Clínicas. Facultad de Medicina, Universidad Nacional de Colombia, Bogotá.3Profesora Asociada, Departamento de Pediatría, Facultad de Medicina, Universidad Nacional de Colombia, Bogotá. Correspondencia:airuizp@unal.edu.co
Recibido para publicación:25/11/09
Aceptado para publicación:01/12/09
Resumen
Antecedentes. La causa más común de resistencia a antibióticos aminoglucósidos en bacterias Gram positivas, especialmente en S. epidermidis, es la enzima modificante AAC(6')-APH(2"), capaz de acetilar y fosforilar un amplio rango de antibióticos.
Objetivo. Determinar la presencia del gen aac(6')-aph(2") en cepas de Staphylococcus coagulasa negativa aisladas en infecciones neonatales, e investigar la concordancia con las pruebas de sensibilidad in-vitro.
Material y métodos. Se determinó la presencia del gen aac(6')-aph(2") en 63 cepas de estafilococos coagulasa negativa provenientes de hemocultivos y puntas de catéter, de la Unidad de Neonatología del Instituto Materno Infantil, en Bogotá.
Resultados. Staphylococcus epidermidis fue el germen más frecuente y el gen aac(6')-aph(2") estaba presente en 55 (87,3%) cepas; de éstas, 42 (73,4%) cepas provenían de hemocultivos y 13 (23,6%) cepas de punta de catéter. La susceptibilidad a gentamicina se determinó mediante concentración inhibitoria mínima y para amikacina por difusión en disco. No se encontró asociación estadísticamente significativa entre la presencia del gen aac(6')-aph(2") y la resistencia a gentamicina y amikacina.
Conclusión. La presencia del gen aac(6')-aph(2") de resistencia a aminoglucósidos es muy alta en cepas de Staphylococcus epidermidis. Diferencias en la expresión del gen pueden explicar parcialmente la falta de consistencia con las pruebas de susceptibilidad utilizadas en la clínica.
Palabras clave: enzimas, infecciones estafilocócicas, Staphylococcus epidermidis, recién nacido, farmacorre¬sistencia bacteriana.
Muñoz-M L, Pinilla-B G, Ruíz-Parra AI, Cifuentes-Cifuentes Y, Gallego EA. Determinación del gen aac(6')-aph(2") asociado con resistencia a aminoglucósidos en cepas de Staphylococcus coagulasa negativa en una unidad neonatal en Bogotá. rev.fac.med. 2009; 57: 326-333.
Summary
Background. The most common cause of resistance to aminoglucosides is the acetilation and phosphorilation of the antibiotic by the enzyme AAC(6')APH(2").
Objetive. To determine the presence of the gene aac(6')-aph(2") in strains of coagulase-negative Staphylococci isolated from infected neonates and to investigate the concordance with the susceptibility tests in-vitro.
Materials and methods. the aac(6')-aph(2") gene was determined in 63 coagulase-negative Staphylococci strains isolated from blood cultures and catheter tips obtained from the neonatal care unit at the Instituto Materno Infantil in Bogotá, Colombia.
Results. Staphylococcus epidermidis was the most frequently identified microorganism. The aac(6')-aph(2") gene was detected in 55 out of 63 strains (73,43%), 42 strains (87,5%) isolated from blood cultures, and 13 strains (23,6%) isolated from catheter tips. Susceptibility to gentamycin was determined by minimum inhibitory concentration, and susceptibility to amikacin by the disk diffusion antibiotic sensitivity test. There was no a significant statistical association between the presence of the gene and the microbial susceptibility to either gentamycin or amikacin.
Conclusion. The presence of the aac(6')-aph(2") gene in strains of Staphylococcus epidermidis is very high. Differences in the expression of the gene might explain some cases of inconsistency with the susceptibility tests.
Key words: enzymes, staphylococcal infections, Staphylococcus epidermidis, infant, newborn, drug resistance, bacterial.
Muñoz-M L, Pinilla-B G, Ruíz-Parra AI, Cifuentes-Cifuentes Y, Gallego EA. Detection of gene aac(6')-aph(2") in associated with aminoglucosides resistance in Staphylococcus coagulase-negative strains isolates from one neonatal unit in Bogota. rev.fac.med. 2009; 57: 326-333.
Introducción
Los aminoglucósidos (AG) son antibióticos de amplio espectro, utilizados en la práctica clínica para el tratamiento de infecciones causadas por bacterias Gram positivas y Gram negativas. Su mecanismo de acción es la inhibición de la síntesis de proteínas por la unión del antibiótico a la subunidad pequeña 30S de los ribosomas bacterianos (1,2). La forma más común de resistencia bacteriana a estos antibióticos es la adquisición de genes que codifican enzimas encargadas de modificar covalentemente los grupos amino e hidroxilo del antibiótico, disminuyendo la afinidad de unión a la subunidad 30S (3,4).
El gen aac(6')-aph(2"), que se cree es el resultado de la fusión de los genes aac y aph (5), se puede hallar tanto en plásmidos como en el cromosoma bacteriano (6,7). El gen fue encontrado inicialmente en Staphylococcus aureus y Staphylococcus epidermidis (8,9) y luego en aislamientos clínicos de Enterococcus, Streptococcus y Staphylococcus coagulasa negativa (SCN) (10,11). Este gen codifica la enzima bifuncional ACC(6')-APH(2") (12) que inactiva los antibióticos aminoglucósidos como gentamicina, tobramicina, neomicina y amikacina. La enzima posee dos actividades diferentes que inactivan a los AG; una de ellas es un aminoglucósido-acetiltransferasa (AAC) que acetila grupos amino en la posición 6', y la otra es un aminoglucósido-fosfotransferasa (APH) que fosforila el grupo hidroxilo en la posición 2" (13).
La resistencia a los AG en las infecciones nosocomiales causadas por SCN tiene una alta prevalencia en las unidades de neonatología y se asocia con una elevada tasa de morbimor¬talidad (14-16). La determinación de la suceptibilidad para los AG es usualmente realizada en los laboratorios de microbiología por las pruebas difusión en disco o por la concentración inhibitoria mínima (CIM); sin embargo, es importante conocer también la presencia de genes que confieren resistencia a los AG. El objetivo de este estudio fue el de determinar la presencia del gen aac(6')-aph(2") en cepas de SCN aisladas en infecciones neonatales y establecer la concordancia con las pruebas de sensibilidad in-vitro, con el propósito de contribuir a la caracterización de la resistencia microbiana y el uso racional de los antibióticos.
Material y métodos
Aislamientos
Se investigaron 63 cepas de SCN aisladas a partir de hemocultivos y cultivos de puntas de catéter provenientes de 57 recién nacidos admitidos en las unidades de neonatología del IMI. La identificación y la concentración inhibitoria mínima (CIM) a gentamicina de las especie de los SCN se llevó a cabo mediante el uso de páneles Gram positivos MIC/Combo MicroScan® (P35) siguiendo las instrucciones del fabricante, y la lectura se hizo por el sistema automatizado MicroScan® auto Scan-4 (Roche Biocare, Canadá). La prueba de susceptibilidad a amikacina se determinó por el método de difusión en disco (concentración 30 μg). Las cepas control fueron S. epidermidis ATCC 12228 y S. aureus ATCC 29213; los resultados se interpretaron de acuerdo con las normas NCLSI (17).
Amplificación de gen aac(6')aph(2")
La extracción de ADN se realizó a partir de un cultivo puro, de acuerdo con el protocolo del fabricante (proDNA2003, CorpoGen-Colombia). La amplificación se hizo por PCR múltiplex, usando para la detección del gen aac(6')aph(2") los oligonucleótidos 5'TTGGGAAGATGAAGTTTTTAGA3' 5'CCTTTACTCCAATAATTTGGCT3' (GenBank M13771), como control interno el RNA ribosomal 16S (16SrRNA) 5'GGAGGAAGGTGGGGATGACG3' 5'ATGGTGTGACGGGCGGTGTG3'(Gen Bank J01859), y para confirmar la presencia de S. epidermidis los oligo¬nucleó¬tidos¬ 5'ATCAAAAAGTTGGCGAACCTTTTCA3' y 5'CAAAAGAGCGTGGAGAAAAGTATCA3 (GenBank N/A) (18).
En cada corrido como control positivo se utilizó la cepa S. epidermidis Rescol 13, proporcionada por el Instituto de Genética Molecular Bacteriana de la Universidad del Bosque, Bogotá, como control negativo la cepa de S. epidermidis ATTC 12228 y como control ambiental la mezcla de reactivos, sin ADN diana.
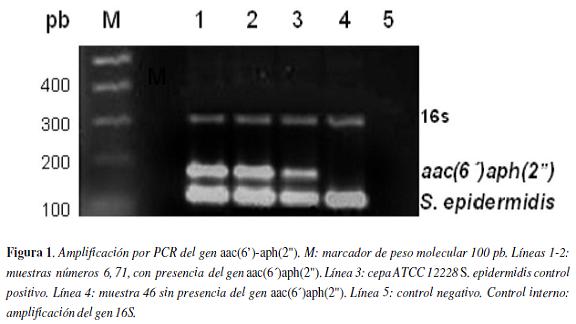
Las condiciones de reacción fueron: dNTPs 200 μM, Go Taq polimerasa 2,5 U (Promega), e iniciadores 0,4 μM para la amplificación del gen aac(6')aph(2"), y 0,1 μM para el control interno y la determinación específica del microorganismo. Las reacciones se llevaron a cabo en el termociclador PTC-100 (MJ Research Inc. Alameda, EE.UU.). Las condiciones de amplificación fueron: desnaturalización inicial a 96° C por tres minutos, seguida de 34 ciclos de desnaturalización a 95° C por 30 segundos, anillamiento a 55° C por 30 segundos, extensión a 72° C por 30 segundos y una extensión final a 72° C por siete minutos. La detección de los productos de amplificación se efectuó mediante electroforesis en gel de agarosa al 2 por ciento, seguido de coloración con bromuro de etidio 0,5 μg/mL figura 1.
Análisis
El análisis de los datos se hizo utilizando el programa STATA 8.0; se aplicó estadística descriptiva informando frecuencias absolutas y relativas y se utilizó la prueba exacta de Fisher para independencia de las variables categóricas.
Consideraciones éticas
El protocolo fue aprobado por los comités de ética de las instituciones participantes. Los aislamientos corresponden a muestras tomadas durante el manejo clínico estándar de los neonatos y no se tomaron muestras exclusivamente para los fines del estudio.
Resultados
Durante el período comprendido entre el 21 de noviembre de 2003 y el 20 de mayo de 2004 se aislaron 48 (76,19%) cepas de SCN provenientes de aislamientos de hemocultivos y 15 (23,81%) cepas de SCN obtenidas de aislamientos de puntas de catéter, de 57 recién nacidos en la Unidad de Neonatología del Instituto Materno Infantil (IMI).
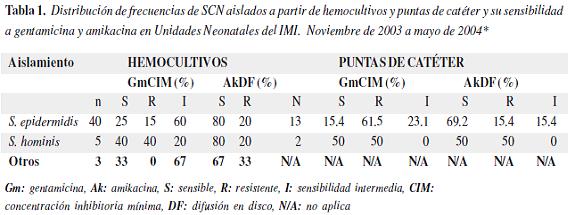
La mayoría de los recién nacidos recibieron tratamiento con vancomicina asociada con gentamicina o amikacina. La tabla 1 muestra la distribución de frecuencias de los SCN aislados de hemocultivo y cultivos de punta de catéter y los resultados de la prueba de susceptibilidad a gentamicina y amikacina.
La interpretación de la prueba de sensibilidad para la gentamicina por el método de CIM fue: cepas sensibles (menor o igual a 4 mg/L), aislamientos con susceptibilidad intermedia (8 mg/L) y cepas resistentes (mayor de 8 mg/L), de acuerdo con las indicaciones del fabricante y las normas NCLSI (17). La interpretación de las lecturas para sensibilidad a la amikacina por difusión en disco fue la siguiente: sensible mayor o igual a 17mm, sensibilidad intermedia: 16-15mm y resistente menor o igual a 14mm.
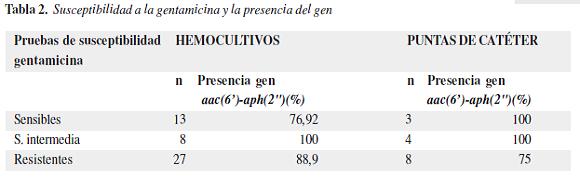
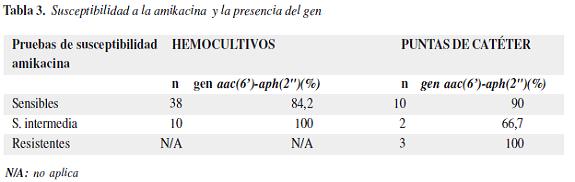
Se buscó el gen aac(6')aph(2") en las cepas en estudio y se identificó en 42 (87,5%) de las 48 cepas de SCN provenientes de hemocultivos y en 13 (86,67%) de las 15 cepas provenientes de cultivos de puntas de catéter. Las tablas 2 y 3 señalan los resultados de la prueba de susceptibilidad y la presencia o ausencia del gen en los aislamientos de hemocultivos y puntas de catéter, respectivamente.No se encontró asociación estadísticamente significativa entre la presencia del gen aac(6')-aph(2") y los resultados de las pruebas de resistencia a gentamicina en cepas de SCN obtenidas a partir de hemocultivos (p = 0,284) y cultivos de punta de catéter (p = 0,364)tabla 2. Tampoco se halló asociación estadísticamente significativa entre la presencia del gen y la resistencia a amikacina en cepas de SCN obtenidas a partir de muestras de hemocultivos (p = 0,179) y de puntas de catéter (p = 0,486)tabla 3.
Discusión
Los SCN, y entre ellos el S. epidermidis aislado con mayor frecuencia en muestras instituciones, son considerados patógenos causantes de bacteremias e infecciones asociadas con catéteres. Esta situación es particularmente importante en pacientes inmunocomprometidos, como los neonatos (14-16), en quienes en la mayoría de los casos se encuentran relacionados con multirresistencia a antibióticos, y pueden servir de reservorio a diversos genes que les confieren resistencia (19).
Aunque la vancomicina es el antibiótico de elección en este tipo de infecciones neonatales, se ha reportado resistencia en Staphylococcus y Streptococcus, por lo cual se ha recomendado controlar su uso en las infecciones causadas por estos microorganismos (20).
El estudio de la sensibilidad a los AG en muestras obtenidas de recién nacidos de la Unidad Neonatal del Instituto Materno Infantil evidenció una alta tasa de resistencia a gentamicina para las cepas aisladas de hemocultivos (56,3%) y de cultivos de puntas de catéter (53,3 %). Estos resultados son similares a los reportados en un estudio que incluyó 19 hospitales europeos, donde los SCN presentaron un 54 por ciento de resistencia a gentamicina (21). La resistencia a amikacina fue menor de 20,8 por ciento para las cepas aisladas de hemocultivos y de 20 por ciento para las cepas aisladas de cultivos de punta de catéter); por lo tanto, en los casos de sepsis neonatal la amikacina debería ser considerada como terapia antimicrobiana inicial en asociación con un antibiótico ?-lactámico.
El gen aac(6')-aph(2") estuvo presente en 55 de las 63 cepas (87,3%) de SCN, prevalencia idéntica a la informada para Staphylococcus por el grupo europeo de resistencia a antibióticos (ESGAR) (22). Udo E. y colaboradores, en el año 2000 reportaron que sobre un total de 70 cepas de Staphylococcus estudiadas, 30 correspondieron a SCN y en 24 de éstas (80%) el gen acc(6')-aph(2") estaba presente (23). Sin embargo, otros autores hallaron la presencia del gen en un porcentaje menor (68%) de todas las cepas de SCN (24). Las discrepancias en los resultados se pueden explicar por diferencias en el uso de antibióticos, por la dispersión y diseminación de cepas intrahospitalarias resistentes y por el tipo de población estudiada, puesto que la mayoría de los trabajos realizados incluyen pacientes adultos con sepsis.
Las pruebas de susceptibilidad in-vitro pueden no reflejar apropiadamente la respuesta in-vivo, lo que supone la necesidad de correlacionar el resultado clínico con la presencia o ausencia del gen. La correlación de los desenlaces clínicos neonatales con los perfiles de susceptibilidad antimicrobiana se dificultaron en nuestro estudio, pues los neonatos con cultivos positivos para SCN recibieron como tratamiento una asociación de vancomicina más AG y no se encontraron cepas resistentes a este antibiótico (16).
La falta de asociación entre la presencia del gen aac(6')-aph(2") y las pruebas de susceptibilidad puede indicar que la prueba microbiológica convencional no es suficientemente sensible ni específica, ya que el 81,25 por ciento de las cepas sensibles a gentamicina presentaban el gen y en el 85,42 por ciento de las cepas sensibles para amikacina se encontró el gen. Sin embargo, esto no implica su expresión ni la función apropiada de la proteína, y por lo tanto se requeriría determinar también los perfiles de expresión del gen y la secuenciación de éste.
Las cepas que presentaron resistencia y no presentaron el gen se podrían explicar por la existencia de otros mecanismos de resistencia, tales como la presencia de integrones y genes cassettes, entre otros, los cuales han sido asociados a la resistencia de AG, y en las diferencias entre las características fenotípica y genotípicas en cepas bacterianas (25).
Dado que los AG hacen parte de los protocolos de tratamiento para la sepsis neonatal la identificación de genes que confieren resistencia a los AG en las cepas aisladas de los pacientes podría ser útil para racionalizar el uso de estos antibióticos.
Agradecimientos
A María Teresa Herrera, por sus aportes académicos a la realización de este trabajo, y al Instituto Materno Infantil de Bogotá.
Financiación
El trabajo fue realizado y financiado por la Universidad Colegio Mayor de Cundinamarca y por la Universidad Nacional de Colombia.
Referencias
1. Kotra L, Haddad J, Mobashery S. Aminoglycoside: Perspectives on mechanisms of action and resistance and strategies to counter resistance. Antimicrob Agents Chemother. 2000; 44: 3249-3256. [ Links ]
2. Francois B, Rusell R, Murray J, Aboul-ela F, Masquida B, Vicens Q, Westhof E. Crystal structures of complexes between aminoglycosides and decoding A site oligonucleotides: role of the number of rings and positive charges in the specific binding leading to miscoding. Nucleic Acids Res. 2005; 33: 5677-5690. [ Links ]
3. Azucena E, Mobashery S. Aminoglycoside-modifying enzymes: mechanisms of catalytic processes and inhibition. Drug Resist Updat. 2001; 4: 106-117. [ Links ]
4. Boehr DD, Daigle DM, Wright GD. Domain-domain interactions in the aminoglycoside antibiotic resistance enzyme AAC(6')-APH(2¨). Biochemistry. 2004; 43: 9846–9855. [ Links ]
5. Daikos GL, Bamias G, Kattamis C, Zervos MJ, Chow JW, Christakis G, et al. Structures, locations, and transfer frequencies of genetic elements conferring high-level gentamicin resistance in Enterococcus faecalis isolates in Greece. Antimicrob Agents Chemother. 2003; 47: 3950-3953. [ Links ]
6. Ferretti JJ, Gilmore KS, Courvalin P. Nucleotide sequence analysis of the gene specifying the bifunctional 6-aminoglycoside acetyltransferase 2-aminoglycoside phosphotransferase enzyme in Streptococcus faecalis and identification and cloning of gene regions specifying the two activities. J Bacteriol. 1986;167: 631-638. [ Links ]
7. Horaud T, de Céspèdes G, Trieu-Cuot P. Chromosomal gentamicin resistance transposon Tn3706 in Streptococcus agalactiae B128. Antimicrob Agents Chemother. 1996; 40: 1085-1090. [ Links ]
8. Ubukata K, Yamashita N, Gotoh A, Konno M. Purification and characterization of aminoglycoside-modifying enzymes from Staphylococcus aureus and Staphylococcus epidermidis. Antimicrob Agents Chemother. 1984; 25: 754-759. [ Links ]
9. Chow JW, Kak V, You I, Kao SJ, Petrin J, Clewell DB, Lerner SA, Miller H, Shaw KJ. Aminoglycoside resistance genes aph(2")-Ib and aac(6')-Im detected together in strains of both Escherichia coli and Enterococcus faecium. Antimicrob Agents Chemother. 2001; 45: 2691-2694. [ Links ]
10. Galimand M, Lambert T, Gerbaud G, Courvalin P. High-level aminoglycoside resistance in the beta-hemolytic group G Streptococcus isolate BM2721. Antimicrob Agents Chemother. 1999; 43: 3008-3010. [ Links ]
11. Ardic N, Sareyyupoglu B, Ozyurt M, Haznedaroglu T, Ilga U. Investigation of aminoglycoside modifying enzyme genes in methicillin-resistant Staphylococci. Microbiol Res. 2006; 161: 49-54. [ Links ]
12. Wright G, Berghuis A, Mobashery S. Aminoglycoside antibiotics. Structures, functions and resistance. Adv Exp Med Biol. 1998; 456: 27-69. [ Links ]
13. Davies, J, Wright GD. Bacterial resistance to aminoglycoside antibiotics. Trends Microbiol. 1997; 5: 234-240. [ Links ]
14. Cifuentes Y, Ruiz A, Leal A, Muñoz L, Herrera M, Jiménez L. Perfil microbiológico en asilamientos en Unidades neonatales en un hospital de tercer nivel de Bogotá, Colombia. Rev Salud Pública Bogotá. 2005; 7: 191-205. [ Links ]
15. Leboucher B, Leblanc M, Berlie I, Savagner C, Lemarie Le Bouedec S. C, Effectiveness of an informative report on the prevention of nosocomial bloodstream infections in a neonatal intensive care unit. Arch Pediatr. 2006; 13: 436-441 [ Links ]
16. Karlowicz M, Buescher E, Surka A. Fulminat late-onset sepsis in a neonatal late-onset sepsis in a neonatal intensive care unit., 1988-1997 and the impact of avoiding empiric vancomycin therapy. Pediatrics. 2000; 106: 137-190. [ Links ]
17. Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing; sixteenth informational supplement. M100-S17. Wayne, PA: CLSI; 2007. [ Links ]
18. Martineau F, Picard F, Grenier L, Roy P, Ouellette M, Bergeron M. Muliplex PCR assay for the detection of clinically relevant antibiotic resistance genes in Staphylococci isolated from patients infected after cardiac surgery. The ESPRIT Trial. J Antimicrob Chemother. 2000;46:527-533. [ Links ]
19. Piette A, Verschraegen G. Role of coagulase-negative staphylococci in human disease. Vet Microbiol. 2009; 134: 45-54. [ Links ]
20. Karlowicz M, Buescher E, Surka A. Fulminat late-onset sepsis in a neonatal late-onset sepsis in a neonatal intensive care unit., 1988-1997 and the impact of avoiding empiric vancomycin therapy. Pediatrics. 2000;106: 137-190. [ Links ]
21. Schmitz F-J, Fluit A, Gondolf M , Beyraua R, Lindenlaufa E, Verhoef J, et al. The prevalence of aminoglycoside resistance and corresponding resistance genes in clinical isolates of Staphylococci from 19 European hospitals. J Antimicrob Chemother. 1999; 43: 253-259. [ Links ]
22. Dornbusch K, Miller G, Hare R, Shaw K. Resistance to aminoglycoside antibiotics in gram-negative bacilli and Staphylococci isolated from blood. Report from a European collaborative study. The ESGAR Study Group (European Study Group on Antibiotic Resistance). J Antimicrob Chemother. 1990; 26: 131-144. [ Links ]
23. Udo EE, Dashti AA. Detection of genes encoding aminoglycoside-modifying enzymes in Staphylococci by polymerase chain reaction and dot blot hybridization. Int J Antimicrob Agents. 2000; 13: 273-279. [ Links ]
24. Culebras E, Marinesz J. Aminoglycoside resistance mediated by the bifunctional enzyme 6'-N-aminoglycoside acetyltransferase-2"-O-aminoglycoside phosphotransferase. Front Biosci. 1999; 4: D1-8. [ Links ]
25. Shi L, Zheng M, Xiao Z, Asakura M, Su J, Li L, Yamasaki S. Unnoticed spread of class 1 integrons in Gram-positive clinical strains isolated in Guangzhou, China. Microbiol Immunol. 2006; 50: 463-467. [ Links ]