Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista de la Facultad de Medicina
versión impresa ISSN 0120-0011
rev.fac.med. vol.63 no.3 Bogotá jul./sep. 2015
https://doi.org/10.15446/revfacmed.v63n3.49352
DOI: http://dx.doi.org/10.15446/revfacmed.v63n3.49352
ARTÍCULO DE REVSIÓN
Genética molecular del alcoholismo
Molecular genetics of alcoholism
Mauricio Rey-Buitrago1
1 Maestría en Genética Humana, Facultad de Medicina. Universidad Nacional de Colombia. Bogotá, D.C., Colombia.
Correspondencia: Mauricio Rey-Buitrago. Carrera 45 No. 26-85, Instituto de Genética, Edificio 426, Universidad Nacional de Colombia. Bogotá, D.C., Colombia. Tel: +57 1 3165000, ext: 11610. Correo electrónico: mrey@unal.edu.co.
Recibido: 25/02/2015 Aceptado: 27/04/2015
Resumen
El alcoholismo es una patología psiquiátrica compleja y de origen multifactorial en la que el factor genético explica alrededor del 50 % del fenómeno. Son numerosos los genes que se han asociado a esta enfermedad, pero su aporte individual es mínimo y contradictorio. Estos genes operan a través de características intermedias como la impulsividad y la sensibilidad al alcohol, lo que hace compleja la definición del fenotipo del alcoholismo. Los estudios de asociación de SNPs, de asociación a todo el genoma, de expresión y epigenéticos han identificado una amplia gama de variantes genéticas y epigenéticas, blancos para los estudios de susceptibilidad, diagnóstico y tratamiento farmacológico. Actualmente se comprenden mucho más estas relaciones y el desarrollo rápido de nuevas metodologías de estudio promete continuar este proceso, así como la generación de algoritmos de diagnóstico, prevención y tratamientos más acertados y confiables.
Palabras clave: Alcoholismo; Genómica; Polimorfismo de nucleótido simple; Farmacogenómica; Epigenética (DeCS).
Rey-Buitrago M. Genética molecular del alcoholismo. Rev. Fac. Med. 2015;63(3):483-94. Spanish. doi: http://dx.doi.org/10.15446/revfacmed.v63n3.49352.
Summary
Alcoholism is a complex and multifactorial psychiatric pathology in which the genetic factor explains about 50% of the phenomenon. Numerous genes have been associated with the disease, but their individual contribution is minimal and contradictory. These genes operate through intermediate characteristics such as impulsivity and sensitivity to alcohol, which makes the definition of alcoholism phenotype a complex one. Association of SNPs, genome-wide association, expression and epigenetic studies have identified a wide range of genetic and epigenetic variants, targets for susceptibility studies, diagnosis and drug treatment. Currently, there is much more understanding of these relationships, and the rapid development of new methods of study promises to continue this process as well as the generation of algorithms for diagnosis, prevention and successful reliable treatments.
Keywords: Alcoholism; Genomics; Polymorphism, Single nucleotide; Pharmacogenomics; Epigenetics (MeSH).
Rey-Buitrago M. [Molecular genetics of alcoholism]. Rev. Fac. Med. 2015;63(3):483-94. Spanish. doi: http://dx.doi.org/10.15446/revfacmed.v63n3.49352.
Introducción
La Organización Mundial de la Salud (OMS) define el alcoholismo o alcohol–dependencia como el consumo crónico y continuado o periódico de alcohol que se caracteriza por un deterioro del control sobre la bebida, episodios frecuentes de intoxicación, obsesión por el alcohol y consumo a pesar de sus consecuencias adversas (1-3). El consumo excesivo de alcohol es uno de los problemas de salud pública de mayor magnitud con relación a psicoactivos de uso legal, siendo una problemática de alcance mundial que pone en peligro el desarrollo individual, la vida familiar y la convivencia social de una persona. Se estima que en el mundo cerca 2600 millones de personas consumen alcohol, sea de forma ocasional, habitual, abusiva o adictiva. Anualmente, 2.5 millones de personas mueren por causas relacionadas con el alcohol (4-6).
En Colombia, alrededor del 86% de los participantes en las últimas encuestas de consumo de sustancias psicoactivas (7,8) manifestó haber consumido alcohol alguna vez en su vida. El 35% había consumido bebidas alcohólicas en los últimos 30 días. Se calcula que 2.4 millones de colombianos tienen problemas con el consumo de alcohol. En el ámbito escolar y universitario, dos de cada tres encuestados declaran haber consumido alguna bebida alcohólica en su vida. El 40% de los estudiantes de 11 a 18 años declararon haber consumido algún tipo de bebida alcohólica durante el último mes (9,10). Estas cifras muestran que el consumo de alcohol es un grave problema de salud pública en Colombia.
El consumo de drogas lícitas e ilícitas, particularmente de alcohol, se relaciona con más de 60 condiciones de salud, que van desde las que son resultado de un consumo excesivo durante el embarazo y que afectan al feto, hasta lesiones intencionales, enfermedades hepáticas y condiciones neuropsiquiátricas, incluyendo la dependencia. De igual forma, se relaciona con el desarrollo de eventos epidémicos —cardiovasculares, cáncer y otros que se están incrementando— , con enfermedades sociales y mentales, con la violencia intrafamiliar, la morbilidad y mortalidad por accidentes de tráfico, el suicidio, el absentismo laboral y escolar, los problemas interpersonales y las conductas sexuales peligrosas (11).
El alcoholismo es una enfermedad psiquiátrica crónica y multifactorial cuyo desarrollo está influenciado por numerosos factores fisiológicos, genéticos, psicosociales y ambientales. Dilucidar el aporte de cada uno de estos factores al fenotipo —tan heterogéneo— de alcoholismo humano es complicado, pues cada uno de ellos, en especial el genético y el ambiental, son complejos de controlar. No todos los individuos que consumen alcohol se vuelven alcohólicos, uno de los factores desencadenantes es la vulnerabilidad o susceptibilidad biológica, que es alta en el alcoholismo. Se entiende por susceptibilidad biológica la totalidad de condiciones con las que un individuo nace, que le hacen más o menos predispuesto al desarrollo de una condición patológica (12,13).
Las investigaciones que emplean muestras de individuos de una misma familia, hermanos gemelos, hijos adoptados y personas no emparentadas revelan más del 50% del componente genético involucrado en las conductas que conllevan a una adicción al alcohol (14). De acuerdo con lo anterior, la alcohol–dependencia es una enfermedad de herencia compleja y de amplia heterogeneidad, en la que múltiples y diversos genes y alelos pueden estar involucrados con aportes mínimos si se toman de manera individual, pueden ser diferentes de una población a otra, y comunes con otras patologías (15). En la identificación de estos genes se ha enfocado la atención en los asociados con el metabolismo del ¬alcohol —como las alcohol y aldehído deshidrogenasas (ADH y ALDH, respectivamente), los citocromos P450, genes de resistencia al estrés, genes de neuroinflamación, genes de ejecución del control cognitivo— y en los sistemas reforzadores del consumo de alcohol que están involucrados con la adicción —como los sistemas dopaminérgico, serotoninérgico, opioide, acetilcolinérgico, glutamaérgico y gabaérgico, que están relacionados con múltiples neuroadaptaciones que ocurren en el proceso de transición hacia la adicción— (12,13).
Actualmente, con la implementación de estudios de genómica total, transcriptómica, epigenómica, de respuesta a fármacos con herramientas como microarreglos y de secuenciación de ADN y ARN de siguiente generación, los blancos genéticos se han ampliado, diversificado y han permitido analizar la expresión de genes, su estado de activación entre controles y alcohólicos, y la elaboración de redes de genes (14).
Los individuos que empiezan a abusar tempranamente de las drogas se constituyen en el grupo con mayor riesgo de desarrollar una adicción. Si se pudiera reconocer la predisposición en una fase precoz por medio de un perfil genético, se podría dirigir la intervención en prevención hacia esa población, e incluso, en fases posteriores, se podrían proponer alternativas de diagnóstico y tratamiento psíquico o farmacológico, de acuerdo al perfil genético y/o patrones de expresión génica particulares.
Instrumentos de clasificación
El tamaño de la muestra y la identificación correcta de un fenotipo son fundamentales para los estudios genético-moleculares; desafortunadamente, no existen pruebas específicas para el diagnóstico del alcoholismo. Además de la exploración clínica adecuada, se emplean diferentes instrumentos como cuestionarios, encuestas, escalas, etc., que son de gran ayuda en la identificación de consumo de riesgo, consumo perjudicial y alcoholismo. Los cuestionarios CAGE, CBA y AUDIT, por su brevedad y sencillez, son los instrumentos más útiles. Se recomienda emplear aquellos que hayan sido adaptados, flexibles y validados para la población objeto de estudio (17,18).
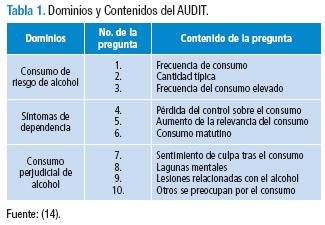
Particularmente, se ha empleado el cuestionario AUDIT (Alcohol Use Disorders Identification Test), desarrollado por la OMS como un método simple de tamizado del consumo excesivo de alcohol y como un apoyo en la evaluación breve. Éste comprende 10 preguntas que indagan sobre el consumo reciente, síntomas de la dependencia y problemas relacionados con el alcohol. AUDIT es un test de tamizado que identifica el consumo de riesgo y perjudicial de alcohol, así como una posible dependencia. Es breve, rápido, flexible y de gran utilidad en atención primaria (2,3,16,17) (Véase Tabla 1).
Cada una de las preguntas tiene una serie de respuestas a elegir y cada respuesta tiene una escala de puntuación de 0-4. Cuando se obtienen puntuaciones totales iguales o mayores a 8 se toman como indicadores de consumo de riesgo; puntuaciones iguales o mayores a 13 se toman como consumo perjudicial y puntuaciones iguales o mayores a 20 se toman como problemas físico-químicos con la bebida y/o posible dependencia alcohólica.
Fenotipos y endofenotipos
El fenotipo de las enfermedades neuropsiquiátricas, y particularmente del alcoholismo, es muy heterogéneo, puede ser modulado por factores ambientales pasando por diversas fases a lo largo de un muy extenso período de tiempo que llevan a la confusión en el diagnóstico clínico (20). Para facilitar su abordaje y con base en estudios de adopción y modelos de neuroaprendizaje, el alcoholismo se puede dividir en dos subtipos: Tipo I, caracterizado por inicio tardío, baja heredabilidad, sentimiento de culpa, preocupación y baja dependencia psicológica. Tipo II, caracterizado por inicio temprano (menos de 25 años), presencia de comportamiento antisocial y reducción del sentimiento de culpa (21). Se han registrado otros intentos de clasificar los fenotipos de alcoholismo atendiendo a otras características, no solamente la edad de inicio, sino también, por ejemplo, teniendo en cuenta su historia familiar o respuesta al tratamiento. Es así que se ha generado otro tipo de clasificación como la siguiente realizada en Estados Unidos: 1. Adultos jóvenes (32%); 2. Funcionales (19%); 3. Familiar intermedio (19%); 4. Joven antisocial (21%); 5. Crónico severo (9%) (21,22).
Aun así, la definición del fenotipo puede ser difícil y, por tanto, se recurre a los endofenotipos. Los endofenotipos de las enfermedades neuropsiquiátricas corresponden a alteraciones bioquímicas, neurofisiológicas, neuroanatómicas o cognitivas, que están determinadas por factores genéticos y ambientales, y que en conjunto muestran procesos fisiopatológicos subyacentes a la enfermedad y expresivos de su vulnerabilidad. En otras palabras, los endofenotipos son intermedios medibles observados entre el desorden y el proceso biológico responsable de la manifestación del desorden (20).
Una estrategia para su selección ha sido el uso de endofenotipos basados en medidas electrofisiológicas y estudios de imágenes. Estas técnicas, por sus costos, naturaleza invasiva, requerimiento de equipos especializados y tiempo de espera para obtener los resultados, es prácticamente imposible de llevar a cabo en estudios genético-poblacionales.
Genes involucrados en la patología
La susceptibilidad genética al consumo de alcohol puede tener su origen en características de personalidad tales como la ansiedad, la disforia y la impulsividad, que predisponen al comportamiento de búsqueda de alcohol, al distinto nivel de respuesta en cada individuo al consumo del psicoactivo y a factores relacionados con respuesta al estrés. Los desórdenes de conducta pueden incrementar el riesgo en el consumo abusivo de alcohol. Muchos de los polimorfismos en genes involucrados en los circuitos cerebrales descritos más adelante están asociados a problemas de conducta (23).
Los polimorfismos de nucleótido simple (SNPs, por sus siglas en inglés), que son sustituciones de una base por otra en la secuencia de un gen, son marcadores genéticos ideales en la búsqueda de genes de susceptibilidad a una enfermedad o genes que determinan la respuesta a un fármaco determinado. Entre sus características ideales se pueden citar su simplicidad, amplia distribución (alta frecuencia en zonas de regulación génica), estabilidad (menos susceptibles de presentar mutaciones) y alta frecuencia a lo largo del genoma (aproximadamente 10 millones en todo el genoma humano) (24-26).
Los hallazgos más robustos de la influencia genética sobre el alcoholismo están relacionados con SNPs de genes que codifican enzimas para el metabolismo del etanol. La primera vía de su metabolismo es la hepática y, en menor proporción, el estómago e intestino. Ambos casos involucran la conversión del etanol a acetaldehído por la alcohol deshidrogenasa (ADH), y este último a acetato por la aldehído deshidrogenasa (ALDH). En el sistema nervioso, la citocromo P4502E1 del sistema microsomal cobra relevancia en bebedores fuertes. Existen 7 genes de ADH y 18 genes de ALDH, pero sólo 4 clases de genes ADH (ADH1A, ADH1B, ADH1C y ADH4) y una de ALDH (ADLH2) juegan un papel importante en su biotransformación. En Asia, las variantes ADH1B*2, que generan una enzima con 100 veces más actividad, y ALDH2*2, con menor actividad, conllevan al fenotipo que presenta enrojecimiento facial, hipotensión, dolor de cabeza y náuseas, por tanto, se consideran un factor de protección (25). El citocromo P4502E1 posee varios polimorfismos, uno de ellos situados en la región reguladora 5' del gen con la variante c1 —la más frecuente— y c2 —inusual—, que posee una tasa de transcripción mayor relacionada con el consumo de alcohol.
En Colombia, la combinación de genotipos correspondientes a ADH1B, ADH1C, ALDH2 y CYP2E1 podría estar llevando a la población a un posible riesgo; dado que, al presentar alelos cuya presencia permite una tasa de oxidación mayor (ADH1B*2, ADH1C*1, CYP2E1c2) combinados con el genotipo homocigoto de ALDH2*1, aumentaría las tasas de aclaramiento del alcohol y acetaldehído, dando como resultado una mayor tolerancia al consumo de mayores cantidades de alcohol y, por ende, generando un factor de riesgo de dependencia (27). Lo anterior se suma a otros factores como los sociológicos, psíquicos y ambientales, que contribuyen fuertemente a la susceptibilidad. En general, las frecuencias obtenidas para los cuatro alelos se acercan más a lo reportado en poblaciones como las europeas, las indoamericanas y las méxico-americanas. De alguna manera, estos hallazgos eran de esperar, ya que en Colombia los primeros asentamientos fueron de amerindios, seguidos de europeos —especialmente españoles— y finalmente de africanos (27).
Los estudios realizados con modelos animales y humanos en los últimos años indican que las adicciones traen como resultado neuroadaptaciones en el sistema nervioso central que van progresando desde la fase de consumo inicial, pasando al consumo compulsivo, hasta llegar al crónico y las recaídas. Los modelos de estudio en roedores han permitido identificar numerosos genes relacionados con la transmisión de señales en el sistema nervioso central, especialmente con los circuitos de recompensa. La euforia experimentada por el humano posterior al consumo de alcohol está asociada con el incremento de dopamina en la sinapsis en las vías de recompensa, además de incluir la alteración de otras vías de neurotransmisores como GABA, acetilcolina, glutamato, serotonina, péptidos opioides y canabinoides (14). En población colombiana, recientemente se evaluó la posible relación de algunos de los polimorfismos de genes involucrados en estas vías con la alcohol-dependencia. Se halló que, para el grupo de pacientes con un puntaje AUDIT entre 14 y 19, la frecuencia del alelo-141C Ins del gen DRD2 (receptor de dopamina tipo 2) arrojó diferencias significativas al compararlo con el grupo control. De igual manera, con el mismo gen se encontró diferencia significativa para el alelo IB del polimorfismo TaqIB en el grupo de mujeres con alcohol-dependencia (28).
El sistema dopaminérgico juega un papel muy importante en la regulación del movimiento, la conducta y la liberación de hormonas. El circuito dopaminérgico cerebral se puede dividir en nigroestriado, mesolímbico-mesocortical y tuberohipofisiario. El sistema mesolímbico–mesocortical tiene su origen en el área tegmental ventral (ATV), con proyecciones hacia la corteza frontal y el lóbulo límbico. En este último, se proyecta hacia el núcleo accumbens (NAc), el tubérculo olfatorio, la amígdala, entre otros. Se ha propuesto que la liberación de dopamina en NAc desde ATV, como parte de un circuito de recompensa cerebral, se asocia con estados de ánimo positivos como el placer o la euforia. El sistema límbico contiene circuitos que no sólo codifican para la magnitud de la recompensa sino también para su almacenamiento en la memoria, al establecer las pertinentes asociaciones entre estímulos —externos e internos— y la recompensa que inducen, lo cual podría estar directamente relacionado con los estados de adicción (29-31).
Existen muchas proteínas involucradas en el metabolismo (síntesis y degradación), el transporte y la acción de la dopamina. El precursor de la dopamina es el aminoácido tirosina, que es convertido en dihidroxifenilalanina (L-DOPA) por acción de la tirosina hidroxilasa. Luego, la L-DOPA es descarboxilada, convirtiéndose en dopamina. Se han identificado varias enzimas involucradas en su vía sintética y en su vía degradativa, así como cinco receptores membranales, una proteína recaptadora y proteínas de vesículas sinápticas. Los polimorfismos en genes que codifican para estas proteínas podrían estar involucrados en la susceptibilidad al alcoholismo (32).
El gen que codifica DRD2 se localiza en el brazo largo del cromosoma 11, específicamente en la región q22-q23, consta de 8 exones y 7 intrones. Se han identificado tres polimorfismos de éste:
Polimorfismo DRD2 TaqIA: con dos alelos 1 y 2, y cambio en la región 3' dentro del exón 8. Actualmente, dicho polimorfismo se ubica en el gen vecino ANKK1.
Polimorfismo DRD2 TaqIB: con dos alelos 1 y 2, y cambio en el extremo 5' junto al exón 2.
Polimorfismo DRD2-141C INS/Del: presencia o ausencia de citosina en la posición -141 en la región promotora del gen.
Existen dos subfamilias de receptores de dopamina: la subfamilia D1, que incluye los receptores D1 y D5, y la subfamilia D2, que incluye los receptores D2, D3 y D4. Todos son proteínas de membrana acopladas a proteínas G, con un extremo amino extracelular, 7 dominios transmembranales y un extremo carboxilo intracelular. Existen diferencias en las asas que se forman entre segmentos transmembranales que originan variantes proteicas estructural y funcionalmente distintas. De los receptores para dopamina, todos tienen localización postsináptica, excepto el DRD2, que también es autorreceptor (ubicación presináptica). Éste es el que más se ha relacionado con el alcoholismo, específicamente el polimorfismo Taq1A1, que presenta una disminución postsináptica de la función y disponibilidad. Esta disminución de dopamina en el sistema de recompensa puede causar disforia, depresión o anhedonia, y como consecuencia, se podría consumir alcohol para mejorar el estado de ánimo negativo (30). También se ha reportado la asociación de polimorfismos en la región 5' UTR del gen DRD1, en casos con severidad de alcoholismo. Asimismo, en el gen DRD4 se encuentra un polimorfismo VNTR (Variable Number of Tandem Repeats) de 48 pares base en el exón 3 cuya repetición de 7 da una reducida respuesta a la dopamina. Este polimorfismo parece más relacionado con la búsqueda compulsiva de la bebida alcohólica y el establecimiento de relaciones sociales (33).
El transportador de dopamina DAT1 (SLC6A3) es el encargado de recaptar la dopamina en ambas direcciones en el punto de sinapsis, y es normal que lo haga hacia la terminal nerviosa. Los DAT son proteínas sodio-cloro dependientes que poseen 12 dominios transmembranales. El gen que codifica DAT1 está localizado en el cromosoma 5q15.3, se compone de 15 exones y 14 intrones, y posee formas polimórficas en su región 3' UTR junto a la cola de poli-A. Este gen es un minisatélite —o VNTR— de 40 bases que se repiten de 3 a 13 veces, siendo las de 9 y 10 las más frecuentes. Algunos polimorfismos de 9 repeticiones (A9) se han asociado con problemas de abstinencia en la adicción al alcohol. Se ha demostrado que portadores del alelo A9 tienen una reducida densidad de la proteína DAT1 en comparación con otros genotipos, hecho causado probablemente por un descenso en su expresión (34).
La enzima catecol-O-metiltrasferasa (COMT) participa en la vía metabólica de degradación de la dopamina transformándola en 3-metoxitiramina, la cual luego se convierte en ácido homovanílico, el metabolito inactivo más importante de esta vía. El gen se ubica en 22q11.1-q11.2. La COMT presenta variables que se diferencian en el residuo de aminoácido de la posición 158, que puede ser metionina o valina. Entre los alcohólicos se ha hallado una alta frecuencia de individuos met/met, que tienen una menor respuesta a la β-endorfina y son más sensibles al estrés, lo que incrementa su ansiedad y, por tanto, su riesgo de alcoholismo (35).
El sistema de serotonina (5-hidroxitriptamina o 5-HT) podría participar en respuesta al consumo excesivo de alcohol, ya que modula la excitación glutaminérgica e inhibe la gabaérgica. Se han reportado polimorfismos en el gen del transportador de serotonina (5-HTT), que consisten en una deleción/inserción de 44 pares de bases en su región promotora. Esto se traduce en dos variantes con diferente actividad transcripcional: la corta (S) y la larga (L). Los alelos largos de 5-HTT (5-HTTLPR) con baja actividad transcripcional estarían asociados a la tolerancia y dependencia al alcohol (36). De igual forma, se han reportado polimorfismos en el receptor de serotonina, siendo la isoforma HTR3B la que representa riesgo para alcohol y drogodependencia (14).
El aminoácido ácido γ-amino-butírico (GABA) es el principal neurotransmisor inhibitorio que actúa sobre dos receptores conocidos como A y B. En el cromosoma 4 se encuentran los genes GABRA2, GABRA4, GABRB1 y GABG2, en el cromosoma 5 los genes GABRA1, GABRA6, GABRB2 y GABRG2 y en el cromosoma 15 los genes GABRA5, GABRB3 y GABRG3. El alcohol actúa sobre los receptores tipo A conocidos como GABRA. El receptor de GABA Aα6 presenta dos variantes en la posición 385 pro/ser, posiblemente asociadas con problemas de consumo de alcohol. El consumo de alcohol podría aliviar el estado de hiperexcitabilidad del organismo normalizándolo, pero su efecto es temporal, lo que lleva a su uso frecuente y creciente, poniendo al individuo en peligro de caer en dependencia. Según los trabajos de genómica total, el gen candidato asociado con más frecuencia al consumo de alcohol es el GABRA2 (37,38).
De igual forma, existen estudios que asocian a los opioides y a sus receptores (µ y δ) con sensibilidad inicial y efectos reforzadores del consumo y abuso de alcohol. Numerosas líneas de evidencia indican que los mecanismos de refuerzo del etanol implican, al menos parcialmente, la activación inducida por el psicoactivo del sistema opioide endógeno. El etanol puede alterar la transmisión opioidérgica a diferentes niveles, incluyendo la biosíntesis, la liberación y la degradación de los péptidos opioides, así como la unión de los ligandos endógenos a los receptores opioides. Estos estudios señalan a las encefalinas y beta-endorfinas como mediadores fisiológicos de las acciones de etanol en el cerebro. La importancia de los receptores opioides en el alcoholismo humano se pone de manifiesto por la asociación de los polimorfismos de los receptores µ y δ, que podrían estar involucrados en la liberación de dopamina en el núcleo accumbens de los alcohólicos. Ha sido reportado un polimorfismo en el residuo aminoacídico 40 del receptor µ con cambio de asparagina por aspartato, activando tres veces más este receptor por acción de la β-endorfina (39).
El glutamato se encarga de modular la actividad y liberación dopaminérgicas en el núcleo accumbens. El bloqueo de los receptores de glutamato-NMDA aumenta la liberación de dopamina dependiente del estímulo del estriado. Mientras que la dopamina es determinante para la recompensa aguda y el inicio de la adicción, la adicción en la fase final se debe principalmente a las adaptaciones celulares en la corteza cingulada anterior y a las proyecciones glutamatérgicas orbitofrontales hacia el núcleo accumbens. La plasticidad en la transmisión excitatoria reduce la capacidad de la corteza prefrontal para iniciar conductas en respuesta a recompensas y, por consiguiente, para proporcionar un control ejecutivo sobre la búsqueda de drogas (40).
Expresión génica en el alcoholismo
Algunos ensayos con alcohol en animales de laboratorio muestran cambios en la expresión y función de ciertos genes y proteínas. Asimismo, se han obtenido resultados similares en humanos (41,42). El fenotipo de adicción puede persistir durante toda la vida de un individuo e incluso generar recaídas después de muchos años de abstinencia. Esto sugiere que las sustancias psicoactivas causan alteraciones en el cerebro que perduran por largos períodos. Estos cambios, correspondientes a estados de adaptación que inducen los psicoactivos, incluyen activación o inhibición de factores de transcripción, modificación de la cromatina y estructura del ADN, e inducción de ARN no codificante (43).
Aunque las diferentes drogas recreativas difieren en su mecanismo de acción, virtualmente todas las sustancias psicoactivas incrementan la actividad dopaminérgica desde VTA a NAc y a otras regiones límbicas del cerebro. Entre los principales factores de transcripción afectados por el consumo de psicoactivos tenemos:
ΔFosB: factor de transcripción inducible por efecto de sustancias psicoactivas más estable de todos. Es indispensable y suficiente para generar los cambios en los circuitos de recompensa por exposición crónica a las drogas.
CREB: (cAMP response element-binding) factor de transcripción inducible por efecto de sustancias psicoactivas.
NF-κB: factor de transcripción que se encuentra en casi la totalidad de células y que se encarga de dar respuesta a los estímulos de estrés. En el sistema nervioso, está involucrado en plasticidad sináptica y memoria.
MEF2: factor de transcripción involucrado en diferenciación de células musculares. También se expresa en cerebro, se ve afectado por la acción del etanol y regula enzimas tipo HDACs (histonas deacetilasas).
La exposición aguda al alcohol estimula la liberación de dopamina en el NAc, que activa los receptores de dopamina, y estimula la señalización de la proteína quinasa A (PKA) y la posterior expresión génica mediada por CREB. Por otro lado, la exposición crónica al alcohol conduce a la regulación de la baja de adaptación de esta vía, en particular de la función de CREB. La función de CREB disminuido en el NAc puede promover el consumo de drogas de abuso para lograr un aumento en la recompensa y, por lo tanto, puede estar involucrado en la regulación de los estados afectivos positivos de la adicción. La señalización de PKA también afecta la actividad del receptor NMDA y puede desempeñar un papel importante en la neuroadaptación como respuesta a la exposición crónica al alcohol (43,44).
Los estudios con genes candidatos, particularmente los ensayos con SNPs en humanos, no son suficientes para explicar los mecanismos tan complejos del efecto neurotóxico del alcohol y los diversos fenotipos del alcoholismo. A pesar de la complejidad del panorama, los estudios de genómica total —de expresión génica (transcriptómica y proteómica)— nos muestran algunas redes íntimamente ligadas con los cambios funcionales que se presentan en tejidos y momentos específicos. Mediante técnicas de microarreglos y neuroproteómica, se ha analizado la expresión génica de distintos tejidos humanos de individuos con adicción a distintos psicoactivos, incluido el alcohol. Así, se ha encontrado una alteración significativa en genes de distinta localización y función celular. Además de los ya mencionados de metabolismo y sistema de recompensa, se han descrito genes de transducción de señales (CREB1, MAPK1, NTRK2, SOD2), genes de neuroinflamación, genes de la vía de señalización de calmodulina, genes de tránsito del retículo endoplasmático y aparato de Golgi, genes estructurales del citoesqueleto y adhesión celular, receptores nucleares, y moléculas que regulan la plasticidad neuronal, la memoria y la arquitectura dendrítica (14,42,45,46,47,48,49,50,51).
Aspectos epigenéticos del alcoholismo
La concordancia incompleta entre gemelos monocigóticos como respuesta al alcohol sugiere que los factores ambientales y epigenéticos son importantes en la susceptibilidad a su abuso. Además, los resultados tan distintos y contradictorios en estudios de polimorfismos genéticos asociados al alcoholismo nos hacen pensar que quizás, además de la heterogeneidad alélica y génica propia de las enfermedades de herencia multifactorial, las diferencias se deban a alteraciones en un nivel diferente como su expresión (transcripción y traducción) o su regulación (52,53).
Entre los mecanismos epigenéticos, se puede citar la metilación del ADN, la ADP-ribosilación, la sumoilación, la acetilación, la metilación, la fosforilación, deaminación, isomerización, ubiquitinación de las histonas, los ARN no codificantes, entre otros. Casi el 50% de los estudios epigenéticos hacen referencia a la metilación del ADN por ser un cambio más estable, mientras que la obtención de muestras adecuadas para los otros tipos de estudios epigenéticos es más compleja (54,55).
La modificación de histonas es un complejo código de acceso a genes individuales por parte de maquinaria transcripcional. La acetilación de histonas por las acetiltransferasas (HTAs) está asociada a la activación de genes, mientras la metilación por histonas metiltransferasas (HMTs) está asociada a activación y represión. Ambos procesos pueden ser reversibles por acción de las histonas deacetilasas (HDACs) y de las histonas demetilasas (HDMs), respectivamente (43).
El genoma de mamíferos cuenta con alrededor de un 75% de dinucleótidos CpG metilados, que se encuentran principalmente en elementos repetitivos y de regulación génica. Además, alrededor de la mitad de los genes están asociados a estas denominadas islas CpG y su estado de metilación depende de factores externos como dieta, estilo de vida y condiciones específicas clínicas (56).
La regulación epigenética es fundamental en el alcoholismo, como lo muestran las evidencias que se enumeran a continuación:
- En los últimos años, se han desarrollado estudios caso/control que muestran la alteración en estados de metilación en islas CpG de numerosos genes de pacientes alcoholdependientes. Por ejemplo, los genes de la α-synucleina (SNCA), la monoaminoxidasa A (MAO A), el transportador de serotonina (SLC6A4), la subunidad 2B del receptor de N-metil-D-aspartato, la proopiomelacortina y la prodinorfina muestran diferencias de metilación entre controles y pacientes con dependencia al alcohol (53).
- Los medicamentos zebularine y 5-aza-2-deoxicitidina bloquean las DNA metiltransferasas (DNMT). Su inhibición altera la metilación del promotor de BDNF y reelina implicados en la plasticidad en el hipocampo como respuesta al alcohol (57).
- Las drogas de abuso tienen sus efectos porque producen modificaciones en la remodelación de la cromatina; por ejemplo, la disminución en la acetilación de la histona H3 y el incremento en la expresión mRNA de HDAC2 (58).
- Durante el consumo excesivo de alcohol la relación NADH/NAD+, la concentración de ROS (especies reactivas de oxígeno) y la concentración de acetato pueden verse alteradas y afectar enzimas de acetilación dependientes de NAD+ —por ejemplo HDAC—, que controlan la expresión génica de diversas vías, incluso las de muerte celular programada (59).
- La capacidad del alcohol para alterar los patrones de metilación del ADN genera hipometilación global, causada por la reducción de los niveles de adenosilmetionina (SAM). Esto concuerda con lo que se observa en otras patologías como el cáncer (51).
- Enzimas como las DNA metiltransferasas (DNMTs), las histona acetiltransferasas (HATs), histona metil transferasas( HMTs) e histona demetilasas (HDMs) son reguladas por metabolitos (cofactores) como el NAD+, ATP, acetilCoA y S-adenosilmetionina (SAM). Éstos actúan modificando las histonas, que, a su vez, actúan finalmente como sensores metabólicos (59).
- Estudios en humanos y animales de laboratorio han permitido demostrar que tanto el consumo crónico como agudo de alcohol afecta muchos aspectos del ciclo circadiano como las funciones fisiológicas, endocrinas y de comportamiento (60).
- Los cambios epigenéticos explican la progresión de la enfermedad mejor que las mutaciones. Además, estos cambios son dinámicos y reversibles y, por tanto, potentes candidatos para intervenciones terapéuticas (61,62).
Tratamiento personalizado del alcoholismo
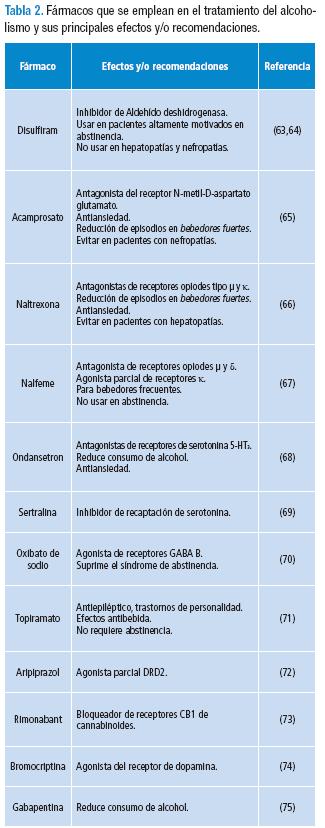
Teniendo en cuenta todo lo expuesto anteriormente, es claro entender porque no existe un medicamento ideal para todo tipo de paciente con problemas de abuso de alcohol. Además, debido a la importancia del factor étnico, los estudios de genética molecular del alcoholismo son esenciales para profundizar en el conocimiento sobre la prevención (consejería genética), el diagnóstico (susceptibilidad) y el tratamiento (farmacogenómica) en una población específica. La farmacogenómica —como el área que estudia la variabilidad genética en cualquier nivel como respuesta a tratamientos farmacológicos— puede ser empleada en el tratamiento personalizado del alcoholismo, única alternativa viable, según parece. Son bien conocidas las deficiencias en los tratamientos farmacológicos de las dependencias en general. Son relativamente más exitosos los tratamientos de tipo conductual, lo que puede explicarse por deficiencias en la investigación básica y aplicada. A pesar de esto, existen algunos tratamientos farmacológicos aceptados por las comunidades médicas mundiales, que se resumen en la Tabla 2.
Los últimos trabajos científicos relacionados con el tema se han enfocado en el receptor opiode tipo µ y el transportador de serotonina. Existen tres tipos de receptores de opiodes µ, κ y δ, que producen efectos biológicos. El exón 1 del gen del receptor opiode µ (OPRM1) posee variantes que hacen que la respuesta al alcohol y los tratamientos cambien de manera modesta. Existe un polimorfismo A118G que codifica una sustitución Asn40Asp en el dominio extracelular del receptor y hace que cambie su funcionalidad con pérdida o ganancia, según estudios controversiales (76).
La frecuencia del polimorfismo varía según la población que se estudie. Los poseedores del polimorfismo Asp40 experimentan, en el momento de administración de alcohol, una mayor intoxicación, estimulación, sedación y placer que los homocigotos poseedores de la forma Asn40. Entre los bebedores fuertes, los poseedores de la variante Asp40 experimentan mayor ansiedad que quienes no la poseen. Los pacientes con al menos un alelo que codifique para Asp40, bajo tratamiento con naltrexona, tuvieron menos recaídas que los otros (76).
Las investigaciones también se han centrado en el gen SLC6A4, que codifica el transportador de serotonina (5-hidroxitriptamina o 5-HTT). Se ha hallado un polimorfismo en la región del promotor de 5-HTT: una inserción de repeticiones de 44 nucleótidos de longitud que genera dos posibles alelos —uno largo (L) y el otro corto (S)— que están asociados a una gran y baja actividad transcripcional, respectivamente. También se presenta un SNP en la versión larga: un cambio de A/G conocido como el alelo LG, cuya actividad es similar a la del alelo S. Pacientes tratados con ondansetron y sertralina, con los alelos LL y SNP TT en la región 3' UTR, consumían menos alcohol por día y tenían días de abstinencia mayores que los demás genotipos (77).
Finalmente, se han reportado algunos análisis bioinformáticos asociados a estudios con humanos y ensayos de expresión en ratones que revelan modulación edad-específica del consumo de alcohol por vía neuroinmune con minociclina (78). Este trabajo generó gran expectativa, pues sus resultados muestran que el análisis bioinformático de la expresión génica puede generar nuevas hipótesis capaces de ser trasladadas y reflejadas en un tratamiento individualizado para el elevado consumo de alcohol.
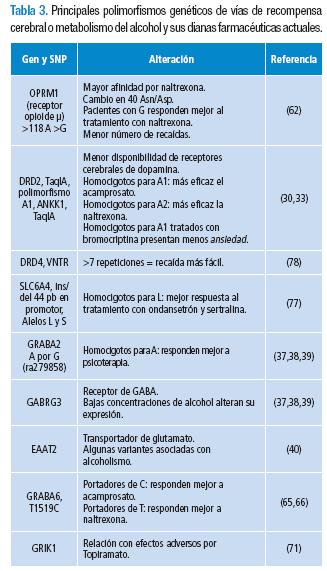
En la Tabla 3 se resumen los principales polimorfismos genéticos blanco de la personalización de los tratamientos farmacológicos.
Conclusiones
El alcoholismo en humanos es una enfermedad con alto grado de heterogeneidad, con componentes biológicos y psicosociales que sólo pueden abordarse plenamente de manera multidisciplinar. Desde la genética humana, se deben seguir identificando los genes en los que la variación afecta el riesgo, para realizar un diagnóstico temprano y encontrar las claves del tratamiento farmacológico. Los estudios en animales también son muy importantes, ya que permiten el control de genes y entorno que no son posibles en los estudios humanos. Además, estos estudios genómicos —transcriptómicos y epigenómicos—, apoyados en la bioinformática, deberán seguir generando aportes sobre los mecanismos particulares de la enfermedad.
Conflicto de intereses
Ninguno declarado por el autor.
Financiación
Ninguna declarada por el autor.
Agradecimientos
A todas aquellas personas que aportaron con sus comentarios a mejorar este escrito.
Referencias
1. Organización Mundial de la Salud. Glosario de términos de alcohol y drogas. Madrid: Ministerio de Sanidad y Consumo, Centro de Publicaciones; 1994. [ Links ]
2. Babor TF, Higgins-Biddle JC, Saunders JB, Monteiro MG. AUDI: Cuestionario de Identificación de los Trastornos debidos al Consumo de Alcohol. Ginebra: Organización Mundial de la Salud, Departamento de Salud Mental y Dependencia de Sustancias; 2001. [ Links ]
3. Organización Panamericana de la Salud. Clasificación Estadística Internacional de Enfermedades y Problemas Relacionados con la Salud. 10ª revisión. Washington, D.C.: OPS, Oficina Sanitaria Panamericana; 1995. [ Links ]
4. Herran OF, Ardila MF, Barba DM. Consumo problemático de alcohol en Bucaramanga, Colombia: diseño y validación de una prueba. Biomédica. 2008 [cited 2015 Jun 29];28(1):25-37. Spanish. http://doi.org/5s9. [ Links ]
5. Organización Mundial de la Salud. Estadísticas sanitarias mundiales. Francia; 2011. [ Links ]
6. Tellez MJ, Cote MM. Alcohol etílico: un tóxico de alto riesgo para la salud humana socialmente aceptado. Rev. Fac. Med. 2006 [cited 2015 Jun 29];54(1):32-47. Spanish. Available from: http://goo.gl/7L9DCT. [ Links ]
7. Colombia. Ministerio de Justicia y Derecho, Observatorio de Drogas de Colombia, Ministerio de Salud y Protección Social, Ministerio de Educación. Estudio nacional del consumo de sustancias psicoactivas en población escolar Colombia. Bogotá; 2011. [ Links ]
8. Colombia. Ministerio de Justicia y Derecho, Observatorio de Drogas de Colombia, Ministerio de Salud y Protección Social. Estudio nacional del consumo de sustancias psicoactivas en Colombia 2013. Bogotá: ALVI Impresores S.A.S; 2014. [ Links ]
9. Programa Anti-Drogas Ilícitas de la Comunidad Andina (PRADICAN). Estudio epidemiológico Andino sobre el consumo de drogas sintéticas en la población universitaria. Lima; 2009. [ Links ]
10. Programa Anti-Drogas Ilícitas de la Comunidad Andina (PRADICAN). II Estudio epidemiológico Andino sobre consumo de drogas en la población universitaria; Informe regional. Lima; 2012. [ Links ]
11. Rehm J, Monteiro M. Alcohol consumption and burden of disease in the Americas-implications for alcohol policy. Rev Panam Salud Publica. 2005 [cited 2015 Jun 29];18(4-5):241-8. http://doi.org/ccx9n4. [ Links ]
12. Morozova TV, Mackay TF, Anholt RR. Genetics and genomics of alcohol sensitivity. Mol Genet Genomics. 2014 [cited 2015 Jun 29];289(3):253-69. http://doi.org/5tb. [ Links ]
13. Secades R, Fernández JR. Modelos de adicción. In: García E, Mendieta S, Cervera G, Fernández JR, editores. Manual SET de Alcoholismo. Madrid: Editorial Médica Panamericana; 2003. p. 19-47. [ Links ]
14. Monteiro MG, Maristela G. Alcohol y salud pública en las Américas: un caso para la acción. Washington, D.C.: OPS; 2007. [ Links ]
15. Salcedo MA. Aspectos genéticos relacionados con el alcoholismo. Típica, Boletín Electrónico de Salud Escolar. 2007 [cited 2015 Jun 29];3(2):1-8. Spanish. Available from: http://goo.gl/tuRJ2K. [ Links ]
16. Enoch MA. Genetic influences on the development of alcoholism. Curr Psychiatry Rep. 2013 [cited 2015 Jun 29];15(11):412. http://doi.org/5tc. [ Links ]
17. Sáiz PA, G-Portilla MP, Paredes MB, Bascaran MT, Bobes J. Instrumentos de evaluación en alcoholismo. Adicciones. 2002 [cited 2015 Jun 29];14 Supp 1:387-403. Spanish. Available from: http://goo.gl/E3hLMR. [ Links ]
18. World Health Organization (WHO). Global status report on alcohol and health. Library Cataloguing-in-Publication Data. Italia; 2011. [ Links ]
19. Cannon TD, Gasperoni TL, Van-Erp TG, Rosso IM. Quantitative neural indicators of liability to schizophrenia: implication for molecular genetics studies. Am J Med Genet. 2001 [cited 2015 Jun 29];105(1):16-9. http://doi.org/cdgqk4. [ Links ]
20. Chloe CY, Wong JM, Fernandes C. Drugs and addiction: an introduction to epigenetics. Addiction. 2011 [cited 2015 Jun 29];106(3):480-9. http://doi.org/b3qwcm. [ Links ]
21. Moss HB, Chiung MC, Hsiao Y. Subtypes of alcohol dependence in nationally representative sample. Drug Alcohol Depend. 2007 [cited 2015 Jun 29];91(2-3):149-58. http://doi.org/dgp98j. [ Links ]
22. Moss HB, Kirisci L, Gordon HW, Tarter RE. A neuropsychologic profile of adolescent alcoholics. Alcohol Clin Exp Res. 1994 [cited 2015 Jun 29];18(1):159-63. http://doi.org/fsxpvs. [ Links ]
23. Kreek MJ, Nielsen DA, Laforge KS. Genes associated with addiction: alcoholism, opiate, and cocaine addiction. Neuromolecular Medicine. 2004 [cited 2015 Jun 29];5(1):85-108. http://doi.org/frpmmz. [ Links ]
24. Daudén E. Farmacogenética II. Métodos moleculares de estudio, bioinformática y aspectos éticos. Actas Dermosifiliogr. 2007 [cited 2015 Jun 29];98(1):3-13. Spanish. http://doi.org/djv5sf. [ Links ]
25. Higuchi S, Matsushita S, Masaki T, Yokoyama A, Kimura M, Suzuki G, et al. Influence of genetic variations of ethanol-metabolizing enzymes on phenotypes of alcohol-related disorders. Ann N Y Acad Sci. 2004 [cited 2015 Jun 29];1025(1):472-80. http://doi.org/d36sfz. [ Links ]
26. López M, Mayorquin P, Vega M. Aplicación de los microarrays y biochips en salud humana. Madrid: Fundación Española para el Desarrollo de la Investigación en Genómica y Proteomica; 2005. [ Links ]
27. Méndez C. Tipificación de polimorfismos de los genes alcohol deshidrogenasa, aldehído deshidrogenasa y citocromo p4502E1 en una población de la ciudad de Bogotá y la evaluación de su posible asociación con el consumo problemático de alcohol [Tesis]. Bogotá: Universidad Nacional de Colombia; 2009. [ Links ]
28. Castro TX. Determinación de los polimorfismos genéticos TaqIA (ANKK1), TaqIB (DRD2), -141c ins/del (DRD2) y VNTR 40pb (DAT1) en población colombiana y evaluación de su asociación con el alcoholismo [Trabajo de Grado de Maestría]. Bogotá: Universidad Nacional de Colombia; 2013. [ Links ]
29. Hodgkinson CA, Yuan Q, Xu K, Shen PH, Heinz E, Lobos EA, et al.Addictions biology: haplotype-based analysis for 130 candidate genes on a single array. Alcohol Alcohol. 2008 [cited 2015 Jun 29];43(5):505-15. http://doi.org/bqn4dn. [ Links ]
30. Heinz A. Dopaminergic dysfunction in alcoholism and schizophrenia-psycopathological and behavioral correlates. Eur Psychiatry. 2002 [cited 2015 Jun 29];17(1):9-16. http://doi.org/b5nkwx. [ Links ]
31. Volkov ND, Fowler JS, Wang GJ. The addicted human brain viewed in the light of imaging studies: brain circuits and treatment strategies. Neuropharmacology. 2004 [cited 2015 Jun 29];47 Suppl 1:3-13. http://doi.org/dttmvm. [ Links ]
32. Flatscher BT, Harrison E, Matsumoto I, Wilce PA. Genes associated with alcohol abuse and tobacco smoking in the human nucleus accumbens and ventral tegmental area. Alcohol Clin Exp Res. 2010 [cited 2015 Jun 29];34(7):1291-302. http://doi.org/b2fxn5. [ Links ]
33. Kimura M, Higuchi S. Genetics of alcohol dependence. Psychiatry and Clinical Neurosciences. 2011 [cited 2015 Jun 29];65(3):213-25. http://doi.org/ffzbcn. [ Links ]
34. Köhnke MD, Batra A, Kolb W, Köhnke AM, Lutz U, Schick S, et al. Association of the dopamine transporter gene with alcoholism. Alcohol Alcohol. 2005 [cited 2015 Jun 29];40(5):339-42. http://doi.org/fmdq6v. [ Links ]
35. Enoch MA, Xu K, Ferro E, Harris CR, Goldman D. Genetic origins of anxiety in women: a role for a functional catechol-O-methyltransferase polymorphism. Psychiatr Genet. 2003 [cited 2015 Jun 29];13(1):33-41. http://doi.org/ctvgdg. [ Links ]
36. Choi IG, Kee BS, Son HG, Ham BJ, Yang BH, Kim SH, et al. Genetic polymorphisms of alcohol and aldehyde dehydrogenase, dopamine and serotonin transporters in familial and non-familial alcoholism. Eur Neuropsychopharmacol. 2006 [cited 2015 Jun 29];16(2):123-8. http://doi.org/dsrfg7. [ Links ]
37. Izquierdo M. Intoxicación alcohólica aguda. Adicciones. 2002 [cited 2015 Jun 29];14 Supp 1:175-93. Available from: http://goo.gl/08Ai1q. [ Links ]
38. Xuei X, Dick D, Flury-Wetherill L, Tian HJ, Agrawal RG, Bierut LJ, et al. Association of the kappa-opioid system with alcohol dependence. Mol Psychiatry. 2006 [cited 2015 Jun 29];11(11):1016-24. http://doi.org/b26kdx. [ Links ]
39. Méndez M, Morales M. Role of mu and delta opioid receptors in alcohol drinking behaviour. Curr Drug Abuse Rev. 2008 [cited 2015 Jun 29];1(2):239-52. http://doi.org/fpgs3h. [ Links ]
40. Kalivas PW, Volkow ND. The neural basis of addition: A pathological of motivation and choice. Am J Psychiatry. 2005 [cited 2015 Jun 29];162(8):1403-13. http://doi.org/cb3rdc. [ Links ]
41. Singh SM, Treadwell J, Kleiber ML, Harrison M, Uddin RK. Analysis of behavior using genetical genomics in mice as a model: from alcohol preferences to gene expression differences. Genome. 2007 [cited 2015 Jun 29];50(10):877-97. http://doi.org/ccvdgf. [ Links ]
42. Tabakoff B, Saba L, Printz M, Flodman P, Hodgkinson C, Goldman D, et al. Genetical genomic determinants of alcohol consumption in rats and Humans. BMC Biol. 2009 [cited 2015 Jun 29];7(1):70. http://doi.org/djc8z6. [ Links ]
43. Robison AJ, Nestler EJ. Transcriptional and epigenetic mechanism of addition. Nat Rev Neurosci. 2011 [cited 2015 Jun 29];12(11):623-37. http://doi.org/fbncv5. [ Links ]
44. Fei SS. Integrating genetics and protemics to study alcohol-drinking behavior [dissertation]. School of Medicine, Oregon Health & Science University: Student Scholar Archive; 2011. Paper 588. [ Links ]
45. Gelernter J, Kranzler HR, Sherva R, Almasy L, Koesterer R, Smith AH, et al. Genome-wide association study of alcohol dependence: significant findings in African- and European-Americans including novel risk loci. Mol Psychiatry. 2014 [cited 2015 Jun 29];19(1):41-9. http://doi.org/5td. [ Links ]
46. Kelley KW, Dantzer R. Alcoholism and inflammation: Neuroimmunology of behavioral and mood disorders. Brain Behav Immun. 2011 [cited 2015 Jun 29];25 Supp 1:S13-20. http://doi.org/csrv54. [ Links ]
47. Lehrmann E, Colantuoni C, Deep-Soboslay A, Becker KG, Lowe R, Huestis M, et al. Transcriptional changes common to human cocaine, cannabis and phencyclidine abuse. PLoS One. 2006 [cited 2015 Jun 29];1(1):e114. http://doi.org/fpb55r. [ Links ]
48. Levey DF, Le-Niculescu J, Ayalew M, Jain N, Kirlin K, Learman R, et al. Genetic risk prediction and neurobiological understanding of alcoholism. Transl Psychiatry. 2014 [cited 2015 Jun 29];4(5):e391. http://doi.org/5tf. [ Links ]
49. Mayfield RD, Lewohl JM, Dodd PR, Herlihy A, Liu J, Harris RA. Patterns of gene expression are altered in the frontal and motor cortices of human alcoholics. J Neurochem. 2002 [cited 2015 Jun 29];81(4):802-13. http://doi.org/fjn7dx. [ Links ]
50. Sohma H, Hashimoto E, Shirasaka T, Tsunematsu R, Ozawa H, Boissl K, et al. Quantitative reduction of type I adenylyl cyclase in human alcoholics. Biochim Biophys Acta. 1999 [cited 2015 Jun 29];1454(1):11-8. http://doi.org/fskqj7. [ Links ]
51. Zhang R, Miao Q, Wang C, Zhao R, Li W, Haile CN, et al. Genomic-wide DNA metilation analysis in alcohol dependence. Addict Biol. 2013 [cited 2015 Jun 29];18(2):392-403. http://doi.org/5tg. [ Links ]
52. Franklin TB, Mansuy IM. Epigenetic inheritance in mammals: evidence for the impact of adverse environmental effects. Neurobiol Dis. 2010 [cited 2015 Jun 29];39(1):61-5. http://doi.org/d6pt46. [ Links ]
53. Zhang H, Herman AI, Kranzler HR, Anton RF, Zhao H, Zheng W, et al. Array-based profiling of DNA methylation changes associated with alcohol dependence. Alcohol Clin Exp Res. 2013 [cited 2015 Jun 29];37 Supp 1: E108-15. http://doi.org/5th. [ Links ]
54. Girardot M, Feil R, Llères D. Epigenetic deregulation of genomic imprinting in humans: causal mechanisms and clinical implications. Epigenomics. 2013 [cited 2015 Jun 29];5(6):715-28. http://doi.org/5tj. [ Links ]
55. Razvi E. Epigenetic research classes and assay trends. GEN Reports: Market & Tech Analysis. 2013 [cited 2015 Jun 29];33(3):1-10. Available from: http://goo.gl/dcl9vW. [ Links ]
56. Tatarinova T, Owain K. DNA Methylation-from genomics to technology. Rijeka, Croatia: InTech; 2012. [ Links ]
57. Zhou L, Cheng X, Connolly BA, Dickman MJ, Hurd PJ, Hornby DP. Zebularine: a novel DNA methylation inhibitor that forms a covalent complex with DNA methyltransferases. J Mol Biol. 2002 [cited 2015 Jun 29];321(4):591-9. http://doi.org/cs27bj. [ Links ]
58. Shakharka AJ, Zhang H, Tang L, Shi G, Pandey SC. Histone deacetylases (HDAC)-induced histone modifications in the amygdala: a role in rapid tolerance to the anxiolytic effects of ethanol. Alcohol Clin Exp Res. 2012 [cited 2015 Jun 29];36(1):61-71. http://doi.org/dh9rc3. [ Links ]
59. Zakhari S. Alcohol metabolism and epigenetics changes. Alcohol Res. 2013 [cited 2015 Jun 29];35(1):6-16. Available from: http://goo.gl/F9kPSh. [ Links ]
60. Wasielewski JA, Holloway FA. Alcohol's Interactions with Circadian Rhythms. A focus on body temperature. Alcohol Res Health. 2001 [cited 2015 Jun 29]; 25(2):94-100. Available from: http://goo.gl/30sh2a. [ Links ]
61. Andrés G, Ashour N, Sánchez-Chapado M, Ropero S, Angulo JC. The study of DNA methylation in urological cancer: present and future. Actas Urol Esp. 2013 [cited 2015 Jun 29];37(6):368-75. http://doi.org/f2jkfj. [ Links ]
62. Shukla SD, Zakhari S. Epigenetics: new frontier for alcohol research. Alcohol Res. 2013 [cited 2015 Jun 29];35(1):1-2. Available from: http://goo.gl/wqTBaB. [ Links ]
63. Fuller RK, Branchey L, Brightwell DR, Derman RM, Emrick CD, Iber FL, et al. Disulfiram treatment of alcoholism: a Veterans Administration cooperative study. JAMA. 1986 [cited 2015 Jun 29];256(11):1449-55. http://doi.org/bzhvsf. [ Links ]
64. Caputo F, Vignoli T, Grignaschi A, Cibin M, Addolorato G, Bernandi M. Pharmacological management of alcohol dependence: From mono-therapy to pharmacogenetics and beyond. Eur Neuropsychopharmacol. 2014 [cited 2015 Jun 29];24(2):181-91. http://doi.org/5tk. [ Links ]
65. Whitworth AB, Oberbauer H, Fleischhacker WW, Lesch OM, Walter H, Nimmerrichter A, et al. Comparison of acamprosate and placebo in long-term treatment of alcohol dependence. Lancet. 1996 [cited 2015 Jun 29];347(9013): 1438-42. http://doi.org/fp46fd. [ Links ]
66. Kranzler HR, Van-Kirk J. Efficacy of naltrexone and acamprosate for alcoholism treatment: A meta-analysis. Alcohol Clin Exp Res. 2001 [cited 2015 Jun 29];25(9):1335-41. http://doi.org/c59s2h. [ Links ]
67. Selinn J. Implementation of substitution treatment in Finland: Beyond rationalisation and medicalisation. Nordic Studies on Alcohol and Drugs. 2011 [cited 2015 Jun 29];28(1): 29-42. http://doi.org/fgqr4c. [ Links ]
68. Camilleri M. Pharmacogenomics and serotonergic agents: research observations and potential clinical practice implications. Neurogastroenterol Motil. 2007 [cited 2015 Jun 29];19 Supp 2:40-5. http://doi.org/dv8c36. [ Links ]
69. Pettinati HM, Volpicelli JR, Kranzler HR, Luck G, Rukstalis MR, Cnaan A. Sertraline treatment for alcohol dependence: interactive effects of medication and alcoholic subtype. Alcohol Clin Exp Res. 2000 [cited 2015 Jun 29];24(7):1041-9. http://doi.org/cwdwsc. [ Links ]
70. Caputo F, Francini S, Brambilla R, Vigna-Taglianti F, Stoppo M, Del Re A, et al. Sodium oxybate in maintaining alcohol abstinence in alcoholic patients with and without psychiatric comorbidity. Eur Neuropsychopharmacol. 2011 [cited 2015 Jun 29];21(6): 450-6. http://doi.org/fgxhsd. [ Links ]
71. Johnson BA, Ait-Daoud N, Bowden CL, DiClemente CC, Roache JD, Lawson K, et al. Oral topiramate for treatment of alcohol dependence: a randomised controlled trial. Lancet. 2003 [cited 2015 Jun 29];361(9370):1677-85. http://doi.org/dv6cvm. [ Links ]
72. Martinotti G, Di Nicola M, Di Giannantonio M, Janiri L. Aripiprazole in the treatment of patients with alcohol dependence: a double-blind, comparison trial vs. naltrexone. J Psychopharmacol. 2009 [cited 2015 Jun 29];23(2):123-9. http://doi.org/dzk3pq. [ Links ]
73. Colombo G, Orrù A, Lai P, Cabras C, Maccioni P, Rubio M, et al. The cannabinoid CB1 receptor antagonist, rimonabant, as a promising pharmacotherapy for alcohol dependence: preclinical evidence. Mol Neurobiol. 2007 [cited 2015 Jun 29];36(1):102-12. http://doi.org/b7dfbk. [ Links ]
74. Lawford BR, Young RM, Rowell JA, Qualichefski J, Fletcher BH, Syndulko K. Bromocriptine in the treatment of alcoholics with the D2 dopamine receptor A1 allele. Nat Med. 1995 [cited 2015 Jun 29];1(4):337-41. http://doi.org/fb5mz3. [ Links ]
75. Furieri FA, Nakamura-Palacios EM. Gabapentin reduces alcohol consumption and craving: a randomized, double-blind, placebo-controlled trial. J Clin Psychiatry. 2007 [cited 2015 Jun 29];68(11):1691-700. http://doi.org/d8kcv9. [ Links ]
76. Kranzler HR, McKay JR. Personalized treatment of alcohol dependence. Curr Psychiatry Rep. 2012 [cited 2015 Jun 29];14(5):486-93. http://doi.org/5tm. [ Links ]
77. Becerril VE, Moreno AJ, Mendieta CD, González CD, Natera RG, Pavón RL, et al. Estudio preliminar de la expresión del mensaje genético del transportador de serotonina en células mononucleares de sangre periférica en pacientes con dependencia al alcohol con y sin depresión mayor comórbida. Salud Mental. 2011 [cited 2015 Jun 29];34(2):139-47. Spanish. Available from: http://goo.gl/WZT5dk. [ Links ]
78. Agrawal RG, Owen JA, Levin PS, Hewetson A, Berman AE, Franklin SR, et al. Bioinformatics analyses reveal age-specific neuroimmune modulation as a target for treatment of high ethanol drinking. Alcohol Clin Exp Res. 2014 [cited 2015 Jun 29];38(2):428-37. http://doi.org/5tn. [ Links ]
79. Ray L, Bryan A, MacKillop J, McGeary J, Hesterberg K, Hutchison KE. The dopamine D4 receptor (DRD4) gene exon III polymorphism, problematic alcohol use and novelty seeking: direct and mediated genetic effects. Addict Biol. 2009 [cited 2015 Jun 29];14(2):238-44. http://doi.org/fn7wzt. [ Links ]