Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de la Facultad de Medicina
Print version ISSN 0120-0011
rev.fac.med. vol.64 no.1 Bogotá Jan./Mar. 2016
https://doi.org/10.15446/revfacmed.v64n1.53037
DOI: http://dx.doi.org/10.15446/revfacmed.v64n1.53037
REPORTE DE CASO
Enfoque diagnóstico molecular utilizando secuenciación exómica en las distrofias musculares cintura-cadera
Molecular diagnosis approach through the use of whole exome sequencing in limb-girdle muscular dystrophies
Andrés Felipe Ramírez-Botero1, Leidy Johanna Posso-Gómez1, Andrés Castillo2, Carmen Collado3, Victoria Fernández-Pedrosa3, Oscar Rodríguez-Cruz3, Sergio Lois3, María Teresa Gil3, Jairo Alonso Quiñones4, Harry Pachajoa1,4
1 Universidad Icesi - Facultad de Ciencias de la Salud - Centro de Investigaciones en Anomalías Congénitas y Enfermedades Raras CIACER - Santiago de Cali - Colombia.
2 Universidad del Valle - Facultad de Ciencias Naturales y exactas - Departamento de Biología - Santiago de Cali - Colombia.
3 Sistemas genómicos - Valencia - España.
4 Fundación Clínica Valle del Lili - Santiago de Cali - Colombia.
Correspondencia: Harry Pachajoa. Centro de Investigaciones en Anomalías Congénitas y Enfermedades Raras CIACER, Facultad de Ciencias de la Salud, Universidad Icesi. Calle 18 No. 122-135, edificio L, oficina 5025. Teléfono: +57 2 5552334, extensión: 8075. Santiago de Cali. Colombia. Correo electrónico: hmpachajoa@icesi.edu.co
Recibido: 14/09/2015 Aceptado: 04/11/2015
Resumen
Antecedentes. La distrofia muscular cintura-cadera tipo 1B es una enfermedad con herencia autosómica dominante y secundaria a una mutación en el gen LMNA. Esta enfermedad se caracteriza por su afectación a nivel neuromuscular y cardiaco.
Objetivo. Realizar diagnóstico clínico y confirmatorio molecular en una paciente con debilidad muscular proximal y sintomatología cardíaca a través de secuenciación exómica.
Materiales y métodos. Se presenta el caso de una paciente de 57 años de edad con cuadro de debilidad muscular proximal progresiva principalmente en extremidades y posterior afectación cardíaca; adicionalmente, la paciente tiene múltiples familiares con la misma sintomatología. Se realizó estudio de secuenciación exómica con confirmación, por método de Sanger, de la mutación hallada y posteriormente el análisis bioinformático de esta.
Resultados. La detección de la mutación R377L en el gen LMNA por secuenciación exómica con confirmación por Sanger, junto con la sintomatología clínica de la paciente y el análisis bioinformático de la mutación hallada, permitió realizar diagnóstico confirmatorio de distrofia muscular cintura-cadera tipo 1B.
Conclusión. Es difícil realizar un diagnóstico clínico debido a la heterogeneidad genética del fenotipo de distrofias musculares cintura-cadera. La aproximación diagnóstica es compleja y requiere clasificar las distrofias musculares según el patrón de afectación y el patrón de herencia de la enfermedad. Adicionalmente, debido a los múltiples genes que pueden generar clínica semejante a las diferentes distrofias musculares, se recomienda realizar secuenciación exómica solicitando especial énfasis en los genes candidatos de distrofias musculares cintura-cadera.
Palabras clave: Genética; Distrofias musculares; Cardiomiopatía dilatada; Polimorfismo de nucleótido simple; Exoma (DeCS).
Ramírez-Botero AF, Posso-Gómez LJ, Castillo A, Collado C, Fernández V, Rodríguez-Cruz O, et al. Enfoque diagnóstico molecular utilizando secuenciación exómica en las distrofias musculares cintura-cadera. Rev. Fac. Med. 2016;64(1):159-64. Spanish. doi: http://dx.doi.org/10.15446/revfacmed.v64n1.53037.
Summary
Background. Limb-girdle muscular dystrophy type 1B has a dominant autosomal inheritance pattern and is caused by a mutation in the LMNA gene. This disease has a major neuromuscular and cardiac compromise; furthermore, it belongs to the limb-girdle muscular dystrophies.
Objective. To make a clinical and molecular confirmatory diagnosis in a patient with proximal muscular weakness and cardiac symptoms using whole exome sequencing.
Materials and Methods. This is the case of a 57 year old patient with a slowly progressive proximal muscular weakness and cardiac compromise; furthermore, the patient has many relatives with the same clinical history. Whole exome sequencing with Sanger confirmation and bioinformatics analysis was performed on the found mutation.
Results. The detection of mutation R377L in the LMNA gen by whole exome sequencing with Sanger confirmation, the bioinformatic analysis of the mutation and the symptoms exhibited by the patient allowed the confirmatory diagnosis of limb-girdle muscular dystrophy type 1b.
Conclusion. Due to genetic heterogeneity in the phenotype of limb-girdle muscular dystrophies it is difficult to make a clinical diagnosis. The diagnostic approach is complex and requires classification of the muscular dystrophies according to the pattern of muscular weakness and to identify the disease inheritance pattern. Additionally, due to the multiple genes that can generate similar symptoms in the different muscular dystrophies, the authors recommend the use of whole exome sequencing with a special emphasis on the candidate genes for limb-girdle muscular dystrophies.
Keywords: Genetics; Muscular Dystrophies; Cardiomyopathy, Dilated; Polymorphism, Single Nucleotide; Exome (MeSH).
Ramírez-Botero AF, Posso-Gómez LJ, Castillo A, Collado C, Fernández V, Rodríguez-Cruz O, et al. [Molecular diagnosis approach through the use of whole exome sequencing in limb-girdle muscular dystrophies]. Rev. Fac. Med. 2016;64(1):159-64. Spanish. doi: http://dx.doi.org/10.15446/revfacmed.v64n1.53037.
Introducción
Las distrofias musculares están conformadas por un grupo diverso y heterogéneo de enfermedades que afectan al sistema musculoesquelético; debido a esto, es común determinar el patrón de debilidad muscular para clasificarlos en grupos más específicos: cintura-cadera, compromiso distal, humero-fibular, entre otras (1). El término distrofia muscular cintura-cadera (por su nombre en inglés Limb-girdle muscular dystrophy LGMD) fue introducido por primera vez por Stevenson (2) en 1953 y ampliado luego por Walton & Nattrasss (3), quienes hacían referencia a un grupo de distrofias cuya manifestación más temprana era debilidad de los músculos de la cintura escapular y pélvica; desde estas descripciones tempranas de la enfermedad se reconocía la variabilidad clínica en la historia natural de este grupo de enfermedades.
Debido a la heterogeneidad genética, la clasificación fenotípica de las LGMD ha mostrado limitaciones, y en 1995 se propuso una clasificación de acuerdo al patrón de herencia: las de tipo 1 son aquellas con un patrón de herencia autosómico dominante, mientras que las de tipo 2 son aquellas con un patrón de herencia autosómico recesivo (4).
Dentro del grupo de las LGMD tipo 1 se encuentra la distrofia muscular cintura-cadera tipo 1B (LGMD1B; OMIM 159001); esta es una enfermedad lentamente progresiva de herencia autosómica dominante, secundaria a una mutación en el gen LMNA que codifica para la proteína laminina A (5). La LGMD1B se caracteriza por presentar debilidad en los músculos de la cintura escapular y pélvica y posterior afectación cardíaca. Usualmente los primeros síntomas se presentan alrededor de los 20 años con debilidad de cadera, que en ocasiones es tan leve que pasa desapercibida, posteriormente debilidad de hombros y más tardíamente desórdenes de la conducción atrioventricular, bradicardia, cardiomiopatía dilatada y muerte cardíaca súbita. Es importante destacar que cursa rigidez articular leve con preservación de la movilidad en los codos; adicionalmente, la biopsia muscular muestra distrofia leve, la electromiografía muestra cambios de miopatía y puede haber aumento de creatina fosfoquinasa (5-7).
Es importante conocer que el gen LMNA está ubicado en el cromosoma 1q21-22 y está compuesto por 12 exones (5); por medio del splicing alternativo, este codifica las lamininas tipo A y tipo C (5). Las anteriores, junto con las lamininas tipo B —codificadas por el gen LMNB1— y las proteínas asociadas a la envoltura nuclear, forman la lámina nuclear, que es una red subyacente a la membrana nuclear interna. Además, también se encuentran distribuidas en el nucleoplasma donde cumplen funciones por asociación a cromatina, histonas nucleares y varios factores de transcripción, dando así soporte estructural al núcleo y encargándose del mantenimiento de la arquitectura nuclear, migración nuclear y apoptosis. Por último, forman parte en la transcripción, regulación del ciclo celular, desarrollo y diferenciación celular (8-10).
Para este estudio se presenta el caso de una paciente de 57 años, quien desde alrededor de los 20 años inicia un cuadro de debilidad muscular progresiva principalmente en las extremidades, y a quien a los 42 años se le diagnosticó cardiomiopatía dilatada; además, la paciente cuenta con múltiples familiares en primer, segundo, tercer y cuarto grado de consanguinidad con la misma sintomatología (Figura 1).
Se consideró que la paciente presentaba clínica compatible con el grupo de LGMD; sin embargo, debido al compromiso cardíaco también era necesario descartar otras condiciones genéticas como la distrofia muscular Emery-Dreiffus autosómica dominante (EDMD2; OMIM 181350), la cardiomiopatía dilatada tipo 1A (CMD1A; OMIM 115200) y la enfermedad Charcot-Marie-Tooth tipo 2B1 (ECMT2B1; OMIM 605588). Por lo tanto, se realizó una secuenciación del exoma (SE) como método de identificación de la causa genética debido al gran número de genes que podrían generar un fenotipo compatible con la clínica de la paciente, siendo esta una estrategia diagnóstica más costo-efectiva (1,11).
Materiales y métodos
Sujeto
Se describe el caso de una paciente del sur occidente colombiano con historia de distrofia muscular iniciada en la segunda década de la vida y asociada a cardiomiopatía dilatada y valvulopatía mitral. La paciente refiere tener múltiples familiares afectados con la misma sintomatología, entre ellos el padre y dos hermanos, pero sin diagnóstico clínico-molecular
Estudios moleculares
El exoma fue capturado usando el kit de captura de SureSelect All Human Enrichment Target Exon (Agilent's) de 51Mb y luego secuenciado usando la tecnología Illumina Hiseq 2000. En el proceso de secuenciación se obtuvieron lecturas emparejadas de tamaño 101 nucleótidos.
Resultados
Caracterización clínica
Paciente de 57 años de edad con cuadro clínico que inicia alrededor de los 20 años con una leve debilidad muscular a nivel de cintura escapular y pélvica y que ha ido aumentando con el paso del tiempo. A los 42 años consulta al cardiólogo por sensación de disnea, el profesional realiza ecocardiograma hallando cardiomiopatía dilatada y valvulopatía mitral; además, con el tiempo se hace diagnóstico de fibrilación auricular con alto riesgo cardioembólico. A los 50 años se le toma biopsia del músculo cuádriceps izquierdo sin hallazgos concluyentes para distrofia muscular.
Las patologías de base han sido de carácter progresivo y en la actualidad la paciente presenta falla cardíaca en estadio C-NYHA III con último ecocardiograma realizado a los 55 años, el cual revela fracción de eyección del ventrículo izquierdo en 21%, insuficiencia mitral y dilatación severa de aurícula izquierda y moderada del ventrículo izquierdo. A la paciente se le realizó una electromiografía con velocidad de neuroconducción a los 57 años, con la que confirma una miopatía crónica no inflamatoria y síndrome del túnel del carpo moderado bilateral. La creatinina fosfoquinasa se ha encontrado dentro de rangos de normalidad.
Concomitantemente, la paciente presenta osteoporosis diagnosticada por densitometría ósea a sus 54 años. Ese mismo año se le realizó una tomografía computarizada de columna lumbar, que evidenció fractura por compresión de la primera vértebra lumbar, y una tomografía computarizada de cerebro simple con cambios involutivos corticales.
Al examen físico se encuentra una paciente femenina que aparenta edad cronológica, examen mental normal, signos vitales normales, marcha inestable, pérdida de la fuerza muscular en las cuatro extremidades —4-/5—, arreflexia generalizada y pie cavo bilateral.
Hasta el momento, la paciente ha presentado cuatro accidentes cerebrovasculares isquémicos secundarios a eventos cardio-embólicos y un bloqueo atrioventricular de segundo grado. La paciente se encuentra recibiendo tratamiento farmacológico para fibrilación auricular, falla cardíaca y osteoporosis, al tiempo que tiene un cardiodesfibrilador implantable con resincronizador.
Teniendo en cuenta la historia clínica de debilidad muscular a nivel de los grupos musculares de la cintura escapular y pélvica, el posterior compromiso cardíaco, el patrón de herencia autosómico dominante y la miopatía crónica no inflamatoria, se consideró que el diagnóstico más probable es una LGMD. Por lo tanto, se realizó una SE con especial énfasis en genes compatibles para las LGMD (LMNA, SGCA, SGCB, SGCG, SGCD, CAPN3, DYSF, TCAP, TRIM32, FKRP, TTN, POMT1, ANO5, FKTN, POMT2, POMGNT1, PLEC, MYOT, CAV3, DES, DNAJB6) (12).
Secuenciación del exoma
Se realizó secuenciación del exoma detectando variante nucleótida c.1130G>T en el gen LMNA; esta variante produce el cambio del aminoácido arginina por leucina en la posición 377 de la cadena polipeptídica (R377L). El resultado fue confirmado posteriormente mediante secuenciación Sanger; en la Figura 2 se observa un cambio de codón CGC por CTC con un cambio de aminoácido de arginina por leucina en la posición 377.
Análisis bioinformático del SNP R377L
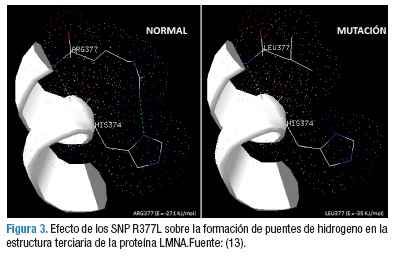
Los análisis del efecto de los polimorfismo de un solo nucleótido (SNP) R377L se realizaron bajo condiciones ideales, con pH=7.0 y a una temperatura de 27oC. Al realizar el análisis, utilizando la estructura cristalina de la proteína humana LMNA 1X8Y, la estabilidad de la proteína decreció 22%, lo que sugiere un aumento en la inestabilidad de la estructura tridimensional debido al cambio del aminoácido. Igualmente, el análisis estructural de la proteína LMNA mostró la pérdida de un enlace de hidrógeno entre el aminoácido leucina 377 con histidina 374 posterior a la mutagénesis in silico (Figura 3) con un cambio de la energía libre a nivel local en el cambio de aminoácido Arginina (R) por Leucina (L) de 271KJ/mol a -35KJ/mol, y a nivel global del cristal la LMNA de -4094KJ/mol a -3855KJ/mol (13). El análisis fue realizado con el programa DeepView-Swiss-PdbViewer y los puentes de hidrogeno se representaron en color verde.
Diagnóstico definitivo
Teniendo en cuenta la clínica de la paciente, el análisis in silico confirma la patogenicidad de la mutación y que la mutación hallada estaba previamente reportada como causante de LGMD1B (14,15); se confirma el diagnóstico molecular de LGMD1B.
Discusión
En Colombia, este es el primer caso de LGMD1B reportado y diagnosticado molecularmente, y también es la primera vez que se reporta la mutación R377L en el gen LMNA en población latinoamericana. En el 2002, Ki et al. (14) reportaron el caso de una mujer coreana de 41 años con contracturas en ambos tobillos, alteraciones en la marcha y debilidad proximal de ambas extremidades, además de taquicardia atrial con bloqueo atrioventricular de tercer grado, cardiomiopatía dilatada y fibrilación auricular. Al secuenciar el gen LMNA se encontró la mutación R377L y se realizó diagnóstico de LGMD1B.
Es importante recalcar que aunque la secuenciación del exoma no es considerada actualmente el examen de primera línea para el diagnóstico de distrofias musculares, recientemente se ha demostrado que su resultado es costo-efectivo: Ghaoui et al. (11) demostraron que es una herramienta útil en pacientes con fenotipo compatible con miopatías y debilidad muscular; en este estudio se le realizó SE en genes candidatos a 60 familias —100 pacientes— con distrofia muscular cadera-cintura y se lograron identificar mutaciones patogénicas en genes de miopatía en 27 familias. De estos, 12 genes estaban relacionados con LGMD y se identificaron 15 variantes en genes patológicos no relacionados con las LGMD. Con la SE, los autores lograron una tasa diagnóstica de 45% para las distrofias, siempre analizando estos estudios junto con el resto del contexto clínico de cada paciente (11).
Los análisis bioinformáticos demostraron que el cambio del aminoácido arginina por leucina genera efectos negativos con relación a la estabilidad de la proteína, generando la pérdida de un enlace de hidrogeno entre los aminoácido 377 (L) y 374 (H). Los resultados corroboran a nivel molecular que el SNP R377L genera efectos negativos a la láminina A, lo que colaboraría a explicar el fenotipo de LGMD1B en la paciente.
En pacientes con clínica semejante a la de la paciente, además de considerar el grupo de las LGMD, es fundamental tener como diagnósticos diferenciales la EDMD2, la CMD1A y la ECMT2B1. Contrario a la LGMD1B, la EDMD2 presenta contractura en los codos y tendón calcáneo, restricción en los movimientos cervicales, cardiomiopatía dilatada con defectos de conducción cardíaca y arritmias cardíacas. Además puede presentar escápula alada, rigidez espinal, atrofia y debilidad de extremidades de predominio superior, aumento de la creatina fosfoquinasa, compromiso muscular en la primera década de la vida y compromiso cardíaco después de los 20 años (5,16-20).
La CMD1A puede presentarse como cardiomiopatía congestiva, defectos en la conducción, fibrilación o flutter auricular, arritmia ventricular o incluso efusión pericárdica y su sintomatología usualmente inicia alrededor de los 50 años; se diferencia de la LGMD1B ya que esta no presenta compromiso osteomuscular (5,16). Por último, los pacientes con ECMT2B1 presentan pie cavo, alteraciones en la marcha y debilidad en extremidades; sin embargo, se diferencia de la LGMD1B en que su patrón de herencia es autosómico recesivo, tiene compromiso de fuerza distal, los síntomas inician entre los 10 y 20 años y presenta ausencia de afectación cardíaca (5,16).
Conclusiones
La secuenciación del exoma es una herramienta útil y costo-efectiva para el diagnóstico molecular de pacientes con clínica de distrofia muscular y especialmente LGMD; así mismo, es fundamental caracterizar el patrón de debilidad muscular, determinar el tipo de herencia y solicitar otros exámenes como la creatinina fosfoquinasa, la biopsia muscular y la electromiografía con velocidad de neuroconducción para poder determinar el tipo de genes y patologías que pueden generar dicho compromiso. No obstante, debido a la heterogeneidad genética de las distrofias musculares hay limitación en los diagnósticos fenotípicos y por eso se debe recurrir a técnicas moleculares para realizar un diagnóstico confirmatorio y definitivo (11); por lo tanto, los autores recomiendan la SE para este tipo de patologías como una herramienta valiosa y además sugieren la implementación del análisis in silico de las mutaciones halladas para predecir su posible patogenicidad.
Conflicto de intereses
Ninguno declarado por los autores.
Financiación
Universidad Icesi.
Agradecimientos
Ninguno declarado por los autores.
Referencias
1. Narayanaswami P. Dismantling Limb-Girdle Muscular Dystrophy: The Role of Whole-Exome Sequencing. JAMA Neurol. 2015;72(12):1409-11. http://doi.org/bbfj. [ Links ]
2. Stevenson AC. Muscular dystrophy in Northern Ireland, I. An account of the condition in fifty-one families. Ann. Eugen. 1953;18(1):50-93. [ Links ]
3. Walton JN, Nattrass FJ. On the classification, natural history and treatment of the myopathies. Brain. 1954;77(2):169-231. http://doi.org/brqhq8. [ Links ]
4. Bushby KM, Beckmann JS. The limb-girdle muscular dystrophies-proposal for a new nomenclature. Neuromuscul. Disord. 1995;5(4):337-43. http://doi.org/cwrmwn. [ Links ]
5. Online Mendelian Inheritance in Man. Muscular Dystrophy, Limb-Girdle, Type 1B; LGMD1B. Baltimore: OMIM; 1993 [updated 2014 Jun 1; cited 2015 julio 13]. Available from: http://goo.gl/ANEaA3. [ Links ]
6. van der Kooi AJ, Ledderhof TM, de Voogt WG, Res CJ, Bouwsma G, Troost D, et al. A newly recognized autosomal dominant limb girdle muscular dystrophy with cardiac involvement. Ann. Neurol. 1996;39(5):636-42. http://doi.org/fhhjwb. [ Links ]
7. van der Kooi AJ, van Meegen M, Ledderhof TM, McNally EM, de Visser M, Bolhuis PA. Genetic localization of a newly recognized autosomal dominant limb-girdle muscular dystrophy with cardiac involvement (LGMD1B) to chromosome 1q11-21. Am. J Hum. Genet. 1997;60(4):891-5. [ Links ]
8. Luo YB, Mastaglia FL, Wilton SD. Normal and aberrant splicing of LMNA. J. Med. Genet. 2014 Apr;51(4):215-23. http://doi.org/bbfk. [ Links ]
9. Bonne G, Quijano-Roy S. Emery-Dreifuss muscular dystrophy, laminopathies, and other nuclear envelopathies. Handb. Clin. Neurol. 2013;113:1367-76. http://doi.org/bbfm. [ Links ]
10. Tesson F, Saj M, Uvaize MM, Nicolas H, Płoski R, Bilińska Z. Lamin A/C mutations in dilated cardiomyopathy. Cardiol. J. 2014;21(4):331-42. http://doi.org/bbfn. [ Links ]
11. Ghaoui R, Cooper ST, Lek M, Jones K, Corbett A, Reddel SW, et al. Use of Whole-Exome Sequencing for Diagnosis of Limb-Girdle Muscular Dystrophy: Outcomes and Lessons Learned. JAMA Neurol. 2015;72(12):1424-32. http://doi.org/bbfp. [ Links ]
12. Pegoraro E, Hoffman EP. Limb-Girdle Muscular Dystrophy Overview. In: Pagon RA, Adam MP, Ardinger HH, Wallace SE, Amemiya A, Bean LJH, et al., editors. GeneReviews®. Seattle: University of Washington; 1993-2015 [updated 2012 Aug 30; cited 2015 Dec 29]. Available from: http://goo.gl/36pH26. [ Links ]
13. Guex N, Peitsch MC. SWISS-MODEL and the Swiss-PdbViewer: an environment for comparative protein modeling. Electrophoresis. 1997;18(15):2714-23. http://doi.org/bhf2w6. [ Links ]
14. Ki CS, Hong JS, Jeong GY, Ahn KJ, Choi KM, Kim DK, et al. Identification of lamin A/C (LMNA) gene mutations in Korean patients with autosomal dominant Emery-Dreifuss muscular dystrophy and limb-girdle muscular dystrophy 1B. J. Hum. Genet. 2002;47(5):225-8. http://doi.org/fdnkv2. [ Links ]
15. Hong JS, Ki CS, Kim JW, Suh YL, Kim JS, Baek KK, et al. Cardiac dysrhythmias, cardiomyopathy and muscular dystrophy in patients with Emery-Dreifuss muscular dystrophy and limb-girdle muscular dystrophy type 1B. J. Korean Med. Sci. 2005;20(2):283-90. http://doi.org/dxmqj4. [ Links ]
16. Vigouroux C, Bonne G. Laminopathies: One Gene, Two Proteins, Five Diseases. In: Madame Curie Bioscience Database. Austin: Landes Bioscience; 2000-2013 [cited 2015 Dec 29]. Available from: http://goo.gl/sx1TNZ. [ Links ]
17. Online Mendelian Inheritance in Man. Emery-Dreifuss Muscular Dystrophy 2, Autosomal Dominant; EDMD2. Baltimore: OMIM; 1986 [updated 2015 Aug 8; cited 2015 dec 29]. Available from: http://goo.gl/YusIsz. [ Links ]
18. Jennekens FG, Busch HF, van Hemel NM, Hoogland RA. Inflammatory myopathy in scapulo-ilio-peroneal atrophy with cardiopathy. A study of two families. Brain. 1975;98(4):709-22. http://doi.org/fdxjj5. [ Links ]
19. Bonne G, Mercuri E, Muchir A, Urtizberea A, Bécane HM, Recan D, et al. Clinical and molecular genetic spectrum of autosomal dominant Emery-Dreifuss muscular dystrophy due to mutations of the lamin A/C gene. Ann Neurol. 2000;48(2):170-80. http://doi.org/bvzbgd. [ Links ]
20. Mercuri E, Brown SC, Nihoyannopoulos P, Poulton J, Kinali M, Richard P, et al. Extreme variability of skeletal and cardiac muscle involvement in patients with mutations in exon 11 of the lamin A/C gene. Muscle Nerve. 2005;31(5):602-9. http://doi.org/fbcm4w. [ Links ]