Introducción
La sepsis, ya sea grave, en choque séptico o síndrome de disfunción multiorgánica, constituye en la actualidad la primera causa de mortalidad en los pacientes ingresados a unidades de cuidado intensivo (UCI), causando más del 60% de las muertes en este servicio 1-4.
En Colombia, la prevalencia de pacientes con sepsis en la UCI es del 12% y la tasa de mortalidad alcanza el 33.6% 4. En EE. UU., la incidencia de sepsis grave posoperatoria se ha triplicado, pasando del 0.3% para el año 2000 al 0.9% en el 2010 2. En Europa, se reporta alrededor de un 29% de mortalidad en pacientes que desarrollaron sepsis y sepsis grave 1,3.
La sepsis no presenta manifestaciones clínicas específicas y su evaluación tiene limitaciones al establecer la severidad y al predecir el pronóstico de la enfermedad de manera efectiva. En la actualidad, los indicadores que se emplean para seguir la clínica de la infección incluyen: fiebre, recuento de leucocitos (WBC), determinación de proteína C reactiva (PCR) y procalcitonina (PCT), entre otros parámetros 5,6.
La búsqueda de nuevos biomarcadores de alta sensibilidad y especificidad que ayuden a mejorar el diagnóstico y pronóstico de sepsis es un campo de estudio de la medicina molecular 7. Una aproximación a este tipo de investigaciones se basa en establecer como biomarcador a la variación en la concentración plasmática de las proteínas que participan en la respuesta inflamatoria durante la enfermedad 7-10. Sin embargo, también se propone el polimorfismo de los genes que codifican por estas proteínas como un biomarcador más confiable 11,12.
En el presente artículo se revisa el estado del arte de los biomarcadores moleculares propuestos hasta el momento como potenciales marcadores para el diagnóstico y pronóstico de la sepsis, sepsis grave y choque séptico.
Materiales y métodos
Se analizaron los registros de los últimos 14 años que se encontraban en PubMed, en The New England Journal of Medicine (NEJM) y en Illinois Automatic Computer (ILLIAC), clasificando los artículos por año de publicación e incluyendo solo artículos de investigación y de revisión que evaluaran o trataran las palabras clave: sepsis, genetic polymorphisms, genetic variation y molecular marker. Solo se tuvieron en cuenta los artículos publicados durante los últimos 10 años.
Resultados
La búsqueda arrojó 3 370 referencias que cubren las generalidades de los biomarcadores de sepsis, las bases teóricas del polimorfismo genético y la publicación de más de 30 genes con polimorfismos considerados marcadores de esta enfermedad. Sin embargo, en la revisión se tuvieron en cuenta 84 artículos que describieron los aspectos epidemiológicos y las bases teóricas de la sepsis; así como los biomarcadores que incluyen el polimorfismo en 17 genes candidatos, considerados como potenciales marcadores moleculares de susceptibilidad para el desarrollo de sepsis, choque séptico y de mortalidad.
Biomarcadores moleculares en sepsis
La variación en los niveles plasmáticos de las moléculas generadas por las células efectoras durante la respuesta inflamatoria a la sepsis incluye: especies reactivas de oxígeno (ROS), metabolitos de nitrógeno (RNS), citocinas, quimiocinas y aumento en la expresión de receptores de superficie. Estos han sido considerados biomarcadores moleculares de gran utilidad para confirmar o descartar la enfermedad, así como para evaluar la evolución del paciente en una terapia específica 7-10.
Entre los mediadores, se destacan las citocinas cuya secreción se detecta desde los primeros momentos de la infección 11,12. Entonces, los altos niveles séricos de las interleucinas IL-1β, IL-4 IL-6, IL-8, la proteína inflamatoria de macrófagos (MCP-1) y la osteopontina (OP) se consideran indicadores de un mal pronóstico de sepsis 13-17. En la Tabla 1 se describen posibles biomarcadores identificados en la búsqueda bibliográfica.
En modelos animales se ha demostrado que la IL-1β es capaz de inducir varios de los síntomas del choque séptico y disfunción multiorgánica 10,13, mientras que las interleucinas IL-2, IL-6, IL-10, IL-12, IL-13, IL-18 y el factor de necrosis tumoral (TNF-α) participan en la patogénesis de la sepsis grave 14-17.
El TNF-β (también llamado linfotoxina alfa) es una citocina que tiene efectos similares a los descritos para el TNF-α, aunque se ha encontrado que el nivel de esta citocina en el suero de pacientes que desarrollaron sepsis es significativamente mayor comparado con el de pacientes que no la desarrollaron durante un proceso infeccioso. Sin embargo, no es clara su función en la patogénesis de la sepsis 10,14.
La IL-1β, OP y MCP-1 se encuentran en altos niveles plasmáticos en los pacientes sépticos 7-9 y el incremento de las IL-2 e IL-4 se relaciona con la severidad y el desarrollo de sepsis 14. El alto nivel plasmático de las IL-6, IL-10, IL-12, IL-13, IL-18, MCP-1 y TNF-a se considera un predictor de desarrollo fatal de la enfermedad 10,11,16,17.
La proteína de alta movilidad B1 (HMGB1) induce la activación de los macrófagos y monocitos y desencadena la liberación de citocinas proinflamatorias y la regulación de moléculas de adhesión. Esta proteína se presenta en altos niveles en pacientes con sepsis 18; sin embargo, tiene una reactividad más lenta que el TNF y la IL-1β, por lo que se desestima como biomarcador para pronosticar la enfermedad 19.
En los estudios realizados en torno a la función de los marcadores celulares de superficie, se destacan las proteínas del clúster de diferenciación (CD). La determinación sérica de las proteínas CD48 y CD69 en alto nivel se relaciona con el desarrollo de la sepsis y los bajos niveles de CD10 y CD11 con un mal pronóstico de la enfermedad (20). Del mismo modo, los altos niveles plasmáticos de CD14, CD18, CD25, CD28, CD40 y CD80 en pacientes con sepsis grave se reportan como un indicador de mal pronóstico de la enfermedad y conllevan casos fatales 21-24.
Otras moléculas de interés que se analizan como biomarcadores de sepsis son los receptores de reconocimiento de patrones (PRR) que reconocen moléculas de patógenos (PAMP) y hacen parte de la inmunidad innata 25. Entre estos, se destaca la familia de receptores similar a Toll (TLR), con al menos 10 proteínas reconocidas en humanos; esta familia se expresa en macrófagos, células dendríticas, neutrófilos y en otras poblaciones celulares y desempeña un papel central en la respuesta inmune innata a la infección mediante el reconocimiento de distintos antígenos bacterianos 26. Por ejemplo, el TLR4 identifica el lipopolisacárido (LPS) de las bacterias Gram negativas, mientras que el TLR2 es esencial en el reconocimiento del péptidoglucano de las bacterias Gram positivas 25,26. El incremento en los niveles séricos de TLR2 y TLR4 se detecta en pacientes con sepsis y se relaciona con el mal pronóstico de la enfermedad 26.
Tabla 1 Posibles marcadores humorales en la sepsis.
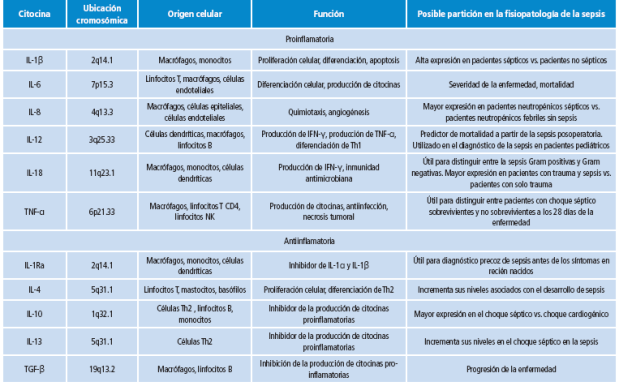
IL: Interleucina; TGF: Factor de crecimiento tumoral.
Fuente: Elaboración propia.
Otro marcador es el receptor soluble de las células mieloides (sTREM-1) que hace parte de la superfamilia de receptores de las inmunoglobulinas. En los estudios de Arts et al.27, se propone como biomarcador predictivo del desarrollo de sepsis grave.
Los altos niveles séricos del receptor soluble de la IL-1 se detectan en pacientes sépticos, mientras que los receptores solubles de la IL-2α y el TNF-β se relacionan con desarrollo de choque séptico y disfunción multiorgánica 13,14.
Las moléculas inflamatorias que actúan sobre el endotelio también han sido estudiadas como biomarcadores. En este caso, el péptido adrenomedulina (ADM), regulado por el sistema del complemento, actúa como un potente vasodilatador con efectos bactericidas y ha sido señalado por algunos estudios como un buen predictor de la gravedad y la evolución de la sepsis, de acuerdo a la variación de sus niveles plasmáticos en pacientes sépticos 28,29. Los estudios también señalan a la molécula de adhesión leucocitaria del endotelio (ELAM-1) y el factor de crecimiento derivado de plaquetas (PGDF) con un desarrollo fatal de la enfermedad 7,8,14.
Las selectinas (proteínas de superficie expresadas por las células endoteliales y que facilitan el rodamiento leucocitario) se han relacionado con el desarrollo de sepsis, en especial la selectina S, la cual se ha propuesto como predictora de disfunción multiorgánica 7,8.
En los últimos años, se han estudiado las moléculas microARN (miARN) como potenciales marcadores de sepsis. Se trata de un tipo de pequeños RNA endógenos no codificantes con alrededor de 22 nucleótidos de longitud que participan en importantes funciones biológicas al inhibir la expresión del ARN mensajero (ARNm) 30. Las miARN están involucradas en el desarrollo y la función específica de los tejidos; además, debido a su patrón de expresión único, son moléculas consideradas como potenciales marcadores en el diagnóstico o como blancos terapéuticos de muchas enfermedades 30,31. La expresión alterada de esta molécula se detecta en la circulación de pacientes con sepsis, por lo que se ha sugerido su utilidad como biomarcador de diagnóstico y pronóstico de la enfermedad 31.
A pesar del importante papel de las miARN en la patogenia de la sepsis, estas representan un valor limitado como biomarcadores ya que su expresión se induce también en otras enfermedades no infecciosas. Entonces, es necesario enfocarse en otros marcadores más específicos. De acuerdo a esta aproximación, los estudios se enfocan en estudiar los polimorfismos genéticos como marcadores moleculares de sepsis.
Polimorfismo genético
El polimorfismo genético corresponde al cambio en la secuencia de un gen y ocurre con una frecuencia superior a 1% en la población. Los polimorfismos pueden producirse en regiones codificantes y no codificantes del genoma 32. Existen dos clases de polimorfismos genéticos: el que se genera por la sustitución de un nucleótido y se origina por la inserción o deleción de uno o más nucleótidos, single nucleotide polymorphism (SNP), y el que se presenta cuando en un grupo un nucleótido se repite en bloques, variable number tandem repeat (VNTR) 30,31.
Una forma de clasificar los SNP se basa en el efecto que causan en la función de la proteína; de esta forma existen los ligados (SNP indicativos), que son variaciones que ocurren en regiones no traducidas a proteínas y que no afectan la función de estas. No obstante, pueden afectar la susceptibilidad de contraer una determinada enfermedad 32. Por ejemplo, los SNP en las regiones promotoras de un gen pueden alterar la afinidad de la unión de factores de transcripción y afectar la concentración de la proteína, mientras que los SNP generados en los extremos terminales del ARNm que no se traducen pueden alterar la estabilidad de estos últimos y, en consecuencia, la síntesis de la proteína 32.
Los SNP causativos son los que afectan de forma directa la función de la proteína, provocan una enfermedad, se presentan en la región codificante de un gen y pueden cambiar la secuencia de aminoácidos del producto proteico con la probabilidad que afecte su estructura y función. Este tipo de SNP es candidato para ser empleado como alelo modificador de la enfermedad 32,33.
En el caso de los VNTR, los que se detectan en la región codificante pueden generar proteínas no funcionales, mientras que, si se encuentran en regiones no codificantes, la alteración puede resultar en fragilidad cromosómica, silenciamiento de genes y modulación en los procesos de transcripción y traducción 32-34.
Se ha planteado que la presencia de algunos de estos polimorfismos genéticos estaría relacionada con la susceptibilidad del individuo al desarrollo de ciertas enfermedades 34-37. Así, por ejemplo, variaciones nucleotídicas en los genes PARK-2 o PACRG se relacionan con la progresión de la lepra 38. Algunas mutaciones específicas en el gen que codifica para el TLR parecen estar relacionadas con el desarrollo de la tuberculosis 39. Las mutaciones en los genes a-globina, P-globina, SLC4A1, mal/tirAP y darC se relacionan con la susceptibilidad a la malaria 40.
Variaciones nucleotídicas específicas también han sido detectadas por las proteínas que participan en la respuesta inflamatoria durante la infección en los genes que codifican. Tales variaciones se relacionan con susceptibilidad al desarrollo de la sepsis y progresión de la enfermedad 12,41,42.
Polimorfismos genéticos como biomarcadores de sepsis
Diversos estudios relacionan el polimorfismo genético de más de 30 genes con el desarrollo de sepsis, sepsis grave y choque séptico. En esta revisión se describen 20 de estos genes que codifican para las citocinas proinflamatorias, receptores celulares, enzimas, mediadores químicos, entre otros 41,42.
Polimorfismos estudiados en citocinas
Las citocinas se consideran mensajeras fisiológicas de la respuesta inmune, son activas en bajas concentraciones y se unen a receptores específicos en diferentes células, lo que ocasiona la liberación de mediadores secundarios y de otras citocinas, así como la expresión de múltiples moléculas que permiten la activación de diferentes poblaciones celulares 6-9,43. Diversos estudios han relacionado la presencia de ciertos polimorfismos en los genes que las codifican con la susceptibilidad a infecciones, las diferentes formas clínicas de la sepsis y la mortalidad 44-57.
IL-1
Las IL-1 son potentes citocinas proinflamatorias liberadas por los macrófagos que participan en la respuesta inflamatoria sistémica. Esta familia se encuentra conformada por dos agonistas (Il-1α e Il-1β) y por una antagonista (receptor antagonista IL-1: ILra) 6.
Los genes IL-1A, IL-1B e IL-1RN codifican para IL-1α, IL-1β e IL-1ra, respectivamente, y están localizados en los genes que codifican para los antígenos leucocitario humano (HLA) en el área q13-21 el cromosoma 2 6. Se han identificado cinco polimorfismos relacionados con el riesgo de desarrollar sepsis: un SNP en la posición -889 de la región promotora del gen L-1A, dos SNP en la posición -31C y -511 de la región promotora y un SNP en la posición +3954 del exón 5 del gen IL-1B 44,45.
En el gen IL-1RN se ha detectado una región polimórfica en el intrón 2 con un VNTR de 86 pb que origina cinco alelos (A1, A2, A3, A4 y A5), los cuales son marcadores pronóstico para desarrollar sepsis grave (46). El estudio realizado por Fang et al.47 encontró que el 54% de los individuos sanos presentan cuatro copias de las 86 pb repetidas (alelo A4) y el 34% dos copias (alelo 2). El alelo 2 se asocia con altos niveles séricos de IL-1ra en numerosas enfermedades donde la inflamación juega un papel central.
Algunos estudios analizados establecen la asociación entre el polimorfismo en IL-1 con el desarrollo de la sepsis; sin embargo, los resultados de estos son inconsistentes y no concluyentes 43,44. Por su parte, Zhang et al.45 realizaron un metaanálisis con la literatura publicada relacionada con el polimorfismo en el gen IL-1 como factor de riesgo para el desarrollo de sepsis en las bases de datos de PubMed y Embase y en la web hasta junio del 2013; estos autores también encontraron que para el SNP en -889 IL-1A (rs1800587) una asociación significativa (OR=1.47; IC95%: 1.01-2.13; p=0.04), mientras que con los polimorfismos -511 IL-1B (rs16944) o -31 IL-1B (rs1143627) no se evidenció asociación. En el caso del SNP +3594 IL-1B (rs143634), el genotipo TT representó menor riesgo de sufrir la enfermedad (OR=0.59; IC95%: 0.36-0.97; p=0.04).
Para el VNTR en el gen IL-1RN se encontró una asociación significativa con el desarrollo de sepsis (OR=1.40; IC95%: 1.011.95; p=0.04); además, el incremento del VNTR estuvo relacionado con un mayor riesgo en la severidad de la enfermedad 45.
IL-6
La IL-6 es una citocina pleiotrópica secretada por varias células, entre las que se destacan macrófagos, fibroblastos, células endoteliales y células T 6. El gen que codifica para la IL-6 se localiza en el cromosoma 7, generado por el cambio de una G por C en la posición -174 G/C de la región promotora, y se relaciona con una menor mortalidad por sepsis cuando los pacientes presentan el genotipo GG 48. También se han identificado los SNP en la posición+1753C/G y +2954 G/C, a los cuales no se les ha demostrado en forma concluyente su relación con el riesgo de desarrollar sepsis 7.
En un estudio realizado con pacientes quirúrgicos, la mortalidad en la sepsis fue significativamente inferior en los sujetos homocigóticos GG (OR=0.11; IC95%: 0.02-0.57). En esta misma investigación, los pacientes que fallecieron tenían niveles séricos de IL-6, es decir, significativamente superiores comparados con los de aquellos pacientes que sobrevivieron (genotipo GG) 49. Estos resultados fueron concordantes con los obtenidos en un estudio realizado en un hospital alemán, en los que los pacientes portadores del genotipo homocigoto GG se asociaron con una mejora de la supervivencia en la sepsis en forma significativa 50.
También se ha encontrado un SNP en la posición +5174 de la región promotora de este gen y una asociación positiva con sepsis neonatal 51. Sin embargo, se necesita un mayor número de estudios para confirmar estos resultados.
IL-10
La IL-10 es la citoquina antiinflamatoria más potente que regula la disminución de las citocinas proinflamatorias y quimiocinas secretadas por monocitos, neutrófilos y eosinófilos; previene la activación de células T; inhibe la expansión de células T, y potencia la liberación del modulador inflamatoria IL-1ra 6.
El gen de la IL-10 está localizado en el cromosoma 1, en la posición 1q31-1q32 20. En la región de este gen se han determinado tres SNP: -1082A/G, -819C/T y -592C/A 52-55. El alelo A esta relacionado con el polimorfismo -1082A/G y se asocia con susceptibilidad al desarrollo de sepsis 52; por el contrario, el genotipo -1082G/G se ha asociado con menor mortalidad 53,54, mientras que en el alelo G (-819T/G) se observó que los pacientes presentan mayores niveles de IL-10, con un notable incremento en la mortalidad causada por la sepsis grave 54.
El alelo generado por el SNP en -592A se asocia con bajos niveles de IL-10 y un mayor riesgo de mortalidad como consecuencia de la sepsis 55,56. Shu et al.57 llevaron a cabo un estudio en China para investigar los SNP en las posiciones -592, -819 y -1082 y encontraron que estos se asociaban con una mayor incidencia de sepsis grave. Los pacientes con sepsis grave eran más propensos a tener el alelo A (-1082A) y los que sobrevivieron a esta presentaron menores niveles de IL-10 y mayor frecuencia del alelo G (-1082G), comparado con los controles (17% vs. 47.2%; p=0.012). En cuanto a las formas alélicas de los SNP -592 y -819 no se encontró asociación con el riesgo de mortalidad 57.
TNF
La TNF-α es una citocina proinflamatoria multifuncional secretada predominantemente por monocitos/macrófagos y células T 6.
La TNF-β, también llamada linfotocina a (LT-α), es una citoquina producida por los linfocitos T que activa las células endoteliales y los neutrófilos y actúa como mediador de la respuesta inflamatoria aguda y la activación de células T.
Los genes que codifican para la proteínas TNF-α y TNF-β están localizados dentro de los genes del antígeno leucocitario humano (HLA) clase III, en el cromosoma 6 posición 6p21.3 6. El papel de los polimorfismos de estos genes con susceptibilidad a la sepsis se ha evaluado en diferentes poblaciones de todo el mundo 7,8. También se han reportado varios SNP en la región promotora del gen que codifica para el TNF-α y algunos han sido relacionados con el desarrollo de sepsis. Los polimorfismos más estudiados se encuentran en el sitio -308 que genera el cambio nucleotídico G/A 58,59. Los alelos resultantes del SNP -308 G/A se denominan TNF1 y TNF2; las copias que contienen G corresponden al alelo TNF1 y las que poseen A corresponden al alelo TNF2 58. Los mayores niveles plasmáticos del TNF-a se detectan en pacientes que murieron con un SNP T/A -308 y tenían una mayor susceptibilidad para desarrollar sepsis grave y choque séptico 59.
Un segundo polimorfismo fue detectado en la posición -238G/A en la unión con el represor, ubicado en la región promotora donde el alelo A sustituye al alelo más común G y se asocia con bajos niveles de expresión del gen 60. La variación 250 G/A se asocian con un alto nivel de mortalidad en choque séptico en adultos y bacteremia en niños 61.
Dwyer et al.62 examinaron la asociación de los polimorfismos en la región promotora del gen TNF-α y los niveles de ARNm en una cohorte de pacientes con sepsis grave y encontraron que en los pacientes homocigotos había mayor producción de ARNm para el alelo G (SNP en la posición -308) que en los que portaban el alelo A (SNP en la posición -863). Los pacientes portadores del haplotipo 1 (alelo A y G) también presentaron mayor nivel de ARNm, mientras que los portadores del haplotipo 4 (con C en la posición -863 y A en la posición -308) presentaron disminución en los niveles de ARNm. Los pacientes homocigotos para el alelo A en la posición -308 presentaron mayor mortalidad en la sepsis grave que los portadores del alelo G.
En el caso del gen que codifica para el TNF-β, se ha detectado un SNP G/A en la región +252 ubicada en un intrón. La variante alélica con G se denomina TNFB1 y está relacionada con una mayor producción de TNF-P, mientras que la variante con A se denomina TNFB2 y está relacionada con mayor producción de TNF-α 43).
Estudios llevados a cabo en alemanes caucásicos con shock séptico han demostrado que los pacientes con el alelo TNFB2 tienen una mayor capacidad secretora de TNF-a respecto al alelo TNFB1 y que el alelo TNFB2 también se asocia con un mayor riesgo de muerte. Entre los pacientes posoperatorios con sepsis severa, el 65% de los que no sobrevivieron eran homocigóticos para el alelo variante (TNFB2/B2) 9. Sin embargo, un estudio realizado en Brasil con 60 pacientes con sepsis y 148 donadores sanos de sangre estableció la relación del alelo TNFB2 con la susceptibilidad a la sepsis, pero no se encontró que este estuviera asociado con los biomarcadores inmunológicos y clínicos de la enfermedad 63.
Polimorfismos en receptores celulares
TLR
Existen estudios que han tratado de cuantificar los efectos en la variación genética en el TLR durante la respuesta inflamatoria a los PAMP y el riesgo a sufrir disfunción de órganos o muerte en pacientes con sepsis. Wurfel et al.64 realizaron un estudio de cohorte en pacientes con sepsis analizando el polimorfismo del TLR-1 en la posición -7202A/G (rs5743551) y encontraron que el alelo G se relacionó con la elevada producción de citocinas. Este SNP se genera en la región codificante del gen y causa una mayor inducción del gen por la activación de NF-kB. En consecuencia, el LTR1 se expresa en mayor medida en la superficie de la célula 64. Los pacientes con sepsis que presentaron el alelo G tuvieron un mayor riesgo de disfunción multiorgánica y de muerte (OR=1.82; IC95%: 1.7-3.9). En esta misma investigación, se realizó un estudio de casos y controles y se encontró que el alelo G estuvo asociado con el riesgo de sufrir lesión pulmonar aguda relacionada con sepsis (OR=3.40; IC95%: 1.59-7.27). Además, este alelo se encontró en mayor proporción en pacientes con sepsis por bacterias Gram positivas.
El polimorfismo del TLR-2 en la posición -16.933A, que provoca el cambio de arginina por glicina en la posición 753 del receptor (Arg753Gln), se relaciona con bacteremia ocasionada por bacterias Gram positivas con desarrollo de sepsis y choque séptico 65.
El SNP en la posición +896G del gen que codifica para el TLR-4 resulta en una sustitución de glicina por ácido aspártico en la posición 299 de la secuencia de aminoácidos (Asp299Gly) y se asocia con una mayor mortalidad en pacientes con síndrome de respuesta inflamatoria sistémica (SIRS) y con el desarrollo de sepsis en pacientes con traumas o quemaduras 66. El otro SNP resulta en un cambio de treonina a isoleucina en la posición 399 de la secuencia de aminoácidos (Thr399Ile), este SNP desencadena una mayor prevalencia de infecciones por bacterias Gram negativas 67. Las variantes Asp299Gly e Ile399Thr reducen los niveles de IL-1α y la respuesta a la endotoxina del lipopolisacárido (LPS) en pacientes infectados.
El estudio de Schroder & Schumann 68 identificó una asociación positiva entre el polimorfismo Thr399Ile y los nacimientos prematuros. Sin embargo, como la frecuencia de alélicas de este polimorfismo TLR4 es baja (6-10%), es necesario un mayor número de estudios epidemiológicos para comprobarlo.
CD14
La molécula CD14 es una glicoproteína receptora de 53 KDa que se expresa en la superficie de las células monocíticas. Además de esta forma celular, existe la forma soluble (CD14s), que induce la cascada inflamatoria cuando se une al LPS de las bacterias Gram negativas 6.
El gen que codifica para la molécula CD14 presenta un SNP en la región promotora en -159T/C y se establece como un marcador predictivo de mortalidad y de sepsis grave en pacientes quemados 65. El alelo T se encontró relacionado con un incremento de choque séptico y el genotipo TT fue un factor de riesgo para la mortalidad (OR=5.3) 69.
TREM-1
La proteína TREM hace parte de la superfamilia de las inmunoglobulinas que se expresan en los granulocitos polimorfonucleares y en monocitos maduros. Su expresión se induce por la infección por bacterias y hongos 7-9.
El VNTR rs2234237, detectado en el gen que codifica para la proteína TREM-1, se asocia con mortalidad en pacientes sépticos; los VNTR rs7768162 y rs9471535 del gen TREM-1 se relacionan con sepsis grave y el rs2234237 se asocia con una elevada mortalidad a los 28 días en los pacientes con sepsis 70. Se plantea entonces que el TREM-1 puede ser empleado como un biomarcador ideal de fatalidad para el diagnóstico y pronóstico de sepsis.
Polimorfismos genéticos en efectores moleculares de la inflamación
Lectina de unión a mañosa (MBL)
La MBL es una lectina que reconoce los motivos de polisacáridos de varios patógenos y su función principal es participar en la opsonización 6.
Los SNP en posición -221C y -550C en la región promotora del gen que codifica para la MBL están relacionados con bajos niveles de la proteína en pacientes que desarrollaron sepsis y choque séptico 71. También se han identificado polimorfismos en la región codificante del gen que generan tres cambios aminonoacídicos en las posiciones 52TGT, 54GAC, y 57GAA de la proteína MBL (se denominan variantes D, C y B, respectivamente), lo que ocasiona la inestabilidad en el plegamiento de la proteína y el desarrollo de pielonefritis, bacteremia y choque séptico a causa de la infección por Escherichia coli72.
HSP70
La familia de la proteína Hsp70 actúa como inhibidor de mediadores inflamatorios producidos por la vía de activación del factor nuclear kappa, beta (NF-κβ). Esta se encuentra codificada por tres genes (HSPA1B, HSPA1A, y HASA1L) ubicados en la región HLA en el cromosoma 6, cercanos a la región codificante por las proteínas TNF-α y del complemento 6.
Se han detectado los polimorfismos en la región codificante del gen HSPA1B en la posición +1267 (alelo A) y en la posición -179 (alelo C). El haplotipo -179C/+1267A se relaciona con la producción significativa de HSPA1B y TNF-a como respuesta a la exposición a bacterias Gram negativas 73. Sapru et al.74 demostraron que los pacientes con el genotipo AA en la posición +1267 presentaron mayor riesgo de desarrollar choque séptico que aquellos quienes presentaron el alelo G, ya fuera homocigoto o heterocigoto.
β-defensina humana 1 (DEFB1)
El DEFB1 es un péptido con actividad microbicida y citotóxica, secretado por los neutrófilos y mediador multifuncional en la infección y la inflamación. Este péptido se ha explorado en gran medida en los estudios ex vivo6.
En el gen DEFB1 se detecta un SNP en la posición -44G que genera sepsis grave y mayor mortalidad por esta enfermedad 75. Fang et al.76 investigaron las variaciones en el gen asociadas con el desarrollo de la sepsis y encontraron seis polimorfismos: el genotipo con los alelos -44G/-44G se asoció significativamente con la incidencia de sepsis grave; el alelo -20G y el genotipo GG se relacionaron con la susceptibilidad a la sepsis grave, mientras el genotipo con los alelos -1816G/-1816G-alelo influyó en el resultado de la sepsis grave; los haplotipos -20A/-44C/-52G mostraron un papel protector contra la sepsis grave, y el haplotipo -20G/-44G/-52G fue un factor de riesgo para el desarrollo fatal de la sepsis grave.
Inhibidor del activador del plasminógeno tipo 1 (PAI-1)
Se trata de una glicoproteína de 50 kilodalton que pertenece a la familia de inhibidores de serina proteasa que promueve la coagulación; el PAI-1 actúa como una proteína de fase aguda durante la inflamación y sus niveles se relacionan con aumento de la gravedad de los pacientes con sepsis 6,7,10.
En el gen que codifica para esta proteína se han encontrado variaciones nucleotídicas en su región promotora; lo que produce el genotipo 4G/4G, que se relaciona con un incremento de los niveles plasmáticos del PAI, desarrollo de sepsis, disfunción orgánica y mortalidad 77. Binder et al.78 demostraron que la predisposición genética para producir altos niveles de PAI-1 (la 4G/4G genotipo) se asocia con un mal pronóstico de la sepsis grave. El alelo 4G de PAI-1 se vincula a concentraciones plasmáticas elevadas de PAI-1 y a una baja sobrevida de los pacientes, mientras que el polimorfismo 4G/5G se vincula con el desarrollo de meningococcemia sistémica y coagulación intra-vascular diseminada 78.
Factor inhibitorio de migración del macrófago (MIF)
Este factor es producido por diversas células y juega un papel importante en la patogénesis de enfermedades inflamatorias agudas y crónicas, así como en desórdenes autoinmunes 6. En pacientes con sepsis, se han detectado niveles séricos altos de MIF y se correlacionan con la severidad de la enfermedad 7,8.
El SNP detectado en la posición -173C del gen que codifica para MIF se relaciona con el desarrollo de sepsis en pacientes con neumonía asociada a la comunidad, al igual que una secuencia repetida detectada en la posición -794 CATT, lo que provoca un aumento en los niveles de la proteína 79.
Caspasa 12
Las caspasas son enzimas claves en la mediación de los eventos proteolíticos en las cascadas de inflamación y muerte celular por apoptosis. En este grupo se encuentra la caspasa 12 (Csp-12), la cual se relaciona filogenéticamente con las caspasas de maduración de citocinas (caspasas 1, 4 y 5), conocidas como caspasas inflamatorias6.
Saleh et al.80 detectaron un SNP en el exón 4 del gen que codifica para la caspasa 12 con un cambio 125T>C, el cual conduce a la alteración de un codón de terminación por una arginina, lo que a su vez origina la síntesis de una proteína truncada (Csp-12S) o una proteína más larga (Csp-12L). Esta forma larga se relaciona con desarrollo de sepsis grave.
Un estudio realizado por Mejía et al.81 en 81 pacientes de Medellín con diagnóstico de sepsis, 23 individuos sanos de una población afroamericana del Chocó y 24 individuos sanos provenientes de Medellín buscó analizar el polimorfismo 125T>C de la Csp-12 y encontró que el alelo L es más frecuente en individuos afroamericanos y, en una menor proporción, en los mestizos, lo que indica que la población afroamericana de Colombia podría tener mayor susceptibilidad a sepsis grave que las poblaciones mestizas.
Polimorfismo genético en el sistema de coagulación del hospedero
Proteína C
Los niveles endógenos y plasmáticos de la proteína C en pacientes con sepsis se asocian en forma inversa con la evolución de la enfermedad y la mortalidad 6.
El gen que codifica para la proteína C se encuentra localizado en el cromosoma 2q13-14 y los polimorfismos se han identificado en la región no traducida 5' en las posiciones -1654CT y -1641AG, ambos afectan la transcripción y se relacionan con severidad de sepsis 82. El haplotipo CA en este SNP se ha asociado con un mayor riesgo de disfunción orgánica y muerte, mientras que el haplotipo CG se relaciona con mayor frecuencia en los pacientes con meningococcemia 83.
Selectina E
La selectina E es una proteína de superficie que se expresa en las células endoteliales y que media el rodamiento de los leucocitos 6.
Jilma et al.84 investigaron el efecto del cambio Ser128Arg de la selectina E que se produce en la inflamación y la coagulación en voluntarios humanos que recibieron dosis de LPS. Los marcadores de la coagulación fueron más altos en individuos con el polimorfismo en comparación con los controles con el genotipo normal. El cambio se relacionó con un incremento en la inflamación y la coagulación de los pacientes infectados con bacterias Gram negativas.
En la Tabla 2, se resumen los principales SNP determinados como marcadores.
Susceptibilidad genética a sepsis en las poblaciones
Debido a los SNP y los VNTR, el polimorfismo genético ocasiona una heterogeneidad alélica y de locus entre las poblaciones. La acción combinada de estos biomarcadores determina el grado de respuesta a la sepsis, el cual varía en cada persona. El fenotipo final encontrado en el paciente puede ser el reflejo de los SNP que se han heredado juntos durante miles de años de evolución (haplotipos) y que han evolucionado interaccionando entre sí.
Los SNP o VNTR han estado asociados a diversas poblaciones como la asiática y europea, así como algunos países de África y diversos lugares de América. Ciertos SNP relacionados con sepsis se pueden acentuar en algunas poblaciones que presentan una mayor gravedad y mortalidad cuando la enfermedad se ha manifestado. Por ejemplo, los SNP -1641 A/T y 1654C/G en el gen Il-6 se relacionan con un incremento en los niveles de IL-6, desarrollo de sepsis y disfunción orgánica en la población china 85,86. Una variante detectada en la posición -572 con un cambio de C por G en la región promotora de este gen reduce su actividad transcripcional y se asocia con un bajo riesgo de desarrollar sepsis, específicamente en el grupo étnico han de China 87.
Chen et al. (75) y Chen et al.88 encontraron SNP en los genes Trem-1 y Defb1 en este grupo han con predisposición a desarrollar sepsis grave. También se estudiaron los polimorfismos en las regiones codificantes de los genes ltr4, cd14, TNF e ll-10 en la población japonesa 89 y un VNTR (rs2069912) en el gen que codifica para la proteína C reactiva en poblaciones de indígenas de Norteamérica y de Asia Oriental con un desarrollo fatal en sepsis grave 90.
En poblaciones africanas y americanas se ha encontrado la variación del gen de la IL-6 en la posición -174 G/C y en la posición -260 C/T de la molécula CD14 como marcadores predictivos de mortalidad en sepsis inducida por ventilación mecánica prolongada 87. En las poblaciones caucásicas se ha identificado el alelo -376G/A del gen TNF-a que se relaciona con una mayor producción de la citocina 91.
En el caso del SNP 125T/C, en el gen que codifica para la caspasa 12 (proteína implicada en la muerte celular programada), la frecuencia del alelo L es mucho mayor en poblaciones afroamericanas, donde este alelo se ha detectado abriendo la posibilidad que esta población pudiese tener mayor susceptibilidad a desarrollar sepsis grave 81. Todas estas asociaciones permiten un análisis más profundo a la hora de relacionar el genotipo de un paciente con la susceptibilidad a la enfermedad. Sin embargo, es necesario evaluar todos estos marcadores con el fin de obtener un patrón de susceptibilidad que permita definir los marcadores con un alto porcentaje en el desarrollo de la sepsis.
Conclusión
La sepsis es un problema de salud pública que debe ser intervenido con estrategias oportunas para disminuir el porcentaje de mortalidad. La búsqueda de nuevos biomarcadores para evaluar la severidad de la enfermedad y predecir el pronóstico de los pacientes es un desafío importante que proporciona una nueva idea para enfrentar la sepsis.
El empleo de los biomarcadores moleculares tiene su fundamento en el hecho que cada paciente tiene su propio acervo genético con distinto grado de respuesta a las enfermedades, por lo que constituye un papel importante en la determinación de la susceptibilidad para el resultado de enfermedades tan complejas como la sepsis.
Conflicto de intereses
Ninguno declarado por los autores.
Financiación
Ninguna declarada por los autores.















