Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Colombiana de Entomología
versión impresa ISSN 0120-0488versión On-line ISSN 2665-4385
Rev. Colomb. Entomol. v.29 n.1 Bogotá ene./jun. 2003
Estudio preliminar de la estructura genética de poblaciones domésticas, peridomésticas y silvestres de Triatoma dimidiata (Hemiptera: Reduviidae)
Preliminary study of the genetic structure of domestic, peridomestic and sylvatic populations of Triatoma dimidiata (Hemiptera: Reduviidae)
CARLOS JARAMILLO1, CAROLINA RAMÍREZ M.2, PILAR DELGADO3, NÉSTOR PINTO4, GERMÁN AGUILERA5, FELIPE GUHL6
1Autor para correspondencia: M. Se. Universidad de l.os Andes, Departamento de Ciencias Biológicas, Centro de Investigaciones en Microbiología y Parasitología Tropical, CIMPAT. Carrera 1 No. 18 A - 10 Bloque A. Fax. 3324540, Tel. 3394949 Ext. 2774. E-mail: cjaramil@uniandes.edu.co
2Microbióloga. Universidad de Los Andes, Departamento de Ciencias Biológicas, CIMPAT. E-mail: carolin-@uniandes.edu.co
3M. Se. Universidad de Los Andes, Departamento de Ciencias Biológicas, CIMPAT. E-mail: mdelgado@uniandes.edu.co
4M. Se. Universidad de Los Andes, Departamento de Ciencias Biológicas, CIMPAT. E-mail: npinto@uniandes.edu.co
5M. Se. Universidad de Los Andes, Departamento de Ciencias Biológicas, CIMPAT. E-mail: geragui25@hotmail.com
6M. Se. Universidad de Los Andes, Departamento de Ciencias Biológicas, CIMPAT. E-mail: fguhl@uniandes.edu.co
Resumen. Con el fin de estudiar la estructura genética de poblaciones silvestres, peridomésticas y domésticas de Triatoma dimidiata se empleó la técnica de amplificación azarosa de polimorfismos de ADN (RAPD). Se calculó el grado de flujo genético existente entre las poblaciones para estimar el riesgo epidemiológico que representan las poblaciones no domiciliadas en la transmisión de la enfermedad de Chagas. Se analizaron 14 individuos silvestres, 10 individuos peridomésticos y 15 domésticos. Se obtuvo un Fst de Wright de 0.071 que denota que hay poca diferenciación genética, y una tasa efectiva de migración (Nm) de 3.3 que indica que por lo menos 3 individuos migran por generación. Estos hallazgos sugieren que las poblaciones no domiciliadas de T. dimidiata representan un riesgo epidemiológico en la transmisión de la enfermedad de Chagas dado que pueden colonizar las viviendas. Por lo tanto deben plantearse: la vigilancia epidemiológica no sólo de la población domiciliada sino también de la peridomiciliada; nuevas y mejores alternativas de control encaminadas a los reservónos en el peridomicilio, y un programa de educación permanente de las personas en riesgo de adquirir la enfermedad.
Palabras clave: Enfermedad de Chagas. RAPD. Flujo genético. Fst. Tasa de migración. Programas de control.
Summary. In order to study the genetic structure of sylvatic, peridomestic and domestic populations of Triatoma dimidiata, we used the randomly amplified polymorphic DNA (RAPD) technique. The genetic flow among populations was estimated to determine the epidemiológica! risk of nondomiciliated populations in the transmission of Chagas disease. Fourteen sylvatic, 10 peridomestic and 15 domestic insects were analyzed. The estimated Wright 's Fst was 0.071 showing low genetic differentiation and an effective migration rate Nm of 3.3 suggesting a movement of at least tnree individuáis per generation. These findings suggest that nondomiciliated populations of T. dimidiata represent an epidemiológica! menace for the transmission of Chagas disease, due to their ability to colonize dwelling. It is necessary to perform: an epidemiológica! surveillance not only of the domestic populations but also on the peridomestic ones. It is also important to carry out new and better control alternatives for reservoirs, and to offer a permanent education program to people at risk of infection.
Key words: Chagas disease. RAPD. Genetic flow. Fst. Migration rate. Control programs.
Introducción
La enfermedad de Chagas es exclusiva del continente americano, y se considera la enfermedad parasitaria más importante en América (Worl Bank 1993). Muestra una prevalencia de 16 millones de personas, una incidencia de 200.000 nuevos casos cada año (estadísticas de 1999) y una mortalidad de 50.000 personas por año (OMS). El rn/panosoma cruzí, agente causal de esta enfermedad, es transmitido principalmente por insectos vectores que han mostrado una fuerte estructuración geográfica que puede ser detectada por marcadores morfométricos, citogenéticos e isoenzimáticos (Guhl y Schofield 1996). Igualmente pueden emplearse marcadores moleculares generados por amplificación azarosa de polimorfismos de ADN que permiten determinar la estructura genética de las poblaciones de vectores (Williams et al. 1991; Black et al. 1992). En Colombia, se consideraba que Rhodnius prolixus (Stal) vector principal de esta enfermedad, presentaba dos poblaciones distribuidas en el domicilio y en las palmeras. Moreno et al. (1999), mediante métodos morfométricos y bioquímicos, demostró que no eran dos poblaciones de Rhodnius prolixus, sino dos especies diferentes similares morfológicamente, y definió a los insectos que se encuentran en el ambiente silvestre como una nueva especie, Rhodnius colombiensis. Paralelamente, usando técnicas moleculares (Jaramillo et al. 2001) estableció que no se presentaba flujo genético entre Rhodnius silvestres y domiciliados, corroborando lo hallado por Moreno et al. (1999).
El análisis de polimorfismos en el ADN ya ha probado ser útil en la determinación de flujo genético en poblaciones de insectos como Aedes aegypti (Gorrochotegui-Escalanle et al. 2002).
Estudios como los realizados en Rhodnius, evidencian la necesidad de analizar otras especies vectoras a través del análisis de su estructura genética y por tanto del entendimiento del comportamiento de sus poblaciones, con el fin de controlar a mediano y largo plazo los vectores, focalizar los programas de control, y por tanto prevenir la transmisión de la enfermedad de Chagas.
En esta investigación se muestran resultados preliminares sobre la estructura genética de poblaciones de Triatoma dimidiata (Latreille), el segundo vector en importancia en Colombia de la transmisión de la enfermedad. A diferencia de otras especies estrictamente domiciliadas como T. infestans, este insecto se ha encontrado en tres hábitats o ambientes diferentes, uno silvestre (cuevas y rocas), uno peridoméstico (gallineros y camas de animales), y uno doméstico. Se. empleó la técnica de KAPD (Williams el al. 1991), con el fin de detectar variabilidad genética entre las poblaciones silvestre, peridoméstica y doméstica de esta especie y calcular el grado de flujo genético que se presenta entre ellas, para determinar el posible riesgo que representan las poblaciones no domiciliadas en la transmisión de la enfermedad. Este estudio se llevó a cabo en el Centro de Investigaciones en Microbiología y Parasitología Tropical - CIMPAT. Universidad de Los Andes, Bogotá - Colombia, año 2001.
Materiales y Métodos
Recolección de insectos
Los insectos evaluados se capturaron en la vereda Río de Abajo, municipio Boavita del departamento de Boyacá, a 1.970 msnm, 6o 19' 33.5" N; 72° 37' 10.61" W. Las capturas se realizaron en tres hábitats diferentes: en cuevas (habitat silvestre), en el peridomicilio (gallineros, cocinas, bodegas, camas de animales) y dentro de las viviendas.
Extracción y purificación de ADN
Se retiraron las 6 patas de cada insecto y se sometieron a un proceso de ruptura mecánica en un volumen de 300 ul de "Genomic DNA lysis solution" del kit AquaPure Genomic DNA de BIORAD. Se continuó con el procedimiento recomendado por el fabricante para aislamiento de ADN a partir de tejidos.
Amplificación azarosa de polimorfismos de ADH (RAFD)
Se empleó el kit Ready To Qo RAPD de Pharmacia, rehidratando cada perla con 19 ul de agua destilada y agregando 25 pmol de iniciador. Se dividió el contenido en cuatro tubos, a cada uno de los cuales se le agregó 2 ul de ADN templado, para un volumen final de reacción de 8 ul. Se analizaron 14 individuos silvestres, 10 peridomésticos y 15 domésticos con los iniciadores RTG de Pharmacia 2 y 3 (Tabla 1), con el siguiente perfil térmico: un paso inicial de denaturación a 95.5"C por 5 minutos, seguido de 44 ciclos de 95°C por 1 min, 36°C por 1 min, 72°C por 2 min y un paso final de elongación a 72°C por 7 min.

Visualización de los productos de RAPD
Los productos de amplificación se visualizaron en geles de poliacrilamida al 6%. Posteriormente, se tiñeron con el kit Silver Stain Plus de Pharmacia siguiendo el protocolo recomendado por el fabricante, empleando la mitad del volumen indicado para la tinción de cada gel.
Análisis de resultados de los RAPD
Se construyeron matrices binarias comparando las bandas más intensas de cada gel e indicando la presencia de una banda como 1 y la ausencia de la misma como 0. Las matrices se construyeron de acuerdo con Welsh eí al. (1992) y se analizaron con los programas SYNTAX (Podani 1993), POPGENE (Yeh eí al. 1999) y RAPDPLOT (Kambhampati eí al. 1992).
Para el análisis, se partió del supuesto de que las tasas de evolución o sustitución son constantes para las poblaciones; es decir, que se encuentran en un equilibrio de Hardy-Weinberg y que por tanto no hay presiones de selección que favorezcan una u otra población o algún genotipo particular.
Los programas RAPDPLOT y POPGENE producen una matriz que contiene las distancias genéticas calculadas usando el índice de similitud de Nei y Li (1985) que se define de la siguiente manera: S= 2NAB / (NA+NB). donde NAB es el número de bandas que comparten los individuos A y B, NA es el número de bandas de A y NB el número de bandas de B.
Dado que los RAPD segregan independientemente y más del 95% de los alelos son dominantes (Williams eí al. 1991), RAPDPLOT emplea una segunda medida que involucra tanto la presencia compartida de una banda como la ausencia compartida. lo que permite tomar ventaja del fenotipo recesivo (o ausencia compartida de una banda). Se estima el número de concordancias M que permite determinar relaciones genéticas entre individuos mediante el cálculo M = NAB/ NT, donde NAB es el número total de bandas compartidas (presentes y ausentes) entre los individuos A y B. y NT es el número total de loci en estudio.
Los programas RAPDPLOT y SYNTAX permiten utilizar el algoritmo UPGMA, introducido por Sneath y Sokal (1973), el cual toma la distancia más pequeña de la matriz de distancias y elabora un cluster con los individuos que tengan esa distancia. Luego calcula la similitud promedio entre las poblaciones y el cluster ya evaluado, generando así una nueva matriz de distancias, repitiéndose el proceso hasta analizar todos los individuos.
Resultados
Con los 14 individuos silvestres, 10 peridomésticos y 15 domésticos de Triatoma dimidiata evaluados, se observa que no hay un patrón de bandeo definido para cada población (Fig. 1). Se aprecia una homogeneidad en los patrones de amplificación que sugiere una diferenciación genética baja entre las poblaciones de este insecto.
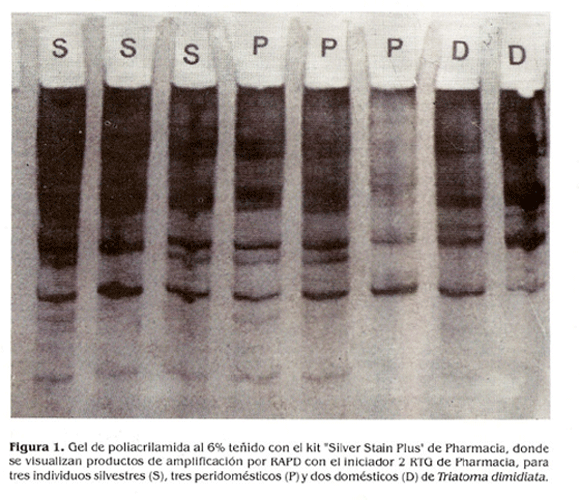
Al realizar un análisis mediante los programas RAPDPLOT, POPGENE y SYNTAX, se obtuvo un dendrograma donde no se define un cluster para cada población, sino que se ve una distribución heterogénea de los individuos de las diferentes poblaciones (Fig. 2).

Se generaron dendrogramas de las poblaciones silvestre y doméstica (fig 3a), peridoméstica y doméstica (fig 3b), y silvestre y peridoméstica (fig 3c), observándose que para los pares de poblaciones evaluados, se mantiene el patrón de distribución heterogénea advertido para las tres poblaciones en conjunto.
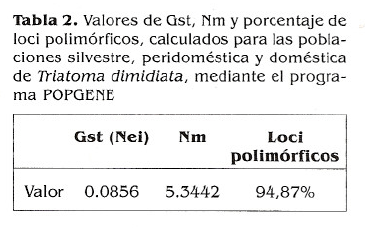
Al realizar el análisis con POPGENE se obtuvo el valor de Gst (Nei 1973), el cual combina información de múltiples loci en una sola medida de la subestructura poblacional y es un estimativo del Fst o índice de fijación. También se obtuvo el Mm y el porcentaje de loci polimórficos (Tabla 2).
Se calculó el valor de identidad genética y el de distancia genética de Nei entre las poblaciones, tanto para la distancia original de 1973 (Tabla 3), como para la distancia de nei corregida con el algoritmo UPGMA (1978) (Tabla 4). Con ambas medidas se observa que hay una identidad genética alta entre las poblaciones, siendo mayor para las poblaciones silvestre y peridoméstica.

El valor de distancia genética es mayor entre las poblaciones peridoméstica- doméstica y Silvestre-doméstica.
Se obtuvo un dendrograma de las tres poblaciones evaluadas donde se evidencia una diferenciación genética menor entre la población silvestre y la peridoméstica con respecto a la doméstica (Fig. 4), dado que el cluster que agrupa a las dos primeras agrupa a una menor distancia genética.
Con el programa RAPDPLOT se calculó el Fst de Wright (1951), de Weir y Cockerham (1984), y de Lynch y Milligan (1994) (Tabla 5), tanto para las tres poblaciones en conjunto, como para los pares de poblaciones. Para todos los casos, el Nm fue suficiente para mantener homogeneidad genética, y el Fst indicó moderada diferenciación genética.
Discusión
Los resultados obtenidos sugieren un flujo genético mayor que el registrado para especies de mariposas como Papilo cjlaucus en Oblo, Georgia y Florida donde se logró un tim de -0.0032 (Bossart y Scriber 1995). Previamente se ha descrito flujo genético entre poblaciones de insectos como Aedes aegypti (el principal vector de fiebre amarilla y dengue) en Puerto Rico, con un Mm de 11.7 (Apóstol et ai. 1993).
El método de estimación de flujo genético es indirecto, dado que se basa en una distribución geográfico-espacial de unos genotipos o individuos, que posteriormente han sido sometidos a un análisis genético molecular para que permita inferir un comportamiento poblacional que concuerde con el parámetro genético (Slatkin 1985, 1993). El patrón encontrando con los individuos evaluados, sugiere que hay un nivel elevado de flujo genético entre las poblaciones, que no permite clasificarlas como unidades discretas, sino que las define como poblaciones de una especie que ¡nteractúan constantemente entre sí. Hay migración de los insectos (Tabla 2), es decir movimiento de los individuos desde un ambiente silvestre hacia uno peridoméstico y hacia uno doméstico, incluyendo movimientos que puedan hacer que regularmente un individuo retorne a su localización original (Baker 1978). Parece no darse entre las poblaciones de Triatoma dimidiata un proceso de dispersión pues no se presenta un incremento significativo en las distancias genéticas entre las poblaciones (Tablas 3 y 4), aunque en los dendrogramas se observa una variabilidad mayor de la población doméstica, Siete de 15 individuos domésticos evaluados presentan un mayor grado de disimilitud o variabilidad genética con respecto a individuos silvestres o peridomésticos (Fig. 2), observándose también una relación de individuos silvestres y peridomésticos más estrecha pero no definitiva, lo que podría indicar que los individuos domésticos una vez que logran colonizar la vivienda es poco probable que retornen a su habitat natural silvestre. Esto se observa igualmente en el dendrograma de poblaciones (Fig. 4), donde hay una mayor relación genética entre la población silvestre y la población peridoméstica, con respecto a la doméstica.

Los valores de Gst y Fst obtenidos indican que hay una diferenciación genética moderada entre las poblaciones de T. dimidiata; es decir, que hay un intercambio genético entre las poblaciones que permite mantener una heterocigocidad importante. Por otro lado, los valores de Nm (Tabla 5) denotan una migración representativa de individuos por generación suficiente para mantener homogeneidad genética y se muestran como cifras significativamente elevadas si se comparan con valores de Nm descritos para insectos (Slatkin 1985).
Se obtuvo un porcentaje de loci polimórFicos de 94,87%, es decir 37 de 39 loci estudiados (Tabla 2). Esto concuerda con un modelo de efecto Wahlund (Wahlund 1928), en donde la homocigocidad promedio decrece cuando las subpoblaciones se mezclan, es decir, que hay un aumento de heterocigotos y por tanto de loci polimórficos, generando así más alelos en una población.
Puede plantearse que las poblaciones de T. dimidiata estarían encaminándose hacia un proceso lento de domesticación, como ya está establecido para otras especies como T. infestans en países del cono sur o R. prolixus en los Andes o en Centro América. En años recientes, ya se han señalado establecimientos de colonias de T. dimidiata en viviendas urbanas y periurbanas (Schofield et al. 1999). La mayor diferenciación genética de la población domiciliada con respecto a la peridomiciliada y a la silvestre, podría ser consecuencia de una permanencia de los insectos dentro de las viviendas, en donde encuentran nuevas fuentes de alimento, resguardo y mejores condiciones climáticas (Schofield et al. 1999).
Conclusiones
- Mediante la técnica de RAPD se obtuvieron patrones de bandeo homogéneos para las poblaciones silvestre, doméstica y peridoméstica de Triatoma dimidiata, sin poder discriminar con los 39 marcadores obtenidos, una u otra población. Esto deja establecer que hay un flujo genético elevado entre las poblaciones, que no permite que se diferencien.
-El Fst y el Nm obtenidos mediante las metodologías de Wright (1978), Lynch y Milligan (1994) y Weir y Cockerhan (1984), son suficientes para mantener una homogeneidad genética entre las poblaciones.
-Las poblaciones no domiciliadas de 7". dimidiata estarían representando un riesgo epidemiológico en la transmisión de la enfermedad de Chagas dada la elevada movilidad de los individuos de las poblaciones no domiciliadas. Sería necesario un monitoreo periódico de la tasa efectiva de migración hacia el domicilio, al igual que una vigilancia y control del peridomicilio, y mejores programas educativos frente a las personas en riesgo de adquirir la enfermedad.
- Se evidencia la importancia de estudiar especies vectoras secundarias en la transmisión de esta enfermedad ya que pueden contribuir significativamente en su transmisión o en la reinfestación de casas tratadas con los insecticidas de acción residual.
- Estudios como éste brindan un beneficio social, económico y científico a la comunidad, ya que permiten conocer el comportamiento de poblaciones de vectores importantes en el país y así mismo, implementar programas de control mejores y más efectivos.
Agradecimientos
Este proyecto se llevó a cabo gracias al apoyo financiero de Colciencias y el B1D, proyecto Do. 276-2000.
Literatura citada
APÓSTOL, B. L.; BLACKIV, W. C; MILLER, B. R.; REITER, P.; BEATY, B. J. 1993. Estimation of the number of fullsiblings family at an oviposite site using RAPD-PCR markers: applicationsto the mosquito Aedes aegypti. Theor. Appl. Genet. 86: 991-1000. [ Links ]
BAKER, R. R. 1978. The Evolutionary Ecology of Animal Migration. rtodder & Stoughton, London. 1012 p. [ Links ]
BLACK IV, W. C; DUTEAU, N. M.; PUTERKA, G. J.; MECMOLS, J. R.; PETTORIMI, J. M. 1992. Use of the random amplifíed polymorphic DNA polymerase chain reaction (RAPD-PCR) to detect DNA polymorphisms in aphids. Bull. Entornol. Res. 82: 151-159. [ Links ]
BOSSART, J. L.; SCRIBER, J. M. 1995. Maintenance of ecologically significant genetic variation in the tlger swallowtail butterfly through differential selection and gene flow. Evolution 49: 1163-1171. [ Links ]
GORROCHOTEGUI-ESCALANTE, II.; GÓMEZ-MACHORRO, C; LOZANO-FUENTES, S.; FERNÁNDEZ-SALAS. L.; DE LOURDES MUÑOZ, M.; FARFAN-ALE, J. A.; GARCÍA-REJON, J.; BEATY, B. J.; BLACK, W. C. 2002. Breeding structure of Aedes aegypti populations in México varíes by región. Am, J. Trop. Med. Hyg. 66 (2): 213-22. [ Links ]
GUHL, F; SCHOFIELD, C.J. 1996. Population genetics and control of triatominae. Parasitology Today 12 (5): 169-170. [ Links ]
JARAMILLO, C; MONTAÑA, M. F; CASTRO, L. R.; VALLEJO, G. A. 2001. Differentiation and Genetic Analysis of Rhodnius prolixus and Rhodnius colombiensis by rDNA and Random amplified polymorphic DNA arnplification. Memorias do Instituto Oswaldo Cruz 96 (8): 1043-1048. [ Links ]
KAMBHAMPATI, S.; BLACK, W.C. IV; RAÍ, K. S. 1992. RAPD-PCR of mosquito species and populations: techniques, statistical analysis and applications. Journal of Medical Entomology 29: 939-945. [ Links ]
LYNCH, M.; MILLIGAN, B. G. 1994. Analysis of population genetic structure with RAPD markers. Molecular Ecology 3: 91-99. [ Links ]
MORENO, J.; GALVAO, C; JURGERG, J. 1999. Rhodnius colombiensis sp. N. Da Colombia com quadros comparativos entre estructuras fálizas do genero Rhodnius Stal. 1859 (Hemiptera, Reduviidae, Triatominae). Entomología y Vectores 6: 601-616. [ Links ]
NEI, M. 1973. Analysis of gene diversity in subdivided populations. Proc. Nati. Acad. Sci. USA 70: 3321-3323. [ Links ]
NEI,M.1978. Estimation of average heterozy-gosity and genetic distance from a small number of individuáis. genetics 89: 583-590. [ Links ]
NEI, M.; LI, W. H. 1985. Mathematical model for studying genetic variation ¡n terms of restriction endonucleases. Proc Nati Acad Sci USA 76: 5269 - 5273. [ Links ]
PODAMI, J. 1993. SYNTAX. Computer programs for multivariate data analysis in Ecology and Systematics. Scienta Publishing. Budapest. [ Links ]
SCHOFIELD, C. J.; DIOTAIUTI, L.; DUJARDIN, J.P. 1999. The Process of Domestication in Triatominae. Memorias do Instituto Oswaldo Cruz 94, Suppl. I: 375-378. [ Links ]
SLATKIN, M. 1985. Rare alíeles as indicators of gene flow. Evolution 39: 53-65. [ Links ]
SLATKIN, M. 1993. Isolation by distance in equilibrium and non-equilibrium populations. Evolution 47: 264-279. [ Links ]
SNEATH, P. A.; SOKAL, R. R. 1973. Numerical Taxonomy. Freeman. San Francisco. [ Links ]
WAHLUND, S. 1928. Zusanimersetung von Populationen und Korrelation-serchei-nungen von Standpunkt der Verebungs-lehre aus betrachtet. Hereditas 11:65-106. [ Links ]
WEIR, B. S.; COCKERHAM, C. C. 1984. Estimating F-statistics for the analysis of populations structure. Evolution 38: 1358-1370. [ Links ]
WELSH, J.; PRETZRNAN, C; POSTIC, D.; GIRONS, I.S.; BARANTON, G.; McCLELLAND, M. 1992. Genomic fingerprinting by arbitrary primer polymerase chain reaction resolves Borrelia burgeri into three distinct phylogenetic groups. International Journal Of Systematic Bacteriology 42: 370 - 377. [ Links ]
WILLIAMS, J. K.; KUNELIK, A. R.; LIVAK, K. J.; RAFALSKI, J. A.; TINGEY, S. V. 1991. DNA polymorphism amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research 18: 6531-6535. [ Links ]
World Bank World Development Report. 1993. Investing in Health, Oxford University Press. [ Links ]
WRIGHT, S. 1951. The genetical structure of populations, Ann. Eugen. 15: 323-354. [ Links ]
YEH, F; YANG, R.; BOYLE, T. 1999. University of Alberta. Centre for International Forestry Research. August. [ Links ]
Recibido: Jun. 30 / 2002 Aceptado: Ago. 16 / 2002















