Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Colombiana de Entomología
versión impresa ISSN 0120-0488versión On-line ISSN 2665-4385
Rev. Colomb. Entomol. v.30 n.1 Bogotá ene./jun. 2004
Análisis electroforético de las relaciones evolutivas en las especies de Drosophila (Diptera: Drosophilidae) del enjambre martensis: D. martensis, D. starmeri y D. uniseta
Electrophoretic analysis of the evolutionary relations in Drosophila species (Diptera: Drosophilidae) of the cluster martensis: D. martensis, D.starmeri and D. uniseta
MARINA ORDÓÑEZ1, ANGÉLICA ARENAS2, LUZ ANGELA BETANCOURT3, MARÍA MAGDALENA DE POLANCO4
1 Autor para correspondencia: M. Se. Biología. Instituto de Genética, Universidad de Los Andes. Cra. 1 No. 18A -10. Tel:3394949 Ext. 2794. Bogotá, Colombia. E-mail: marordon@uniandes.edu.co
2 Bióloga. Universidad del Tolima. Av. 5 No. 17-84. Ibagué, Colombia. E-mail: pitaya01@hotmail.com
3 Bióloga. Universidad del Tolima. Cra. 115 No. 89A-31 Ciudadeia Colsubsidio. Bogotá, Colombia. E-mail: canoluzangela@hotmail.com
4 Ph D. Genética. Lab. de Citogenética, Lab. de Ciencias, Universidad del Tolima. Calle 42 con cra 4 Santa Helena. Ibagué, Colombia. E-mail: mmpol@eudoramail.com
Resumen. En geles de almidón fue inferida electroforéticamente la variación genética de doce loci isoenzimáticos, entre seis poblaciones alopátricas colombianas del enjambre martensis (D.martensis, D. starmeri y D. uniseta). provenientes de Santa Marta (departamento del Magdalena), Camarones (departamento de la Guajira) y del desierto de la Tatacoa (departamento del Huiia). Esta estimación fue confrontada con una especie filogenéticamente relacionada (D. buzzatii) y una distante (D. melanogaster). Las divergencias enzimáticas se infirieron a partir de distancias genéticas y los resultados permitieron identificar seis niveles de divergencia taxonómica: Población local (D = 0,1031), subrazas (D = 0,1412), subespecies o razas (D = 0,1870), especies de un mismo enjambre o cluster (D = 0,7907), especies de enjambres diferentes (D = 1,0055) y especies de grupos diferentes (D = 1,4327). A través de este estudio preliminar, se confirmó la utilidad de los datos electroforéticos como estimadores de divergencia evolutiva y se sugiere un posible ejemplo de especiación alopátrica.
Palabras clave: Especiación alopátrica. Variabilidad alozímica. Niveles divergencia. Sinmórficas.
Summary. Starch gel electrophoresis was used to elucídate genetic variation at 12 isoenzyme loci among six allopatric Colombian populations of the cluster martensis (D. martensis, D. starmeri and D. uniseta), from Santa Marta (Magdalena Department), Camarones (Guajira Department) and from the Tatacoa desert (Huila Department). This estimation was compared to a phylogenetically related (D. buzzatii) and distant (D. melanogaster) species. The enzymatic divergences were inferred from genetic distantes and the results allowed the identification of six levéis of taxonomic divergence: Local population (D = 0,1031), subraces (D = 0,1412), subspecies or races (D = 0,1870), species of a same cluster (D = 0,7907), species of different clusters (D = 1,0055) and species from different groups (D = 1,4327). Through this preliminary study, the utility of electrophoretic data as estimators of evolutionary divergence was confirmed and a possible example of allopatric speciation is suggested.
Rey words: Allopatric speciation. Allozyme variability. Levéis of divergence. Siblings.
Introducción
El subgénero Drosophila se originó en la zona tropical del viejo mundo. La radiación inicial para los linajes del nuevo mundo surgieron a partir de la rama ancestral virilis/ repleta. Uno de los linajes más conspicuos y que ha presentado sucesivas radiaciones de Drosophila es el grupo repleta. La característica más notable de este grupo es que las especies utilizan como nicho trófico los cactus en descomposición, condición que le ha permitido establecerse en ambos hemisferios en zonas templadas, desérticas y semiáridas de los trópicos y semi - trópicos (Wasserman 1962; Sánchez 1987; Powell 1997; Castañeda 2000).
Las 89 especies del grupo repleta se caracterizan por poseer una cerda en cada punto negro del mesonotum, aspecto que las distingue de ios otros. Los estudios morfológicos y citogenéticos han permitido dividirlo en seis subgrupos o complejos: repleta, mercatorum, hydei, mulleri, fasciola y buzzatii (Patterson y Stone 1952; Wasserman 1962, 1967; Ruiz y Wasserman 1993). El subgrupo buzzatii está compuesto por los enjambres o clusters stalkeri, buzzatii y martensis. El enjambre martensis está conformado por las especies: D. martensis (Wasserman, Wilson en Vuela 1983), D. uniseta (Wasserman; Koepfer; Ward en Vuela 1983), D. starmeri (Wasserman, Koepfer, Ward en Vuela 1983) y D. venezolana (Ruiz y Fontdevila 1981).
Las especies del enjambre martensis son sinmórficas y están estrechamente relacionadas, porque comparten una serie de inversiones que les son únicas (Xabcj, 2abmnz7, 5d2). Su ancestro debió presentar un alto polimorfismo, que ha evolucionado a D. starmeri. Las demás emergieron de éste fijando sus propias inversiones; muchas de las cuales han permanecido polimórficas en D. starmeri (Wasserman 1962; Ruiz y Wasserman 1993; de Fblanco 1998; Prada 2002).
Estas especies se encuentran distribuidas en las zonas semiáridas del norte de Sudamérica (Colombia, Venezuela y Antillas holandesas). Se conoce que ocupan recursos diferenciados (Benado et al. 1984). D. uniseta utiliza exclusivamente el cactus columnar Ritterocereus griseus. D. martensis emerge tanto de los columnares como de opuntias; D. starmerí prefiere los columnares y ocasionalmente explota los opuntias (Ruizy Fontdevila 1981; Sánchez 1987).
En Colombia estas especies han sido detectadas en los departamentos de la Guajira, Magdalena, César y Cundinamarca (Ruiz y Fontdevila 1981; Sánchez 1987; Hoenigsberg et al. 1987). En el Hulla se coleccionaron por primera vez en el desierto de la Tatacoa, por de Folanco (1998).
En el ámbito reproductivo, Rojas (2001) confirma que todas las especies del enjambre maríensis se comportan como verdaderas. Las poblaciones geográficamente aisladas de la costa norte y el desierto de la Tatacoa tanto en D. starmerí como en D. martensis, han manifestado divergencias significativas a nivel morfológico y reproductivo (Castañeda 2000; Rojas 2001). En la especie D.starmerí la población de Santa Marta presenta mayor polimorfismo cromosómico que las poblaciones de Camarones y Rosalía (Prada 2002). Desafortunadamente en D. uniseta no se han realizado estudios comparativos entre las poblaciones de estas dos localidades.
Sin embargo, no se tiene ningún conocimiento sobre la diferenciación enzimática que presentan estas poblaciones alopátricas. En este estudio se pretendió llenar este vacío utilizando geles de almidón para inferir, en doce loci, el grado de divergencia alozímica existente entre ellas, con la intención de aportar evidencia molecular que pueda ser utilizada para descifrar su estado evolutivo.
Materiales y Métodos
MuestrasSe estudiaron tres especies del enjambre martensis (D. martensis, D. starmerí y D. uniseta), una relacionada a este enjambre, D. buzzatii y otra filogenéticamente más distante, D. melanogaster. El número de individuos promedio utilizado por especie fue de 18, coleccionados desde 1998 en las poblaciones de Santa Marta y Camarones (costa norte) y en Rosalía (vereda el "Cuzco", desierto de la Tatacoa). La población de D. melanogaster proviene de una colección realizada en el año 2001, en la localidad de Rosalía. D. buzzatii es una cepa procedente del laboratorio de genética de la Universidad de Barcelona (España) que ha sido mantenida en el Instituto de Genética, de la Universidad de Los Andes desde el año 1996.
Análisis isoenzimáticoSistemas enzimáticos. Se analizaron los siguientes sistemas: a-Esterasa (E.C.3.1.1.2.EST), a-Glicerol fosfato deshidrogenasa (E.C.1.1.1.8.-GFD), Deshidrogenasa málica, (E.C.1.1.1.37.-MDH), Aldehido oxidasa (E.C.1.2.3.1.-AO), Enzima málica (E.C. 1.1.1.40.-ME), Peptidasa-S-leucil tirosina (E.C.3.4.13.18.-PEP), Fosfoglucomutasa (E.C.5.4.2.2.-PGM), Fumarasa (E.C.4.2.1.2.-FUM), Isocitrato deshidrogenasa (E.C.1.1.1.42.-1DM), Fosfogluconato deshidrogenase (E.C. 1.1.1.44.-PGD).
Procedimiento. E
n esta investigación se compararon las variaciones de diez sistemas enzimáticos que corresponden a doce loci enzimáticos, en geles de almidón al 10%. El estudio electroforético se realizó utilizando los protocolos de corrido y tinción descritos en Sánchez (1987) y Beltrán (1999).
Análisis genético
Las frecuencias alélicas, heterocigosidad, la distancia genética e Identidad Genética de Del (1978, 1987), se calcularon con el programa PopGene versión 1.31 (Yeh et al. 1999). Las relaciones filogenéticas entre los grupos estudiados se establecieron a través del dendograma construido por este programa, que emplea el algoritmo UPGMA. Además, se calcularon los siguientes parámetros:
• el porcentaje de bandas comunes o de similitud  ,
,
• la frecuencia de alelos comunes  y comunes (C = l-Q) como se describe en Zouros (1973) y Nair et al. (1971) por parejas de poblaciones y
y comunes (C = l-Q) como se describe en Zouros (1973) y Nair et al. (1971) por parejas de poblaciones y
• el error estándar asociado a la distancia genética de Nei  , Zouros 1973)
, Zouros 1973)
Resultadosy Discusión
Variabilidad enzimática en las especies analizadas
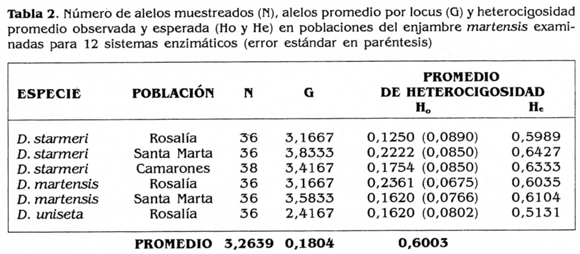
En la tabla 1 se aprecian las frecuencias alélicas de los sistemas analizados los cuales fueron todos polimórficos. Los sistemas enzimáticos que presentaron mayor número de alelos, en el enjambre martensis, son la PGD (10 alelos) y a - EST (8 alelos). D. martensis y D. uniseta exhibieron mayor variabilidad en el sistema MDH y D. starmeri en PGD.
Los valores de heterocigosidad de los loci analizados difieren significativamente de los esperados en todas las poblaciones analizadas del enjambre martensis, excepto para las enzimas Isocitrato deshidrogenasa (D. starmeri - Santa Marta, X2= 5,48; df = 3; P< 0,05) y Malato deshidrogenasa (D. uniseta - Rosalía, X2= 3,31; df= 1; P< 0,05). Los valores de heterocigosidad promedio observada para cada sistema son una tercera parte del valor esperado en todas las poblaciones estudiadas (Tabla 2). Aunque este fenómeno es típico en muchas especies de Drosophila (Ayala y Tracey 1974; Sánchez 1987), se debe tener en cuenta que el número de individuos es relativamente bajo (x = 18) y que no han sido recientemente coleccionados, por tanto, en el laboratorio estas poblaciones han podido sufrir sucesivos cuellos de botella.
Las especies del enjambre martensis presentaron un valor medio de alelos por locus de 3,2 y una heterocigosidad media de 0,1804. El valor medio más alto de alelos por locus lo manifestó D. starmerí (3,8) y el más bajo lo mostró D. uniseta con 2,4 (Tabla 2). Las poblaciones de D. martensis presentan, en general, valores de variabilidad análogos a los de las poblaciones de D. starmerí y también mayores que los de D. unisefa (especie monófoga). En cierta manera este resultado puede estar reflejando la especificidad de nicho que presenta D. uniseta, en contraste con D. starmerí y D. martensis que son especies polífogas (Barker y Mulley 1976); quizás esta diferenciación enzimática sea una insinuación de la importancia del sustrato utilizado por cada una de las especies.
Las poblaciones de D. starmerí de la costa norte (Santa Marta y Camarones) presentaron un valor medio de heterocigosidad mayor (0,1988) que la población de Rosa-lía en el desierto de la Tatacoa (0,1250). En la especie de D. martensis se observó lo contrario, un valor de heterocigosidad mayor en la población de Rosalía que en la población de Santa Marta. Estos resultados reflejan parte de la diferenciación genética que existe entre estas poblaciones alopátricas.
Niveles de identidad intra e inter específico
Identidad genética de Nei (1978, 1987). A partir de los valores de frecuencias alélicas se estimó el nivel de divergencia (Mei 1978, 1987) en las especies estudiadas. El programa PopGene arrojó la matriz de identidad, resumida en tabla 3. Las especies analizadas en este estudio presentaron una diferenciación enzimática que se ajusta al grado de divergencia taxonómica. De tal manera que se observan valores de identidades genéticas de Nei (1978,1987) que van desde 0,902 hasta 0,1537 (Ī = 0,4434). Las poblaciones de D. starmeri de Santa Marta y Camarones (costa norte) muestran el valor más alto de identidad genética (0,902), al comparar estas poblaciones con la de Rosalía (desierto de Tatacoa), la identidad promedio se disminuye notablemente (Ī = 0,8503). La divergencia es menor en Camarones (0,8712) que en Santa Marta (0,8295). D. martensis mostró en estas poblaciones un valor de 0,8654.
Las comparaciones entre las especies del enjambre martensis (D. martensis, D. starmeri y D. uniseta) aún cuando son morfológicamente similares, dieron un buen índice de divergencia genética entre ellas (Ī = 0,4559), el cual se reduce a 0,3682 cuando se comparan especies de enjambres diferentes (D. buzzatii vs. especies del enjambre martensis) y como es esperado, la identidad más baja se da entre las especies morfológicamente más diferenciadas 0,2462 (D. melanogaster vs. las demás). Los valores de identidad genética entre las poblaciones se pueden observar más fácilmente en la figura 1. En donde se destaca, en color negro, el valor de la identidad genética en las poblaciones de la costa norte y en gris las poblaciones de las distintas zonas geográficas para las especies de D. martensis y D. starmeri.
Porcentaje de similitud (S). Los porcentajes promedios de bandas comunes o de similitud entre las poblaciones estudiadas se calcularon utilizando la ecuación de Nair et al. (1971). Al comparar todas las poblaciones geográficamente aisladas de D. starmeri y D. martensis se encontró que solo comparten un 78,9% de bandas comunes, denotando que son unidades diferentes. Detallando las diferencias por especie se observó que S decrece de la siguiente manera: 84,44%, entre las poblaciones de Santa Marta y Rosalía (D. martensis), 83,33% entre las poblaciones de Santa Marta y Camarones (D. starmeri), 76,09% entre Santa Marta y Rosalía (D. starmeri) y 71,74% entre Camarones y Rosalía(D. starmeri).
Los resultados también permitieron observar que el valor de similitud, como es lógico, disminuye de acuerdo con el nivel evolutivo: 35,75% al comparar especies del mismo enjambre, 23,18% especies de diferentes enjambres y 17,31% especies de grupos diferentes. De manera coherente, los porcentajes de S se aproximan a los valores obtenidos de las identidades genéticas.
Niveles de diferenciación genética intra e ínter específica
Distancia genética de Nei (1978, 1987). En la tabla 3 se muestran los valores de distancia genética con sus errores estándar y las identidades genéticas. La distancia menor corresponde a las comparaciones realizadas entre poblaciones de una misma especie: 0,1031 (D. starmeri, Santa Marta - Camarones), 0,1870 (D. starmeri, Santa Marta - Rosalía), 0,1379 (D. starmeri, Camarones - Rosalía) y 0,1446 (D. martensis, Santa Marta - Rosalía). La divergencia entre los taxa es congruente con un aumento de los valores de distancia: 0,7907 especies del mismo enjambre, 1,0055 entre diferentes enjambres y 1,4327 entre especies de grupos diferentes.
Frecuencia promedio de alelos comunes (C) y no comunes (Q). En la figura 2 se ilustra la frecuencia de alelos comunes (C) y no comunes (Q). En la cual se puede apreciar la tendencia inversa de estos parámetros, mientras C decrece, Q aumenta de acuerdo con la divergencia taxonómica. En las especies del enjambre martensis los valores más altos de C lo exhibieron las poblaciones de una misma especie. En D. starmeri las poblaciones de la costa norte (Santa Marta y Camarones) presentaron un valor de C = 0,9474, el cual se disminuye a 0,9168 entre las poblaciones alopátricas Camarones - Rosalía y a 0,9097 entre Santa Marta - Rosalía. Contrariamente, las poblaciones de D. martensis presentaron valores similares a los de la costa norte. El valor de C se disminuye sensiblemente cuando las comparaciones son ínter específicas: 0,563 especies del enjambre martensis, 0,4161 especies de distintos enjambres y 0,3647 especies de grupos diferentes.
DendogramaEn la topología del árbol generado, se evidencia el valor sistemático de los resultados electroforéticos, los cuales permiten separar claramente las distintas especies. Por un lado el enjambre martensis aparece relacionado con la especie evolutivamente más cercana, D. buzzatii y como la más distante del grupo surge D. melanogaster. Las especies del enjambre maríensis se pueden diferenciar visiblemente en dos grupos, uno reúne las poblaciones de D. starmeri y otro está conformado por D. martensis y D. uniseta. Además, es apreciable la separación en el árbol, entre las poblaciones de la costa norte y el desierto de la Tatacoa, tanto para D, martensis como D. starmeri. Contrariamente, las poblaciones de Santa Marta y Camarones (costa norte) en D. starmeri aparecen agrupadas (Fig. 3 a).
Las relaciones evolutivas insinuadas anteriormente podrían ser desvirtuadas sí la tasa de cambio evolutivo, a la cual se ha dado la alta variabilidad observada en el segundo cromosoma de estas especies, no es constante (Sánchez 1987; Beltrán et al. 1995; de Polanco 1998). Por tal motivo se realizó un fenograma excluyendo los loci ubicados en este cromosoma (Fig. 3 b). La topología del árbol generado cambió la relación de D. buzzatii agrupándola con las especies del enjambre martensis y relacionándola más con D. martensis, y no con D. starmeri como se observó al incluir to dos los loci (Fig. 3 a). Vale la pena destacar que las relaciones de las poblaciones alopátricas no se alteran ni en D. starmeri ni en D. martensis. Este resultado insinúa que los loci que determinan estas diferencias también se encuentran en otros cromosomas.
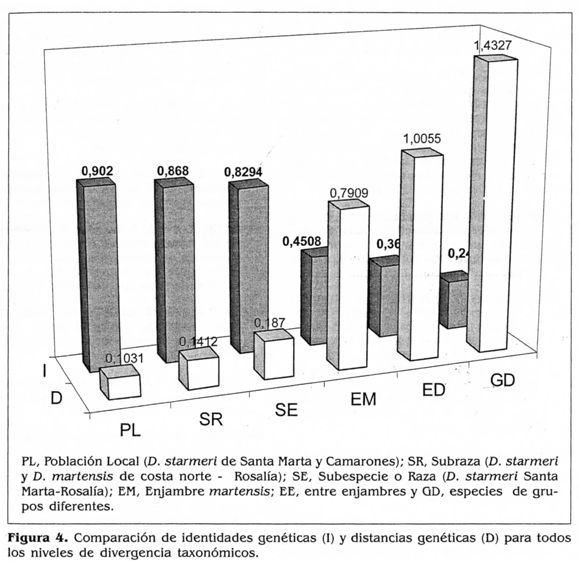
Los altos valores de errores estándar de las distancias genéticas pueden estar asociados tanto al número bajo de muestreo como de loci analizados. El análisis de las divergencias genéticas de las poblaciones alopátricas en estudio se confrontó con la información de diferenciación a nivel morfométrico, reproductivo y citogenético, que éstas han manifestado (de Polanco 1998; Castañeda 2000; Rojas 2001; Prada 2002). Con la matriz de distancia genética de Mei se sugieren los siguientes seis niveles de diferenciación, que se ajustaron al grado de divergencia taxonómica (Tabla 3).
Población local. El par de poblaciones Santa Marta y Camarones (costa norte), en la especie D. starmeri exhibió una distancia genética propia de poblaciones locales (D = 0,1031). Aunque es ligeramente superior a la informada por Sánchez (1987) para las poblaciones locales venezolanas de la misma especie (D = 0,069). Esta diferencia puede ser un reflejo de procesos ecológicos propios de cada localidad. Castañeda (2000) comprobó que las poblaciones de la costa norte tienen su propia estructura morfométrica, pero conservan su identidad como especie.
Subrazas. En D. starmeri y D. martensis las comparaciones entre las poblaciones de la costa norte y el desierto de la Tatacoa mostraron un valor promedio de distancia genética de 0,1412 ± 0,002. Es decir, se han acumulado en el tiempo 14 sustituciones alélicas por cada cien loci; valor que es 1,4 veces superior a la divergencia manifestada en D. starmeri en las poblaciones locales de la costa norte. Esta divergencia es mayor en 0. martensis (0,1446) que en D. starmeri (0,1379). La magnitud de esta divergencia cae dentro del intervalo encontrado para subespecies por otros autores (Ayala et al. 1972; Zouros 1973; Sánchez 1987). Sin embargo, cuando se comparan los promedios de las frecuencias de átelos no comunes, se observa que tales poblaciones (Q =0,0679), no difieren sustancialmente de lo detectado en D. starmeri para poblaciones locales (Q = 0,0509). Zouros (1973) tampoco encontró diferencias entre las poblaciones locales (0,0016) y subrazas (0,002) del grupo mulleri. Por lo tanto, se considera que las poblaciones de D. starmeri y D. martensis de estas zonas alopátricas posiblemente presentan una divergencia típica de subrazas.
Entre las poblaciones de la costa norte y el desierto de la Tatacpa de las especies anteriormente mencionadas, también se ha evidenciado diferenciación a otros niveles. Rojas (2001), al realizar cruces circulares entre las poblaciones de Santa Marta, Camarones y Rosalía, observó que la viabilidad de los híbridos de la F1 (en las dos vías) y la descendencia de los retrocruces disminuyó significativamente tanto en D. starmeri como en D. martensis, siendo este efecto más acentuado en esta última especie; sin embargo, esta diferenciación no se reflejó a nivel alozímico para D. martensis.
No obstante para poder inferir adecuadamente al respecto es necesario analizar un mayor número de loci y de individuos. Además, Castañeda (2000) entre las poblaciones de D. starmeri de estas dos zonas geográficas evidenció que son morfológicamente diferentes (D. mahalanobis = 13,383).
Subespecie o raza. El valor de distancia genética más alto entre estas poblaciones alopátricas se detectó en D. starmeri, para las poblaciones de Santa Marta y Rosalía (0,1870). El cual es análogo al informado por otros autores para la categoría de subespecie (Hubby y Throckmorton 1965; Ayala et al. 1972; Yang et al. 1972; Zouros 1973; Sánchez 1987). Es decir, han ocurrido aproximadamente 19 sustituciones alélicas, acumuladas en el tiempo, por cada 100 loci entre estas poblaciones; divergencia que es aproximadamente 2,8 veces más grande que la distancia que exhibieron las poblaciones locales venezolanas, de la misma especie y 1,8 más grande que la población local de la costa norte.
Estas poblaciones manifestaron una reducción significativa de la descendencia en la F1 (Rojas 2001). En la población de Santa Marta, Prada (2002) observó un mayor polimorfismo y la presencia de heterocariotipos en híbridos cuando realizó cruces entre esta población y Rosalía. Además, Castañeda (2000) determinó morfométricamente, mediante distancias de Mahalanobis, que son diferentes (D.M. = 14,021). Aunque a nivel de aislamiento precigótico de selección múltiple, Pérez (2002) no encontró diferencias significativas entre estas poblaciones, sí se manifestó una tendencia hacia el reconocimiento de individuos de una misma localidad. Se denota que el aislamiento reproductor entre estas poblaciones es aún insuficiente, pero que ha comenzado a insinuarse. Por lo anteriormente mencionado se podría sugerir que dichas poblaciones están iniciando la primera etapa de especiación
Enjambre martensis.Las distancias genéticas entre las especies del enjambre martensis (D. martensis. D. starmeri y D. uniseta) exhibieron valores que oscilan entre 0,6387 y 0,9777, con un valor medio de D = 0,7907±0,0315. Sánchez (1987), para estas mismas especies, analizando 22 sistemas enzimáticos, encontró valores de divergencia que fluctúan entre 0,647 y 0,840, con un valor medio de 0,7225. Este nivel de diferenciación cae dentro del intervalo de distribución observado en otros enjambres de grupos diferentes (Ayala y Tracey 1974; Eisses et al. 1979; Cabrera et al. 1980; Cabrera et al. 1983).
Enjambres o clusters diferentes. El nivel de divergencia genética entre las poblaciones del enjambre martensis y D. buzzatii (D =1,0055±0,0366), se incrementó con respecto a lo obtenido para el enjambre martensis. se incrementó con respecto a lo obtenido para el enjambre martensis. Este resultado es concordante con los referenciados por Ayala y Tracey (1974) y Eisses et al. (1979) para enjambres del grupo willístoni y melanogaster.
Especies de grupos diferentes. La divergencia genética entre especies de grupos diferentes, como es lógico, manifiesta los valores más altos de distancia genética (Cabrera et al. 1980; Cabrera et al. 1983).
Hecho que se confirmó en este estudio al comparar D. melanogaster con las poblaciones de D. martensis, D. starmeri, D. uniseta, y D. buzzatü, las cuales presentaron un valor medio de 1,4327 ± 0,0934.
En la figura 4 se grafican las identidades y distancias entre los diferentes niveles taxonómicos detectados. Se aprecia un incremento gradual, desde el nivel de población local hasta el nivel de especies completamente diferenciadas. Las comparaciones entre las frecuencias de alelos comunes (C) y no comunes (Q), en los distintos niveles taxonómicos, se ajusta a lo encontrado por Zouros (1973) en especies del enjambre mullen. En la figura 2 se muestra la relación inversa de Q y C para los diferentes niveles de divergencia.
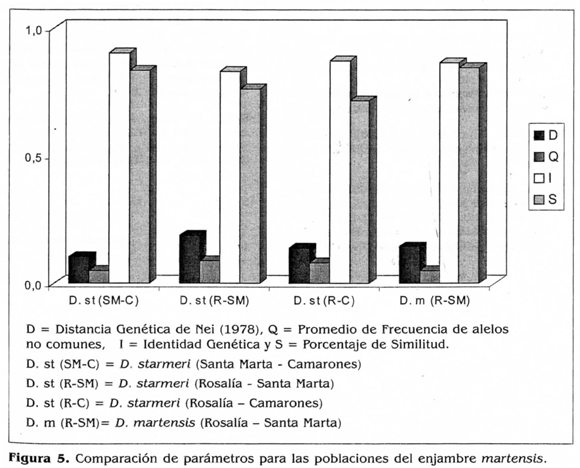
En la figura 5 se grafican todos los parámetros analizados en este estudio para las especies del enjambre martensis, en la cual se puede apreciar que D. starmeri fue la especie más divergente. En esta especie el par de poblaciones Rosalía - Santa Marta (R-SM) presenta mayor diferenciación respecto al par de poblaciones locales, de Santa Marta y Camarones (SM-C).
Conclusiones
• Las especies del enjambre martensis (D. martensis, D. starmeri y D. uniseta) exhibieron bajos valores de heterocigosidad, fenómeno ya evidenciado en otras especies cactofílicas del grupo repleta.
• El análisis de los doce loci enzímáticos permitió sugerir seis niveles de divergencia taxonómica entre las especies: Población local (D =.0,1031), subrazas (D = 0,1412), subespecies o razas (£> = 0,1870), especies de un mismo enjambre o cluster (= 0,79£7), especies de enjambres diferentes (D =JL,0055) y especies de grupos diferentes (D = 1,4327).
• El dendograma construido, a partir de la matriz de distancias genéticas, separó acertadamente las especies analizadas de acuerdo con su divergencia evolutiva.
• Con esta investigación se pudo establecer que en D. starmeri las poblaciones geográficamente más cercanas (costa norte) no se diferenciaron alozímicamente, mientras que las poblaciones más distantes (costa norte y desierto de la Tatacoa) tanto en D. starmeri como en D. martensis, son diferentes alozímicamente. La divergencia más contundente se manifestó en starmeri entre las poblaciones de Santa Marta y Rosalía. Así en este estudio preliminar se sugiere un posible ejemplo de especiación alopátrica incipiente.
Recomendaciones
Realizar un análisis más riguroso ampliando tanto el número de individuos (los cuales deben ser recién coleccionados) cómo el número de loci analizados (mínimo 24), para evaluar el estado real evolutivo de las especies objeto de estudio.
Confrontar el grado de diferenciación genética obtenido a partir de las distancias genéticas de Nei, en este estudio, con otros métodos igualmente potentes como distancia de Mahalanobisy transformaciones angulares como las de Cavalli-Sforza y Eduarws.
Extender los estudios morfométricos, cromosómicos, de cortejo y selección múltiple en las poblaciones colombianas de D. martensis y D. uniseta.
Agradecimientos
Se agradece a los profesores Gonzalo Palomino y Francisco Villa que colaboraron en las salidas de campo y a los estudiantes de biología de la Universidad del Tblima, por la ayuda en el muestreo que permitió obtener los individuos objeto de estudio. Al Director del Instituto de Genética, Mauricio Linares, por permitir realizar esta investigación en el laboratorio de electroforesis de la Universidad de los Andes. A la secretaria del Instituto de Genética, Cielo Rocío de Oro por su asistencia incondicional. A los comités de investigaciones de la Universidad de los Andes (Decanatura de Ciencias - Investigaciones varias, centro de costo 280) y de la Universidad del Tolima (465) por sus aportes económicos para la realización de este proyecto.
Literatura citadaAVALA, F.; TRACEY, M. 1974. Genetic differentiation within and between species of the Drosophila willistoni group. Proceeding. Nature. Academic. Science. USA 71: 999-1003. [ Links ]
AVALA, F.; POWELL, J.; TRACEY, M.; MAURAO, C.; PÉREZ, S. 1972. Enzyme variability in the Drosophila wiUistoni group. IV Genic variation in natural population of Drosophila willistoni. Genetics 70 (1): 113-139. [ Links ]
BARKER, J.; MULLEY, J. 1976. Isozyme variation in natural populations of Drosophila buzzatii. Evolution 30: 213-233. [ Links ]
BELTRÁN, M. 1999. Evidencia genética (Alozimas), para evaluar el posible origen híbrido de Heliconius heuripa (Lepidóptera: Nymphalidae). Tesis. Magíster Ciencias Biológicas. Universidad de los Andes, Facultad de Ciencias Biológicas, Instituto de Genética. Bogotá, D. C. 112 p. [ Links ]
BELTRÁN, J.; QUEZADA-DIAZ, E.; RUIZ, A. SANTOS, M.; FONTDEVILA, A. 1995. The evolutionary history of Drosophila buzzatii. XXXII Linkage disequilibrium between allozymes and chromosome inversions in two colonizing populations. Heredity 74: 188-199. [ Links ]
BENADO, M.; FONDEVILA, A.; CERDA, H.; GARCÍA, G.; RUIZ, A.; MONTERO, C. 1984. On the distribution and the cactophilic niche of D. martensis in Venezuela. Biotropica 16: 120-124. [ Links ]
CABRERA, V. M.; GONZÁLEZ, A. M.; GULLÓN, A. 1980. Enzymatic polymorphism in Drosophila subobscwa populations from the Canary Islands. Evolution 34 (5): 875-887. [ Links ]
CABRERA, V. M.; GONZÁLEZ, A. M.; LARRUGA, J. M.; GULLÓN, A. 1983. Genetic Distance and evolutionary relationships in the Drosophila obscura group. Evolution 37 (4): 675-689. [ Links ]
CASTAÑEDA, H. 2000. Análisis morfológico comparado en Drosophila starmeri de las poblaciones de Camarones (Guajira), Santa Marta (Magdalena) y la Rosalía (desierto de la Tatacoa - Huila). Tesis. Magíster Ciencias Biológicas. Universidad de Los Andes, Facultad de Ciencias Biológicas, Instituto de Genética. Bogotá, D. C. 118 p. [ Links ]
DE POLANCO, M. M. 1998. Estudios cromosómicos comparados de Drosophila repleta (cepa de Siboney Cuba) Vs Drosophila repleta (Wharton 1942) y Drosophila martensis (Valledupar, Barrancas y Riohacha). Tesis. Doctorado Genética de Poblaciones. Universidad de Los Andes, Facultad de Ciencias Biológicas, Instituto de Genética Bogotá. 272 p. [ Links ]
EISSES, K.; DIJK. H.; DELDEN, W. 1979. Genetic differentiation within the melanogaster species group of the genus Drosophila (Sophophora). Evolution 33 (4): 1063-1068. [ Links ]
HOENIGSBERG, H. F.; MONTANO, A.; MORENO, R., SANZ DE LA ROSA, M.; ORDÓÑEZ, M.; GRIGORIA DE BUENDIA, P. 1987. Genética de poblaciones en el trópico americano XXXIV. Las enzimas diagnosticas del enjambre Martensis del grupo repleta de Drosophila de la Guajira Colombiana. Instituto de Genética, Universidad de Los Andes, Bogotá, D. E. Colombia. Evolución Biológica 1: 297-334 [ Links ]
HUBBY, J. L.; THROCKMORTON, L. 1965. Protein differences in Drosophila. II. Comparativo species genetics and evolutionary problems. Genetics 52: 203-215. [ Links ]
NAIR, Y. S.; BRNCIC, D.; ROJIMA, J. 1971. Isozyme variations and evolutionary relationships in the mesophragmatica species group of Drosophila. University Texas Publications 7103: 17-28. [ Links ]
NEI, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuáis. Genetics 89:583-590. [ Links ]
NEI, M. 1987. Molecular Evolutionary Genetic. Columbia University Press. EEUU. 125 p. [ Links ]
PATTERSON, J. T.; STONE, W. S. 1952. Evolution in the genus Drosophila. MackMillan Co., New York. 89 p. [ Links ]
PÉREZ, C. 2002. Cortejo y selección sexual entre poblaciones colombianas de Drosophila starmeri. Trabajo de grado. Bióloga. Universidad de los Andes. Facultad de Ciencias Biológicas, Bogotá, D. C. 42 p. [ Links ]
POWELL, J. 1997. Progress and prospects in evolutionary biology. The Drosophila model. Oxford University Press. New York. 562 p. [ Links ]
PRADA, C. 2002. Citogenética comparada de Drosophila starmeri (Díptera: Drosophilidae) de dos ecosistemas áridos aislados Colombianos. Trabajo de grado. Biólogo, Universidad del Tolima. Facultad de Ciencias. Ibagué. 124 p. [ Links ]
ROJAS, A. 2001. Análisis del aislamiento reproductivo en las especies cactofílicas del enjambre martensis, en dos regiones colombianas (costa norte y desierto de la Tatacoa). Tesis. Magíster Ciencias Biológicas, Universidad de los Andes. Facultad de Ciencias. Santa fe de Bogotá, D. C. 70 p. [ Links ]
RUIZ, A; FONTDEVUA, A. 1981. Ecología y evolución del subgrupo mullen de Drosophila en Venezuela y Colombia. Acta Científica Venezolana 32: 338-345. [ Links ]
RUIZ, A.; WASSERMAN, M. 1993. Evolutionary cytogenetics of the Drosophila buzzatii species complex. Heredity 70: 582-596. [ Links ]
SÁNCHEZ, A. 1987. Relaciones filogenéticos en los clusters buzzatii (Grupo repleta) y martensis de Drosophila. Tesis doctoral. Universitat Autónoma de Barcelona. Facultad de Ciéncies. Bellaterra. 458 p. [ Links ]
VILELA, C.R. 1983. A revisión of the Drosophila repleta species group (Díptera, Drosophilidae). Review Brasilian Enthomology 27(1): 1-114. [ Links ]
WASSERMAN, M. 1962. Cytological studies of the repleta group. University Texas, Publications. 5721:132-156. [ Links ]
WASSERMAN, M. 1967. Colletionsof Drosophila from Central México. Drosophila Information Service 42: 67-68. [ Links ]
YANG, S.; WHEELER, L. L.; BOCK, R. 1972. Isozyme variations and phylogenetic relationships in the Drosophila peptinata. Complex. University Texas Publications. 7213: 213-227 [ Links ]
YEH, F. C.; YANG, R.; BOYLE, T. 1999. PopGene versión 1.31. Microsoft Window- based freeware for Population Genetic Analysis. Quick user guide. http://www.ualberta.ca/~fyeh/download.htm. Fecha última revisión: septiembre 2001. Fecha ultimo acceso: Agosto 30 1999. [ Links ]
ZOUROS, E. 1973. Genic differentiation associated with the early stages of speciation in the mu/Jen subgroup of Drosophila. Evolution 27: 601-621. [ Links ]
Recibido: Ago. 22 / 2002 • Aceptado: Feb. 21 / 2003