Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Entomología
Print version ISSN 0120-0488On-line version ISSN 2665-4385
Rev. Colomb. Entomol. vol.32 no.1 Bogotá Jan./June 2006
Sección Médica
Estructura genética de Anopheles darlingi Root, An. nuneztovari Gabaldon y An. marajoara Galvão & Damasceno de Colombia mediante RAPD-PCR
Genetic structure of Anopheles darlingi Root, An. nuneztovari Gabaldon and An. marajoara Galvão & Damasceno from Colombia using RAPD-PCR
CARMEN ELISA POSSO G.1, RANULFO GONZÁLEZ O.1, HEIBER CÁRDENAS H.1 Y RICARDO TASCÓN1
1. Biólogos, Departamento de Biología, Universidad del Valle, A.A. 25362 Cali. E-mail: ceposso@univalle.edu.co, ranulfo@univalle .edu.co, heiber@univalle.edu.co.
Resumen. La variación y estructura genética de poblaciones colombianas de tres vectores de malaria fue analizada mediante RAPD-PCR. Los análisis incluyeron poblaciones de los mosquitos de diferentes áreas geográficas: (1) Para An. darlingi, poblaciones de Medio Atrato, Granada y Tierralta; (2) para An. nuneztovari, poblaciones de Buenaventura, Tieralta y Tibú y (3) para An. marajoara, poblaciones de Fuente de Oro, San Carlos de Guaroa, Yaguará y Cúcuta. Se siguieron protocolos similares de colecta con cebo humano protegido y para la extracción de ADN. La heterocigosidad esperada de las tres especies varió de 0,28 a 0,34. Las tasas de migración/generación entre poblaciones de cada especie, variaron entre 1,7 y 30,4. El AMOVA reveló poca estructura genética; entre 8,30% y 11,31% de la variación fue explicada por diferencia entre las poblaciones de cada especie. En general, las poblaciones de cada una de estas especies en Colombia presentan apareamientos al azar, con un mayor flujo de genes dependiendo del grado de separación geográfica. Los valores de FST y ΦST para An. darlingi y An. nuneztovari confirmaron mayor flujo genético entre las poblaciones del Occidente (Buenaventura, Medio Atrato y Tierralta) y para An. marajoara entre las poblaciones del Oriente (Cúcuta, Fuente de Oro y San Carlos de Guaroa).
Palabras clave: Culicidae. Diversidad genética. Marcadores moleculares. Malaria.
Abstract. The genetic variation and structure of Colombian populations of three malaria vectors were analyzed by RAPD-PCR. Analysis included mosquito populations from different geographical areas: (1) For An. darlingi, populations from Medio Atrato, Granada and Tierralta; (2) for An. nuneztovari, populations from Buenaventura, Tierralta and Tibú, and (3) for An. marajoara, populations from Fuente de Oro, San Carlos de Guaroa, Yaguará and Cúcuta. Similar collection protocols were followed for using protected human bait and for DNA extraction. The expected heterozygosity of the three species varied from 0,28 to 0,34. The migration/generation rates among populations of each species varied between 1,7 and 30,4. The AMOVA values revealed little genetic structure; between 8,30 and 11,31% of the variation was explained by differences between populations of each species. In general, the populations of each of these species in Colombia display random mating, with a greater gene flow according to degree of geographical separation. FST and ΦST values for An. darlingi and An. nuneztovari confirmed that the higher genetic flow occurred among western populations (Buenaventura, Medio Atrato, and Tierralta) and for An. marajoara among the eastern populations (Cúcuta, Fuente de Oro, and San Carlos de Guaroa).
Key words: Culicidae. Genetic diversity. Molecular markers. Malaria.
Introducción
La malaria, una de las enfermedades de mayor importancia en salud pública en América Tropical y el mundo, produce trastornos a la salud y puede ocasionar la muerte sino se trata adecuadamente. En Colombia la malaria representa un grave problema, estimando que 18 millones de personas se encuentran en riesgo de enfermar o morir por esta causa (OPS/OMS 2000). La malaria es transmitida por vectores del género Anopheles (Fleming 1986) entre los que figuran las especies objeto de estudio.
Los programas para el control de los vectores de malaria, requieren un conocimiento exacto de la determinación taxonómica de éstos, ya que se registran en ellos, diferencias en su ecología, capacidad vectorial y respuesta a las medidas de control. Estudios taxonómicos registran en Colombia 41 especies de anofelinos incluido el subgénero Nyssor-hynchus (Suarez et al. 1987), al cual pertenecen las principales especies vectores primarios de malaria en nuestro país. Estos vectores se caracterizan por presentar variaciones en la actividad de picadura hematófaga y en el comportamiento de picadura y de reposo post-hematofágico, sumado a un polimorfismo fenotípico, lo cual dificulta y podría ocasionar orientaciones erróneas en las medidas de control, por lo cual la importancia de una correcta determinación de las especies vectoras es indispensable especialmente en lugares donde ocurren especies morfológicamente similares que difieren en su desarrollo y ecología. Esta determinación taxonómica se dificulta cuando se presentan complejos de especies enmascarados en especies isomórficas con importancia epidemiológica variable. Tal es el caso de Anophe-les albitarsis, un complejo conformado por cuatro especies (An. albitarsis s.s., An. deaneorum, An. marajoara y la especie C aún sin describir) (Wilkerson et al. 1995) ó por variaciones citotípicas como es el caso de An. nuneztovari para la que se reportan tres citotipos (A, B y C), (Conn et al. 1993, 1998).
El entendimiento de la variación genética en poblaciones vectoras, ofrece la base para el entendimiento del papel de estos en la epidemiología de enfermedades. Los factores que conforman la variación genética en una especie vectora también influyen en su capacidad y competencia vectorial (Tabachnick y Black IV, 1996).
Para propósitos taxonómicos, sistemáticos y de poblaciones, el uso de marcadores morfológicos ha sido reforzado y en muchos casos reemplazado por los marcadores de tipo molecular, herramientas muy útiles para analizar individuos de diferentes grupos naturales. La aplicación de técnicas moleculares ha permitido nuevas interpretaciones en la sistemática de vectores de enfermedades y ha estimulado el descubrimiento y caracterización de complejos de especies crípticas (Munstermann y Conn 1997). Los parámetros de estructura genética obtenidos a partir de un marcador molecular permiten además, determinar variabilidad genética inter ó intrapoblacional y ayudan al entendimiento de la variación en el comportamiento hematofágico de las especies vectoras.
Los marcadores moleculares RAPD-PCR generan información que podrían diferenciar subespecies ó especies crípticas; aunque con limitaciones, como ser considerados un carácter dominante y su baja reproducibilidad, han sido utilizados invariablemente por diferentes investigadores de vectores para estudios taxonómicos, relaciones genéticas entre poblaciones y para explicar el papel epidemiológico de diferentes poblaciones geográficas de una especie (Ferreira y Grattapaglia 1998). Para definir el estatus taxonómico y/o estructura genética en cada una de las siguientes especies vectoras: An. darlingi, An. nuneztovari y An. marajoara en Colombia, se utilizó este marcador. Se determinó dentro de cada una de las especies, su diversidad genética y flujo de genes entre las poblaciones estudiadas.
Materiales y Métodos
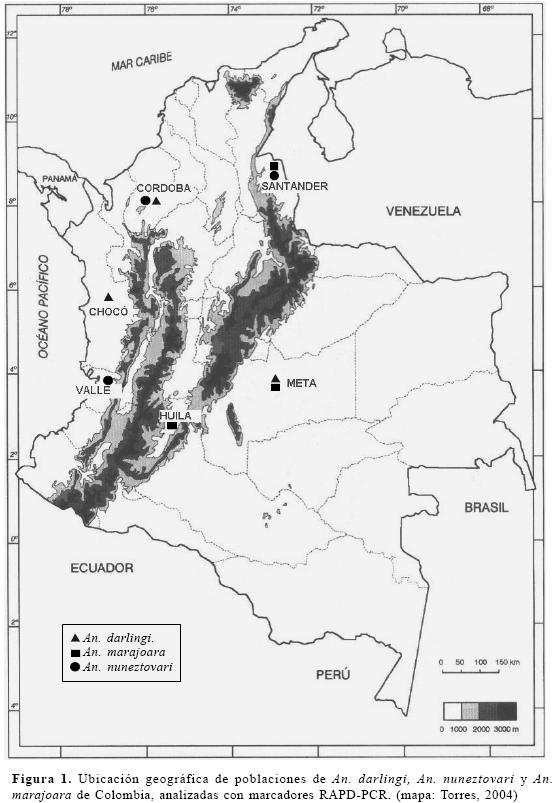
Colección de mosquitos. Para cada especie se seleccionaron mosquitos de zonas geográficas de Colombia donde se constituyen vectores primarios de malaria, considerando en este estudio como poblaciones, las localidades donde se efectuaron las colectas. Para An. darlingi, el municipio de Tierralta (8010´N, 76004´W), Departamento de Córdoba; el municipio de Granada (3032´N, 73043´ W), Departamento del Meta y el municipio de Medio Atrato (5041´N, 76040´W), en el Departamento del Chocó. Los especimenes de An. nuneztovari se colectaron en el municipio de Tierralta; en el municipio de Tibú (8039´N, 72059´W) en Norte de Santander y el municipio de Buenaventura (3054´N, 77005´W) en el Departamento del Valle. Para An. mara-joara el municipio de Cúcuta (7054´N, 72030´W) en el Departamento de Norte de Santander; los municipios Fuente de Oro (3028N, 73038´W) y San Carlos de Guaroa (3043N, 73015´W) en el Departamento del Meta, y el municipio de Yaguará (2040N, 75031´W) en el Departamento del Huila (Fig. 1).
La recolección de los mosquitos se realizó mediante capturas con cebo humano protegido. Los insectos se llevaron vivos al laboratorio de Entomología de la Universidad del Valle para la identificación de las especies por medio de caracteres morfológicos (Faran 1980; Delgado y Rubio-Palis 1993; Linthicum 1988) y preservados individualmente a –200C en tubos de microcentrifuga de 1,5ml con etanol al 100%.
Extracción de ADN y condiciones del RAPD-PCR. Se realizó en los laboratorios de Biotecnología del Centro Internacional de Agricultura Tropical, CIAT (An. darlingi y An. nuneztovari) y el laboratorio del Instituto de Inmunología de la Universidad del Valle (An. mara-joara). Para la extracción del ADN se utilizó el método de Coen et al. (1982) con las modificaciones de Pat Romans (Black IV y Du Teau 1997). Con el propósito de lograr patrones de bandas bien definidas, consistentes y reproducibles se estandarizaron las condiciones del RAPD-PCR para cada uno de las especies, obteniendo para An. darlingi y An. nuneztovari: 1ng/µl de ADN, Buffer PCR 1X, 2,5mM de MgCl2, 0,2mM de oligonucleótidos y 1U de taq polimerasa, ajustados a 25 µl con agua destilada ultrafiltrada. Para An. marajoara las condiciones variaron, utilizando 5ng/µl de ADN, Buffer PCR 1X, 2,0mM de MgCl2, 0,1mM de oligonu-cleótidos y 0,5U de taq polimerasa, ajustados a 25 µl con agua destilada ultrafiltrada. Los productos de amplificación se visualizaron en geles de agarosa al 1,5% de acuerdo con el método de Sambrook et al. (1989). Los geles se corrieron a 300V durante una hora aproximadamente. En cada gel se utilizó una muestra del fago l digerido con la enzima PstI como referencia para la determinación de los tamaños moleculares aproximados de los productos de amplificación. Las bandas se visualizaron con bromuro de etidio (0,8µg/ml). Los geles y patrones de bandas se visualizaron y registraron en un equipo Eagleye II (Stratagene).
Selección de oligonucleótidos polimór-ficos. Se utilizaron cebadores de Operon, Technologies®, (Alameda California). La selección de los oligonucleótidos poli-mórficos se hizo a partir de 70 y 540 cebadores para An. darlingi y An. nunez-tovari, respectivamente. Para An. mara-joara se utilizaron los cuatro cebadores que produjeron marcadores diagnósticos para la especie C del complejo An. albitarsis s.l. (Wilkerson 1995). Para descartar artefactos de la técnica y verificar la producción de un proceso de amplificación confiable, se emplearon controles negativos (muestra sin ADN) y positivos (la misma muestra de ADN en todos los geles), para cada población y especie. Seis oligonucleótidos fueron seleccionados para evaluar 64 individuos de An. darlingi (14 de Tierralta, 24 de Medio Atrato y 26 de Granada); diez cebadores fueron empleados para evaluar 119 individuos de An. nuneztovari (42 de Buenaventura, 39 de Tierralta y 38 de Tibú). (Posso et al. 2003). Para An. marajoara se evaluaron 99 individuos (45 de Cúcuta, 42 de Fuente de Oro y San Carlos de Guaroa y 12 de Yaguará).
Análisis de los fragmentos RAPD-PCR.
Análisis taxonómico. Mediante un análisis independiente, para cada especie se derivó una matriz de distancia (1-M) generada con el programa RAPDPLOT de Black IV (1995) esta matriz se estima de la fracción de emparejamientos (M), mediante la fórmula M = NAB /NT, donde NAB es el número total de emparejamientos entre los individuos A y B (tanto para las bandas ausentes ó presentes) y NT el número total de loci (fragmentos) del estudio. De esta matriz se derivó otra de distancias (1- M) la cual fue analizada con el programa Neighbor en PHYLIP 3.5C (Felsenstein 1993) y se generó un dendrograma con el método UPGMA. Con el propósito de analizar la consistencia con la cual el dataset RAPD de cada una de las especies soportaba la relación fenética entre las posibles taxa, se realizaron tres análisis bootstrap de 100 pseudoréplicas, utilizando el programa RAPDBOOT 1.0 (Black IV 1996). Con estos se generaron los respectivos juegos de 100 matrices para cada especie, que fueron leídas por PHYLIP 3.5C Cada matriz del juego fue colapsada utilizando el algoritmo de Neighbor para generar un juego de dendrogramas a partir de los cuales, por medio de la opción consenso estricto y regla de la mayoría, se obtuvieron los respectivos árboles consenso de cada especie.
Análisis genético. Las distancias genéticas entre las poblaciones de cada una de las especies, fueron analizadas con el programa RAPDDIST 1.0 (Black IV 1997). A partir de cada juego de datos específicos RAPD se generó una matriz de distancia (1-S) (similitud de Nei 1972); S = 2NAB /(NA + NB), donde NAB es el número de fragmentos que los individuos A y B comparten en común, NA es el número de fragmentos del individuo A y NB el número de fragmentos del individuo B. Los valores fueron corregidos por el factor de Lynch y Milligan (1994). La consistencia con la cual el juego de datos RAPD de cada especie, soportaba las relaciones estimadas entre las poblaciones, también fue evaluada por medio de un bootstrap de 100 pseudoréplicas, como fue descrito en el análisis taxonómico.
La estructura genética de las poblaciones de cada una de las especies, se estudió mediante el cálculo de heterocigosidad, los estadísticos FST, las tasas de migración/generación y el análisis molecular de varianza (AMOVA). Para el cálculo de la heterocigosidad se utilizaron dos métodos: el tradicional de raíz cuadrada de la frecuencia del genotipo recesivo, asumiendo Hardy-Weinberg (Apostol et al. 1996) y el método de Lynch y Milligan (1994). Para realizar este cálculo, se asumió: primero, que las regiones genómicas amplificadas por RAPD-PCR segregan como alelos dominantes; segundo, que los genotipos en los loci RAPD están en equilibrio Hardy-Weinberg; tercero, la identidad en estado entre alelos dominantes amplificados y cuarto, la identidad en estado entre alelos nulos. Utilizando los cuatro supuestos, se estimó qj(i), la frecuencia de alelos nulos a en el locus i en la población j.
Para analizar las tasas de migración por generación entre las poblaciones de cada vector estudiado, se calculó el estadístico FST de Wright (1951). El estadístico FST es la proporción entre la varianza observada (S2) de la frecuencia de un alelo entre las poblaciones y su varianza máxima en la población total. Este estadístico toma los valores extremos de cero (0) cuando todas las poblaciones tienen frecuencias genéticas idénticas y uno (1) cuando la población tiene fijados alelos diferentes, reflejando diferenciación poblacional completa. Los cálculos se llevaron a cabo a partir de las matrices utilizadas en el análisis de agrupamien-to, mediante el programa RAPDFST 4.01 (Black IV 1997). La tasa de migración por generación (Nm) entre poblaciones fue estimada de FST, Wright, Theta (q) y Lynch y Milligan, asumiendo un modelo de islas, mediante la fórmula Nm = (1-FST)/4FST. El valor de Nm es el producto de un tamaño de población (N) poten-cialmente grande y la tasa de migración (m) potencialmente pequeña.
La distancia genética entre los individuos de las poblaciones de cada especie se midió también como una distancia métrica euclidiana con base en el número total de bandas polimórficas y el número de bandas compartidas. Los archivos se crearon con el programa AMOVA-PREP 1.01 (Miller 1998) diseñado para ser utilizado únicamente con marcadores dominantes como los RAPDs. Con el programa WINAMOVA, versión 1.5 (Excoffier et al. 1992), se realizó el análisis de varianza molecular (AMOVA) con el cual se estimaron los componentes de las varianzas internas y se calcularon los valores de las estadísticas FIS con el término análogo ΦST. El valor de ΦST estima el grado de estructura de la población.
Resultados y Discusión
Análisis de los fragmentos RAPD-PCR.
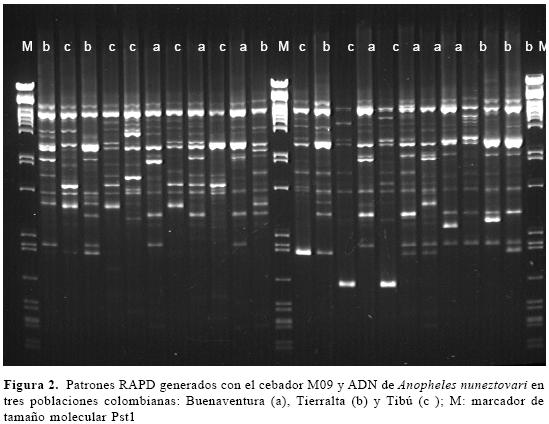
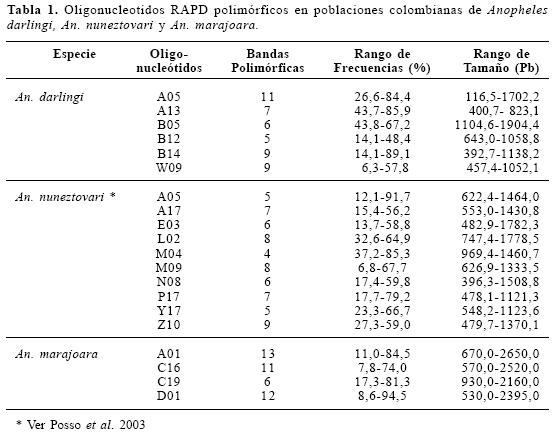
Los seis oligonucleótidos polimórficos para An. darlingi produjeron 46 bandas con frecuencias que oscilaron entre 6,3 y 85,9%. Para establecer este rango de frecuencias se consideró el criterio de restricción de Lynch y Milligan (1994), que permite realizar estimados no sesgados de los parámetros genéticos. La banda de mayor tamaño molecular presentó 1904,4 pares de bases y la de menor tamaño registró 116,5 pb (Tabla 1). Para An. nuneztovari los 10 cebadores que resultaron polimórficos produjeron 65 bandas cuyas frecuencias oscilaron entre 6,8% y 91,7%. La banda de mayor tamaño molecular presentó 1782,3 pares de bases y la de menor tamaño registró 396,3 (Tabla 1). El patrón de amplificación generado por uno de los oligonucleótidos del estudio (M09), se presenta en la figura 2. Para An. marajoara los cuatro cebadores diagnósticos de la especie C de An. albitarsis s. l. produjeron 42 bandas polimórficas, con frecuencias entre 7,8 y 94,5% y tamaños moleculares entre 530,0 y 2650,0 pares de bases (Tabla 1).
En todos los casos y para cada especie analizada, las diferencias en la frecuencias de bandas entre y dentro de los oligonucleótidos evaluados para cada población, indicaron que aunque los individuos pertenecen a la misma población, presentaron diferencias en el patrón RAPD.
Los cuatro cebadores que produjeron marcadores diagnósticos para la especie C del complejo An. albitarsis s. l. en los trabajos de Wilkerson et al. (1995), fueron reproducidos con éxito en este estudio y permitieron establecer que en Colombia, está presente únicamente An. marajoara, descartando las otras tres especies del complejo (An. albitarsis s.s., An. daeneorum y An. albitarsis sp. B). Este resultado adquiere más importancia si se tiene en cuenta que An. marajoara, presente también en el noreste de la Amazonía en Brasil, después de considerarse como especie vectora de menor importancia en la zona, se convirtió en vector principal de malaria, presentando además abundancia poblacional superior al vector considerado primario en la zona, An Darlingi (Conn et al. 2002). De acuerdo con estos resultados se hace necesario realizar un análisis poblacional de estas especies en Colombia, en zonas donde se encuentren simpátricamente y estudiar en ellas, el efecto de factores tales como: migración humana y/o cambios en el uso del suelo, mencionados por Conn et al. (2002) como causantes del cambio en la importancia vectora de estas dos especies en la zona amazónica de Brasil.
El análisis taxonómico realizado con RAPDPLOT entre los individuos y su respectivo Bootstrapping, para cada una de las tres especies, determinó que la constitución genética promedio de los individuos de las diferentes poblaciones no varió con respecto a la de la población total, es decir que no se encuentran sub-divididas en unidades que difieran genéticamente (Chakraborty y Leimar 1987). A partir de los loci RAPD-PCR de cada especie no se observó una clara separación genética entre las poblaciones y se mostraron claramente conespecíficas (Conglomerados no presentados). En los dendrogramas consenso resultantes de cada especie, las ramas formadas contenían individuos de diferentes poblaciones de cada especie, y por otro lado no fueron significativamente diferentes para un gran número de loci. Estos resultados implican que en Colombia no se registran hasta el presente, complejos en ninguna de las tres especies analizadas.
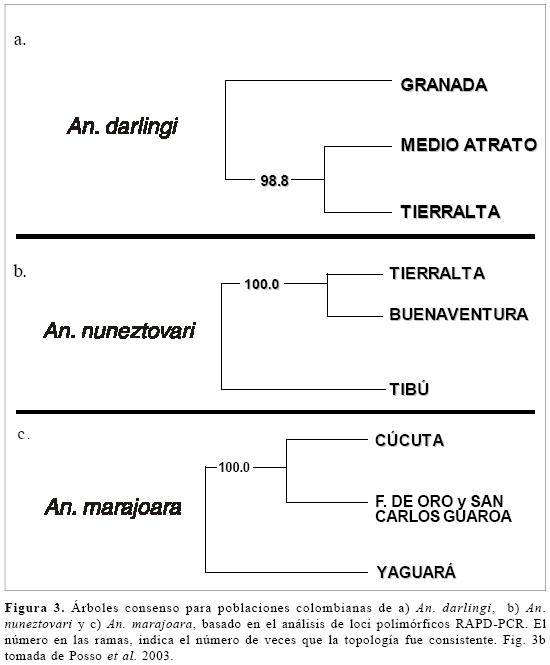
Análisis genético. A pesar de la conespecifidad observada, al computar las distancias genéticas de Nei entre las poblaciones (análisis con el programa RAPDDIST), la frecuencia de alelos de las poblaciones de cada una de las especies, analizadas por separado, fue consistentemente diferente para un gran número de loci ya que el boostrap que soporta las ramas derivadas fue mayor que el 90% (Fig. 3), es decir que a pesar de no ser especies claramente estructuradas, existe una diferenciación genética, explicada por un gradiente de separación geográfica. En An. darlingi y An. nunez-tovari, el árbol consenso estricto determinó un mayor parecido (genético) entre las dos poblaciones del Occidente, mientras que en An. marajoara la rama derivada relacionó a las poblaciones del oriente (Figs. 3a y 3b). En An darlingi, Medio Atrato (Chocó) y Tierralta (Córdoba) conformaron un agrupamiento significativamente separado de Granada (Meta) (Fig. 3a). Para An. nuneztovari la mayor relación genética se observó entre las poblaciones de Buenaventura (Valle) y Tierralta, claramente diferenciada de Tibú (N. De Santander) (Fig. 3b). Para An. marajoara la rama derivada agrupó a las poblaciones de Cúcuta y Meta, diferenciada significativamente de la población de Yaguara (Huila), (Fig. 3c). Los análisis genéticos siguientes soportan estas observaciones.
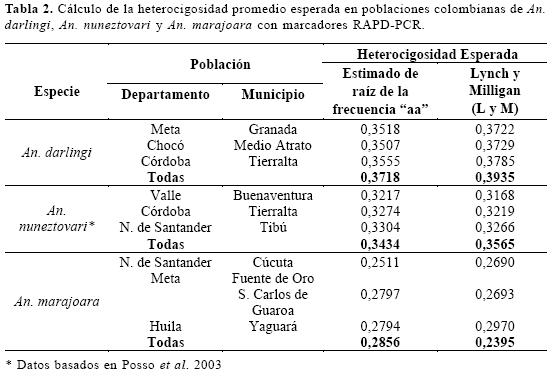
La heterocigosidad promedio esperada, calculada para cada vector con el método estimado de la raíz de la frecuencia de homocigotos recesivos (Tabla 2), indicó para las poblaciones de An. darlingi y An. nuneztovari un alto polimorfismo genético de loci-RAPD, con valores similares para cada población de cada vector y una heterocigosidad total de 0,3718 en el caso de An. darlingi y 0,3434 para An. nuneztovari . Para An. marajoara, se obtuvo un polimorfismo genético menor que osciló entre 0,2511 y 0,2797 para las poblaciones y un valor total de 0,2935, indicando con este resultado una variabilidad genética menor, muy probablemente por la utilización de oligonucleótidos que definen bandas diagnósticas, haciéndose necesario la evaluación de otros oligos que generen otras bandas polimórficas. El hecho de obtener en todos los casos, valores similares de heterocigo-sidad para cada una de las poblaciones, indica que la heterocigosidad obtenida para cada subpoblación, puede representar la heterocigosidad de la población general y validan la efectividad del muestreo de las poblaciones de mosquitos.
Los valores de heterocigosidad obtenidos con el método de Lynch y Milligan Tabla 2, indicaron también la existencia de poblaciones heterogéneas de An. darlingi y An. nuneztovari con un alto nivel de polimorfismo. Aunque para An. marajoara se obtuvo un menor valor (0,2936) estos niveles de polimorfismo, indican que existe un flujo genético dentro y entre las poblaciones, el cual permite la expresión de un rango de caracteres y comportamientos que aunque variables, confirman características que definen la especie de cada uno de estos vectores.
Comparar estos resultados de heteroci-gosidad (Tabla 2), con los obtenidos por otros investigadores, implica una comparación con trabajos en los que la heterocigosidad se haya obtenido a partir de este mismo tipo de marcador molecular pues carece de sentido la comparación con heterocigosidades obtenidas a partir de marcadores bioquímicos, siempre menores. Para evitar comparaciones innecesarias, vale la pena enfocar esta discusión en la implicación de calcular esta heterocigosidad con base en un marcador de tipo dominante de acuerdo con lo expuesto por Yan et al. (1999) quienes implican la viabilidad de utilizar este marcador dominante para calcular este parámetro de diversidad genética pobla-cional, al obtener para Aedes aegypti Linnaeus, 1762, una heterocigosidad mayor con datos RFLPs que con datos AFLPs y datos similares a los obtenidos por Apóstol et al. (1996) para la misma especie con marcadores RAPDs.
Al analizar el estadístico FST (Tabla 3) aunque los coeficientes de correlación de Pearson para todas las poblaciones combinadas a través de los loci RAPD fueron altos, los promedios estimados para An. darlingi y An. nuneztovari fueron variables, siendo ligeramente menores para An. nuneztovari con el estadístico de Wright que con el estadístico de Theta y Lynch-Milligan. También para An. marajoara se obtuvieron promedios menores con el estadístico de Wright lo que indica un grado relativamente bajo de diferenciación de las tres poblaciones de este vector.
Al comparar los valores FST (Tabla 3) entre grupos de pares de poblaciones para cada especie, se confirmó una menor variabilidad genética entre las poblaciones occidentales de An. darlingi y An. nunez-tovari, y las poblaciones orientales de An. marajoara, al presentar valores más bajos de FST comparados con los valores entre estas poblaciones y las poblaciones localizadas geográficamente en el oriente u occidente, respectivamente para cada especie. Lo anterior se reflejó en las tasas de migración por generación entre las poblaciones de cada especie. En el caso de An. darlingi, indicaron un mayor flujo genético entre las poblaciones occidentales de Tierralta y Medio Atrato con los tres tipos de estadísticos FST, (Tabla 3). Para An. nuneztovari, los valores de Nm fueron mayores entre las poblaciones de Buenaventura y Tierralta, que entre cada una de estas dos poblaciones y la de Tibú donde el número de migrantes por generación fue relativamente menor. Para An. marajoara fue más alto el número efectivo de migrantes por generación entre las poblaciones del oriente, Fuente de Oro-San Carlos de Guaroa y Cúcuta. Comparados con los otros dos vectores estudiados los migrantes por generación entre pares de poblaciones de An. marajoara fueron menores, probablemente subestimados por la utilización de cebadores diagnósticos.
Lo anterior confirmó la existencia de un flujo genético diferencial entre las poblaciones de estos tres vectores en Colombia y es muy probable que estas tasas de migrantes por generación estén asociadas con la presencia o ausencia de barreras geográficas (cordilleras) entre las regiones en las cuales se ubican las diferentes localidades, más que por un aislamiento geográfico por distancia. De igual modo, los análisis de componentes de varianza de los AMOVAS indicaron poca diferencia entre las poblaciones de cada una de las especies, ya que únicamente entre el 8,43% y el 11,31% de la variación es atribuible a varianza entre las poblaciones, mientras que el resto de la variación se presentó entre los individuos de cada población (Tabla 4). Este resultado indicó que An. darlingi, An. nunez-tovari y An. marajoara de Colombia presentan poca subdivisión geográfica entre poblaciones de la respectiva especie. La diferencia observada es comparable con apareamientos al azar, con un flujo de genes suficiente para mantener un grado bajo de estructuración genética, el cual puede depender del grado de separación geográfica entre sus poblaciones. Esto es posiblemente algo diferente a como lo plantean Lounibos y Conn (2000) los cuales citan aislamientos geográficos por distancia en poblaciones de An. albima-nus de Guatemala (De Mérida et al. 1999). Para An. nuneztovari Conn et al. (1998) reportaron valores de FST que aunque obtenidos con otro tipo de marcador molecular, indican también variaciones en las frecuencias génicas entre y dentro de poblaciones de Bolivia, Brasil y Venezuela, coincidentes con los resultados obtenidos para los vectores analizados en este estudio.
También Conn et al. (1999), utilizando un marcador codominante, demostraron un aislamiento significativo por distancia entre poblaciones de An. darlingi de Bolivia, Brasil y Venezuela, debido a la restricción de flujo genético a un nivel local, sugiriendo que las poblaciones aisladas pueden poseer fenotipos distintivos para factores que afectan su capacidad vectorial. En este estudio no se observó una marcada restricción al flujo genético, incluso entre poblaciones geográficamente separadas por la barrera de las cordilleras, pero esto puede ser atribuido también a que la información fue obtenida con un marcador dominante (RAPD).
Conclusiones
Las poblaciones de An. darlingi, y An. nuneztovari presentaron en Colombia un alto polimorfismo genético de loci RAPD-PCR, mientras que para las poblaciones de An. marajoara, el polimorfismo fue menor.
Los cuatro cebadores que produjeron marcadores diagnósticos para la especie C de Anopheles albitarsis s.l. en los trabajos de Wilkerson et al. (1995) fueron reproducidos con éxito en este estudio, reportando con esto la presencia en Colombia de únicamente An. marajoara. El análisis taxonómico y genético, determinó que las otras dos especies (An.darlingi y An. nuneztovari) conforman independientemente, una sola población.
Los análisis de distancia genética entre individuos de las poblaciones de An. darlingi, indicaron subdivisión geográfica entre las del occidente (Medio Atrato y Tierralta) y la población del oriente (Granada). Para An. nuneztovari sugieren evidencia de separación geográfica entre las poblaciones occidentales (Buenaventura y Tierralta), con la del nororiente (Tibú). En An. marajoara se presentó una mayor relación genética entre las poblaciones orientales de Cúcuta, Fuente de Oro y San Carlos de Guaroa.
En general, para cada uno de estas tres especies vectoras de malaria en Colombia se registraron diferencias intrapoblacio-nales que podrían indicar apareamientos al azar, con un flujo de genes suficiente para mantener un bajo grado de estructuración genética, el cual puede depender del grado de separación geográfica entre las poblaciones de cada especie.
Literatura citada
APOSTOL, B. L.; BLACK IV W.C. ; REI-TER, P.; MILLER B. R. 1996. Population genetics with RAPD-PCR markers: The breeding structure of Aedes aegypti in Puerto Rico. Heredity 76: 325-334. [ Links ]
BLACK IV, W. C. 1993. PCR with arbitrary primers: Approach with care. Insect Molecular Biology 2 (1):1-6. [ Links ]
BLACK IV, W. C. 1995. Statistical analysis of arbitrary primed PCR patterns in molecular taxonomic studies. En Clapp JP (ed.), Methods in Molecular Biology, vol 50:Species Diagnostic Protocol PCR and Other Nucleic Acid Methods, Human Press, Totowa, N Y, p. 39-55. [ Links ]
BLACK IV, W. C. 1996. Tutorial of RAPDFST 4.01 Program. Department of Microbiology. Colorado State University. [ Links ]
BLACK IV, W. C.; ANTOLIN, M. 1997. Tutorial of RAPDBOOT 1.0 Program. Department of Microbiology. Colorado State University. [ Links ]
BLACK IV, W. C.; DU TEAU, N. M. 1997. RAPD-PCR and SSCP analysis for insect population genetic studies. En Molecular Biology of Insect Disease Vectors: A methods manual. Edited by J. M. Cramp-ton, C. B. Beard and C. Louis. Chapman & Hall. 362-363pp. [ Links ]
COEN, E. S.; STRACHA, T.; DOVER, G. 1982. Dynamics of concerted evolution of ribosomal DNA and histone gene families in the Melanogaster species subgroup of Drosophila. Journal of Molecular Biology 158: 17-35. [ Links ]
CONN, J.; WILKERSON, R. C.; NAZARÉ, M.; SEGURA, O.; DE SOUZA, R. T. L.; SCHLICHTING, C. D.; WIRTZ, R. A.; PÓVOA, M. M. 2002. Emergence o a new neotropical malaria vector facilitated by human migration and changes in land use. The American Journal of Tropical Medicine and Hygiene 66(1): 18-22. [ Links ]
CONN, J. E.; ROSA-FREITAS, M. G.; LUZ, L. B.; MOMEN, H. 1999. Molecular population genetics of the primary neotropical malaria vector Anopheles darlingi using mtDNA. Journal of the American Mosquito Control Association 15(4): 468-474. [ Links ]
CONN, J.; MITCHELL, S.E.; COCKBURN, A. F. 1998. Mitochondrial DNA analysis of the Neotropical malaria vector Anopheles nuneztovari. Genome 41(3): 313-327. [ Links ]
CONN, J.; RANGEL, Y.; SEAWRIGHT, J. A. 1993. A new cytotype of Anopheles nuneztovari from western Venezuela and Colombia. Journal of the American Mosquito Control Association 9(3): 294-301. [ Links ]
CHAKRABORTY, R.; LEIMAR, O. 1987. Ge-netic variation within a subdivided population. In: Population genetics and fishery mana-gement. N. Ryman y F. Ulter (Eds) Washington Sea Gant Program. University of Washington Press. Pp. 87-120. [ Links ]
DELGADO, N.; RUBIO-PALIS, Y. 1993. Morphometric characterization of the malaria vector Anopheles nuneztovari (Dipte-ra: Culicidae) from western Venezuela. Mosquito Systematics 24: 231-241. [ Links ]
DE MERIDA, A. M. ; PALMIERI, M. ; YURRITA, M. ; MOLINA, A.; MOLINA E. ; BLACK, W. C. IV. 1999. Mitochondrial DNA variation among Anopheles albima-nus populations. The American Journal of Tropical Medicine and Hygiene 61(2): 230-239. [ Links ]
EXCOFFIER, L.; SMOUSE, P.E.; QUA-TTRO, J. M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction sites. Genetics 131: 479-491. [ Links ]
FARAN, M. E. 1980. Mosquito studies (Diptera: Culicidae) XXXIV. A revision of the Albimanus Section of the subgenus Nysorrhynchus of Anopheles. Contribu-tions of the American Entomological Institute 15(7): 1-215. [ Links ]
FELSENSTEIN, J. 1993. PHYLIP (Phyloge-ny Inference Package) version 3.5c. Department of Genetics. University of Seattle, Washington. [ Links ]
FERRREIRA, M. E.; GRATTAPAGLIA, H. 1998. Introducción al uso de marcadores moleculares en el análisis genético. Ministerio do Agricultura e do Abastacimiento EMBRAPA, Cenargen. Brasilia, D.F. [ Links ]
FLEMING, G. 1986. Biología y Ecología de los vectores de la malaria en las Américas. Publicación 86-72 de la Organización Panamericana de la Salud, Washington, D.C. 54 pp. [ Links ]
LINTHICUM, K. J. 1988. A revision of the Argyritarsis Section of the Subgenus (Nyssorhynchus) of Anopheles (Diptera: Culicidae) Mosquito Systematics 20: 98-271. [ Links ]
LOUNIBOS, L. P.; CONN, J. E. 2000. Malaria vector heterogeneity in South America. American Entomologist 46(4): 238-249. [ Links ]
LYNCH, M.; MILLIGAN, B. G. 1994. Analysis of population genetic structure with RAPD markers. Molecular Ecology 3: 91-99. [ Links ]
MILLER, M. P. 1998. AMOVA-PREP. 1.01. A program for the preparation of AMOVA input files from dominant-marker row data. Department of Biological Sciences, Northern Arizona University, Flagstaff, AZ. [ Links ]
MUNSTERMANN, L. E.; CONN, J. .E 1997. Systematics of mosquito disease vectors (Diptera: Culicidae): Impact of molecular biology and cladistic analysis. Annual Review of Entomology 42: 351-369. [ Links ]
NEI, M. 1972. Genetic distances between po-pulations. American Naturalist 106: 283-292. [ Links ]
OPS/OMS. 2000. Informe sobre la implemen-tación de la iniciativa hacer retroceder la malaria en Colombia. 6-12 p. [ Links ]
POSSO, C. E.; GONZALEZ, R.; CARDE-NAS, H.; GALLEGO, G.; DUQUE, M. C.; SUAREZ, M. F. 2003. Random amplified polymorphic DNA analysis of An. nuneztovari (Diptera: Culicidae) from western and northeastern Colombia. Memorias of Institute Oswaldo Cruz. (98)4: 469-476. [ Links ]
SAMBROOK, J. C.; FRITSCH, R.; MANIATAS, T. 1989. Molecular Cloning: A Laboratory Manual, 2nd ed., Cold Spring Harbor Laboratory Press, N.Y, p. 6.3-6.34. [ Links ]
SUAREZ, M. F.; QUIÑONEZ, M. L.; FLEMING, G. A.; ROBAYO, M. 1987. Clave gráfica de los anofelinos de Colombia. Ministerio de Salud Pública de Colombia, 57pp. [ Links ]
TABACHNICK, W. J.; BLACK IV, W. C. 1996. Population genetics in vector biology. In Beaty BJ, Marquet WC. The Biology of the Disease Vectors, University Press of Colorado, Niwot Colorado, p. 417-437. [ Links ]
TORRES, C. 2004. La tribu Pentatomini (Hemiptera: Pentatomidae) en Colombia. En: Fernández, et al. (eds), Insectos de Colombia. Vo l 3, Universidad Nacional de Colombia, p. 61-128. [ Links ]
WILKERSON, R. C.; PARSONS, T.J.; KLEIN, T. A.; GAFFIGAN, T. V.; BERGO, E.; CONSOLIN, J. 1995. Diagnosis by random amplified polymorphic DNA polymerase chain reaction of four cryptic species related to Anopheles (Nyssorrhynchus) albitarsis (Diptera: Culicidae) from Paraguay, Argentina and Brazil. Journal of Medical Entomology 32: 697-704 . [ Links ]
WRIGHT, S. 1951. The genetical structure of populations. Annals of Eugenics. 15: 323-354. [ Links ]
YAN, G.; ROMERO-SEVERSON, J.; WAL-TON, M.; CHADEE, D. D.; SEVERSO, D. D. 1999. Population genetics of the yellow fever mosquito in Trinidad: Comparisons of amplified fragment length polymorphism (AFLP) and restriction fragment length polymorphism (RFLP) markers. Molecular Ecology (8): 951-963. [ Links ]
Recibido: 01-oct-04 Aceptado: 05-dic-05