Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Entomología
Print version ISSN 0120-0488On-line version ISSN 2665-4385
Rev. Colomb. Entomol. vol.33 no.2 Bogotá June/Dec. 2007
Sección Agrícola
Análisis y aplicaciones de la tipificación molecular del genoma mitocondrial
de Beauveria bassiana
Analysis and applications of the molecular typification of the mitochondrial genome of
Beauveria bassiana
DANIEL URIBE1,2 y GEORGE KHACHATOURIANS1
1 Ph. D., BioInsecticide Research Laboratory, Department of Applied Microbiology and Food Science, University of Saskatchewan, Saskatoon, SK S7N 5A8, Canada. khachatouria@skyway.usask.ca
2 Autor para Correspondencia: Ph. D., Universidad Nacional de Colombia, Instituto de Biotecnología, A.A. 14-490, Bogotá, D.C. Colombia. duribev@unal.edu.co
Resumen: Beauveria bassiana es uno de los hongos entomopatógenos más ampliamente usados a nivel mundial para el control de plagas en el sector agrícola. Su importancia desde el punto de vista comercial ha planteado la necesidad de desarrollar estrategias de caracterización molecular, con el objeto de realizar seguimiento de las cepas en campo y eventualmente lograr la protección de aquellas comercialmente importantes. El genoma mitocondrial es una herramienta que se presta para la caracterización taxonómica, la realización de estudios filogenéticos y la identificación de cepas a nivel molecular, ya que es una molécula haploide, de rápida evolución, y con similitud funcional en diferentes niveles taxonómicos. En este trabajo se evaluó la diversidad de DNA mitocondrial en 20 cepas del hongo entomopatógeno B. bassiana. Esta caracterización se realizó mediante el uso de la técnica de RFLP empleando el DNA total de la cepa de referencia GK2016 digerido con la enzima de restricción Hpa II como sonda. Se encontró una gran diversidad representada por la presencia de 10 mito-tipos dentro de la muestra estudiada. No se encontró relación específica entre patrones de mito-tipificación y el origen geográfico o de hospedero. Así mismo se realizó un análisis filogenético de diferentes especies de hongos filamentosos incluyendo algunos entomopatógenos empleando el gen que codifica para el fragmento 16s srRNA de origen mitocondrial. El análisis de estos resultados y nuestra interpretación de las implicaciones en la investigación de B. bassiana serán discutidos.
Palabras clave: Hongos entomopatógenos. Mitocondria. RFLP. Caracterización molecular.
Abstract: Beauveria bassiana is one of the entomopathogenic fungi most widely used world wide for the control of insect pests in the agricultural sector. Its importance from the commercial point of view has lead to the necessity to develop strategies of molecular characterization, with the aim of following the fate of strains in the field and eventually protecting those that are most commercially important. The mitochondrial genome is a tool that is appropriate for taxonomic characterization, the development of phylogenetic studies and the characterization of strains at the molecular level because it is a haploid molecule, fast evolving and with functional similarity at different taxonomic levels. In this work the mitochondrial DNA diversity of 20 strains of the entomopathogenic fungi B. bassiana were evaluated. This characterization was done by using the RFLP technique with the total mitochondrial DNA of the standard strain GK2016 digested with the restriction enzyme Hpa II as a probe. A great diversity was found, represented by the presence of 10 mito-types within the sample studied. No specific relationship was found between the patterns of mito-typification and either the geographic origin or the host. Similarly a phylogenetic analysis was conducted on several filamentous fungi species including some entomopathogenic fungi using the mitochondrial gene 16s srRNA. The analysis of these results and our interpretation of the implications for research on B. bassiana are discussed.
Key words: Entomopathogenic fungi. Mitochondria. RFLP. Molecular characterization.
Introducción
Las moléculas de ADN mitocondrial (ADN mt) son genomas relativamente pequeños que coevolucionan a su propia taza evolutiva en relación con genoma nuclear de los organismos en los cuales ellos están alojados. Particularmente en animales y hongos dichos genomas evolucionan más rápidamente que su contraparte de ADN nuclear (Kouvelis et al. 2004). El ADN mt es ampliamente empleado para la determinación de relaciones taxonómicas y filogenéticas entre los grupos de organismos eucarióticos que poseen dicho organelo. Las razones para su popularidad reside en características intrínsecas de esta molécula tales como su reducido tamaño, la alta tasa evolutiva, la falta de bases metiladas, el alto contenido de residuos adenina-timina (AT) y el hecho de ser una molécula haploide donde la mayoría de los alelos poseen la misma función e inclusive poseen regiones universalmente conservadas (Burns et al. 1991). Dentro del genoma mitocondrial las secuencias de ARN ribosomal (ARNr) han sido empleadas como herramienta molecular para identificar relaciones filogenéticos a diferentes niveles taxonómicos (Hillis et al. 1996).
El genoma mitocondrial en hongos filamentosos es usualmente una molécula circular de ADN, de tamaño variable que oscila entre 18,9 kpb en Torulopsis glabatra y 176 Kpb en Agaricus bitorquis (Quel) (Taylor 1986). A pesar de las diferencias en tamaños, todos los genomas mitocondriales pertenecientes a hongos examinados hasta la fecha poseen un número similar de genes, incluyendo una serie de genes de ARNr, 23-26 ARN de transferencia (ARNt) y varios genes codificantes de proteínas (Khachatourians y Uribe 2004). La diferencia en tamaño en relación con los genomas más grandes está determinada principalmente por intrones y regiones ricas en A-T (Cummings et al. 1990).
En los hongos entomopatógenos (HE) el genoma mitocondrial de los organismos donde dicha molécula ha sido estudiada es relativamente pequeño (Khachatourians y Uribe 2004). Los tamaños oscilan entre 24,5 kpb en Lecanicillium muscarium (Petch) (Sinónimo Verticillium lecanii) (Kouvelis et al. 2004) y 32 kpb en Metarhizium anisopliae (Metschnikoff) (Mavridou y Typas 1998), pasando por 28,5 kpb en Beauveria bassiana (Bálsamo) (Pfeifer et al. 1993). Su reducido tamaño sugiere moléculas con pocas secuencias intergenéticas redundantes, así como poca frecuencia de secuencias tipo intrones y por ende poca variabilidad genética a nivel intraespecífico (Khachatourians y Uribe 2004; Kouvelis et al. 2004). Sin embargo, a pesar del reducido tamaño de los genomas mitocondriales en los HE, Mavridou y Typas (1998) demostraron un alto grado de variabilidad genética asociada al genoma mitocondrial de M. anisopliae. En su trabajo estos investigadores encontraron 16 patrones de RFLP mitocondrial, luego de analizar 25 cepas de M. anisopliae obtenidas de 15 países, demostrando así un alto grado de variabilidad genética asociada al genoma mitocondrial de este HE. Así mismo, Kouvelis et al. (1999) identificaron una alta variabilidad genética presente en el genoma del HE L. muscarium al encontrar 17 mitotipos diferentes luego de analizar 51 cepas.
Beauveria bassiana es uno de los hongos entomopatógenos más ampliamente usados a nivel mundial en el control de insectos plaga en la industria agrícola y forestal (Inglis et al. 2001). La razón para esta preferencia reside en su amplio rango de acción correspondiente a cerca de 750 especies de insectos, el alto grado de conocimiento a nivel molecular de la interacción hospedero-patógeno y el desarrollo del sistema de producción de este hongo (Feng et al. 1994; Inglis et al. 2001; Khachatourians et al. 2002). El éxito comercial de B. bassiana ha llevado a la necesidad de desarrollar sistemas de tipificación molecular, no únicamente con el objeto de proteger algunas cepas de interés comercial, sino para lograr el seguimiento de las mismas en campo con el objeto de entender su capacidad de residualidad, así como la estructura poblacional del genero a nivel molecular (Gaitán et al. 2002; Padmavathi et al. 2003; Wang et al. 2003). Diferentes aproximaciones se han llevado a cabo para la detección de polimorfismos genéticos en B. bassiana, entre las técnicas mas sobresalientes se pueden mencionar la de RFLP (Kosir et al. 1991; Maurer et al. 1997; Coates et al. 2001), polimorfismos asociados a la conformación de cadena sencilla (Hegedus y Khachatourians 1996), análisis telomérico (Couteaudier y Viaud 1997; Padmavathi et al. 2003) e inclusive combinación de técnicas como PCR anidado del gen de la Pr1, marcadores de microsatélites y análisis de 28s rADN (Wang et al. 2003). Estos análisis han proporcionado una mejor comprensión de la taxonomía y la tipificación molecular de la especie, sin embargo, estas aproximaciones no incluyen el análisis de ADN mt, el cual, como bien se mencionó antes, es una molécula altamente informativa para este tipo de análisis.
El DNA mitocondrial de B. bassiana ha sido caracterizado con respecto a los rasgos físico-químicos más relevantes, tales como densidad boyante, temperatura de fusión, contenido de G+C, mapa genómico y secuencia de aproximadamente 60% del genoma (Pfeifer y Khachatourians 1989; Hegedus et al. 1991; Pfeifer et al. 1993; Hegedus et al. 1998). El análisis de polimorfismos del DNA mt de B. bassiana, fue abordado por primera vez por Hegedus y Khachatourians (1993). En dicho estudio fueron empleadas 15 cepas principalmente de norte América, mostrando únicamente dos genotipos mitocondriales, lo cual sugirió un genoma altamente conservado en relación con otros HE como M. anisopliae y L. muscarium (Mavridou y Typas 1998; Kouvelis et al. 1999).
En este estudio se pretende evaluar nuevamente el polimorfismo de ADN mt de B. bassiana. Para tal fin se empleó la técnica de RFLP en 18 aislamientos de la especie pertenecientes a cuatro continentes. Digestiones simples y dobles del ADN total de cada cepa se confrontaron contra el ADN mt total de la cepa GK2016 empleada como sonda. Así mismo, se presenta un análisis filogenético de la especie empleando secuencias de 16sRNA del ADN mt, con lo cual se busca establecer relaciones de los hongos entomopatógenos con otro grupo de hongos filamentosos.
Materiales y Métodos
Hongos entomopatógenos. Veinticinco aislamientos identificados como B. bassiana, B. amorpha (Hohn), B. nivea (Syn Tolypoladium inflatum (W. Gams), B. cylindrospora (Syn. Tolypocladium cylindrospurum (W. Gams), Beauveria spp, P. farinosus (Holmsk) y M. anisopliae fueron estudiados; sus insectos hospederos y orígenes geográficos están descritos en la Tabla 1. Los hongos fueron propagados en medio Sabouraud- Dextrosa incubados por 72 horas a 27°C y 150 rpm, y almacenados en glicerol al 12,5% de acuerdo al procedimiento de Pfeifer y Khachatourians (1989).
Sondas de ADNmt. Las sondas de ADN mt usadas en este estudio fueron el fragmento BbmtE2 descrito por Hegedus y Khachatourians (1993), el cual contiene el gen NAD1 y el extremo 5’ del gen ATP6. Este fragmento fue obtenido a partir de ADN mt de la cepa de B. bassiana GK2016 (Hegedus y Khachatourians 1993; Pfeifer et al. 1993). Así mismo el ADN mt completo de la misma cepa fue aislado usando la técnica de gradiente de CsCl-bis-benzimida descrita por Pfeifer y Khachatourians (1989). Una vez obtenido el genoma mitocondrial éste fue digerido con la enzima de restricción Hpa II para posteriormente ser marcado y empleado como sonda.
Preparación del ADN mt. El análisis de RFLP para estudiar los polimorfismos del DNA mt de B. bassiana, se realizó empleando ADN total de cada una de las cepas a analizar. Dicho ADN fue aislado siguiendo la técnica de micelio liofilizado descrita por Pfeifer y Khachatourians (1989). Las técnicas de aislamiento de plásmidos, digestión enzimática de ADN, hibridización, transferencia y revelado, así como la elaboración de soluciones tampón fueron realizados de acuerdo con los protocolos estándar descritos por Sambrook y Russell (2001). A continuación se describen brevemente los procedimientos asociados a la preparación DNA mt, para llevar a cabo los análisis de polimorfismos. Entre siete y diez microgramos de ADN genómico total aislado de cada aislamiento fúngico fueron digeridos con la enzima de restricción EcoR I. Adicionalmente, el ADN fue sometido a digestión doble empleando las enzimas EcoR I- Hind III y EcoR I – Bgl II cuando pBbmtE2 y ADN mt total respectivamente, fueron usados como sonda. El ADN fue separado electroforéticamente en geles de agarosa-TBE (0,09 M Tris-HCl, 0,09 M ácido bórico, 0,02 M EDTA) al 0,8% (peso/vol). Posteriormente el ADN fue transferido a membranas de nylon Hybond-N (Amersham Pharmacia Biotech, Buckinghamshire, Inglaterra) por el método de transferencia por capilaridad (Sambrook y Russell 2001).
Análisis de polimorfismos mitocondriales por RFLP. Las membranas de nylon con el ADN total fueron hibridizadas a 52°C con sondas no radioactivas marcadas con digoxigenina por la técnica de incorporación de nucleótidos modificados por iniciadores aleatorios (“random primers”), de acuerdo al manual de instrucciones del productor (Roche Molecular Biochemicals, Mannheim 1999). Las condiciones de astringencia durante el procedimiento de lavado consistieron en dos lavados durante 30 min. cada uno 0,5x SSC (20X SSC es 175,3 g de cloruro de sodio y 88,2 g de citrato de sodio por litro) y 0,5% de sodio dodecil sulfato a 52°C luego de dos lavados previamente realizados a 2xSSC, 0,1% SDS a 25°C en agitación constante, siguiendo las indicaciones del productor del kit de marcación y detección. La capacidad de las bandas de ADN para hibridizar con las sondas mitocondriales fueron registradas con caracteres binarios donde 1 = presencia y 0 = ausencia de hibridización. Las relaciones filogenéticas entre los polimorfismos mitocondriales obtenidos fueron estimadas usando el método de la parsimonia de Wagner luego de 1000 reorganizaciones usando la opción “bootstrap” con el programa PHYLIP (Felsenstein 1989).
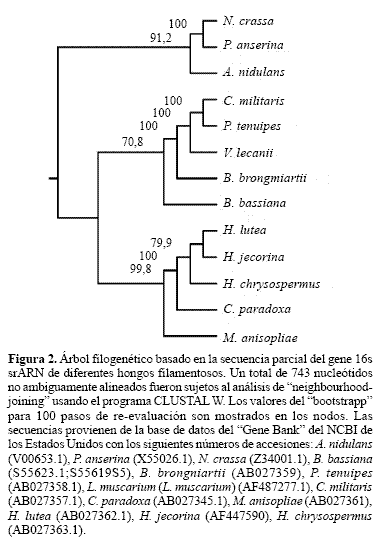
Análisis filogenético empleando secuencias de ADN mt 16sRNA. Un árbol filogenético basado en las secuencias génicas mitocondriales de 16sRNA provenientes de trece hongos filamentosos fue construido usando 743 nucleótidos no ambiguamente alineados. Estas secuencias fueron sometidas al análisis de “neighborhood-joining” usando el programa CLUSTAL W (Higgings et al. 1994), luego de 1.000 pasos de reorganizaciones usando la opción “bootstrap”.
Resultados
Análisis de genotipos mitocondriales empleando la sonda BbmtE2. Se realizaron tres aproximaciones para caracterizar los patrones de RFLP obtenidos al analizar el ADN mt de B. bassiana. La primera consistió en el aislamiento del ADN total de 18 cepas de la especie. El ADN de estas cepas fue digerido con la enzima de restricción EcoR I y confrontado con la sonda de ADN mt BbmtE2. Se encontraron cinco genotipos mitocondriales (mito-tipos) (Tabla 1) con diferencias entre una y dos bandas entre si. Los mito-tipos A y B, descritos previamente por Hegedus y Khachatourians (1993) prevalecieron dentro de la población analizada. Seis de los 18 aislamientos de B. bassiana mostraron genotipo A, mientras que ocho aislamientos mostraron genotipo B cuando son digeridos con EcoR I. Estos mito-tipos se caracterizan por presentar una banda de 4,6 Kb y 4,1 Kb respectivamente. Dos aislamientos presentaron el mito-tipo C el cual consiste en dos bandas de 4,4 y 2,1 Kb respectivamente. Finalmente, un aislamiento representa los mito-tipos D y E con una banda de 5,4 Kb y dos bandas de 4,4 kb y 0,8 Kb respectivamente (Tabla 1).
La segunda aproximación, que consistió en la digestión doble del ADN total con las enzimas EcoR I y Hind III y confrontado contra la sonda BbmtE2, básicamente confirmó los resultados descritos arriba al mantener los cinco mito-tipos con la digestión sencilla (EcoR I) para cada aislamiento. La única excepción fue la cepa DAOM 144746, la cual, luego de la digestión sencilla, fue identificada dentro del mito-tipo A (con una banda de 4,6 Kb), pero la doble digestión con EcoR I y Hind III mostró únicamente dos bandas de 3,1 y 0,6 Kb, como las cepas caracterizadas como mito-tipo B, en lugar de las tres bandas obtenidas para los aislamientos del mito-tipo A (resultados no mostrados). Este resultado sugiere que la cepa DAOM 144746 posee un sitio de corte extra para la enzima de restricción Hind III, produciendo bandas de tamaño molecular más pequeño que no lograron ser detectadas en la membrana de hibridización. Las diferencias en el genoma mitocondrial de esta cepa fueron confirmadas más adelante cuando el ADN total fue doblemente digerido con las enzimas de restricción EcoR I - Bgl II y confrontada contra el ADN mt total de la cepa GK2016 como sonda (Tabla 2). En conclusión, la estrategia de doble digestión ayudó a mejorar el nivel de resolución de la sonda BbmtE2, evidenciando seis mito-tipos en lugar de cinco dentro de la población estudiada. Sin embargo esta estrategia posee menos capacidad de resolución cuando es comparada con la utilización del ADN mt total como sonda.
Las otras especies de Beauveria usadas en este estudio fueron B. amorpha, B. nivea, B. cylindrospora y Beauveria spp. Las primeras tres especies fueron fácilmente diferenciadas de las cepas de B. bassiana al ser confrontadas con la sonda BbmtE2 luego de la digestión simple y doble del ADN total (Tabla 1). Este resultado confirma que el análisis de RFLP mitocondrial es una herramienta confiable para diferenciar cepas a nivel de especie como fue reportado por Hegedus y Khachatourians (1993). Teniendo en cuenta esa consideración, nuestros resultados sugieren que los aislamientos de Beauveria spp. ARSEF 32 y ARSEF 3289 pertenecen a B. bassiana ya que ambos aislamientos poseen el mitotipo B al ser sometidos a digestión simple y doble y confrontados contra la sonda BbmtE2. Adicionalmente, un análisis filogenético empleando el DNA mt total como sonda (resultados no mostrados) agrupó estas dos cepas con el grupo principal de 15 cepas de B. bassiana.
Otras dos especies de HE, Paecilomyces farinosus (Holmsk) y Metarhizium anisopliae fueron también incluidas. Estos dos géneros presentaron claramente patrones de hibridización diferentes en comparación con los aislamientos de Beauveria (Tablas 1-2; Fig. 1). Estos resultados confirman nuevamente que este sistema de tipificación molecular puede ser usado para diferenciar entre al menos algunos géneros principales de HE. Sin embargo, la cepa de P. farinosus ATCC 1360 presentó una banda de 4,6 Kb, similar a los aislamientos del mito-tipo A de los aislamientos de B. bassiana, cuando el ADN total fue digerido con la enzima de restricción EcoR I. A pesar de estas similitudes, cuando el ADN total fue sometido a digestión doble, con las enzimas de restricción EcoR I y Hind III, únicamente una banda de 3,8 Kb fue encontrada, en lugar de dos o tres bandas como en los mito-tipos A y B de los aislamientos de B. bassiana (Tabla 1). Estos resultados sugieren que el fragmento complementario con la sonda de BbmtE2 en P. farinosus no posee sitios de reconocimiento para la enzima de restricción Hind III hacia el segmento central de este fragmento. Sin embargo, si debe poseer un par de estas secuencias hacia uno o ambos extremos del segmento produciendo fragmentos más pequeños que no pudieron ser detectados en la membrana de nylon.
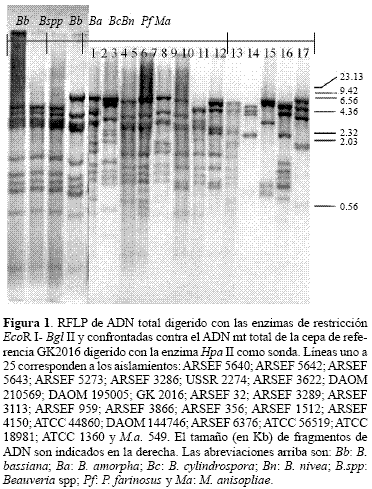
Análisis de genotipos mitocondriales empleando ADN mt total como sonda. La última aproximación para identificar mito-tipos diferentes en cepas de B. bassiana consistió en la hibridización de ADN total digerido con las enzimas de restricción EcoR I - Bgl II, con el ADN mt total de la cepa GK2016 empleado como sonda. El ADN mt fue previamente digerido con la enzima Hpa II y marcado con digocigenina. En este caso, nueve patrones de hibridización diferentes fueron obtenidos con las 18 cepas de B. bassiana o diez patrones cuando los 20 aislamientos, incluyendo los dos aislamientos de Beauveria spp., son considerados. Si este es el caso, cuatro aislamientos fueron agrupados como mito-tipo I, seis como II, dos como mito-tipos III y VI y los otros seis (IV, V, VII-X) fueron mito-tipos representados por una sola cepa (Tabla 2). Una característica muy importante en este caso fue la identificación de secuencias variables y conservadas dentro de el ADN mt de B. bassiana. Por ejemplo, todas las cepas de B. bassiana, Beauveria spp., B. nivea y P. farinosus presentaron la banda de 1Kb, Adicionalmente, una cepa de B. bassiana (ARSEF 959) no mostró las bandas de 1,3 y 1,8 Kb y dos cepas (ARSEF 4150 y DAOM 144746) no mostraron la banda de 1,2 Kb. Estos resultados sugieren que las regiones que contienen estos sitios de restricción particulares son altamente conservados en el genoma mitocondrial de B. bassiana.
Polimorfismos mitocondriales de B. bassiana y su relación con otros hongos filamentosos. En los hongos entomopatógenos se han hecho algunos esfuerzos para obtener secuencias completas de ADN mt. De hecho en B. bassiana alrededor del 60% del genoma ha sido secuenciado (Pfeifer et al. 1993; Hegedus et al. 1991) y para L. muscarium la secuencia completa del genoma ha sido reportada (Kouvelis et al. 2004). Sin embargo, la única secuencia del ADN mt disponible para otros HE diferentes a estos dos es la equivalente al gen de la subunidad pequeña del RNA ribosomal (srRNA). Es bien sabido que esta secuencia ofrece ventajas muy particulares como herramienta para el análisis filogenético a nivel de especie y grupos taxonómicos superiores (Burns et al. 1991). Teniendo en cuenta esta información, se realizó un análisis filogenético (Fig. 2), incluyendo diferentes HE pertenecientes al grupo de hongos deuteromicetes (B. bassiana, B. brongniartii (Petch), L. muscarium, Paecilomyces tenuipes (Samson) y M. anisopliae) y ascomicetos (Cordyceps militaris (LinK) y C. paradoxa (Kobayasi)). Otros ascomicetos fueron considerados incluyendo los hongos mico parásitos Hypocrea lutea (Tode - Petch), H. jecorina (Syn. Trichoderma reesei) y Hypomyces chrysospermus (Tulasne). Las secuencias de Neurospora crassa (Shear & Dodge), Podospora anserina (Ces - Rehm) y Aspergillus nidulans (Tieghem) fueron también incluidas como punto de referencia. Todos los HE, con la excepción de C. paradoxa y M. anisopliae hacen parte de un clado monofilético. Vale la pena anotar la alta significancia estadística de estos resultados según los valores de “bootstrap” en los nodos del árbol filogenético. Interesantemente, las dos especies de Cordyceps se ubicaron en grupos separados, e inclusive más estrechamente relacionadas con otros grupos anamórficos que con ellas mismos. Este resultado sugiere no únicamente un alto grado de variabilidad genética en el género Cordyceps, sino también el grado de similitud entre algunas especies de este género con algunas especies de hongos entomopatogenos deuteromicetos. Este hecho soporta estudios previos que reclaman el hecho de que algunas especies de HE conidiogénicos como Metarrhizium taii, P. tenuipes, y B. bassiana son la fase anamorfa de especies de hongos ascomicetos del genero Cordyceps (Zong-Qui et al. 1991; Huang et al. 2002).
Discusión
Debido a las características del ADN mt, éste ha sido usado en la tipificación de cepas y análisis de poblaciones de HE con diferentes niveles de éxito. Por ejemplo Bidochka et al. (2001) no encontraron diferencias en el análisis por RFLP de ADN mt total entre 83 aislamientos de M. anisopliae obtenidos de suelos agrícolas y forestales de la provincia de Ontario (Canadá). Por otra parte, Mavridou y Typas (1998) reportaron 16 patrones diferentes de ADN mt al analizar 25 aislamientos de M. anisopliae obtenidos de 15 países cuando el ADN mt total se usó como sonda. Estos resultados demuestran que para este tipo de análisis no es importante únicamente tener en cuenta las herramientas moleculares a utilizar, sino que el tamaño y la naturaleza de la población es también importante. Hegedus y Khachatourians (1993) analizaron el genoma mitocondrial de 15 aislamientos de B. bassiana usando diferentes porciones del genoma mitocondrial como sondas. Los resultados de dicho trabajo sugirieron un genoma altamente conservado con únicamente dos mito-tipos. Nosotros en este estudio usamos una población más variada de aislamientos en términos del origen geográfico. Así mismo se emplearon dos tipos de sondas, una porción de 4,6 kb del genoma mitocondrial reconocido anteriormente como relativamente polimórfica para la especie (Hegedus y Khachatourians 1993) y el ADN mt total. La aproximación presentada aquí demostró la presencia de un genoma mitocondrial altamente polimórfico con nueve mitotipos dentro de las 18 cepas de B. bassiana analizadas.
El mapa de restricción de B. bassiana descrito por Pfeifer et al. (1993), es una herramienta adecuada para identificar la localización física no únicamente de las regiones variables sino también de las regiones conservadas dentro del ADN mt de B. bassiana. Por ejemplo, las bandas altamente conservadas de 1,0 y 1,3 Kb producidas después de digerir el ADN mt con Bgl II, están localizadas en el fragmento denominado XS1 en el genoma mitocondrial de la cepa GK2016 de B. bassiana (Pfeifer et al. 1993). Este fragmento contiene el fragmento 5’ de los genes de la subunidad larga del ARN ribosomal (LrARN); sin embargo, el análisis de restricción de la secuencia de dicho fragmento indica que los tres cortes de la enzima Bgl II caen fuera del gen. La alta frecuencia de estas bandas en las cepas de B. bassiana sugieren que los sitios de restricción con Bgl II pertenecen a una sección muy conservada presente en este fragmento. Además, la banda de 1,8 Kb, que corresponde al fragmento E3B en el ADN mt de la cepa GK2016, es producido por dos cortes de la enzima EcoR I. Esta banda contiene la región correspondiente al extremo 3’ de la subunidad 6 de la ATPasa y el extremo 5’ del gen srARN (Pfeifer et al. 1993). Un análisis de alineamiento de secuencias de ADN de diferentes hongos filamentosos usando el programa BLAST 2.2.5 (Altschul et al. 1997) del National Center for Biotechnology Information (NCBI) de los Estados Unidos, mostró que las secuencias de ambos genes son altamente conservadas en este grupo de organismos. Sin embargo, los sitios de restricción de la enzima EcoR I al interior de estos dos genes son aparentemente específicos para el grupo filogenético que incluye las especies de Beauveria y otros HE relacionados sugiriendo la presencia de una marca molecular para este grupo de hongos (resultados no mostrados).
A pesar de la naturaleza altamente conservada de los genes de la subunidad 6 de la ATPasa y el srARN, nuestra evaluación del genoma mitocondrial total de B. bassiana mostró que hay alguna variabilidad a nivel de cepa dentro de la especie, que permite el reconocimiento de diferentes patrones de RFLP con el ADN mt total como sonda. Usando esta estrategia, diez mitotipos de las 20 cepas de B. bassiana analizadas (incluyendo las dos Beauveria spp.), fueron identificadas y separadas de las otras cinco especies de hongos entomopatógenos incluidas en este estudio. Nosotros especulamos que la fuente de esta variabilidad es probablemente las secuencias intergénicas, ya que secuencias no codificantes tipo intrones no están dentro de las secuencias reportadas de los pequeños genomas mitocondriales de HE (Grossman y Hudspeth 1985; Khachatourians y Uribe 2004; Pfeifer et al. 1993; Kouvelis et al. 2004). La alta variabilidad geonómica mitocondria encontrada en este estudio en B. bassiana y en M. anisopliae (Mavridou y Typas 1998) y en L. muscarium (Kouvelis et al. 1999), indican que los genomas mitocondriales están expuestos a mecanismos ambientales de variación de secuencias en la naturaleza (i.e. radiación UV) que puede producir eventos mutacionales aislados ya que no existe ninguna indicación de intercambios de genomas mitocondriales entre estos hongos (Kawano et al. 1995; Viaud et al. 1998). Adicionalmente errores en la replicación, eventos de recombinación intermolecular y la presencia de un sistema de reparación de ADN mt muy simple, en el cual inclusive dímeros de pirimidina no son reparados (Kang y Hamasaki 2002), deben ser algunas de las razones para perpetuar tal variabilidad dentro de los genomas mitocondriales de hongos filamentosos.
Estudios filogenéticos en hongos filamentosos no entomopatógenos han permitido establecer correlaciones entre patrones de ADN mt y características que no están asociadas con las funciones mitocondriales (Gordon y Okasoto 1992; Egger et al. 1991; Jeng et al. 1991). Sin embargo, en HE parece ser que este no es el caso. Diferentes estudios empleando el genoma mitocondrial han conducido a la conclusión que no existe correlación al menos entre especificidad, origen geográfico, hábitat y polimorfismos de ADN mt (Hegedus y Khachatourians 1993; Mavridou y Typas 1998; Kouvelis et al. 1999; Bidochka et al. 2001). Nuestros datos confirman y extienden esta conclusión. Inclusive partiendo del modesto número de cepas de este estudio, nosotros encontramos dos mito-tipos principales (I y II) en B. bassiana. Las cepas del mito-tipo I fueron colectadas de regiones tan separadas entre si como Rusia, Francia y Canadá; mientras aquellas pertenecientes al mito-tipo II pertenecen a regiones de Canadá, Etiopia y USA. Esto sugiere que la distribución geográfica no es un determinante primario para la selección de estos genotipos mitocondriales. En relación con los hospederos, dos grupos principales son identificables, las cepas aisladas originalmente de lepidópteros y ortópteros. Es importante mencionar que el análisis filogenético en este caso muestra unos agrupamientos interesantes tales como mito-tipos III, V y IX, conformados por cepas aisladas únicamente de ortópteros. Mito-tipo I y VI están igualmente asociados y sus cepas fueron originalmente aisladas de lepidópteros o provienen de hospederos desconocidos. Sin embargo, el mito-tipo II que posee cepas aisladas de ortópteros y coleópteros está agrupado con los mitotipos IV y X, los cuales poseen cepas colectadas del suelo y lepidópteros respectivamente. Todos estos resultados sugieren que no existe realmente una relación entre genotipos mitocondriales y origen geográfico u hospederos. La distribución mundial de los mitotipos I y II en B. bassiana deben sugerir una reproducción clonal y una dispersión exitosa de estas esporas independientemente de que el crecimiento natural de esta especie en la naturaleza sea primordialmente
entomopatogénica o saprofítica (St. Leger et al. 1992; Bidochka et al. 2002). Lo que es claro es que hay una gran variabilidad dentro del genoma mitocondrial de esta especie, la cual está representada en este estudio por los nuevos siete mito-tipos aquí descritos. Sería muy interesante entender los eventos naturales que conducen a tal variabilidad bien sean dados por recombinación intra-molecular o mutaciones puntuales.
El análisis de agrupamientos del bandeo del ADN mt después de ser cortado con EcoR I y Bgl II y confrontado con el ADN mt total como sonda (Fig. 1, Tabla 2) mostró que B. amorpha, B. nivea e incluso P. farinosus, no fueron claramente resueltos de la diversidad del genoma mitocondrial de B. bassiana. Este resultado puede ser interpretado como que estas son especies altamente relacionadas compartiendo un genoma mitocondrial ancestral o que B. amorpha y B. nivea están relacionadas con B. bassiana como sub-species. Vale la pena mencionar que esta similitud no es del todo sorprendente ya que todos los HE discutidos aquí son miembros de la familia Clavicipitacea. Más análisis a nivel del genoma nuclear y mitocondrial son necesarios para soportar una u otra hipótesis.
La metodología presentada aquí mostró un mayor grado de variabilidad en el genoma mitocondrial de B. bassiana que la variabilidad antes reportada, indicando además que deben existir aun más mito-tipos en la naturaleza que no han sido descritos todavía. El poder de discriminación de la técnica de RFLP mitocondrial (10 mito-tipos luego del análisis de 20 cepas) fue más grande que las técnicas previamente reportadas para analizar el polimorfismo genómico de B. bassiana. Por ejemplo el análisis usando ITS PCR-RFLP, mostró 24 haplotipos de 96 aislamientos de B. bassiana analizados (Coates et al. 2001) y seis haplotipos de 95 cepas (Gaitán et al. 2002). Por otra parte la utilización de marcadores iso-enzimáticos presentaron 39 haplo-tipos luego de analizar 138 cepas de B. basssiana (St. Leger et al. 1992) y 11 haplo-tipos dentro de 45 aislamientos analizados (Poprawski et al. 1988). Grupos de compatibilidad vegetativa (Couteaudier y Viaud 1997) y el análisis con RAPDS (Gaitán et al. 2002), han sido empleadas también como técnicas de alta resolución para identificar polimorfismos en B. bassiana. Catorce grupos obtenidos del análisis de 25 cepas de B. bassiana fueron reportados por Couteaudier y Viaud (1997). Mientras que Gaitán et al. (2002) reportaron que no encontraron ninguna cepa con un patrón similar al ser analizadas con cinco iniciadores dentro del protocolo de RAPDS. Sin embargo, la necesidad de aislar mutantes auxotróficos para el análisis de los grupos de compatibilidad vegetativa hace de éste un procedimiento muy largo y tecnológicamente inadecuado para ser empleado a nivel industrial. Similarmente, la falta de reproducibilidad inherente al procedimiento de RAPDS (Dietrich et al. 1999) hace esta técnica no viable para la identificación y protección de cepas de interés comercial para la industria. Finalmente, vale la pena mencionar que la utilización de marcadores de polimorfismo empleando secuencias teloméricas parece tener un gran potencial para la tipificación de B. bassiana como bien lo demostraron Viaud et al. (1996) y Padmavathi et al. (2003) al obtener nueve y 16 haplo-tipos luego del análisis de nueve y 17 cepas de B. bassiana respectivamente.
Vale la pena mencionar que la utilización de técnicas diseñadas para la determinación de los patrones polimórficos que pueda presentar una especie tan importante en el control biológico de plagas como es Beauveria bassiana, es muy importante en el contexto de la entomología aplicada, por dos razones fundamentalmente: En primer lugar este tipo de técnicas nos permiten proteger una cepa particular que tenga un interés comercial, ya que se podría determinar la huella molecular única de cada cepa permitiendo su identificación y posterior protección. Por otra parte, la creciente inquietud de las agencias reguladoras de los registros para la venta y comercialización de productos comerciales, basados en agentes de control biológico en general y hongos entomopatógenos en particular, hace cada vez más necesarios los estudios relacionados con efectos adversos de estos agentes en el ecosistema. Dichos estudios necesitan metodologías que nos permitan rastrear las cepas que estamos empleando en campo para poder monitorear tales efectos, lo cual es posible realizar mediante la identificación de patrones polimórficos específicos para cada cepa evaluada. La determinación de patrones únicos, nos permitirá confirmar la permanencia de una cepa en campo por un periodo de tiempo específico, o el aislamiento o no de una cepa de hongo entomopatógeno de interés de hospederos no blanco, solo por citar un par de ejemplos.
Diferencias en el ADN mt entre especie de Metarhizium y otras especies de HE fueron previamente mencionadas en el trabajo de Hegedus y Khachatourians (1993), al indicar que sondas de ADN mt de B. bassiana hibridizaron fácilmente con aislamientos de Beauveria, Lecanicillium y Paecylomices pero no con Metarhizium. El análisis filogenético entre diferentes hongos filamentosos empleando la secuencia del gen mitocondrial 16s srARN agrupó a M. anisopliae y C. paradoxa con algunos hongos micoparásitos (Fig. 2). Es de esperar que especies de hongos compartiendo la estrategia de entomopatogenicidad dentro de su ciclo de vida estén más estrechamente relacionadas entre si que con hongos micoparásitos. Dado que este estudio mostró que éste no es el caso, nos atrevemos a sugerir que la estrategia de parasitismo de insectos proviene de por lo menos dos raíces ancestrales en hongos filamentosos. Sin embargo, un análisis incluyendo un rango más amplio de grupos taxonómicos y genes de origen nuclear debe ser llevado a cabo, con el objeto de obtener una conclusión más robusta en este sentido.
Conclusiones
Contrario a reportes previos (Hegedus y Khachatourians 1993), este estudio demostró que el ADN mt de B. bassiana es altamente variable. Por lo tanto, esta nueva valoración de la variabilidad del genoma mitocondrial de B. bassiana nos permite concluir que la metodología presentada aquí posee una alta capacidad de resolución para llevar a cabo una tipificación genómica en Beauveria. Su utilización en conjunción con aproximaciones similares desde el punto de vista tecnológico (e. g. marcadores teloméricos) deben servir para diferenciar entre la mayoría sino entre todas las cepas de las diferentes especies del genero Beauveria. Dicho sistema debe ser del interés de productores comerciales para proteger derechos de propiedad de cepas de importancia comercial. Adicionalmente, el alto nivel de polimorfismo detectado en cepas de B. bassiana también sugiere que la mito-tipificación, en conjunto con el análisis de secuencias como el gen 16s srARN es una herramienta adecuada al momento de elucidar relaciones evolutivas dentro de los hongos entomopatógenos.
Agradecimientos
Los autores manifiestan su agradecimiento a la Facultad de Estudiantes Graduados así como al programa de becas del Research and Agricultural Biotechnology Initiative de la Universidad de Saskatchewan por el apoyo financiero ofrecido a Daniel Uribe. Así mismo queremos agradecer muy especialmente al Dr. Richard H. Humber (Colección de Hongos Entomopatogenos de la USDA) por proporcionar todas las cepas ARSEF usadas en este estudio con la excepción de la cepa ARSEF 3113 que fue muy amablemente proporcionada por Dr. Leslie C. Lewis.
Literatura Citada
ALTSCHUL, S. F.; MADDEN, T. L.; SCHAFFER, A. A.; ZHANG, J.; ZHANG, Z.; MILLER, W.; LIPMAN, D. J. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Research 25: 3389-3402. [ Links ]
BIDOCHKA, M. J; MENZIES, F. V.; KAMP, A. M. 2002. Genetic groups of the insect-pathogenic fungus Beauveria bassiana are associated with habitat and thermal growth preferences. Archives in Microbiology 178: 531-537. [ Links ]
BIDOCHKA, M. J.; KAMP, A. M.; LAVENDER, T. M.; DEKONING, J.; CROOS, J. N.A. DE. 2001. Habitat association in two genetic groups of the insect pathogenic fungus Metarhizium anisopliae: Uncovering cryptic species. Applied and Environmental Microbiology 67: 1335-1342. [ Links ]
BURNS, T. D.; WHITE, T. J.; TAYLOR, J. W. 1991. Fungal molecular systematics. Annual Review of Ecology and Systematics 22: 525- 564. [ Links ]
COATES, B. S.; HELLMICH, R. L.; LEWIS, L. C. 2001. Beauveria bassiana haplotype determination based on nuclear rDNA internal transcribed spacer PCR-RFLP. Mycological Research 105: 40-50. [ Links ]
COUTEAUDIER, Y.; VIAUD, M. 1997. New insights into population structure of Beauveria bassiana with regard to vegetative compatibility groups and telomeric restriction fragment length polymorphisms. FEMS Microbiology Ecology 22: 175-182. [ Links ]
CUMMINGS, D. J.; MCNALLY, K. L.; DOMENICO, J. M.; MATSUURA, E. T. 1990. The complete DNA sequence of the mitochondrial genome of Podospora anserina. Current Genetics 17: 375-402. [ Links ]
DIETRICH, W. F.; WEBER, J. L.; NICKERSON, D. A.; KWOK, P. Y. 1999. Identification and analysis of DNA polymorphisms, pp. 135-154. En: Birren, B.; Green, E.C.; Hieter, P.; Klapholz, S.; Myers, R. M.; Rietman, H.; Roskams, J. (eds.). Genome analysis: A laboratory manual. Cold Spring Harbor Laboratory Press. N.Y. 494 p. [ Links ]
EGGER, K. N.; DANIELSON, R. M.; FORTIN, J. A. 1991. Taxonomy and population structure of E-strain mycorrhizial fungi inferred from ribosomal and mitochondrial DNA polymorphisms. Mycological Research 95: 866-872. [ Links ]
FELSENSTEIN, J. 1989. PHYLIP- Phylogeny Interference Package (Version 3.2). Cladistics 5: 164-166. [ Links ]
FENG, M. G.; POPRAWSKI, T. J.; KHACHATOURIANS, G. G. 1994. Production formulation application of the entomopathogenic fungus Beauveria bassiana for insect control worldwide. Biocontrol Sciences and Technology 4: 3-34. [ Links ]
GAITÁN, A.; VALDERRAMA, A. M.; SALDARRIAGA, G.; VÉLEZ, P.; BUSTILLO, A. 2002. Genetic variability of Beauveria bassiana associated with the coffee berry borer Hypothenemus hampei and other insects. Mycological Research 106: 1307-1314. [ Links ]
GROSSMAN, L. I.; HUDSPETH, M. E. S. 1985. Fungal mitochondrial plasmids, pp. 65-103. En: Bennett, J. W.; Lasure, L. L. (eds.). Gene Manipulations in Fungi. Academic Press, New York. [ Links ]
GORDON, T. R.; OKASOTO, D. 1992. Variation in mitochondrial DNA among vegetatively compatible isolates of Fusarium oxysporum. Experimental Mycology 16: 245-250. [ Links ]
HEGEDUS, D. D.; KHACHATOURIANS, G. G. 1993. Identification of molecular variants in mitochondrial DNAs of members of the genera Beauveria, Verticillium, Paecilomyces, Tolypocladium, and Metarhizium. Applied and Environmental Microbiology 59: 4283-4288. [ Links ]
HEGEDUS, D. D. ; KHACHATOURIANS, G. G. 1996. Identification and differentiation of the entomopathogenic fungus Beauveria bassiana using polymerase chain reaction and single-strand conformation polymorphism analysis. Journal of Invertebrate Pathology 67: 289-299. [ Links ]
HEGEDUS, D. D.; PFEIFER, T. A; MACPHERSON, J. M.; KHACHATOURIANS, G. G. 1991. Cloning and analysis of five mitochondrial tRNA-encoding genes from the fungus Beauveria bassiana. Gene. 109: 149-154. [ Links ]
HEGEDUS, D. D.; T. A. PFEIFER; D. S. MULYK; G. G. KHACHATOURIANS. 1998. Characterization and structure of the mitochondrial srRNA gene of the entomopathogenic fungus Beauveria bassiana. Genome 41: 471-476. [ Links ]
HIGGINGS, D.; THOMPSON, J.; GIBSON, T.; THOMPSON, J. D.; HIGGINGS, D. G.; GIBSON, T. J. 1994. Clustal W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acid Research 22: 4673-4680. [ Links ]
HILLIS, D. M.; MABLE, B. K.; LARSON, A.; DAVIS, S. K.; ZIMMER, E. A. 1996. Nucleic acids IV: Sequencing and cloning, pp. 321-381. En: Hillis D. M; Moritz, C.; Mable, B. K. (eds.). Molecular Systematics. Sinauer Associates, Massachusetts, USA. [ Links ]
HUANG, B.; LI, CH.; LI, Z.; FAN, M.; LI, Z. 2002. Molecular identification of the teleomorph of Beauveria bassiana. Mycotaxon 71: 229-236. [ Links ]
INGLIS, D. G.; GOETTEL, S. M.; BUTT, M. T.; STRASSER, H. 2001. Use of hypomycetous fungi for managing insect pests, pp. 23-69. En: Butt, T. M.; Jackson, C.; Magan N. (eds.). Fungi as biocontrol agents. CAB International. Wallingford. [ Links ]
JENG, R. S.; DUCHESNE, L. C.; SABOURIN, M.; HUBBES, M. 1991. Mitochondrial DNA restriction fragment length polymorphisms of aggressive and non/aggressive isolates of Ophiostoma ulmi. Mycological Research 95: 537-542. [ Links ]
KANG, H.; HAMASAKI, D. 2002. Maintenance of mitochondrial integrity, repair and degradation. Current Genetics 41: 311-322. [ Links ]
KAWANO, S.; TAKANO, H.; KUROIWA, T. 1995. Sexuality of mitochondria: fusion, recombination, and plasmids. International Review of Cytology 161: 49-110. [ Links ]
KHACHATOURIANS, G. G.; URIBE, D. 2004. Genomics of entomopathogenic fungi, pp. 353-377. En: Arora, D. K; Khachatourians, G. G. (eds.). Elsevier Science. London. Applied Mycology and Biotechnology Vol 4. 434 p. [ Links ]
KHACHATOURIANS, G. G. ; VALENCIA, E.; MIRANPURI, G. S. 2002. Beauveria bassiana and other entomopathogenic fungi in the management of insect pests, pp. 239-275. En: Koul, O.; Dhaliwal, G,S, (eds.). Microbial Biopesticides. Vol. 2. Reading, United Kingdom: Harwood Academic Publishers. [ Links ]
KOSIR, J. M.; MACPHERSON, J. M.; KHACHATOURIANS, G. G. 1991. Genomic analysis of virulent and less virulent strain of the entomopathogenic fungus Beauveria bassiana, using restriction fragment length polymorphisms. Canadian Journal of Microbiology 37: 534-541. [ Links ]
KOUVELIS, V. N.; GHIKAS D. V.; TYPAS, M. A. 2004. The analysis of the complete mitochondrial genome of Lecanicillium muscarium (synonym Verticillium lecanii) suggests a minimum common gene organization in mtDNAs of Sordariomycetes: phylogenetic implications. Fungal Genetics and Biology 41: 930- 940. [ Links ]
KOUVELIS, V. N.; ZARE, R.; BRIDGE, P. D.; TYPAS, M. A. 1999. Differentiation of mitochondrial subgroups in the Verticillium lecanii species complex. Letters in Applied Microbiology 28: 263-268. [ Links ]
MAURER, P.; COUTEAUDIER, Y.; GIRARD, P. A.; BRIDGE, P. D.; RIBA, G. 1997. Genetic diversity of Beauveria bassiana and relatedness to host insect range. Mycological Research 101: 159- 164. [ Links ]
MAVRIDOU, A.; TYPAS, M. A. 1998. Intraspecific polymorphism in Metarhizium anisopliae var. anisopliae revealed by analysis of rRNA gene complex and mtDNA RFLPs. Mycological Research 102: 1233-1241. [ Links ]
PADMAVATHI, J.; UMA DEVI, K.; RAO, U. M.; REDDY, N. N. R. 2003. Telomere fingerprinting for assessing chromosome number, isolate typing and recombination in the entomopathogen Beauveria bassiana. Mycological Research 107: 572-580. [ Links ]
PFEIFER, T. A.; KHACHATOURIANS, G. G. 1989. Isolation and characterization of DNA from the entomopathogen Beauveria bassiana. Experimental Mycology 13: 392-402. [ Links ]
PFEIFER, T. A.; HEGEDUS, D. D.; KHACHATOURIANS, G. G. 1993. The mitochondrial genome of the entomopathogenic fungus Beauveria bassiana: Analysis of the ribosomal RNA region. Canadian Journal of Microbiology 39: 25-31. [ Links ]
POPRAWSKI, T. J.; RIBA, G.; JONES, W. A.; AIOUN, A. 1988. Variation in isosterase profiles of geographical populations of Beauveria bassiana (Deuteromycotina: Hyphomycetes) isolated from Sitona weevils (Coleoptera: Curculionidae). Environmental Entomology 17: 275-279. [ Links ]
SAMBROOK, J.; RUSSELL D. W. 2001. Molecular cloning, a laboratory manual.3rd edn. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. [ Links ]
ST. LEGER, R. J.; ALLEE, L. L.; MAY, B; STAPLES, R. C; ROBERTS, D. W. 1992. World wide distribution of genetic variation among isolates of Beauveria spp. Mycological Research 96: 1007-1015. [ Links ]
TAYLOR, J. W. 1986. Fungal evolutionary biology and mitochondrial DNA. Experimental Mycology 10: 259-269. [ Links ]
VIAUD, M.; COUTEAUDIER, Y.; RIBA, G. 1998. Molecular analysis of hypervirulent somatic hybrids of the entomopathogenic fungi Beauveria bassiana and Beauveria sulfurescens. Applied and Environmental Microbiology 64: 88-93. [ Links ]
VIAUD, M.; COUTEAUDIER, Y.; LEVIS, C.; RIBA, G. 1996. Genomic organization in Beauveria bassiana: electrophoretic karyotype, gene mapping, and telomeric fingerprint. Fungal Genetics and Biology 20: 175-183. [ Links ]
WANG, C-S.; SHAH, F. A.; PATEL, N.; LI, Z-Z.; BUTT, T. M. 2003. Molecular investigation on strain genetic relatedness and population structure of Beauveria bassiana. Environmental Microbiology 5: 908-915. [ Links ]
ZONG-QUI, L.; AI-YING, L.; JIE-LING, L. 1991. A new species of the genus Cordyceps and its Metarhizium anamorph. Acta Mycologica Sinica 10: 257-262. [ Links ]
Recibido: 20-nov-2006 - Aceptado: 23-ago-2007














