Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Entomología
Print version ISSN 0120-0488On-line version ISSN 2665-4385
Rev. Colomb. Entomol. vol.36 no.2 Bogotá July/Dec. 2010
Nota científica
Optimización de un procedimiento de extracción de ADN para mosquitos anofelinos
Optimization of a DNA extraction procedure for anopheline mosquitoes
DORIS A. ROSERO1,2, LINA A. GUTIÉRREZ1,3, ASTRID V. CIENFUEGOS1,4, LUZ M. JARAMILLO1,5 y MARGARITA M. CORREA1,6
1 Grupo de Microbiología Molecular, Escuela de Microbiología, Universidad de Antioquía. Calle 67 No. 53-108, 5- 437. Medellín, Colombia.
2 Estudiante de Maestría en Biología, roserodoris@hotmail.com
3 Ph. D. en Ciencias Básicas Biomédicas. liangutibui@gmail.com
4 M. Sc. en Ciencias Básicas Biomédicas.vanessa.cienfuegos@gmail.com
5 Estudiante de Maestría en Biología. luzmajara@guajiros.udea.edu.co
6 Ph.D, mcorrea@quimbaya.udea.edu.co. Autor paracorrespondencia
Recibido: 29-may-2009 • Aceptado: 5-mar-2010
Resumen: Se optimizó un protocolo para la extracción de ADN a partir de mosquitos anofelinos, evaluando varios tiempos de incubación de la muestra con el acetato de potasio (AcK). Los resultados evidenciaron que la mayor concentración de ADN y frecuencia de amplificación se obtuvieron al utilizar el tratamiento de 1 h, lo que sugiere que es posible reducir el tiempo de incubación con AcK sin afectar la cantidad y calidad del ADN extraído.
Palabras clave: Anopheles. PCR. Acetato de Potasio.
Abstract: A DNA extraction protocol for anopheline mosquitoes was optimized by the evaluation of various incubation times of the sample with Potassium acetate (KAc). The results showed that the highest DNA concentration and amplification frequency were obtained with the 1 h treatment, which suggests that is possible to reduce the incubation time with KAc without affecting the amount and quality of the DNA extracted.
Key words: Anopheles. PCR. Potassium Acetate.
Introducción
Los estudios genéticos con artrópodos requieren ADN de buena calidad. En dichos estudios se deben procesar un gran número de muestras (Nodarse et al. 2004; Gutiérrez et al. 2008), por lo que es indispensable contar con un método de extracción eficiente, rápido y económico. En ocasiones se ha reportado la utilización de partes del mosquito como fuente de ADN para la amplificación por PCR, lo cual permite omitir el proceso de extracción; en dicho caso, las patas o alas son adicionadas directamente a la reacción de amplificación (Zapata et al. 2007; Cienfuegos et al. 2008 ); sin embargo, se ha observado que algunos marcadores, como los mitocondriales, Citocromo Oxidasa Subunidad I (COI) y NADH Deshidrogenasa Subunidad 5 (ND5), no amplifican eficientemente a partir de pata o alas (Gutierrez L, com. pers.).
Se han descrito varios métodos para la extracción del ADN a partir de mosquitos; uno de los más utilizados usa fenol cloroformo (Linton et al. 2001; Zapata et al. 2007); este método es laborioso y requiere usar solventes orgánicos, lo que representa un riesgo para la salud humana y el ambiente, principalmente por las dificultades para la eliminación de estos reactivos (Niu et al. 2008; Valter et al. 2009). Además, se ha reportado que la presencia de los residuos de fenol puede contaminar el producto de extracción e inhibir la amplificación (Rantakokko-Jalava y Jalava 2002). Otros métodos de extracción involucran el uso de aditivos como: Chelex (Angêlla et al. 2007), tampón Cetyl Trimethyl Ammonium Bromide - CTAB, Sarcosil (Lardeux et al. 2008) y proteinasa K (Nodarse et al. 2004), reactivos que pueden aumentar los costos del proceso (Valter et al. 2009).
Un método reportado inicialmente para la extracción de ADN de Drosophila melanogaster Meigen, 1830 (Bender et al. 1983), fue modificado y utilizado para la extracción de material genético de mosquitos Aedes albopictus Skuse, 1894 (Birungi y Munstermann 2002), siendo también de utilidad para mosquitos Anopheles spp. (Gutiérrez et al. 2008, 2009); entre sus ventajas están el no utilizar solventes orgánicos, no requerir reactivos costosos y ser sencillo de ejecutar. Un paso importante en el procedimiento es la precipitación de proteínas con acetato de potasio (AcK), el que se adiciona a la muestra a una concentración de 1M y se incuba en hielo a -20°C durante 12 horas, prolongando el tiempo del proceso. Lo anterior es una desventaja de dicho protocolo pues toma en promedio dos días de trabajo. En la literatura se han reportado protocolos que utilizan otros tiempos de incubación con el AcK: 10 min (Linton et al. 2001; Bisset et al. 2005), 15 min (Beebe et al. 2000; Nodarse et al. 2004), 30 min (Kampen et al. 2003; De Armas et al. 2005) y 60 min (Nodarse et al. 2004; Golczer y Arrivillaga 2008). Estos estudios muestran la posibilidad de modificar el tiempo de incubación con el AcK. Por esta razón, en el presente trabajo se optimizó el protocolo mediante la disminución de dicho tiempo de incubación, con el fin de agilizar la extracción, obteniendo ADN de buena calidad y cantidad, para ser empleado en diferentes estudios moleculares de anofelinos.
Materiales y Métodos
Para la extracción del ADN se utilizaron abdómenes de mosquitos Anopheles (Nyssorhynchus) albimanus Wiedemann, 1820, recolectados entre octubre y noviembre de 2006 en la localidad de Santa Rosa de Lima (Bolívar) y fueron identificados utilizando claves taxonómicas (Faran y Linthicum 1981; Suarez et al. 1988; Wilkerson 1993). Para trabajar con una muestra homogénea, se definió el tamaño de los abdómenes .en 2 mm ± 0,04 DS. Con este criterio, se seleccionaron 54 abdómenes completos que se mantuvieron preservados en etanol al 95%.
Se utilizó un muestreo no probabilístico por conveniencia. Se realizó un diseño experimental de comparación de bloques completamente aleatorizados; se definieron tres bloques, cada uno conformado por 18 abdómenes de mosquitos, y estos se procesaron con una periodicidad de uno por día. Los ensayos se realizaron en días diferentes; con tres repeticiones por cada tratamiento (T), cada uno correspondiente a un tiempo de incubación con AcK 1M, así: T1=15 min, T2=30 min, T3=1 h, T4=3 h, T5=6 h y T6=12 h. El tiempo de 12 h se utilizó como control de tratamientos debido a que es el tiempo reportado en el protocolo (Birungi y Munstermann 2002). Se definió como variable independiente cada uno de los tratamientos evaluados (T1-T6) y como variables dependientes: concentración, calidad de ADN y amplificación por PCR-ITS2.
Para la extracción del ADN, los abdómenes fueron macerados y homogenizados individualmente en viales de 1,5 mL con 100 μL de tampón de lisis (NaCl 0,1 M; Sucrosa 0,1 M; Tris 0,1 M; EDTA 0,05 M; SDS 0,5%, pH 9,1) y se incubaron por 30 min a 65°C, para facilitar la ruptura de los tejidos y membranas celulares. Luego se adicionó 14,5 μL de una solución de AcK 8M para obtener una concentración final de AcK 1M y se incubaron en los diferentes tiempos: T1=15 min, T2=30 min, T3=1 h, T4=3 h, T5=6 h y T6=12 h. Posteriormente, las muestras se centrifugaron a 13.000 rpm, durante 15 min, a 4°C y el sobrenadante se transfirió a un tubo nuevo. A continuación, se realizaron tres pasos de precipitación y subsiguiente centrifugación a 13.000 rpm y 4°C, así: 100 mL etanol absoluto, centrifugando por 15 min, 100 mL etanol frío al 70% por 5 min, y nuevamente, 100 μL etanol absoluto, por 5 min. Finalmente, se dejó secar el botón por un tiempo mínimo de 30 min a temperatura ambiente y se resuspendió en 25 μL de tampón TE 1X (Tris 10 mM, EDTA 1 mM, pH 7,6). Las muestras se incubaron a 65°C por 10 min, para garantizar la resuspensión del ADN extraído y se almacenaron a 4°C.
Se prepararon tres diluciones 1:100 de cada muestra. La concentración de ADN se determinó a una densidad óptica A260; la pureza de las muestras se estimó utilizando la relación A260/280 (Clark y Christopher 2001). Se amplificó el espaciador interno transcrito 2 (ITS2) del ADN ribosomal, como una forma adicional de valorar y validar la calidad y cantidad del ADN extraído, utilizando 2 uL del producto de extracción y las condiciones de PCR descritas anteriormente (Zapata et al. 2007; Cienfuegos et al. 2008). Los productos de la PCR-ITS2 se visualizaron mediante una electroforesis en gel de agarosa al 0,8%, con tinción de bromuro de etidio (5μg/mL).
La comparación de la concentración de ADN e índice de pureza entre los diferentes tratamientos se realizó calculando promedios, desviaciones estándar (DS) y Análisis de Varianza (ANOVA) de un factor. Para identificar los tratamientos que presentaron diferencias significativas en la concentración de ADN se utilizaron las pruebas de Bonferroni y Dunnett (Díaz 1999; Molina et al. 2006). Para el análisis de las amplificaciones del marcador ITS2, se determinó el porcentaje de amplificación y se utilizó la prueba de Chi-cuadrado (X2) para establecer si existían diferencias significativas entre los porcentajes de amplificación obtenidos en los diferentes tratamientos (Halos et al. 2004). Para el análisis estadístico se utilizó el software SPSS para Windows, versión 15 (Inc. SPSS 2008).
Resultados y discusión
Se logró reducir el tiempo de procesamiento de las muestras al disminuir el tiempo de incubación con el AcK. Este logro es importante dado que en la actualidad muchos de los estudios con vectores de enfermedades involucran la extracción de ADN a partir de los especímenes; material que es utilizado para la realización de pruebas moleculares que permitan llevar a cabo diversos tipos de análisis, entre ellos: la confirmación molecular de especies anofelinas que presentan dificultades en su identificación por caracteres morfológicos (Zapata et al. 2007; Cienfuegos et al. 2008), estudios de genética poblacional (Gutiérrez et al. 2009) y filogenia (Beebe et al. 2000).
La cuantificación de los productos de extracción mostró que las concentraciones más altas correspondían al tratamiento tres, con valores entre 489 y 597 ng/uL (Tabla 1). Al aplicar la prueba t de Dunnett para comparar los tratamientos T1 a T5 con el tratamiento control (T6), no se observaron diferencias significativas (P > 0,05) en las concentraciones de ADN: T1 (P = 0,116), T2 (P = 0,116), T3 (P = 0,996), T4 (P = 0,107) y T5 (P = 0,645). Sin embargo, al aplicar la prueba de Bonferroni, si se encontró una diferencia significativa entre el tratamiento T3 con respecto a los tratamientos T1 (P = 0,019), T2 (P = 0,019) y T4 (P = 0,017), lo que indicó que el tratamiento T3 o la incubación con AcK por 1 h podría utilizarse en la extracción para obtener una alta concentración de ADN.
Los valores del índice A260/A280 se encontraron entre 1,5 y 2,0 (Tabla 1) y no mostraron diferencias significativas entre los tratamientos (F = 0,937, P = 0,466); lo que sugiere que cualquiera de los tiempos de incubación con el AcK (T1-T6), pueden ser de utilidad para obtener ADN de buena calidad. Lo anterior permite tener una mayor flexibilidad en el desarrollo de los experimentos, al poder ajustar estos tiempos según conveniencia del investigador. La calidad de ADN es una característica importante, dado que de ella depende en gran parte el éxito de los ensayos posteriores, puesto que incide en la estabilidad del ADN durante su almacenamiento por tiempo indefinido, conservando su estructura y propiedades físico-químicas (Sambrook y Russell 2001).
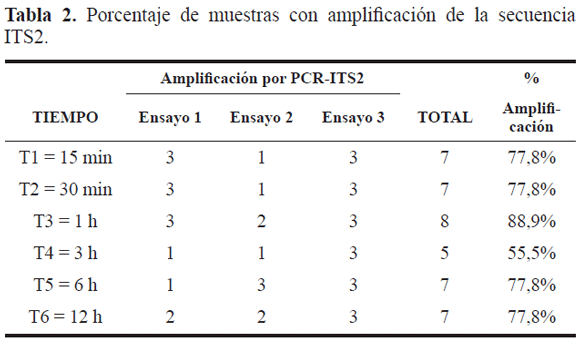
La amplificación de la región ITS2 fue un indicador adicional de la calidad y cantidad del ADN extraído. Se obtuvo un producto de PCR de 500 pb aproximadamente (Fig. 1), que es el tamaño esperado para este marcador en A. albimanus (Zapata et al. 2007; Cienfuegos et al. 2008). Como se observó con los resultados de concentración, el mayor porcentaje de amplificación de la región ITS2 se obtuvo con el tratamiento T3, con el cual amplificaron ocho de las nueve muestras incubadas con el AcK durante 1 h (89,9%, Tabla 2). No hubo diferencia entre los tratamientos para los valores de amplificaciones (X2, P > 0,05). Lo anterior reitera la utilidad de este tiempo de incubación con AcK para disminuir el tiempo de extracción.
En general, la extracción de ADN es un procedimiento sencillo, sin embargo, en el caso de insectos como los anofelinos, el producto final puede ser poco satisfactorio por la escasa cantidad de material obtenida de un solo mosquito. Otros factores que influyen en la obtención de ADN de buena calidad y cantidad son los procedimientos de recolección, transporte y preservación de los especímenes (Rivero et al. 2007). Se han reportado diferentes temperaturas para el almacenamiento del producto de extracción como son: 4ºC, -20ºC y -80ºC (Birungi y Munstermann 2002; Bisset et al. 2005). En ensayos preliminares de la cuantificación del ADN por espectrofotometría, se observó que se obtenían concentraciones más altas de ADN a partir de muestras almacenadas a 4ºC que de aquellas a -20ºC. Aunque se conoce que la congelación es ideal para almacenar muestras de ADN a largo plazo, es importante tener en cuenta que los procesos de congelación y descongelación promueven la contaminación y la degradación del ADN (Gómez y Uribe 2007); por esta razón, una buena estrategia es conservar el material extraído a 4ºC y realizar los análisis posteriores en un tiempo no mayor a una semana y además, almacenar una alícuota a -20°C.
En los protocolos reportados en la literatura para la extracción de ADN a partir de anofelinos y de otros insectos de importancia médica o veterinaria, se pueden encontrar tiempos de incubación con el AcK que son menores o iguales a los evaluados en este estudio (Beebe et al. 2000; Linton et al. 2001; De Armas et al. 2005; Golczer y Arrivillaga 2008); sin embargo, las ventajas del protocolo optimizado consisten principalmente en la disminución del tiempo para el procesamiento de las muestras y el no requerir reactivos adicionales que impliquen el aumento de los costos o riesgos para la salud humana o el ambiente. Este sería de gran utilidad para laboratorios en países endémicos para enfermedades tropicales como la malaria, pues en estos generalmente se tiene mayor acceso a protocolos más sencillos, que a menudo son laboriosos o requieren reactivos nocivos. Este método posibilitaría el procesamiento más rápido de un gran número de mosquitos y podría ser estandarizado para la extracción de ADN a partir de otros artrópodos de importancia médica.
Agradecimientos
Este trabajo fue anidado a proyectos en ejecución del grupo de Microbiología Molecular, códigos: 8700-1416 y 8700- 039, financiados por el Comité para el Desarrollo de la Investigación –CODI– Universidad de Antioquia (a MMC), y contribuyó a la formación en investigación de la Microbióloga DAR, durante sus prácticas académicas y Servicio Social Obligatorio realizado con apoyo de Estrategia para la Sostenibilidad 2007-2008, código BUPP: E01335 y Escuela de Microbiología, Universidad de Antioquia.
Literatura citada
ANGÊLLA, A.; GIL, L.; DA SILVA, L.; RIBOLLA, P. 2007. Population structure of the malaria vector Anopheles darlingi in Rondônia, Brazilian Amazon, based on mitochondrial DNA. Memórias do Instituto Oswaldo Cruz, Rio de Janeiro 102(8): 953-9. [ Links ]
BEEBE, N.W.; COOPER, R.D.; MORRISON, D.A.; ELLIS, J.T. 2000. A Phylogenetic Study of the Anopheles punctulatus Group of Malaria Vectors Comparing rDNA Sequence Alignments Derived from the Mitochondrial and Nuclear Small Ribosomal Subunits. Molecular Phylogenetics and Evolution 17(3): 430-436. [ Links ]
BENDER, W.; SPIERER, P.; HOGNESS, D.S. 1983. Chromosomal walking and jumping to isolate DNA from the ace and rosy loci and the bi thorax complex in Drosophila melanogaster. Journal of Molecular Biology 168: 17-34. [ Links ]
BIRUNGI, J.; MUNSTERMANN, L.E. 2002. Genetic Structure of Aedes albopictus (Diptera: Culicidae) Populations Based on Mitochondrial ND5 Sequences: Evidence for an Independet Invasion into Brazil and United States. Annals of the Entomological Society of America 95(1): 125-132. [ Links ]
BISSET, J.A.; RODRÍGUEZ, M.; DE ARMAS, Y. 2005. Comparación de 2 poblaciones de mosquitos Aedes aegypti de Santiago de Cuba con diferente conducta de reposo. Revista Cubana de Medicina Tropical 57(2): 143-150. [ Links ]
CIENFUEGOS, A.; CÓRDOBA, L.; GÓMEZ, G.; LUCKHART, S.; CONN, J.; CORREA, M. 2008. Diseño y evaluación de metodologías basadas en PCR-RFLP de ITS2 para la identificación molecular de mosquitos Anopheles spp. (Diptera: Culicidae) de la Costa Pacífica de Colombia. Biomédica (México) 19(1): 35- 44. [ Links ]
CLARK, W; CHRISTOPHER, K. 2001. Chapter 5. An Introduction to DNA: Spectrophotometry, Degradation, and the ´Frankengel´ Experiment. Tested studies for laboratory teaching 22: 81-99. [ Links ]
DE ARMAS, Y.; RODRÍGUEZ M.M.; BISSET, J.A. 2005. Modificación de un método de extracción de ADN genómico de Aedes aegypti (Diptera: Culicidae). Revista Colombiana de Entomología 31: 203-6. [ Links ]
DIAZ, A. 1999. Diseño Estadístico de Experimentos. Editorial Universidad de Antioquia, Medellín. 260 p. [ Links ]
FARAN, M.E.; LINTHICUM, K.J. 1981. A handbook of the Amazonian Species of Anopheles (Nyssorhynchus) (Diptera: Culicidae) Mosquito Systematics 13(1): 1-81. [ Links ]
GOLCZER, G.; ARRIVILLAGA, J. 2008. Modificación de un protocolo estándar de extracción de ADN para flebotominos pequeños (Phlebotominae: Lutzomyia). Revista Colombiana de Entomología 34(2): 199-202. [ Links ]
GÓMEZ, M.; URIBE, S. 2007. Preservación y almacenamiento de ADN de mariposas utilizando papel filtro. Revista Colombiana de Entomología 33(2): 191-196. [ Links ]
GUTIÉRREZ, L.; NARANJO, N.; CIENFUEGOS, A.; MUSKUS, C.; LUCKHART, S.; CONN, J.; CORREA M. 2009. Population structure analyses and demographic history of the malaria vector Anopheles albimanus from the Caribbean and the Pacific regions of Colombia. Malaria Journal 8: 259-276. [ Links ]
GUTIÉRREZ, L.; NARANJO, N.; JARAMILLO, L.; MUSKUS, C.; LUCKHART, S.; CONN, J.; CORREA M. 2008. Natural infectivity of Anopheles species from the Pacific and Atlantic Regions of Colombia. Acta Tropica 107: 99-105. [ Links ]
HALOS, L.; JAMAL, T.; VIAL, L.; MAILLARDA, R.; C, SUAUD, A.; LE MENACH, A.; BOULOUISA, H; VAYSSIER-TAUSSATA, M. 2004. Determination of an efficient and reliable method for DNA extraction from ticks. Veterinary Research 35: 709- 713. [ Links ]
KAMPEN, H.; STERNBERG, A.; PROFT, J.; BASTIAN, S.; SCHAFFNER, F.; MAIER, W.; SEITZ, H.M. 2003. Polymerase Chain Reaction-Based differentiation of the mosquito sibling species Anopheles claviger s.s. and Anopheles petragnani (Diptera: Culicidae). The American Journal of Tropical Medicine and Hygiene 69(2): 195-199. [ Links ]
LARDEUX, F.; TEJERINA, R.; ALIAGA, C.; URSIC-BEDOYA, R.; LOWENBERGER, C.; CHAVEZ, T. 2008. Optimization of a semi-nested multiplex PCR to identify Plasmodium parasites in wild-caught Anopheles in Bolivia, and its application to field epidemiological studies. Transactions of the Royal Society of Tropical Medicine and Hygiene 102: 485-492. [ Links ]
LINTON, Y.M.; HARBACH R.E.; MOHSENG, C.; ANTHONY, T.G. AND MATUSOP, A. 2001. Morphological and molecular identity of Anopheles (Cellia) sundaicus (Diptera: Culicidae), the nominotypical member of a malaria vector species complex in Southeast Asia. Systematic Entomology 26: 357-366. [ Links ]
MOLINA, N.B; POLVERINO, D.; MINVIELLE, M.C.; APEZTEGUÍA, M.; AGUILAR, M.; BASUALDO, J.A. 2006. Comparación de métodos de lisis y extracción de ADN de trofozoítos de Giardia lamblia. Parasitología Latinoamericana (Chile) 31: 134. [ Links ]
NIU, C.; KEBEDE, H.; AULD, D.L.; WOODWARD, J.E.; BUROW, G.; WRIGHT, R. 2008. A safe inexpensive method to isolate high quality plant and fungal DNA in an open laboratory environment. African Journal of Biotechnology 7 (16): 2818- 22. [ Links ]
NODARSE, J.F; RODRÍGUEZ, J.; FUENTES, O.; CASTEX, M.; FERNÁNDEZ-CALIENES, A. 2004. Comparación entre 5 métodos para la extracción de ADN de triatomíneos: su utilización en la técnica de ADN polimórfico amplificado al azar. Revista Cubana de Medicina Tropical 56(3): 208-13. [ Links ]
RANTAKOKKO-JALAVA, K.; JALAVA, J. 2002. Optimal DNA Isolation Method for Detection of Bacteria in Clinical Specimens by Broad-Range PCR. Journal of Clinical Microbiology 40(11): 4211-17. [ Links ]
RIVERO, J.; ZOGHBI, N.; RUBIO-PALIS, Y.; URDANETA, L.; HERRERA, F. 2007. DNA degradation of Anopheles darlingi collected at high relative humidity and preserved in isopropanol. Boletín de Malariología y Salud Ambiental, XLVII (1): 149-151. [ Links ]
SAMBROOK, J.; RUSSELL, D. 2001. Molecular Cloning. A Laboratory Manual.Third Edition. Cold Spring Harbor Laboratory Press, New York. [ Links ]
SPSS Inc. SPSS versión 15.0 para Windows. Chicago, Illinois. http://www.spss.com/. Fecha de último acceso: 22 Agosto 2008. [ Links ]
SUÁREZ, M.; QUIÑONES, M.; ROBAYO, M. 1988. Clave gráfica para la determinación taxonómica de los anofelinos de Colombia. Ministerio de Salud Pública de Colombia, 57pp. [ Links ]
VALTER, L.F.; DA LUZ, G.; SILVA, E.L. 2009. Isolation of high quality DNA: a protocol combining "rennet" and glass milk. Electronic Journal of Biotechnology 12 (2): 1-6. [ Links ]
WILKERSON, R.C.; STRICKMAN, D.; FERNÁNDEZ-SALAS, L.; IBÉÑEZ-BERNAL, S.; LITWAK, T.R. 1993. Clave ilustrada para la identificación de las hembras de mosquitos anofelinos de México y Centroamerica (Diptera: Culicidae). Centro de Investigación de Paludismo, Secretaría de Salud, Tapachula, Chiapas, México. pp. 1-46. [ Links ]
ZAPATA, M.; CIENFUEGOS, A.; QUIRÓS, O.; QUIÑONES, M.; LUCKHART, S., CORREA, M. 2007. Discrimination of Anopheles species from San Pedro de Urabá-Antioquia, Colombia, by PCR-RFLP analysis of ITS sequences. The American Journal of Tropical Medicine and Hygiene 77(1): 67-72. [ Links ]