Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Entomología
Print version ISSN 0120-0488On-line version ISSN 2665-4385
Rev. Colomb. Entomol. vol.37 no.2 Bogotá July/Dec. 2011
Nota científica
Análisis de secuencias de ADN mitocondrial (Cytb y ND1) en Lucilia eximia (Diptera: Calliphoridae)
Analysis of mitochondrial DNA sequences (Cytb and ND1) in Lucilia eximia (Diptera: Calliphoridae)
PAULA ANDREA GIRALDO H.1, SANDRA INÉS URIBE S.2 y ANDRÉS LÓPEZ R.3
1 Aspirante a Maestría. Grupo de Investigación en sistemática molecular. Facultad de Ciencias, Escuela de Biociencias. Universidad Nacional de Colombia sede Medellín, Colombia. Calle 59ª # 63-20 - Bloque 16. Teléfono 57-4. 4309394. Fax. 57-4 4309394.E-mail: paulagiraldo07@gmail.com. Autora para correspondencia.
2 Ph.D. Profesora asociada Universidad Nacional de Colombia. Grupo de Investigación en sistemática molecular. Facultad de Ciencias, Escuela de Biociencias. Universidad Nacional de Colombia sede Medellín, Colombia. Calle 59ª # 63-20 - Bloque 16. Teléfono 57-4. 4309394. Fax. 57-4 4309394.E-mail: suribe@unal.edu.co.
3 Ph.D. Profesora asociada Universidad Nacional de Colombia. Grupo de Investigación en sistemática molecular. Facultad de Ciencias, Escuela de Biociencias. Universidad Nacional de Colombia sede Medellín, Colombia. Calle 59ª # 63-20 - Bloque 16. Teléfono 57-4. 4309394. Fax. 57-4 4309394.E-mail: suribe@unal.edu.co.
Recibido: 19-may-2010 - Aceptado: 7-may-2011
Resumen: Lucilia eximia (Diptera: Calliphoridae) es una especie de importancia forense debido a su asociación con cuerpos en descomposición en Antioquia y otras regiones de Colombia. La implementación de metodologías que permitan la identificación rápida y acertada de esta especie es de gran importancia en entomología forense. En la actualidad las secuencias de ADN mitocondrial son consideradas una herramienta complementaria a la taxonomía clásica y son utilizadas en la tipificación de especies morfológicamente similares o indistinguibles como en el caso de las moscas de importancia forense. En el presente trabajo se estudio la variabilidad de la región mitocondrial (Cyt b-RNAt-Ser-ND1) en un fragmento de 480 pb amplificada por PCR y cuyo ADN fue obtenido de larvas de tercer estadio de L. eximia provenientes de Medellín y dos zonas aledañas. Las diferencias en las secuencias en términos de nucleótidos y distancias genéticas permitieron separar los individuos de la especie de interés de otras especies cercanas. Así mismo, al realizar un análisis de NJ (K2P), se observó una agrupación de los haplotipos relacionada con la procedencia geográfica de los individuos.
Palabras clave: Entomología Forense. Citocromo b. Marcadores moleculares.
Abstract: Lucilia eximia (Diptera: Calliphoridae) is a species of forensic importance for its association with decomposing bodies in Antioquia and other regions of Colombia. The implementation of methods to allow the rapid and accurate identification of this species is a matter important in forensic entomology. Currently, the mitochondrial DNA sequences are considered to complement classical taxonomy and are used in the characterization of morphologically similar or indistinguishable species as in the case of forensic importance fies. We evaluated the variability of a mitochondrial region (Cytb –RNAt-Ser-ND1) of 480 bp that was amplifed by PCR, from L. eximia larvae on third stage from Medellin and two other localities. The sequence differences evaluated as nucleotidic differences and genetic distances permitted the separation of individuals from the species of interest when compared with morphologically similar and related species. Furthermore, using a N.J analysis (K2p) the haplotype groupings were related to the geographical origin of individuals from L. eximia.
Key words: Forensic Entomology. Citochrome b. Molecular markers.
Introducción
con base en caracteres morfológicos es difícil. Esta situación es cierta en el caso de los estadios inmaduros debido a la similitud de las características anatómicas. La identificación de éstos con base en herramientas como las moleculares es relevante, dado que son éstos, los estados que predominan en escenas de muerte (Wallman y Donellan 2001; Wells et al. 2007).
Las especies del género Lucilia pueden separarse con base en diferencias sutiles en los adultos, como la coloración de ciertas estructuras, el ancho relativo de la frente y la forma de los genitales en los machos (Withworth 2010). Estas características se consideran válidas en términos taxonómicos pero son difíciles de expresar de un modo objetivo sin la ayuda de un especialista e implicaría la necesidad de criar los estados inmaduros hasta adultos bajo condiciones de laboratorio para su identificación, o disponer de claves taxonómicas y especialistas y un minucioso trabajo con las larvas aumentando la dificultad de su uso potencial en forense.
Entre las metodologías más utilizadas actualmente para complementar estudios de taxonomía clásica, se encuentran aquellas de tipificación molecular a partir de secuencias de ADN. En el caso de los insectos y en particular de aquellos de importancia forense, se considera ventajoso el hecho de que las moléculas posibilitan la identificación de especímenes en cualquiera de sus estados, e incluso de partes de ellos (Benecke 1998). Esto es relevante en entomología forense, no solo para corroborar la identidad de las especies, sino también por la información que puede obtenerse aun en aquellos casos en los cuales los insectos se encuentran muertos, o afectados por los procesos de depredación propios de la sucesión y en los que el ADN es la única fuente disponible para su identificación (Sperling et al. 1994).
En este sentido, el ADN mitocondrial ha sido ampliamente sugerido y utilizado como marcador molecular; su condición haploide, herencia matrilineal, altas tasas de evolución y la presencia de regiones conservadas y variables, facilitan la tipificación molecular de individuos y su uso para la diferenciación de los mismos a diferentes escalas taxonómicas, ventajas que han sido aprovechadas en estudios con diversos grupos de insectos, incluyendo aquellos de importancia médica y forense (Sperling et al. 1994; Behura 2006).
Actualmente se han estudiado alrededor de 12 genes mitocondriales para caracterizar insectos de importancia forense (Lessinger y Azeredo-Ezpin 2000), siendo más utilizados los genes que codifican para Citocromo Oxidasa uno y dos (COI y COII) y validados como marcadores consistentes para la diferenciación a nivel de género y entre grupos monofiléticos de la familia Calliphoridae (Nelson et al. 2007; Wells y Williams 2007; Wells et al. 2007). Sin embargo, en este último se han encontrado bajos niveles de divergencia entre especies hermanas particularmente en los géneros Calliphora y Lucilia (Wallman y Donellan 2001; Wallman et al. 2005; Wells et al. 2007). Estudios que involucren especies de importancia forense con estos géneros y con este marcador no han sido publicados en Latinoamérica.
El gen Citocromo b (Cytb) está involucrado en el transporte de electrones en la cadena respiratoria de la mitocondria. La variabilidad exhibida por el gen a diferentes niveles taxonómicos y su uso exitoso como marcador molecular en conjunto con fragmentos de genes mitocondriales vecinos, lo ha convertido en uno de los más usados para análisis filogenéticos (Irwin et al. 1991). Así mismo, éste ha mostrado su utilidad en el estudio de complejos de especie, especies morfológicamente similares e incluso al interior de especies (Meyer 1994). Aunque el Cytb ha sido principalmente usado para estudiar mamíferos, también se conocen estudios de filogenia y sistemática de insectos, y en especial del orden Diptera (Simmons y Weller 2001; Torgerson et al. 2003).
El objetivo del presente trabajo fue estudiar la variabilidad molecular de un segmento de ADN mitocondrial amplificado mediante PCR, que corresponde a un segmento de la región 3 del gen Citocromo b (Cytb), el RNA de transferencia para Serina (ARNt-Ser), el espaciador intergénico (IG2) y un segmento de la región 3 del gen NADH deshidrogenasa 1 (ND1) en Lucilia eximia (Wiedemann, 1819).
Esta especie de distribución Neotropical, se ha registrado en Colombia en los departamentos de Antioquia, Bolívar, Caquetá, Cundinamarca, Chocó y Santander (Vélez y Wolff 2008), en asocio con estados tempranos de descomposición cadavérica. Estas características la convierten en una especie con gran potencial para ser utilizada en investigaciones legales y en la determinación de intervalos postmortem.
Materiales y Métodos
Obtención de especímenes. Para la obtención de las larvas se realizaron cuatro muestreos con intervalos de dos meses entre Enero y Agosto de 2006, en tres municipios del departamento de Antioquia (Tabla 1). La frecuencia de muestreo se diseñó con el fin de obtener individuos de diferentes cohortes en las poblaciones seleccionadas.
Se utilizó una trampa VanSomeren–Rydon cebada con hígado fresco de cerdo para colectar adultos y huevos de acuerdo a la metodología modificada de Stevens y Wall (1997). Posteriormente se establecieron crías de laboratorio con el fin de obtener adultos para la determinación taxonómica de la especie y larvas para la tipificación molecular. La determinación taxonómica de la especie fue realizada de acuerdo con el trabajo de filorez y Wolff (2009) y verificada por el especialista Eduardo Amat del Instituto de Investigación de Recursos Biológicos - Alexander Vo n Humboldt (Villa de Ley va, Colombia), siguiendo la propuesta de Whitworth (2010) para individuos adultos.
Como punto de comparación y para evaluar el uso potencial de la secuencias en separar los individuos de otras especies morfológicamente similares y cercanas, se utilizó una secuencia de referencia depositada en genbank correspondiente a Lucilia sericata (Meigen, 1826) y un individuo de Lucilia cuprina (Wiedemann, 1830) recolectado en la ciudad de Medellín, previamente identificado con base en la metodología de Amat et al. (2008) y verificado por el especialista Eduardo Amat, del Instituto Alexander von Humboldt (Villa de Leyva).
Análisis molecular Preparación de especímenes. Las secuencias fueron obtenidas a partir de la extracción y amplificación de ADN de larvas en tercer estadío obtenidas a partir de hembras paridas en el laboratorio. Como paso previo al procedimiento de extracción de ADN, cada individuo fue tratado con agua caliente por 10 segundos y posteriormente disectado con el fin de conservar las estructuras consideradas de importancia en la determinación taxonómica como son el esqueleto cefalofaríngeo y el segmento terminal donde se encuentran las placas espiraculares. Estas estructuras, fueron aclaradas en KOH al 10% según la metodología de Stehr (1987) y posteriormente fjadas en placa con bálsamo de Canadá. El resto fue preservado en isopropanol al 100% para la extracción de ADN. Extracción de ADN. La extracción de ADN se realizó a partir de fragmentos de 4 mm de larvas, siguiendo la metodología de buffer de macerado (Collins y Porter 1990) que ha sido utilizado con modificaciones menores en otros insectos del orden Diptera (Uribe et al.1998; Uribe 1999). La extracción del ADN y su calidad fueron confrmadas por medio de electroforesis en gel de agarosa y la concentración se estimó utilizando un nanodrop 1000 de Thermo Scientific ®. Amplificación del ADN. La amplificación se efectuó mediante la reacción en cadena de la polimerasa (PCR) utilizando un termociclador (PTC 100 MJ Researcher). Los oligonucleótidos utilizados para amplificar los fragmentos de los genes fueron CB3fic (CA(T/C)ATTCAACC(A/T)GA-ATGATA) y NINFR (GGTA(C/T)(A/T)TTGCCTCGA(T/A) TTCG(T/A)TATGA) . La reacción de PCR se llevó a cabo empleado entre 0,5 y 1,0 mL de ADN al cual se le adicionaron 4mL de MgCl2 (25mM), 0,8 mL (1mM) de cada deoxinucleótido (Adenina, Timina, Citosina y Timina), 0,4 mL de cada primer (50 mM), y 5mL de Buffer 10X (Fermentas), ajustados con agua bidestilada para un volumen final de 50 mL. Después de la fase de desnaturalización del ADN a 94°C durante tres minutos, los parámetros de amplificación para los siguientes 35 ciclos de la reacción de PCR fueron: desnaturalización por 30 segundos a 93°C, alineamiento por un minuto a 48°C y extensión por un minuto a 70°C. Los productos amplificados fueron verificados mediante electroforesis en gel de agarosa al 1% con bromuro de etidio y empleando un marcador de peso molecular de 100 pb. La visualización de los geles se realizó en cámara de U.V y estos fueron analizados mediante el programa BioDocAnalyze de BIOMETRA ®. Obtención y análisis de secuencias. La secuenciación de ADN se realizó en el Center for Disease Control CDC de Atlanta (Estados Unidos). La edición y el alineamiento de las secuencias se realizó utilizando el programa BIOEDIT Sequence Alignment Editor (Hall 1999). Para la totalidad de las secuencias provenientes de los individuos se estimó, la diversidad haplotípica (h), la diversidad nucleotídica (pi) y el valor neto de sustituciones nucleotídicas (K), por medio del programa DnaSP versión 4 (Rozas et al. 2004). El análisis de la variabilidad de L.eximia provenientes de los tres sitios de muestreo (municipios), fue realizado con base en los polimorfismos de ADN. Las secuencias fueron comparadas teniendo en cuenta la composición en términos de nucleótidos, el porcentaje de transiciones y de transversiones, y el número de sitios polimórficos o sus longitudes. Se exploró la región secuenciada en búsqueda de las regiones más conservadas y variables. Como una medida de la variabilidad entre los individuos y las localidades se calcularon las distancias genéticas (Jukes y Cantor 1969). Finalmente se usó el programa MEGA (versión 5) para observar de forma gráfica las similitudes (agrupaciones) de los haplotipos con base en las distancias genéticas intra e interespecíficas, usando un criterio de Máxima Verosimilitud (M.L). Los resultados y análisis relacionados con la variabilidad de las secuencias se realizaron considerando éste como un estudio exploratorio sobre el uso potencial de la región secuenciada para agrupar individuos de la especie de interés (provenientes de diferentes localidades geográficas) y separarlos de otras especies morfológicamente similares con base en las distancias genéticas y no como un estudio de genética de poblaciones. No obstante, la validación del gen como marcador taxonómico, corresponderá a un estudio con mayor número de individuos, localidades y especies similares como punto de comparación y que se deriva de los resultados del presente estudio.
Resultados
Extracción y amplificación. El búfer de macerado permitió obtener ADN de las larvas en cantidad y calidad suficiente para una amplificación mediante PCR de la región de interés, usando entre 0,5 y 1mL de ADN de extracción en una reacción de PCR de 50mL. Además, se logró extraer exitosamente ADN con calidad para PCR de fracciones de 2, 3, y 4 mm de larvas L1, L2, y L3 respectivamente con una concentración promedio de 86ng/uL de ADN por muestra. Como dato adicional y pensando en las muestras frecuentemente encontradas en los cadáveres, también se usaron para la extracción de ADN otras fuentes o partes del insecto disponibles en la cría al momento de los ensayos moleculares. Al respecto se encontraron extracciones exitosas a partir de tres huevos, pupas completas, dos patas y pequeños fragmentos de tórax extraídos de los adultos. No se recuperó ADN de puparios vacíos, (datos no mostrados).
Análisis de secuencias. El segmento amplificado incluyó 324 pares de bases correspondientes a la región 3 del gen Cytb, además de un segmento de 66 pares de bases de ARNt-ser, 23bp de la región IG2 y un segmento del extremo 3´ del gen ND1 de 66 bp. La identidad de las secuencias se verificó por comparación con la secuencia de referencia de L. sericata en relación con la cual el segmento amplificado corresponde a las posiciones 12338 - 12815 del genoma mitocondrial el cual está depositado en GenBank con número de acceso AJ422212. En total se obtuvieron 18 secuencias para igual número de individuos.
Al observar la distribución de la variabilidad entre las posiciones del alineamiento, se evidenció que los sitios con mayor cantidad de polimorfismos se encuentran en los extremos del alineamiento. Estas posiciones corresponden a las regiones terminales 3 de los genes Cytb y ND1 respectivamente, por lo cual se realizaron análisis detallados en términos de haplotipos con énfasis especial en la porción de ND1, que fue tomada para caracterizar por tipos los sitios de muestreo. Los sitios conservados, se encontraron distribuidos, en las posiciones centrales del alineamiento.
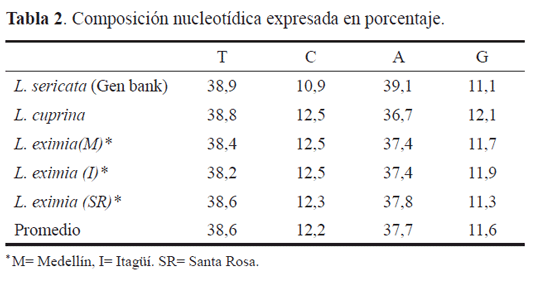
Los porcentajes de la composición nucleotídica (Tabla 2) mostraron una mayor cantidad de Adenina y Timina (76,1%) con respecto a la cantidad de Citosina y Guanina (23,8%) patrón de composición de bases que corresponde a lo observado en la mayoría de los genomas mitocondriales de insectos, especialmente en las regiones codificantes (Hoy 2003).
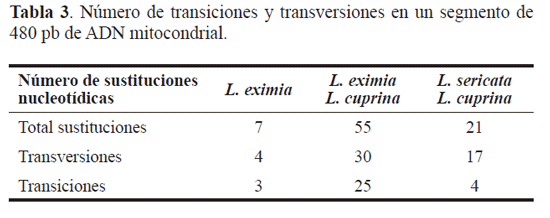
Respecto a las sustituciones nucleotídicas, se encontró un mayor número de transiciones que de transversiones (Tabla 3). L. sericata y L. cuprina se consideran especies morfológicamente similares y diferenciables de L.eximia, lo cual se correlaciona con la variación en el número de sustituciones expresada en porcentajes. Se encontró un porcentaje de 11% de variación entre L. eximia y L. cuprina, 4,2% entre L. cuprina y L. sericata y 1,4% entre individuos de L. eximia.
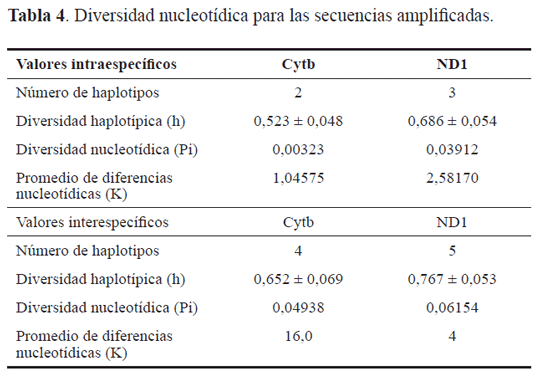
Variabilidad de Cytb. El análisis incluyó 324 nucleótidos de los cuales, 322 sitios fueron conservados y dos polimórficos con mutaciones en las posiciones 23 y 34 de la secuencia que corresponden a las terceras posiciones del codón, este cambio muestra las poblaciones de Medellín e Itagüí con un mismo haplotipo y la población de Santa Rosa como un segundo haplotipo. Los valores de diversidad haplotípica y diversidad nucleotídica para las secuencias de L.eximia (Tabla 4), corresponden con los observados a nivel intraespecífico para datos haploides (Stephan y Langley 1992).
Variabilidad de ND1. El segmento que codifica para ND1 de 66 pb, corresponde a la región 414 - 480 de la matriz. En esta fracción, se encontraron 61 sitios conservados y cinco polimórficos que originaron cambios en las primeras y terceras posiciones del codón. Los sitios variables fueron parsimoniosamente informativos con dos estados en las posiciones dos, tres, 55, 57 (416, 417, 469 y 471 de la matriz general) y un sitio igualmente informativo con tres estados en la posición cuatro (418 de la matriz general) en la primera posición del codón. Este fragmento representó la porción más variable de la región de ADN estudiado y con base en el cual se observó una clara separación de los especímenes por localidad.
Al comparar las distancias genéticas Jukes y Cantor (1969) al interior de la especie con base en las regiones (Cytb, ND1), se encontraron valores entre 0,01 y 0,011, (Tabla 5). Estos resultados son similares a los encontrados a nivel intraespecífico en otras especies del género como L sericata (0,0-2,1) y L. caesar (0,0 -0,9) en los cuales se utilizó una región de 268 pb del gen COI entre individuos provenientes de diferentes provincias de China (Liu et al. 2010). Los valores fueron mayores entre los individuos de Medellín y Santa Rosa y Medellín e Itagüí, que entre los de Itagüí y Medellín, correspondiendo con las mayores distancias geográficas.
De igual forma, los valores de h (Nei 1987) encontrados entre los individuos de las diferentes localidades (0,53 y 0,684) están ligeramente por encima de los valores promedio ya que para marcadores de evolución rápida como el ADN mitocondrial, los valores de diversidad haplotípica in-tra especies generalmente son menores de 0,5. Así mismo, el número promedio de diferencias nucleotídicas entre los haplotipos (K) (Tajima 1983) indicó una diversidad genética media-baja.
La topología obtenida a partir de distancias genéticas de Jukes y Cantor muestra las relaciones entre haplotipos correspondientes a los sitios de muestreo (Fig. 1). En él se observa un único haplotipo por municipio, así como la correspondencia entre las distancias genéticas y las distancias geográficas. El fragmento completo secuenciado permitió discriminar individuos de los tres municipios separados a una distancia mínima de 8 kilómetros.
Discusión
Para las Ciencias Forenses, la aplicabilidad de cualquier método de análisis, depende de varios factores. Entre los más importantes se encuentran la rapidez, eficiencia y costos de la técnica. En cuanto a la obtención y uso de ADN, actualmente se encuentran disponibles y bien documentadas técnicas como RAPDS, RfilPS, y secuenciación de ADN mitocondrial utilizados para análisis de genética de poblaciones, taxonomía molecular y análisis filogenéticos en especies de importancia forense. En la actualidad la obtención directa de secuencias aparece como el método más utilizado y de gran potencial en la sistemática de insectos, mostrando en comparación con otras técnicas excelentes resultados en cuanto a la variabilidad genética de las especies, siendo una información 100% conectable con otros resultados similares y permitiendo a posteriori la implementación de técnicas masivas de caracterización que usen las regiones más variables o conservadas de acuerdo al interés (Infante-Malachias et al. 1999; Litjens et al. 2001; Stevens y Wall 2001).
En este trabajo realizamos una estandarización de la extracción y amplificación de una región del ADN mitocondrial, Cytb y ND1, (480 pb) y verificamos su uso potencial en la asignación de especímenes o sus partes a una especie de interés forense con identificación taxonómica previamente realizada con base en morfología. Esta región además permitió separar los individuos con base en su procedencia. Adicionalmente, exploramos como el polimorfismo en las secuencias de esta región permite separar de forma correcta individuos de otras dos especies cercanas y morfológicamente similares: L. sericata y L. cuprina. Las diferencias en términos de número y tipo de haplotipos y distancias genéticas sugieren un uso potencial de este marcador pero que deberá verificarse en un estudio posterior que incluya muestreos en el rango geográfico de distribución de la especie y en relación con otras especies de forma que se evalué la consistencia del marcador a este nivel taxonómico y de preferencia en el género.
A diferencia de la mayoría de los casos reportados para ciencias forenses, en el presente estudio no se usaron kits comerciales de extracción de ADN que suelen ser costosos en comparación con la solución de lisis con la cual se realizó la extracción. La calidad y cantidad de ADN evaluada con el nanodrop y mediante gel de agarosa, así como la amplificación exitosa de PCR es de resaltar, en especial, si se considera que a partir de pequeñas partes o tejidos de los insectos, con menores costos y en tiempos similares se obtienen resultados adecuados y comparables con los que representa el uso de productos comerciales.
Literatura citada
AMAT, E. 2009. Contribución al conocimiento de las Chrysomyinae y Toxotarsinae (Diptera: Calliphoridae) de Colombia from Colombia. Revista Mexicana De Biodiversidad 80: 693-708. [ Links ]
AMAT, E.; VÉLEZ, C.; WOLFF, M. 2008. Clave ilustrada para la identificación de los géneros y las especies de califóridos ( Diptera : Calliphoridae ) de Colombia. Caldasia 30(1): 231-244. [ Links ]
ANDERSON, G.S. 2000. Minimum and maximum development rates of some forensically important Calliphoridae (Diptera). Journal of Forensic Sciences 45: 824-32. [ Links ]
BEHURA, S. K. 2006. Molecular marker systems in insects: current trends and future avenues. Molecular ecology 15(11): 3087-113. [ Links ]
BENECKE, M. 1998. Six forensic entomology cases: description and commentary. Journal of Forensic Sciences 43:797-805; 43:1303. [ Links ]
COLLINS, F.H; PORTER, C.H. 1990. Comparison of rDNA and mtDNA in the sibling species Anopheles freeborni and A. Hermsi. American Journal of Tropical Medicine and Hygiene 42: 417-423. [ Links ]
FLOREZ, E.; WOLFF, M. 2009. Descripción y clave de los estadios inmaduros de las principales especies de Calliphoridae (Diptera) de importancia forense en Colombia. Neotropical Entomology 38(3): 418-429. [ Links ]
HALL, T.A. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series 41: 95-98. [ Links ]
HOY, M. 2003. Insect molecular genetics; an introduction to principles and applications. USA, Academic Press, 544 p. [ Links ]
INFANTE-MALACHIAS, M.E; YOTOKO, K.S; AZEREDO-ESPIN, A.M. 1999. Random amplifed polymorphic DNA of Screwworm fy populations (Diptera: Calliphoridae) from Southeastern Brazil and Northern Argentina. Genome 42: 772-779. [ Links ]
IRWIN D.M; KOCHER T.D; WILSON, A.C. 1991. Evolution of the cytochrome b gene in mammals. Journal of Molecular Evolution 32: 128-144. [ Links ]
JUKES, T. H.; CANTOR, C. R. 1969. Evolution of protein molecules. (Munro H N, eel.) Mammalian protein metabolism. III. New York: Academic Press. 21-132 p. [ Links ]
LESSINGER, A.C; AZEREDO-ESPIN, A.M.L. 2000. Evolution and structural organization of mitochondrial DNA control region of miasis-causing blow fies. Medical and Veterinary Entomology 14: 71-80. [ Links ]
LIU, Q.L.; CAI, J.F.; CHANG, Y.F.; GU, Y.; GUO, Y.D.; WANG, X.H.; WENG, J.F.; ZHONG. M.; WANG, X.; YANG, L.; WU, K.L.; LAN, L.M; WANG, J.F; CHEN, Y.Q. 2010. Identification of forensically important blow fy species (Diptera: Calliphoridae) in China by mitochondrial cytochrome oxidase I gene differentiation. 18: no. doi: 10.1111/j.1744917.2010.01377.x. [ Links ]
LITJENS, P; LESSINGER, A.C; AZEREDO- ESPIN, A.M. 2001. Characterization of the screwworm fies Cochliomyia homini-vorax and Cochliomyia macellaria PCR-RfilP of mitochondrial DNA. Medical and Veterinary Entomology 15: 183-188. [ Links ]
MARCHENKO, M.I. 2001. Medicolegal relevance of cadaver entomofauna for the determination of the time of death. Forensic Science International 120: 89-109. [ Links ]
MEYER, A. 1994. Shortcomings of the Cytochrome b gene as a molecular marker. Trends in Ecology and Evolution 9: 278-280. [ Links ]
NELSON, L. A.; WALLMAN, J F.; DOWTON, M. 2007. Using COI barcodes to identify forensically and medically important blowfies. Medical and Veterinary Entomology 21(1): 44-52. [ Links ]
NEI, M. 1987. Molecular evolutionary genetics. Columbia University Press. New York. PÉREZ, S. P.; DUQUE, P.; WOLFF, M. 2005. Successional behavior and occurrence matrix of carrion-associated arthropods in the urban area of Medellín, Colombia. Journal of Forensic Sciences 50(2): 448-54. [ Links ] [ Links ]
ROZAS, J; SÁNCHEZ-DEL BARRIO, J.C; MESSEGUER, X; ROZAS, R. 2004. DNASP Dna sequence polimorphism. Version 4.0, Barcelona, Universidad de Barcelona. SALAZAR, J. 2008. Estudio de la entomofauna sucesional asociada a la descomposición de un cadáver de cerdo doméstico Sus scrofa en condiciones de campo. Universitas Scientiarum 13(1): 21-32. [ Links ] [ Links ]
SEGURA, N. A.; USAQUÉN, W.; SÁNCHEZ, M. C;, CHUAIRE, L.; BELLO, F. 2009. Succession pattern of cadaverous entomofauna in a semi-rural area of Bogotá, Colombia. Forensic Science International 187(1-3): 66-72. [ Links ]
SIMMONS, R.B; WELLER, S.J. 2001. Utility and evolution of cytochrome b in insects. Molecular Phylogenetics and Evolution. 20 (2): 196-210. [ Links ]
SPERLING, F.H.A; ANDERSON, G.S; HICKEY. D.A. 1994. A DNA-based approach to the identification of insect species used for postmortem interval estimation. Journal of Forensic Science 39: 418- 427. [ Links ]
STEVENS, J.; WALL, R. 1997. The evolution of ectoparasitism in the genus Lucilia (Diptera: Calliphoridae). International Journal of Parasitology 27(1): 51-59. [ Links ]
STEVENS, J; WALL, R. 2001. Genetic relationships between blowfies (Calliphoridae) of forensic importance. Forensic Science International 120: 116-123. [ Links ]
STEHR, F. W. 1987. Techniques for collecting, rearing, preserving and studying immature insects, vol1, Kendall/Hunt Publishing Company, Iowa, USA.754 p. [ Links ]
STEPHAN, W.; LANGLEY, C.H. 1992. Evolutionary consequences of DNA mismatch inhibited repair opportunity. Genetics 132: 567-574. [ Links ]
TAJIMA, F. 1983. Evolutionary relationships of DNA sequences in finite populations. Genetics 105: 437-460. [ Links ]
TORGERSON, D.G; LAMPO, M; VELÁZQUEZ, Y; WOO, P. 2003. Genetic relationships among some species groups within the genus Lutzomyia (Diptera: Psychodidae). American Journal of Tropical Medicine and Higiene 69: (5) 484-493. [ Links ]
URIBE S, PORTER CH; VÉLEZ ID. 1998 Amplificación y Obtención de Secuencias de rRNA Mitocondrial en Lutzomyia spp (Diptera: Psychodidae) Vectores de Leishmaniosis. Revista Colombiana de Entomología 23 (3-4): 177-185. [ Links ]
URIBE S. 1999 Polimorfismo entre Poblaciones de Lutzomyia longipalpis (Diptera: Psychodidae) Vector de Leishmaniosis Sugiere la Existencia de un Complejo de Especies. Revista Biomédica 19(1): 56-66. [ Links ]
VÉLEZ, M. C.; WOLFF, M. 2008. Rearing fve species of Diptera (Calliphoridae) of forensic importance in Colombia in semicontrolled feld conditions. Papéis Avulsos de Zoologia (São Paulo), 48(6): 41-48. [ Links ]
WALLMAN, J.F; DONELLAN, S.C. 2001. the utility of mitochondrial DNA sequences for identification of forensically important blowfies. (Diptera: Calliphoridae) in southeastern Australia. Forensic Science International 120 (1): 11-115. [ Links ]
WALLMAN, J.F.; LEYS, R., HOGENDOORN, K. 2005. Molecular systematics of Australian carrion-breeding blowfies (Diptera: Calliphoridae) based on mitochondrial DNA. Invertebrate Systematics 19: 1-15. [ Links ]
WELLS, J.; WALL, R.; STEVENS, J. 2007. Phylogenetic analysis of forensically important Lucilia fies based on cytochrome oxidase I sequence: a cautionary tale for forensic species determination. International Journal of Legal Medicine 121(3): 229-33. [ Links ]
WELLS, J.; WILLIAMS, D. 2007. Validation of a DNA-based method for identifying Chrysomyinae (Diptera: Calliphoridae) used in a death investigation. International Journal of Legal Medicine 121(1): 1-8. [ Links ]
WHITWORTH, T. 2010. Keys to the genera and species of blow fies (Diptera: Calliphoridae) of the West Indies and description of a new species of Lucilia Robineau-Desvoidy. Zootaxa 2663: 1-35.
[ Links ]