Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Entomología
Print version ISSN 0120-0488
Rev. Colomb. Entomol. vol.39 no.2 Bogotá July/Dec. 2013
Identificación molecular de califóridos (Diptera: Calliphoridae) de importancia forense en Colombia
Molecular identification of Calliphoridae (Diptera: Calliphoridae) of forensic importance in Colombia
JAIBER J. SOLANO1,2, MARTA WOLFF3 y LYDA R. CASTRO1,4
1 Universidad del Magdalena. Grupo de investigación de Evolución, Sistemática y Ecología Molecular Carrera 32 No. 22-08 Santa Marta, Colombia.
2 Biólogo. jaiber.solano.i@gmail.com.
3 Ph. D. Directora de las Colecciones Entomológicas de la Universidad de Antioquia. calle 70 No. 52-21 Medellín, Colombia. marta_wolff@yahoo.com.
4 Ph. D. lcastro@unimagdalena.edu.co. lydaraquelcastro@hotmail.com. Autor para correspondencia.
Recibido: 15-mar-2013 - Aceptado: 23-oct-2013
Resumen: Reconociendo la importancia de la correcta identificación de los insectos asociados a los cuerpos cadavéricos para estimar el intervalo postmortem de una forma efectiva; teniendo en cuenta la incidencia de las moscas de la familia Calliphoridae en cadáveres y la dificultad existente para su determinación taxonómica de los estadios inmaduros, se evaluó la utilidad de la secuencia del gen mitocondrial COI como herramienta de diagnóstico molecular para la identificación de califóridos de importancia forense en Colombia. Se utilizaron secuencias de muestras colectadas en Colombia y otras tomadas de Genbank. Se realizaron análisis de divergencia de nucleótidos y análisis de "neighbour-joining", con base en las distancias genéticas calculadas con el modelo K2P. Se realizaron análisis bayesianos y se desarrolló una representación gráfica de la variación del fragmento de COI para cada especie. Se obtuvieron fragmentos de 545 pb; los valores de distancia intra-especifica, cuando se incluyen todas las especies secuenciadas, oscilaron entre 0% y 2,904% y los de distancia inter-especifica entre 0,188% y 18,07%, observándose un solapamiento de distancias, ocasionado principalmente por la inclusión de especies con solo una secuencia. Al excluir las especies con una sola secuencia y tratar a L. eximia como dos subgrupos, se logró observar un gap de distancias de 2,292%, el cual es un valor óptimo para la validación de esta técnica. Se observaron monofilias recíprocamente específicas en la mayoría de las especies, concluyendo que el gen COI es un buen marcador para delinear especies de la familia Calliphoridae en Colombia.
Palabras clave: Citocromo oxidasa I. Intervalo Postmortem.
Abstract: Recognizing the importance of the correct identification of insects associated with deceased bodies for an effective estimation of time of death, and taking into account the incidence of flies of the family Calliphoridae in corpses and the difficulty of their taxonomic determination, we evaluated the usefulness of COI mitochondrial gene sequences as a molecular diagnostic tool for identifying forensically important calliphorids in Colombia. Sequences obtained from samples collected in different regions of Colombia and other sequences taken from Genbank were used. Based on the genetic distances calculated using the K2P model, nucleotide divergence and neighbour-joining analyses were performed. Bayesian analyses were also developed and a graphical representation of the variation of the COI fragments for each species. A total 545 pb fragments were obtained; the values of intraspecific distance when all sequenced were included, ranged between 0% and 2.904% and the interspecific distance ranged between 0.188% and 18.07%, with an overlapping distance, caused mostly by the inclusion of species with only one sequence. When we excluded species with a single sequence and L. eximia was analyzed as two subgroups, a distance gap of2.292% was obtained, which is an optimal value for the validation of this technique. Reciprocal monophyly was observed in most species. We conclude that the COI gene is a good marker for delineating species of the family Calliphoridae in Colombia.
Key words: Cytochrome oxidase I. Postmortem interval.
Introducción
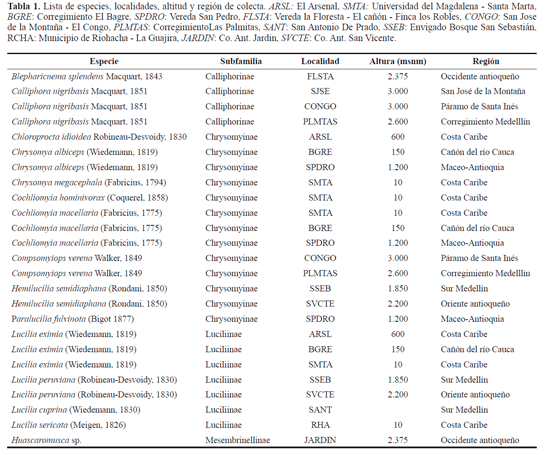
Los califóridos (Diptera:Calliphoridae) han sido objeto de estudios importantes en trabajos realizados en todo el mundo, gracias a su aparición en los cuerpos cadavéricos y su importancia como indicadores del tiempo de muerte y como herramienta para calcular el intervalo post-mortem (IPM) en casos legales (Wolff et al. 2001; Schroeder et al. 2003; Daw-nay et al. 2007; Harvey et al. 2008), de esta manera, juegan un papel fundamental en el estudio de la entomología forense como disciplina (Wolff et al. 2001).
Esta familia presenta alrededor de 1.000 especies en todo el mundo, pero en el Neotrópico sólo se encuentran aproximadamente unas 126 especies, distribuidas principalmente en cinco subfamilias: Calliphorinae, Luciliinae, Chrysomyi-nae, Melanomyinae y Mesembrinellinae (Pape et al. 2004). En Colombia se han registrado 12 géneros y 29 especies de califóridos, donde por lo menos 13 especies presentan importancia forense (Amat 2009; Flórez y Wolff 2009).
La identificación de la especie encontrada en asociación con un cadáver es uno de los primeros pasos utilizados en entomología forense para estimar el IPM (Wells y LaMotte 2001). Sin embargo, el uso de moscas carroñeras en la valoración de IPM ha sido obstaculizado por las dificultades asociadas con la correcta identificación de la especie en las diferentes etapas de desarrollo, huevos, larvas, puparium y adultos (Catts 1992), particularmente cuando sólo hay fragmentos del insecto (e.g. pupas, larvas o adultos muertos, descompuestos o partes de estos), disponibles para el análisis (Stevens y Wall 2001) o cuando se encuentra individuos en estadios larvales. Lo anterior conlleva a la cría en laboratorio a partir de larvas y/o pupas recolectadas, que se traduce a un gasto de tiempo y recursos, debido a que esta es una labor compleja (Catts 1992; Harvey et al. 2008).
Sin duda alguna, la identificación de moscas carroñe-ras requiere del conocimiento taxonómico del especialista, confiando a menudo en el reconocimiento de pequeñas variaciones en características morfológicas y en la disección e inspección de los órganos genitales en los adultos (Wallman 2001). Sin embargo, la semejanza morfológica de las diferentes especies de califóridos, especialmente en sus estadios inmaduros, ocasiona que actualmente exista gran interés por el desarrollo de herramientas alternativas, que ayuden a resolver estos problemas taxonómicos (Schroeder et al. 2003; Ames et al. 2006; Nelson et al. 2007; Alessandrini et al. 2008).
Con el advenimiento de herramientas en biología molecular, se han desarrollado técnicas que facilitan la determinación de especies. Recientemente, la utilización de un sistema de códigos de barras genéticos, que ayuda a la identificación de especies problema, ha sido aplicada para muchos organismos (Catts 1992; Folmer et al. 1994; Herbert et al. 2003; Armb y Ball 2005; Ball et al. 2005; Hebert y Gregory 2005; Dawnay et al. 2007; Yadong et al. 2010; Guo et al. 2012; Boehme et al. 2012).
Los proponentes de los "código de barra genéticos" vi-sionan la construcción de una base de datos universal que incluya todas las secuencias de COI de animales, que además serviría como base para un sistema global de bioidentifica-ción de animales (Hebert et al. 2003), debido a que es un gen que resulta de gran utilidad para la discriminación entre especies. Este gen ya ha sido utilizado en otras partes para la identificación de metazoos (Folmer et al. 1994), gastrópodos (Remigio y Hebert 2003), colémbolos (Hogg y Hebert 2004), insectos como: efemerópteros (Ball et al. 2005), lepidópteros (Hebert et al. 2003), moscas de importancia forense (Meikle-john et al. 2011; Boehme et al. 2012; Jordaens et al. 2013) además de trabajos relacionados con la identificación de califóridos (Stevens y Wall 2001; Nelson et al. 2007; Harvey et al. 2008).
Las ventajas de los códigos de barra genéticos para la identificación y el descubrimiento de especies ha sido bastante discutida (Hebert y Gregory 2005) y el concepto continúa siendo tema de debate. Sin embargo, se cree que la consolidación de un sistema estandarizado para la identiicación de especies va a fortalecer esfuerzos de investigación globales y será beneicioso para la identiicación de especies de importancia médica, económica o ambiental (Armb y Ball 2005).
En la estandarización y validación de esta técnica, es de vital importancia la realización de los análisis de divergencia genética dentro y entre las especies (distancias intra e inter-especificas, respectivamente), donde idealmente se espera la aparición de un gap o una zona donde el valor superior de las distancias intra-específicas se encuentre alejado del valor inferior de las distancias inter-específicas y, de esta manera, no exista un solapamiento entre estos dos parámetros (Meyer y Paulay 2005; Roe y Sperling 2007). Además de esto y de manera complementaria, se han utilizado métodos de agru-pamiento (neigbour-joining) y de reconstrucción filogenética (Bayesiano y máxima verosimilitud, principalmente), para la validación de esta técnica, gracias a la evaluación de las mo-nofílias recíprocamente específicas que se obtienen de forma clara con datos de esta naturaleza (Meyer y Paulay 2005; Nelson et al. 2007; Whitworth et al. 2007; Meiklejohn et al. 2011).
Aun no se ha estandarizado un valor de distancia intra e inter-específica para las especies de la familia Calliphoridae y para el gen COI, pero se han observado valores promedio mínimos de distancia inter-específica y máximos de intra-es-pecífica de 0,480% y 0,295% respectivamente (Nelson et al. 2007), siendo estos los únicos que hemos podido identificar, a pesar de que en esta familia se han desarrollado otros estudios de este tipo (Schroeder et al. 2003; Ames et al. 2006; Whitworth et al. 2007; Alessandrini et al. 2008; Giraldo et al. 2011). Además, de forma general, los defensores de esta herramienta, han propuesto valores estimativos entre 1-2% de variación intraespecíica y valores superiores a estos para separar especies (Lanteri 2007).
Con base en lo anterior, este trabajo tiene como objetivo desarrollar herramientas de diagnóstico molecular para la identiicación de Calliphoridae de importancia forense en Colombia, mediante la ampliicación y secuenciación del código de barra -gen COI-.
Materiales y métodos
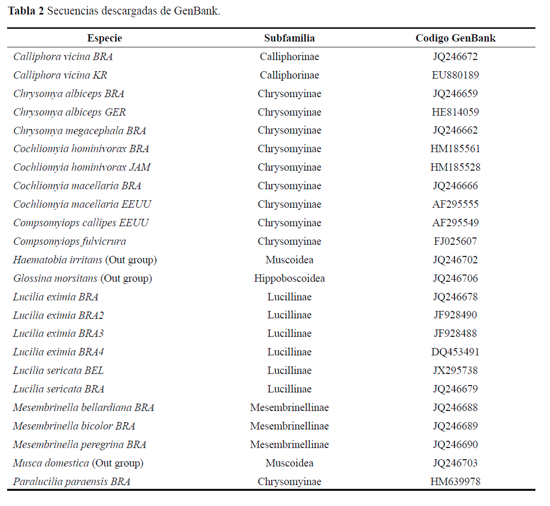
Muestras estudiadas. Las muestras, colectadas en diferentes pisos altitudinales representativos del país, se identificaron en el laboratorio de Colecciones Entomológicas de la Universidad de Antioquia y fueron enviadas al laboratorio de Evolución y Sistemática Molecular de la Universidad del Magdalena, para el desarrollo de las pruebas de diagnóstico molecular, donde están disponibles y preservadas en alcohol al 95%. Los sitios de colecta de las muestras están detallados en la tabla 1. Además, se utilizaron secuencias descargadas de la base de datos de Genbank (Tabla 2).
Extracción de ADN. La extracción de ADN se realizó a partir la disección de la zona torácica de adultos previamente identificados y se utilizo el kit de extracción de ADN de QIA-GEN (DNeasy Blood & Tissue Kit, 250), siguiendo el protocolo del fabricante (DNesay Bloody & TissueHandbook de Qiagen). La extracción del ADN y su calidad se confirmaron por medio de electroforesis en gel de agarosa y con tinción con GelRed (Biotium).
Amplificación y secuenciación de COI. La amplificación se realizo mediante la reacción en cadena de la polimerasa (PCR) por medio de un termociclador (Eppendorf Mastercy-cler® pro); utilizando los cebadores universales para insectos que amplifican este gen, diseñados por Folmer et al. (1994) y modificados por Nelson et al. (2007). La secuencia de los cebadores utilizados fue: LCO1490-L (5' -GTCWACWAATCA-TAAAGATATTGG-3') y HCO2198-L (5'-TAAACTTCWG-GRTGWCCAAARAATCA-3').
Para las reacciones de PCR se utilizaron 2 µL de ADN extraído en reacciones de 25 (íL, usando concentraciones finales de cada reactivo, de la siguiente manera: 0,1 U/mL de Taq Polimerase (0,5 µL de Taq Polimerasa 5U/mL; abm), 3,5 mM de MgCl2 (3,5 µL de MgCL2 a 25mM; abm), 1X de Buffer PCR (No MgCl2; 2,5 µL de Buffer PCR a 10X; abm), 0,2 mM de DNTP's (0,5 µL de DNTPs a 10mM; Bioline), 0,4 mM de cada primer (1 (lL de cada primer a 10mM) y se completa con agua destilada y autoclavada (14 µL de agua), hasta alcanzar 25 µL de reacción de PCR.
Los ciclos de PCR se realizaron de acuerdo con lo sugerido por Nelson et al. (2007) modificado, con una temperatura inicial de desnaturalización de 94 °C durante 2 minutos, seguido de 35 ciclos con los siguientes parámetros: 30 segundos a 94 °C, 30 segundos a 54 °C y 2 minutos a 72 °C, seguido por un paso de extensión final a 72 °C por 5 minutos.
La cuantificación de los productos de amplificación se realizó por corrimientos en gel de electroforesis de agarosa 2% y teñido con GelRed (Biotium).
Los productos obtenidos de la amplificación de COI se purificaron con el kit de purificación de ácidos nucleicos y proteínas de MACHEREY-NAGEL (NucleoSpin® Extract II), de acuerdo con el protocolo del proveedor y estos productos de purificación se enviaron a MACROGEN (Korea) donde se secuenciaron.
Análisis de secuencias. Las secuencias obtenidas, se verificaron mediante la herramienta BLAST del NCBI (www.ncbi.nlm.nih.gov) y Barcoding of life Data (BOLD) System ( www.barcodinglife.org; Ratnasingham y Hebert 2007). Posteriormente se editaron manualmente utilizando los programas BioEdit Sequence Alignment Editor (Version 7.0.9.0; Hall 1999) y se alinearon con el algoritmo ClustalW (Thompson et al. 1994), en el programa MEGA 5 (Tamura et al. 2011), teniendo en cuenta la corrección de los marcos de lectura, para evitar que dentro de la secuencia se encuentren codones de parada, debido a que estas secuencias son codificantes.
Con MEGA 5.0 (Tamura et al. 2011), se realizaron los análisis de divergencia de secuencias de nucleótidos con distancias genéticas inter e intra-específicas (las intra especificas solamente se realizaron con las especies que se contaban conmás de una secuencia), las cuales se calcularon con el modelo de distancia de Kimura 2 parámetros "K2P" (Kimura 1980). También con MEGA 5.0 (Tamura et al. 201l) se realizó el análisis Neighbour-Joining (NJ), utilizando el modelo de evolución molecular de Kimura 2 parámetros, el cual nos provee una representación gráfica de los patrones de divergencia entre especies. En ambos casos se hizo un análisis de bootstrap con 1000 réplicas.
Se realizaron análisis bayesianos de forma particionada con MrBayes 3.1.2. (Huelsenbeck y Ronquist 2001), donde se utilizó un modelo diferente para cada posición del codón de la secuencia de COI, de esta manera, se empleó el modelo GTR+I+G para la primera posición, F81 para la segunda posición, y el modelo GTR+G, para la tercera posición. La selección del mejor modelo para cada posición en este análisis se realizó con el programa jModelTest 0.1 (Posada 2008), utilizando el criterio del mínimo valor de AIC (Criterio de Información de Acaike), como principal parámetro para la elección del modelo.
En todos los análisis se designó a Musca domestica Linnaeus, 1758, Haematobia irritans (Linnaeus, 1758) y Glossina moritans Westwood, 1851 como grupos externos.
Debido a que otros estudios sugieren la importancia de agregar varias secuencias por especies para no generar errores por insuficiencia de muestreo (Lanteri 2007), los análisis de distancia interespecifica se desarrollaron nuevamente excluyendo las especies que presentaron solo una secuencia en los análisis anteriores (excepto los grupos externos). Adi-cionalmente, se hicieron análisis en donde la especie Lucilia eximia (Wiedemann, 1819), fue dividida en dos subgrupos o clados, donde el clado 1 está conformado por L. eximia BRA2, BRA3, BRA4 y SMTA; y el clado 2 por L. eximia ARSL, BGRE y BRA.
A través del programa en línea Fingerprint. http://evol.mcmaster.ca/fingerprint (Lou y Golding 2007), se realizó una representación compuesta de la variación del fragmento de COI para cada una de las especies aquí evaluadas, esta representación ilustra la heterogeneidad en un sitio específico dentro de la región de código de barras, como un porcentaje de la línea vertical trazada para representar la heterogeneidad en cada par de bases en sitios específicos (Fisher y Smith 2008).
Resultados
Características de las secuencias. Todas las secuencias después de editadas y corregidos los marcos de lectura, constaron
de 545 nucleótidos. Estas secuencias se encuentran disponibles en GenBank (http://www.ncbi.nlm.nih.gov/Genbank/) con números de acceso KC568246 - 568278, y en la biblioteca de códigos genéticos del consorcio "Barcoding of the life" (http://www.boldsystems.org/views/login.php), números de acceso SWCCO001-13 - SWCCO033-13 (Tabla 3).
El alineamiento de las secuencias mostró ausencia de inserciones y deleciones, tal como lo observado por otros autores que utilizaron estos genes como códigos de barras (Hebert et al. 2003; Nelson et al. 2007). La composición promedio de nucleótidos observada fue de T = 38,2%, C = 16,1%, A = 29,9% y G = 15,8%.
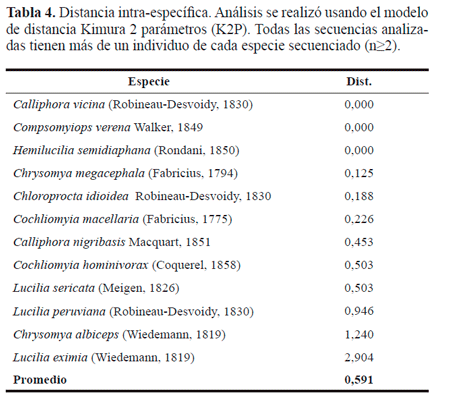
Distancias genéticas. Cuando incluimos todos los taxa se-cuenciados, las variaciones de los valores de distancias genéticas intra-especificas oscilaron entre 0% (Compsomyiops verena Walker, 1849 y Compsomyiops vicina (Robineau-Desvoidy, 1830)) y 2,904% (Lucilia eximia) con una mayor frecuencia de valores inferiores a 1%, encontradas en 10 de las 12 especies analizadas bajo este parámetro, presentando una media de la distancia intra-especifica de 0,591% (Tabla 4). Las variaciones inter-especificas, oscilaron entre 0,188% (C. verena-C. fulvicruca) y 18,077 (G. morsitans- Mesem-brinella bellerdiana Aldrich, 1922). La media de las distancias inter-especificas fue de 10,776%. Como el valor máximo observado en las distancias intra-especificas fue de 2,904%, mientras que el mínimo observado en las variaciones inter-especificas fue de 0,188%, se observa que hubo superposición en los rangos de los valores de divergencia inter e intra-especifica (Fig. 1).
Sin embargo, en los análisis donde se excluyeron las especies con una sola secuencia, se observaron valores de divergencia intraespecificas que oscilan entre 0% (Calliphora vicina (Robineau-Desvoidy, 1830)) y 1,487% (Lucilia peruviana (Robineau-Desvoidy, 1830)); mientras que los valores de divergencia interespecifica oscilaron entre 4,416% (Lucilia eximia clado 1 x Lucilia eximia clado 2) y 17,927% (Cochliomyia macellaria (Fabricius, 1794) x Glossina mori-tans), de esta manera no existe un solapamiento de distancias genéticas observándose un gap de distancias (Fig. 2).
Análisis de "neighbour-joining" (NJ). En el árbol obtenido a través del método NJ (Fig. 3), se lograron visualizar varios grupos, los cuales tienen correspondencia, principalmente, con las subfamilias a quienes pertenece cada una de las especies aquí mostradas. Se observo un primer grupo conformado por los califoridos de los géneros Mesembrinella y Huascaromusca, ambos pertenecientes a la subfamilia Me-sembrinellinae (Guimaraes 1977; Bonatto y Marioni 2005), cada nodo con valores de soporte superiores a 70%. Un segundo grupo, conformado por los géneros Cochliomyia, Pa-ralucilia, Hemilucilia, Compsomyiops y Chrysomya, todas pertenecientes al a subfamilia Chrysomyinae (Flórez y Wolff 2009; Marinho et al. 2012) con un clado muy marcado en el género Chrysomya, soportado por un valor de bootstrap de 82%. Y un tercer grupo polifiletico conformado por los géneros Lucilia, Calliphora y Blepharicnema, pertenecientes a la subfamilia Lucillinae y los otros dos a Calliphorinae (Amat y Wolff 2007; Marinho et al. 2012), aunque el género Lucilia, en algunas claves taxonómicas es considerada perteneciente a la subfamilia Calliphorinae (Flórez y Wolff 2009); en este tercer grupo, las especies del género Lucilia, se presenta como un grupo parafiletico, además se observan dos grupo de L. eximia, donde uno de los subgrupos (BAGRE, ARSL, BRA) está más relacionado con L. peruviana, que con el segundo subgrupo de L. eximia (UNIMAG, USA, BRA2, BRA3), es de gran importancia reconocer que se presentan altos valores de soporte en la definición de este grupo representado por L. eximia (UNIMAG, USA, BRA2, BRA3) y L. peruviana.
Análisis bayesiano (BA). En el análisis bayesiano aparece una distribución diferente, pero a su vez, puede ser dividida en tres grupos fácilmente reconocibles (Fig. 4). Un primer grupo, donde se encuentran representantes de los géneros Chrysomya y Compsomyiops, conservándose un clado dentro del género Chrysomya con un valor de probabilidad posterior de 1.00. Un segundo grupo conformado por los géneros Lucilia, Calliphora, Blepharicnema, Huascaromusca y Mesem-brinella; se mantiene una asociación igual a la observada en el análisis NJ, donde un subgrupo de L. eximia (SMTA, USA, BRA2, BRA3), se encuentra más cercanamente relacionado con L. peruviana, que al otro subgrupo de L. eximia (BRA, ARSL, BAGRE). El tercer grupo está conformado por una politomía entre los géneros Chloroprocta, Cochliomyia, He-milucilia y Paralucilia.
Ilustración de heterogeneidad de COI por especies. Gracias a las ilustraciones obtenidas mediante la herramienta FINGERPRINT (Lou y Golding 2007), podemos observar gráficamente las características de las secuencias, donde se observa, para cada especie, una mayor frecuencia de Adeninas y Timinas (Fig. 5).
Discusión y conclusiones
Es importante destacar que en este trabajo, el valor promedio de la distancia intra-especifica, estuvo por encima de los valores registrados para Lepidópteras por Hebert et al. (2003), Chrysomya (Diptera: Calliphoridae) por Nelson et al. (2007) y para Protocalliphora (Diptera: Calliphoridae) por Whitworth et al. (2007), quienes registran valores promedio inferiores a 1%; aunque en los trabajos realizados en colém-bolos (Hexapoda: Collembola) por Hogg y Hebert (2004) y en Ultracoelostoma spp. (Hemiptera: Margarodidae) por Ball et al. (2005), estos valores llegan a ser superiores a los aquí registrados, presentando valores promedio de distancia intra-especifica, superiores a 7,5%.
Así mismo, se sigue dando una gran discusión sobre la importancia de la aparición del gap entre las distancias intra e inter-especificas, para evaluar de esta manera la utilidad del gen COI para la delineación de especies (Meyer y Paulay 2005). En nuestro análisis de distancias, donde se incluyen todas las especies (inclusive las que solo presentan una secuencia), no es posible la aparición de este gap debido al solapamiento que se observa entre las distancias intra e inter-especificas (Fig. 1), este solapamiento se da principalmente por los valores de distancias inter-específicas entre especies del género Compsomyiops (C. callipes (Bigot, 1877) x C. fulvicruca; C. callipes x C. verena; C. fulvicruca x C. verena); además de las comparaciones entre Mesembrinella bicolor (Fabricáis 1805) x Mesembrinella peregrina Aldrich, 1922 y Lucilia cuprina (Wiedemann, 1830) xLucilia sericata (Meigen, 1826). Cabe resaltar, que en las especies que causan este solapamiento de distancias, solo hay disponible una secuencia, excepto L. cuprina (n = 2) y C. vicina (n = 2), de esta manera, este solapamiento es causado seguramente por el sesgo existente en el número de muestras utilizadas en este análisis (Meyer y Paulay 2005), por lo que se hace importante la realización de análisis que incluyan un mayor número de secuencias por cada especie y que representen en gran medida la variación geográfica de las especies estudiadas (Lanteri 2007).
Ha sido documentado, que especies con recientes procesos de especiación o posibles problemas en la taxonomía, han mostrado problemas en la aplicación de códigos de barras, lo que ocasiona que las distancias inter-especificas sean bajas causando el solapamiento de las distancias (Roe y Sperling 2007). En nuestro análisis, L. eximia presenta valores de distancia intraespecifica relativamente altos en comparación con otros grupos, además de que los análisis de NJ y Bayesianos la dividen en dos subgrupos, uno de ellos más relacionado con la especie L. peruviana que con el segundo subgrupo de L. eximia. De esta manera, al desarrollar el análisis excluyendo las especies con una sola secuencia y al separar a L. eximia en dos subgrupos, se logra observar un gap de distancias de 2,292% (Fig. 2), el cual es un valor óptimo para la validación de esta técnica (Meyer y Paulay 2005).
La monofilia del género Chyrsomya ha sido registrada en varias oportunidades (Nelson et al. 2007; Singh y Wells 2011), lo cual es bien observado en todos los análisis aquí realizados, lo que nos garantiza la gran efectividad de este método en especies pertenecientes a esté género.
Dentro de los análisis realizados, Huascaromusca sp, siempre se muestra cercanamente relacionada con el grupo [M. bicolor-M. peregrina], lo que nos muestra al género Mesembrinella como un grupo parafilético, pero se conserva la monofilia dentro de esta subfamilia. Una desventaja que se presenta, es que la especie Huascaromusca sp. y los demás integrantes de la subfamilia Mesembrinellinae, no han sido muy estudiados y presentan escasos registros en las bases de datos de ADN.
Es importante rescatar, que en todos los casos los valores de probabilidad posterior, como los soportes de bootstrap, en los análisis bayesianos y NJ respectivamente, fueron cercanos a 1 (100%) en los grupos que delinean especies, lo cual es de gran importancia en la validación de la técnica de códigos de barras, porque demuestra su eficiencia en la discriminación entre especies.
¿Lucilia eximia especie criptica? Los análisis de distancias, dentro de la técnica de códigos de barras genéticos, han sido muy aplicados en la detección de especies cripticas (Hebert et al. 2003; De Salle et al. 2005; Ball y Armb 2007;
Roe y Sperling 2007), así pues, teniendo en cuenta que L. eximia muestra un alto valor de distancia intraespecifica, lo cual también se manifiesta en los análisis de NJ y bayesiano realizados, donde L. eximia no es monofilética, se presentan la hipótesis de que exista un proceso de especiación criptica para esta especie y por ende se presentan de forma consistente estos dos grupos de L. eximia. Se hace necesario realizar estudios posteriores que incluyan secuencias que representen los rangos geográficos de distribución de esta especie.
De esta manera, podemos concluir que el gen COI es un buen marcador para delinear especies de la familia Ca-lliphoridae en Colombia, excepto aquellas de la subfamilia Mesembrinellinae, debido a que no se obtuvo evidencia para comprobar la aplicabilidad de esta técnica en estas especies. Se recomienda utilizar un mayor número de secuencias por especie, quienes busquen representar las variaciones geográficas presentes por especie.
Agradecimientos
Al Fondo de Investigaciones de la Universidad del Magdalena (FONCIENCIAS), por la financiación del proyecto. A los miembros del Grupo de investigación de Evolución, Sistemática y Ecología Molecular de la Universidad del Magdalena. A la Bióloga Carolina Vélez del Grupo de Entomología de la Universidad de Antioquia por identificación taxonómica de los especímenes.
Literatura citada
ALESSANDRINI, F.; MAZZANTI, M; ONOFRI, V.; TURCHI, C.; TAGLIABRACCI, A. 2008. mtDNA analysis for genetic identification of forensically important insects. Forensic Science International: Genetics Supplement Series 1 (1): 584-585. [ Links ]
AMAT, E. 2009. Contribución al conocimiento de las Chrysomyinae y Toxotarsinae (Diptera: Calliphoridae) de Colombia. Revista Mexicana de Biodiversidad 80: 693-708. [ Links ]
AMAT, E.; WOLFF, M. 2007. New records of Blepharicnema splendens (Calliphoridae: Calliphorinae, Luciliini) from Colombia. Revista de la Sociedad Entomológica Argentina 66 (12): 187-190. [ Links ]
AMES, C.; TURNER, B.; DANIEL, B. 2006. The use of mitochon-drial cytochrome oxidase I gene (COI) to differentiate two UK blowfly species - Calliphora vicina and Calliphora vomitoria. Forensic Science International 164: 179-182. [ Links ]
ARMb, K. F.; BALL, S. L. 2005. DNA barcodes for biosecurity: invasive species identification. Philosophical Transactions of the Royal Society of London. Biology Sciences 360: 1813-1823. [ Links ]
BALL, S. L.; ARMb, K. F. 2007. Using DNA barcodes to investigate the taxonomy of the New Zealand sooty beech scale insect. DOC Research & Development Series 287. Department of Conservation, Wellington. 14 p. [ Links ]
BALL, S. L.; HEBERT, P. D.; BURIAN, S. K.; WEBB, J. M. 2005. Biological identifications of mayflies (Ephemeroptera) using DNA barcodes. Journal of the North American Benthological Society 24: 508-524. [ Links ]
BOEHME, P.; AMENDT, J.; ZEHNER, R. 2012. The use of COI barcodes for molecular identification of forensically important fly species in Germany. Parasitology Research 110: 2325-2332. [ Links ]
BONATTO, S. B.; MARIONI, L. 2005. Géneros e espécies novos de Mesembrinellinae (Diptera, Calliphoridae) da Costa Rica e Venezuela. Revista Brasileira de Zoologia 22 (4): 883-890. [ Links ]
CATTS, E. P. 1992. Problems in estimating the postmortem interval in death investigations. Journal of Agricultural Entomology 9: 245-255. [ Links ]
DAWNAY, N.; OGDEN, R.; MCEWING, R.; CARVALHO, G. R.; THORPE, R. S. 2007. Validation of the barcoding gene COI for use in forensic genetic species identification. Forensic Science International 173: 1-6. [ Links ]
DE SALLE, R.; EGAN, M.G.; SIDDALL, M. 2005. The unholy trinity: taxonomy, species delimitation and DNA Barcoding. Philosophical Transactions of the Royal Society B: Biological Sciences 360: 1905-1916. [ Links ]
FISHER, B. L.; SMITH, M. A. 2008. A revision of Malagasy species of Anochetus Mayr and Odontomachus Latreille (Hymenoptera: Formicidae). PLoS ONE 3(5): e1787. [ Links ]
FLÓREZ, E.; WOLFF, M. 2009. Descripción y clave de los estadios inmaduros de las principales especies de Calliphoridae (Diptera) de importancia forense en Colombia. Neotropical Entomology 38 (3): 418-429. [ Links ]
FOLMER, O.; BLACK, M.; HOEH, W.; LUTZ, R.; VRIJENHOEK, R. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology 3: 294-299. [ Links ]
GIRALDO, P. A.; URIBE, S. I.; LÓPEZ, A. 2011. Análisis de secuencias de ADN mitocondrial (Cytb y ND1) en Lucilia eximia (Diptera: Calliphoridae). Revista Colombiana de Entomología 37 (2): 273-278. [ Links ]
GUO, Y. D.; CAI, J. F.; XIONG, F.; WANG, H. J.; WEN, J. F.; LI, J. B.; CHEN, Y. Q. 2012. The utility of mitochondrial DNA fragments for genetic identification of forensically important sarcophagid flies (Diptera: Sarcophagidae) in China. Tropical Biomedicine 29 (1): 51-60. [ Links ]
GUIMARÄES, J. H. 1977. A systematic revision of the Mesembri-nellidae, stat. nov. (Diptera, Cyclorrhapha). Arquivos de Zoologia 29: 1-109. [ Links ]
HALL, T. A. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series 41: 95-98. [ Links ]
HARVEY, M. L.; GAUDIERI, S.; VILLET, M. H.; DADOUR, I. R. 2008. A global study of forensically significant calliphorids: Implications for identification. Forensic Science International 177: 66-76. [ Links ]
HEBERT, P. D. N.; GREGORY, T. R. 2005. The promise of DNA barcoding for taxonomy. Systematic Biology 54: 852-859. [ Links ]
HEBERT, P. D. N.; CYWINSKA, A.; BALL, S. L.; DE WAARD, J. R. 2003. Biological identifications through DNA barcodes. Proceedings of the Royal Society of London B Biology Sciences 270: 313-322. [ Links ]
HOGG, I. D.; HEBERT, P. D. 2004. Biological identifications of springtails (Hexapoda: Collembola) from the Canadian Arctic, using mitochondrial DNA barcodes. Canadian Journal of Zoology 82: 749-754. [ Links ]
HUELSENBECK, J. P.; RONQUIST, F. 2001. Mrbayes: Bayesian inference of phylogeny. Biometrics 17: 754. [ Links ]
JORDAENS, K.; SONET, G.; RICHET, R.; DUPONT, E.; BRAET, Y.; DESMYTER, S. 2013. Identification of forensically important Sarcophaga species (Diptera: Sarcophagidae) using the mi-tochondrial COI gene. International Journal of Legal Medicine 127: 491-504. [ Links ]
KIMURA, M. 1980. A simple model for estimating evolutionary rates of base substitutions through comparative studies of nucle-otide sequences. Journal of Molecular Evolution 16: 111-120. [ Links ]
LANTERI, A. A. 2007. Código de barras del ADN y sus posibles aplicaciones en el campo de la Entomología. Revista de la Sociedad Entomológica Argentina 66 (3-4): 15-25. [ Links ]
LOU, M.; GOLDING, G. B. 2007. Fingerprint: visual depiction of variation in multiple sequence alignments. Molecular Ecology Notes 7: 908-914. [ Links ]
MARINHO, M. A. T.; JUNQUEIRA, A. C. M.; PAULO, D. F.; ESPOSITO, M. C.; VILLET, M. H.; AZEREDO-ESPIN, A. M. L. 2012. Molecular phylogenetics of Oestroidea (Diptera: Calyptra-tae) with emphasis on Calliphoridae: Insights into the inter-familial relationships and additional evidence for paraphyly among blowflies. Molecular Phylogenetics and Evolution. 65: 840-854. [ Links ]
MEIKLEJOHN, K. A; WALLMAN, J. F.; DOWTON, M. 2011. DNA-based identification of forensically important Australian Sarcophagidae (Diptera). International Journal of Legal Medicine 125: 27-32. [ Links ]
MEYER, C. P.; PAULAY, G. 2005. DNA Barcoding: Error rates based on comprehensive sampling. Plos Biology 3 (12): E422. [ Links ]
NELSON, L. A.; WALLMAN, J. F.; DOWTON, M. 2007. Using COI barcodes to identify forensically and medically important blowflies. Medical and Veterinary Entomology 21: 44-52. [ Links ]
PAPE, T.; WOLFF, M.; AMAT, E. C. 2004. Los califoridos, estridos, rinoforidos y sarcofagidos (Diptera: Calliphoridae, Oestridae, Rhinophoridae, Sarcophagidae) de Colombia. Biota Colombiana 5 (2): 201-208. [ Links ]
POSADA, D. 2008. jModelTest: Phylogenetic model averaging. Molecular Biology and Evolution 25: 1253-1256. [ Links ]
RATNASINGHAM, S.; HEBERT, P. D. 2007. BOLD: The Barcode of Life Data System (www.barcodinglife.org). Molecular Ecology Notes 7: 355-364. [ Links ]
REMIGIO, E. A.; HEBERT, P. D. 2003. Testing the utility of partial COI sequences for phylogenetic estimates of gastropod relationships. Molecular Phylogenetics and Evolution 29 (3): 641-647. [ Links ]
ROE, A. D.; SPERLING, F. A. 2007. Patterns of evolution of mitochondrial cytochrome c oxidase I and II DNA and implications for DNA barcoding. Molecular Phylogenetics and Evolution 44 (1): 325-345. [ Links ]
SCHROEDER, H.; KLOTZBACH, H.; ELIAS, S.; AUGUSTIN, C.; PUESCHEL, K. 2003. Use of PCR-RFLP for differentiation of calliphorid larvae (Diptera, Calliphoridae) on human corpses. Forensic Science International 132: 76-81. [ Links ]
SINGH, B.; WELLS, J. D. 2011. Chrysomyinae (Diptera: Calliphoridae) is monophyletic: a molecular systematic analysis. Systematic Entomology 36 (3): 415-420. [ Links ]
STEVENS, J.; WALL, R. 2001. Genetic relationships between blowflies (Calliphoridae) of forensic importance. Forensic Science International 120: 116-123. [ Links ]
TAMURA, K.; PETERSON, D.; PETERSON, N.; STECHER, G.; NEI, M.; KUMAR, S. 2011. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution 28 (10): 2731-2739. [ Links ]
THOMPSON, J. D.; HIGGINS, D. G.; GIBSON, T. J. 1994. CLUSTAL W: improving the sensitivity of the progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice. Nucleic Acids Research 22: 4673-4680. [ Links ]
WALLMAN, J. F. 2001. A key to the adults of species of blowflies in southern Australia known or suspected to breed in carrion. Medical and Veterinary Entomology 15: 433-437. [ Links ]
WELLS, J. F.; LAMOTTE, L. 2001. Estimating the postmortem interval. pp. 367-384. En: Jason, H.; Byrd; Castner, J. Forensic entomology: The utility of arthropods in legal investigations, Second edition. CRC Press. 440 p. [ Links ]
WHITWORTH, T. L.; DAWSON, R. D.; MAGALON, H.; BAUDRY, E. 2007. DNA barcoding cannot reliably identify species of the blowfly genus Protocalliphora (Diptera: Calliphoridae). Proceedings of the Royal Society B: Biological Sciences. 274: 1731-1739. [ Links ]
WOLFF, M.; URIBE, A.; ORTIZ, A.; DUQUE, P. 2001. A preliminary study of forensic entomology in Medellin, Colombia. Forensic Science International 120: 53-59. [ Links ]
YADONG, G.; JIFENG, C.; XINGHUA, W.; LINGMEI, L.; QIN-LAI, L.; XIANG, L.; YUNFENG, C.; ZHONG, M.; XIANG, W.; JIFANG, W. 2010. Identification of forensically important sarcophagid flies (Diptera: Sarcophagidae) based on COI gene in China. Romanian Journal of Legal Medicine 18 (3): 217-224. [ Links ]
Citación sugerida:
SOLANO, JAIBER J.; MARTA WOLFF y LYDA R. CASTRO. 2013. Identificación molecular de califóridos (Diptera: Calliphoridae) de importancia forense en Colombia. Revista Colombiana de Entomología 39 (2): 281-290.