Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Entomología
Print version ISSN 0120-0488
Rev. Colomb. Entomol. vol.41 no.2 Bogotá July/Dec. 2015
Código de barras para la tipificación de culícidos inmaduros de Armenia y Circasia (Quindío, Colombia)
DNA barcode for typing of immature mosquitoes from Armenia and Circasia (Quindío, Colombia)
RICHARD HOYOS-LÓPEZ 1,2, STEPHANIE ROMAN PARDO3,4, JHON CARLOS CASTAÑO3,5
y JUAN CARLOS GALLEGO-GÓMEZ1,6
1 Universidad de Antioquia, Facultad de Medicina, Grupo de Medicina Molecular y de Translación. Cra 51 D No 62-29, Edificio MUA - Oficina 303, Medellín - Antioquia, Colombia, teléfono/fax 574+2196924.
2 Biólogo, M. Sc., Ph. D. (c) en Biología. rhoyoslopez@gmail.com. Autor para correspondencia.
3 Universidad del Quindío, Facultad de Ciencias de la Salud, Grupo de Inmunología Molecular. Cra. 15 - Calle 12 Norte, Armenia - Quindío, Colombia, teléfono/fax: 576+7359374.
4 Bióloga. stefy11_79@hotmail.com.
5 Médico, Ph. D. en Ciencias Biomédicas. jhoncarlos@uniquindio.edu.co.
6 Biólogogenetista, Ph. D. en Biología Molecular. juanc.gallegomez@gmail.com.
Resumen: La presencia de mosquitos vectores que explotan hábitats urbanos y generan riesgo epidemiológico en la transmisión de enfermedades tropicales emergentes y reemergentes impulsan el establecer procedimientos rápidos y eficaces que posibiliten la identificación de especies con interés en salud pública. El método código de barras ("DNA barcode") utiliza secuencias de un fragmento del gen citocromo c oxidasa I para la identificación de un amplio rango de especies animales y representa una alternativa a evaluar en grupos de interés en medicina tropical. En nuestro estudio, se realizaron colectas entomológicas en criaderos de áreas urbanas en Armenia y Circasia (Quindío-Colombia) para identificar morfológicamente larvas de culícidos y evaluar el método de código de barras. Secuencias ADN fueron obtenidas para 56 culícidos inmaduros de Aedes (Stegomyia) aegypti, Culex (Culex) quinquefasciatus, Cx. (Phenacomyia) corniger, Cx. (Culex) nigripalpus y Cx. (Culex) grupo Coronator. Se estimó la diversidad genética, distancias genéticas intra/inter-especie y dendrogramas de "neighbor-joining". Los resultados permitieron la separación de los mosquitos y el potencial uso del marcador en genética de poblaciones. Sin embargo, la especie del subgénero Culex, grupo Coronator, no presentó información suficiente para la discriminación taxonómica. A nivel epidemiológico, las especies urbanas identificadas presentan antecedentes en la transmisión de alfavirus y flavivirus emergentes de interés en la salud pública colombiana donde el uso de códigos de barra puede facilitar estudios de ecología de mosquitos en áreas urbanas y la vigilancia epidemiológica de patógenos emergentes.
Palabras clave: Culícidos. Inmaduros. Distancias genéticas. "Neighbor-joining".
Abstract: The exploitation of urban habitats by mosquitoes species contributes to the epidemiological risk of transmission of emerging and re-emerging tropical diseases, indicating the need to establish fast and effective procedures than enable the taxonomic identification of species of interest to public health. DNA-barcode methodology uses sequences of mitochondrial cytochrome c oxidase I genes for the identification of a wide range of animal species and is an alternative evaluation tool for important groups of tropical medical importance. In this study, entomological samplings were performed at breeding sites of urban areas in Armenia and Circasia (Quindío-Colombia) in order to identify the morphology of mosquito larvae and to assess DNA-barcodes. Fifty-six (56) DNA-barcode sequences were obtained for immatures of the Aedes (Stegomyia) aegypti, Culex (Culex) quinquefasciatus, Cx. (Phenacomyia) corniger, Cx. (Culex) nigripalpus, and Cx. (Culex) Coronator groups and estimates of genetic diversity, intra/inter-species genetic distances, and neighbor-joining dendrograms were found. The results allowed for the taxonomic identification of mosquitoes and the separation of potentially useful molecular markers within the genetics of these populations; however, for species belonging to the Culex–Coronatorgroup, the molecular markers had insufficient information for the taxonomic separation of species. Epidemiologically, urban mosquitoes identified have been implicated in the past in the transmission of emerging and re-emerging alphavirus and flavivirus of interest to Colombian public health. Thus, the use of DNA-barcoding may be useful in ecological studies in urban areas and in the epidemiological surveillance of emerging pathogens.
Key words: Culicidae. Immatures. Genetic distances. Neighbor-joining.
Introducción
La familia Culicidae comprende alrededor de 3.490 especies reconocidas en diversos ecosistemas y regiones biogeográficas (Harbach y Howard 2007) de las cuales aproximadamente 150, pertenecientes a los géneros Anopheles (Meigen), Aedes (Meigen) y Culex (Linnaeus), son responsables de la transmisión vectorial de virus, bacterias, helmintos y protozoos de importancia en salud pública humana y veterinaria (Gubler 2002; Harbach 2007). En Colombia, la presencia de protozoos y virus transmitidos por insectos, genera un interés en la diversidad de mosquitos presentes en focos activos de transmisión para patógenos emergentes y re-emergentes, donde la identificación de especies se considera el primer paso en la incriminación, vigilancia y control de estas enfermedades de transmisión vectorial (Krzywinski 2003; Harbach 2007; Ondrejicka 2014). Sin embargo, la identificación de mosquitos, es principalmente realizada con caracteres morfológicos que frecuentemente sufren daños o alteraciones durante el proceso de colecta, almacenamiento y transporte o, probablemente, no se encuentran en todos los estados del ciclo de vida (Wang et al. 2012). Además durante los muestreos entomológicos, por lo general se capturan pocos machos para la confirmación de especies y una alta cantidad de hembras adultas cuyos caracteres diagnósticos varían sutilmente y solo pueden ser reconocidos por taxónomos experimentados o no se encuentran disponibles atributos para la diferenciación taxonómica. La similaridad morfológica dificulta el proceso de identificación de especies, particularmente en grupos que presentan complejos de especies crípticas (Krzywinski 2003; Cywinska et al. 2006; Kumar et al. 2007; Taira et al. 2012; Wang et al. 2012; Versteirt et al. 2014). En este sentido, un acercamiento multidisciplinario e integrativo a la taxonomía de mosquitos vectores que incluya caracteres morfológicos, moleculares y ecológicos (Schlick-Steiner et al. 2010), es necesario para agilizar la identificación a nivel de especie (Besansky et al. 2003).
El uso de secuencias de ADN ha sido propuesto como base fundamental en la clasificación biológica (Tautz et al. 2003) y como herramienta analítica para el reconocimiento e identificación de especies en grupos particularmente complejos (Besansky et al. 2003). Una alternativa metodológica propuesta por Hebert et al. (2003), involucra el análisis de secuencias de un fragmento del gen citocromo c oxidasa I (COI) que sirve para la discriminación de especies de difícil separación morfológica (Besansky et al. 2003; Ondrejicka et al. 2014) y constituye un sistema de bio-identificación por códigos de barras o "DNA barcode" (Valentini 2008). La iniciativa ha sido evaluada en diferentes insectos vectores con resultados significativamente exitosos en la separación de especies presentes en zonas activas de transmisión de enfermedades y en dípteros como Simuliidae (Rivera y Currie 2009), Phlebotominae (Azpurua et al. 2010; Hoyos et al. 2012a; Kumar et al. 2012; Contreras et al. 2014), Tabanidae (Cywinska et al. 2010) y Calliphoridae (Solano et al. 2013). En mosquitos hay un evidente progreso en la tipificación molecular de especies epidemiológicamente importantes en diversas zonas geográficas de países como Canadá (Cywinska et al. 2006), India (Kumar et al. 2007), Bélgica (Versteirt et al. 2014), Pakistán (Ashfaq et al. 2014), Argentina (Díaz-Nieto et al. 2013), Japón (Taira et al. 2012), Ecuador (Linton et al. 2013) y China (Wang et al. 2012). Este método también ha sido usado en la separación de complejos de especies (Rosero et al. 2012; Kumar et al. 2013; Moreno et al. 2013), identificación de potenciales vectores de arbovirus (Golding et al. 2012) y en taxonomía integrativa (Ruiz et al. 2010; Ruiz et al. 2012; Laurito et al. 2013).
Colombia presenta amplias áreas geográficas activas para la transmisión de enfermedades tropicales que involucran mosquitos, entre ellas malaria, dengue, fiebre amarilla, encefalitis equina venezolana, varios arbovirus de baja circulación (mayaro, Ilheus, Oropouche, bussuquara, guaroa, guama) y recientemente chikungunya (Tinker y Olano 1993; Groot et al. 1996; Rodríguez et al. 1995; Rivas et al. 1995; Ferro et al. 2003; Mattar et al. 2005; Montoya-Lerma et al. 2011; González y Mattar 2015). Algunas de estas patologías tropicales se encuentran presentes en zonas peri-urbanas y urbanas por la adaptación y colonización de mosquitos a hábitats artificiales en ecosistemas humanos, cambio climático, la actividad antropogénica en la fragmentación de ecosistemas naturales y el consecuente cambio en la estructura y composición de las comunidades de mosquitos (Gubler 2002; Yanoviak et al. 2006; Jones et al. 2008; Keesing et al. 2010; Jinbo et al. 2011; Hoyos et al. 2012).
La identificación taxonómica permite además determinar aspectos como la transmisión trans-ovárica, la identificación de hábitats y criaderos en remanentes urbanos de vegetación nativa y/o artificial favorecen la colonización de mosquitos vectores en zonas residenciales con alta densidad poblacional humana, cobrando alta relevancia en el manejo y control de enfermedades emergentes y re-emergentes, así como en términos de vigilancia entomológica para identificar posibles re-colonizaciones y re-emergencia de micro-organismos patógenos (Parra et al. 2012; Hoyos et al. 2012). En nuestro estudio, evaluamos el fragmento códigos de barras del gen citocromo c oxidasa I en estadios inmaduros de mosquitos colectados en áreas urbanas de los municipios de Armenia y Circasia del departamento de Quindío para evaluar su utilidad en la vigilancia entomológica y el riesgo epidemiológico en la transmisión de protozoos, bacterias y virus.
Materiales y métodos
Obtención de especímenes. Los inmaduros de culícidos fueron recolectados en salidas de vigilancia entomológica en compañía con la Secretaría de Salud Departamental en septiembre y octubre de 2013 en áreas urbanas de los municipios de Armenia (4°32’20"N, 75°40’21"O) y Circasia (4°37’09"N, 75°38’05"O) del departamento de Quindío. Para el muestreo se siguió el método de búsqueda de Belkin et al. (1969) y los inmaduros fueron capturados, con una pipeta plástica, en criaderos artificiales asociados a viviendas humanas y colocados en recipientes previamente codificados para el transporte de los culícidos al Centro de Investigaciones Biomédicas (CIBM) de la Universidad del Quindío para su seguimiento. Larvas en estadío IV (L4) fueron separadas y guardadas en etanol absoluto y enviadas al laboratorio de Medicina Molecular y Translación de la Universidad de Antioquia. Previo al procesamiento con los protocolos moleculares, las larvas L4 fueron identificadas considerando género y características morfológicas externas con claves dicotómicas (Lane 1953; Cova-García et al. 1966; Bram 1967; Forattini 2002; González y Carrejo 2007). Pupas perdidas por mortalidad fueron usadas para verificar la utilidad del código de barras en la identificación taxonómica de especie.
Protocolos moleculares. Las extracciones de ADN fueron realizadas con el kit DNeasy Blood & Tissue kit (Qiagen, Maryland) y la amplificación del fragmento "DNA barcode" de ~700 pb del gen mitocondrial citocromo oxidasa I fue lograda mediante los oligonucleótidos LCO-1490/HCO-2198 y MTNF/MTNR (Folmer et al.1994; Hebert et al. 2003; Kumar et al. 2007). Cada mix-PCR contenía un amortiguador1x de NH4SO4, 1 mM de cada DNTP, 5 mM de MgCl2, 0,5 µM de oligonucleótidos cebadores, 0,4 U de taq polimerasa (Bioline, Maryland), 4 µL de ADN y volumen final de 50 µL. Los parámetros de amplificación en el termociclador Multigene (Labnet, New Jersey) incluyeron: un ciclo de 94 °C por 10 min, seguido de 35 ciclos de la siguiente forma: 95 °C por 60 s, 50 °C por 60 s y 72 °C por 60 s, una extensión final a 72 °C por 5 min y preservación a 4 °C. Los productos de PCR fueron visualizados en gel de agarosa (1%) con tinción GELSTAR® (Lonza, Rockland) diluido 1/50 y el lector Dark Reader (IMGEN, Alexandria). Los productos de PCR positivos fueron secuenciados mediante los cebadores de amplificación previamente usados en Macrogen (Seul, Corea).
Análisis de datos. Las secuencias fueron editadas manualmente en el software Bioeditv7.2.0 (Hall 1999) (http://www. mbio.ncsu.edu/BioEdit/bioedit.htm) y los consensos en formato fasta fueron alineados en ClustalW (Larkin et al. 2007). Las distancias genéticas fueron estimadas en MEGAv6.0 (Tamura et al. 2013) usando el modelo Kimura 2-parámetros (K2P) (Kimura 1980), y las unidades taxonómicas moleculares operacionales (MOTUs) fueron identificadas de acuerdo a las distancias genéticas intra e inter-especie calculadas y de acuerdo a agrupaciones dentro de un dendrograma inferido por el algoritmo de "neighbor-joining" (NJ) (Saitou y Nei 1987) (modelo K2P, "bootstrap" = 1.000 réplicas) (Felsenstein 1982). Se estimaron parámetros de diversidad genética como número de sitios polimórficos, número de haplotipos, diversidad haplotípica, diversidad nucleotídica y prueba de neutralidad de Tajima con el software DNAspv5.0 (Librado y Rozas 2009).
Resultados
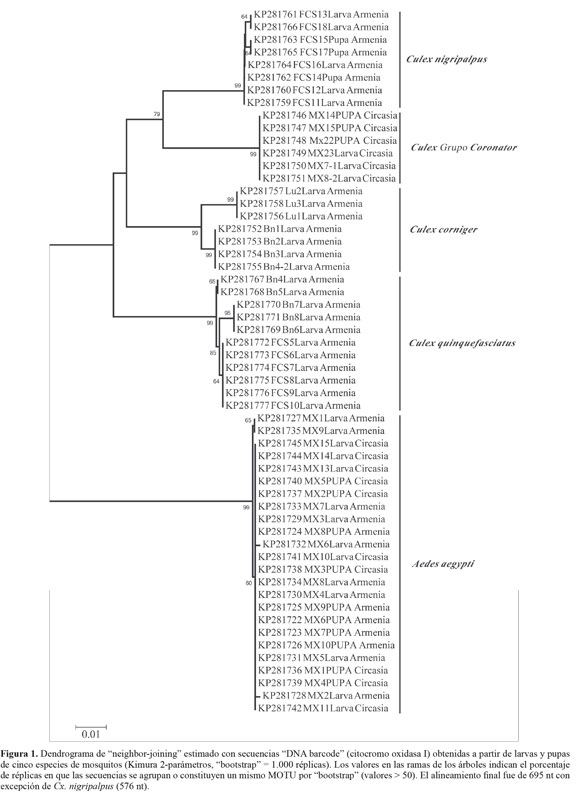
Cinco especies fueron identificadas: Aedes (Stegomyia) aegypti, Culex (Culex) quinquefasciatus, Cx. (Phenacomyia) corniger, Cx. (Culex) nigripalpus y Cx. (Culex) grupo Coronator. Este último grupo no fue identificado al nivel de especie por varias razones: la similaridad morfológica de los estadios inmaduros, la dificultad de separar morfológicamente especies en las hembras adultas que emergen de los mismos criaderos y la ausencia de machos en estos criaderos para realizar disecciones de genitalia y obtener caracteres que permitan una identificación exitosa a nivel de especie. 56 especímenes fueron seleccionados para la extracción de ADN, amplificación por PCR del gen COI - fragmento "DNA barcode", secuenciación y registro en GenBank (Tabla 1).
Las secuencias obtenidas presentan una longitud de 695 nt, con excepción de las pertenecientes a Cx. nigripalpus (576 pb), y corresponden a las posiciones 1541 a 2201 del gen mitocondrial citocromo oxidasa I (secuencia de cia en GenBank EU352212.1 de COI - Aedes aegypti), región consecuente con la ubicación del fragmento código de barras (posiciones 1498 - 2198) (Hebert et al. 2003); no se evidenciaron eventos de inserción-deleción, en las secuencias analizadas o la presencia de codones de parada, característica propia de copias nucleares de genes mitocondriales (NUMT´s) (Black IV y Bernhardt 2009).
Un total de 56 secuencias COI - DNA barcode (10 haplotipos) pertenecientes a cinco especies de dos géneros de Culicidae fueron obtenidos y las distancias intra-específicas en individuos de la misma especie o MOTU´s fueron bajas (0 - 0,00924) (Tabla 2). Las distancias genéticas inter-específicas fueron consistentes en el rango registrado entre especies del mismo género para Culex spp. con rangos de 5,9-7,9%, y entre especies de géneros diferentes (12,5-13,7%) (Tabla 3). Sin embargo, la diversidad genética de las cinco especies fue baja en haplotipos, diversidad haplotípica y diversidad nucleotídica (Tabla 2). La prueba de Tajima fue estadísticamente significativa para Cx. corniger evidenciando evolución no neutral. La caracterización molecular de COI - barcode para Aedes aegypti permitió obtener secuencias para dos poblaciones (Circasia y Armenia), y estimar el índice de estructuración genética (Fst = 0,038) y el flujo de genes (Nm = 12,50) (Hudson, Slatkin y Madison 1992), demostrando un alto intercambio de migrantes y escasa diferenciación genética.
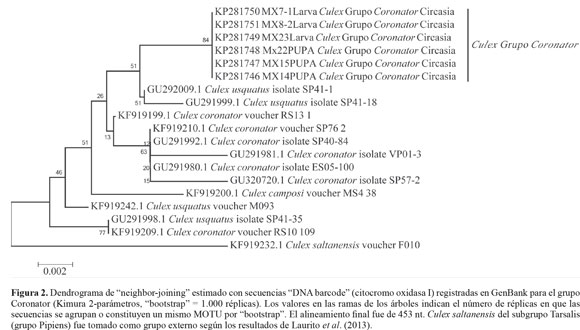
La separación de especies de mosquitos por distancias genéticas fue confirmada por la agrupación de cinco clusters o MOTU´s en el dendrograma de neighbor-joining, y las pupas incluidas en este análisis fueron identificadas (Fig. 1). Los especímenes correspondientes a Culex - grupo Coronator presentan diversidad genética para ser considerados un solo taxón, además se encuentran por debajo del punto de corte (< 2%) considerado por el método "DNA barcode" como propio de una especie (Hebert et al. 2003; Cywinska et al. 2006; Kumar et al. 2007; Taira et al.2012; Wang et al. 2012; DíazNieto et al. 2013; Laurito et al. 2013; Ashfaq et al. 2014). Un alineamiento con secuencias del grupo Coronator disponibles en GenBank y registradas en estudios del género Culex (Demari-Silva et al. 2011; Laurito et al. 2013) fue realizado para identificar la especie de este grupo colectada en Circasia (Quindío), sin embargo, el dendrograma resultante evidenció bajos valores de bootstrap y poca resolución para diferenciar las especies del grupo Coronator (Fig. 2).
Discusión
La colonización de hábitats urbanos por mosquitos de interés médico, constituye una información necesaria para el diseño de un esquema de control y prevención de enfermedades infecciosas emergentes y re-emergentes en ecosistemas humanos (Hoyos et al. 2012b). No obstante, previamente es necesaria la identificación morfológica de especies de la familia Culicidae, la cual es considerada particularmente problemática por el polimorfismo y solapamiento de caracteres asociados a larvas y hembras adultas (Harbach 2007; Demari-Silva et al. 2011; Laurito et al. 2013); usualmente los caracteres pertenecientes a la genitalia masculina que permiten una identificación más aguda a nivel de especie, están condicionados a la colecta de machos, otra dificultad, dado que en los muestreos entomológicos son escasamente colectados. En este sentido, el método "código de barras" o "DNA barcode" fue usado, para evaluar la información genética del fragmento de citocromo oxidasa I en la identificación y separación de especies a partir de especímenes inmaduros de mosquitos urbanos, analizar el potencial uso en estudios ecológicos y la relevancia epidemiológica de las especies presentes.
Cinco MOTU´s fueron separados por distancias genéticas (K2P) y dendrograma estimado por Neighbor-joining, permitiendo diferenciar secuencias pertenecientes a inmaduros colectados en zonas urbanas correspondientes a cinco especies identificadas mediante caracteres morfológicos, evidenciando un patrón de polimorfismos nucleotídicos que posibilita la identificación rápida de mosquitos presentes en hábitats y ecosistemas urbanos. Los resultados del método código de barras en las especies identificadas determinaron un rango intra-especifico de 0 - 0,00316 y 0,059 - 0,137 entre especies; estos valores se encuentran en los rangos intra-especie (0-0,024) e inter-especie (0,022-0,2565) previamente registrados para culícidos presentes en diversas áreas geográficas (Cywinska et al. 2006; Kumar et al. 2007; Taira et al. 2012; Wang et al. 2012; Ashfaq et al. 2014).
Aedes aegypti es responsable de la transmisión del virus dengue, el arbovirus más importante en Colombia (Tinker y Olano 1993), y está comprometido en la transmisión de fiebre amarilla en algunas zonas urbanas a pesar de la erradicación intensiva de este ciclo en grandes zonas del país (Rodríguez et al. 1996). Esta especie presenta un patrón de ingesta sanguínea antropofílico y se encuentra bien adaptado a los hábitats urbanos (Harrington et al. 2001). Es considerado como el actual responsable de la transmisión y epidemia del virus chikungunya en Colombia (González y Mattar 2015). A esta especie correspondió el mayor número de especímenes tipificados molecularmente, y los estimativos de diversidad genética, estructuración y flujo de genes, indican una alta movilidad de individuos en las dos localidades estudiadas, contrastando con lo registrado por Caldera et al. (2013), quienes en dos localidades del departamento de Sucre obtuvieron para la misma especie, secuencias de un fragmento de ND4 con una significativa estructuración genética y limitado flujo de genes. Probablemente la alta similaridad genética en las poblaciones de Armenia y Circasia, sean consecuencia de la dispersión pasiva humana, la alta movilidad poblacional de residentes en la zona y la poca distancia entre las dos localidades (16,5 km) (Scarpassa et al. 2008; Caldera et al. 2013). Cabe destacar la presencia de Ae. albopictus en Barrancabermeja (Gutiérrez et al. 2010), Leticia (Vélez et al. 1998), Cali (Cuellar et al. 2007), Buenaventura (Suárez 2001) y Medellín (Rúa et al. 2011), habitando principalmente en regiones forestales y espacios abiertos con abundante vegetación, teniendo una distribución en simpatría con Ae. aegypti y co-habitando criaderos naturales y artificiales (Rúa et al. 2012), el aporte de secuencias del código de barras de Ae. aegypti permitiría la discriminación de ambas especies y facilitaría estudios en ecología e infección natural de arbovirus de interés médico en las regiones donde presentan simpatría, así como tareas de vigilancia entomológica.
Culex quinquefasciatus, fue hallado en alta proporción en hábitats urbanos de Armenia y se ha encontrado asociado a fragmentos de bosque con baja intervención humana y áreas rurales (Parra et al. 2012), considerándose una especie versátil en la ocupación de hábitats disponibles en ecosistemas humanos, ornitofílicos, pero adaptable al ambiente antrópico, siendo un vector del virus Nilo del occidente (WNV), virus de la encefalitis de San Luis (SLEV), dengue (DENV), virus Sindbis y virus de la fiebre del Valle Rift (Andreadis et al. 2004; Diaz et al. 2013; Turell 2012). La diversidad genética de la secuencia código de barras para Cx. quinquefasciatus fue significativa en diversidad haplotípica, pero limitada en términos de sitios polimórficos y diversidad nucleotídica, al igual que poblaciones venezolanas de esta especie evaluadas mediante un fragmento de 556 nt de COI (Quintero y Navarro 2013), perteneciente a una región homóloga a la secuencia del código de barras, señalando una variabilidad y polimorfismo apropiados para el análisis filogenético y estructuración de poblaciones, en comparación con marcadores mitocondriales y nucleares amplificados y secuenciados en otros estudios (Hasan et al. 2009). A pesar de la importancia de esta especie en la transmisión de arbovirus emergentes y la explotación de hábitats urbanos, en Colombia se desconocen los efectos causados por los insecticidas y métodos de control empleados para Ae. aegypti, así como los patrones de estructuración genética y flujo de genes que pueden originar variabilidad en la competencia vectorial para algunos patógenos virales como WNV y SLEV (Andreadis 2012; Shin 2014) presentes en Colombia (Mattar et al. 2005; Jaramillo et al. 2005; Góes-Rivillas et al. 2010; Mattar et al. 2011; Osorio et al. 2012). Parra et al. (2012) registran a Cx. nigripalpus en la vereda Aguas Claras en el municipio de Turbo (Antioquia), destacando la presencia en una localidad boscosa, la plasticidad ecológica para esta especie y sus hábitos alimenticios ornitofílicos (Guimaraes et al. 2000; Forattini 2002). Esta especie ha sido hallada infectada naturalmente con SLEV, WNV, virus de la encefalitis equina venezolana (VEEV) y linaje III del virus encefalitis equina del este (EEV) (Dow et al. 1964; Day y Curtis 1999; Rutledge et al. 2003; Arrigo et al. 2010).
Los inmaduros colectados en Circasia (Quindío) pertenecientes al grupo Coronator(Cx. coronator, Cx. camposi y Cx. usquatus), no fueron identificados a nivel de especie, por la dificultad en el reconocimiento de caracteres diagnósticos específicos y la ausencia de machos en los especímenes que emergieron de los criaderos evaluados, además el marcador COI-"DNA barcode", presenta bajos valores de distancias genéticas (K2P) y poca información genética que refleje la diferenciación morfológica de Cx. coronator, Cx. usquatus y Cx. camposi, a nivel de la genitalia masculina en las setas apicales del gonocoxito (Bram 1967). Estos resultados son similares a los obtenidos por Demari-Silva et al. (2011), en el análisis de un fragmento de citocromo oxidasa I - homólogo a la región "DNA barcode" (478 nt), donde las distancias genéticas y el dendrograma evidencian baja diferenciación molecular para Cx. coronator y Cx. usquatus. Laurito et al. (2011), describe el mismo patrón de poca discriminación del marcador código de barras para el grupo Coronator incluyendo a Cx. camposi, y la politomía en la agrupación inferido en el dendrograma de "neighbor-joining" y el clado estimado de máxima parsimonia, que observan una alta cercanía con especies del subgrupo Tarsalis del grupo Pipiens (Cx. maxi y Cx. surinamensis) a pesar de las claras evidencias morfológicas. Para el grupo Coronator, es necesaria la caracterización de marcadores moleculares mitocondriales o nucleares que permitan la diferenciación e identificación de estas especies y faciliten estudios en focos epidemiológicos de enfermedades emergentes y re-emergentes. Cx. corniger, miembro del subgénero Phenacomyia, es distinguible de su especie más cercana Cx. lactator, por la quetotaxia del cuarto estadío larval (Strickman y Pratt 1989; Harbach y Peyton 1992) y ha sido hallado infectado naturalmente con el virus Everglades (EVEV), miembro del Complejo - VEEV (subtipo IE) (Farrar et al. 2014). La significancia estadística en la prueba de neutralidad de Tajima, indica posibles procesos poblacionales de cuello de botella por presión humana asociada a insecticidas o intervención antropogénica en esta especie (Tajima 1989; Nielsen 2005; Caldera et al. 2013).
La identificación por COI - código de barras de mosquitos inmaduros en hábitats urbanos, constituye un método alternativo para estudiar la presencia de mosquitos vectores de arbovirus emergentes y re-emergentes en áreas con Ae. aegypti y transmisión activa de dengue. Además permite relacionar la biodiversidad con la heterogeneidad del paisaje en un foco de transmisión (Calderón-Arguedas et al. 2009), establecer los elementos ecológicos y evolutivos que pueden favorecer la aparición de nuevos patógenos o variantes evolutivas virulentas para poblaciones humanas y las interacciones con hospederos y/o reservorios (Hoyos et al. 2012b). Los resultados en el grupo Coronator con el código de barras implican la caracterización y análisis de marcadores moleculares que permitan la identificación de estas especies cercanas evolutivamente.
Finalmente, la identificación con marcadores moleculares y en especial el método código de barras en mosquitos urbanos, permite establecer posibles riesgos epidemiológicos en la transmisión de enfermedades emergentes y re-emergentes, y en consecuencia establecer los elementos ecológicos que favorecen la presencia de una nueva patología, teniendo en cuenta la presión antropogénica sobre los ecosistemas naturales, la adaptación de especies resilientes a los cambios del paisaje, los cambios en la estructura y composición de comunidades de insectos hematófagos, el aumento en la frecuencia de contacto humano-vector, los animales domésticos y las poblaciones humanas susceptibles. Además, es clara la necesidad de tipificar molecularmente con códigos de barras a una mayor cantidad de mosquitos en ecosistemas naturales para construir una base de referencia para futuros estudios de biodiversidad, ecología y predicción de nichos que impliquen la transmisión de arbovirus patógenos.
Agradecimientos
A Colciencias por los recursos aprobados para el proyecto 111549326198 "Rastreo de virus emergentes y re-emergentes en mosquitos silvestres de la costa caribe colombiana" y la convocatoria 528 para Becas de Doctorados Nacionales-Colciencias. A los técnicos de vectores de las secretarías de salud de los municipios de Armenia y Circasia por su acompañamiento en las salidas de campo.
Literatura citada
ANDREADIS, T. G. 2012. The contribution of Culex pipiens complex mosquitoes to transmission and persistence of West Nile Virus in North America. Journal of the American Mosquito Control Association 28 (4): 137-151. [ Links ]
ANDREADIS, T. G.; ANDERSON, J. F.; VOSSBRINCK, C. R.; MAIN, A. J. 2004. Epidemiology of West Nile virus in Connecticut: a five-year analysis of mosquito data, 1999-2003. VectorBorne and Zoonotic Diseases 44: 360-378. [ Links ]
ARRIGO, N.; ADAMS, P.; WEAVER, S. 2010. Evolutionary patterns of Eastern Equine Encephalitis Virus in North versus South America suggest ecological differences and taxonomic revision. Journal of Virology 84 (2): 1014 -1025. [ Links ]
ASHFAQ, M.; HEBERT, P. D. N.; MIRZA, J. H.; KHAN, A. M.; ZAFAR, Y. 2014. Analyzing mosquito (Diptera: Culicidae) diversity in Pakistan by DNA barcoding. PLoS ONE 9 (5): e97268. [ Links ]
AZPURUA, J.; CRUZ, D.; VALDERRAMA, A.; WINDSOR, D. 2010. Lutzomyia sand fly diversity and rates of infection by Wolbachia and an exotic Leishmania species on Barro Colorado Island, Panama. PLoS Neglected Tropical Diseases 4: 1-8. [ Links ]
BENARROCH, E. I. 1931. Studies on malaria in Venezuela. American Journal of Epidemiology 14: 690–693. [ Links ]
BESANSKY, N.; SEVERSON, D.; FERDIG, M. 2003. DNA barcoding of parasites and invertebrate disease vectors: what you don't know can hurt you. Trends in Parasitology 19: 545-546. [ Links ]
BLACK IV, W. C.; BERNHARDT, S. A. 2009. Abundant nuclear copies of mitochondrial origin (NUMTs) in the Aedes aegypti genome. Insect Molecular Biology 18 (6): 705-713. [ Links ]
BLAXTER, M. L. 2004. The promise of a DNA taxonomy. Philosophical Transactions of the Royal Society B: Biological Sciences 359: 669-679. [ Links ]
BRAM, R. 1967. Classification of Culex subgenus Culex (Diptera: Culicidae). Proceedings of the United States National Museum 120: 1-123. [ Links ]
CALDERA, S. M.; JARAMILLO, M. C.; COCHERO, S.; PÉREZDORIS, A.; BEJARANO, E. E. 2013. Diferencias genéticas en- tre poblaciones de Aedes aegypti de municipios del Norte de Colombia, con baja y alta incidencia de dengue. Biomédica 33 (Supl.1): 89-98. [ Links ]
CALDERÓN-ARGUEDAS, O.; TROYO, A.; SOLANO, M.; AVENDAÑO, A.; BEIER, J. 2010. Urban Mosquitoes species (Diptera: Culicidae) of dengue endemic communities in the Greater Puntarenas area, Costa Rica. Revista de Biología Tropical 57 (4): 1223-1234. [ Links ]
CONTRERAS, M. A.; VIVERO, R.; VELEZ, I. D.; PORTER, C.; URIBE, S. 2014. DNA barcoding for the identification of sand fly species (Diptera, Psychodidae, Phlebotominae) in Colombia. PloS ONE 9 (1): e85496. [ Links ]
COVA-GARCÍA, P.; SUTIL, E.; RAUSSEO, J. A. 1966. Mosquitos (Culicinos) de Venezuela: Tomo I and Tomo II. Ministerio de Sanidad y Asistencia Social, Caracas. 816 p. [ Links ]
CYWINSKA, A.; HANNAN, M.; KEVAN, P.; ROUGHLEY, R.; IRANPOUR, M.; HUNTER. F. 2010. Evaluation of DNA barcoding and identification of new haplomorphos in Canadian deerflies and horseflies. Medical and Veterinary Entomology 24: 382-410. [ Links ]
CYWINSKA, A.; HUNTER, F. F.; HEBERT, P. D. 2006. Identifying Canadian mosquito species through DNA barcodes. Medical and Veterinary Entomology 20: 413-424. [ Links ]
DAY, J. F.; CURTIS, G. A. 1999. Blood feeding and oviposition by Culex nigripalpus (Diptera: Culicidae) before, during, and after a widespread St. Louis encephalitis virus epidemic in Florida. Journal of Medical Entomology 36: 176-181. [ Links ]
DIAZ, L. A.; FLORES, F. S.; BERANEK, M.; RIVAROLA, M. E.; ALMIRÓN, W. R.; CONTIGIANI, M. S. 2013. Transmission of endemic St Louis encephalitis virus strains by local Culex quinquefasciatus populations in Cordoba, Argentina. Transactions of the Royal Society of Tropical Medicine and Hygiene 107 (5): 332-334. [ Links ]
DÍAZ-NIETO, L.; MACIÁ, A.; PARISI, G.; FARINA, J.; VIDALDOMÍNGUEZ, M.; PEROTTI, M.; BERÓN, C- 2013. Distribution of mosquitoes in the South East of Argentina and first report on the analysis based on 18S rDNA and COI sequences. PloS ONE 8 (9): e75516. [ Links ]
DOW, R. P.; COLEMAN, P. H.; MEADOWS, K. E.; WORK, T. H. 1964. Isolation of St. Louis encephalitis viruses from mosquitoes in the Tampa Bay area of Florida during the epidemic of 1962. The American Journal of Tropical Medicine and Hygiene 13: 462-474. [ Links ]
FARRAR, J.; HOTEZ, P.; JUNGHANSS, T.; KANG, G.; LALLO, D.; WHITE. N. 2014. Manson´s Tropical Diseases, 23rd Edition. Saunders Ltda. Oxford, EEUU. 1552 p. [ Links ]
FELSENSTEIN, J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39: 783-791. [ Links ]
FOLMER, O.; BLACK, M.; HOEH, W.; LUTZ, R.; VRIJENHOEK, R. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology 3: 294-299. [ Links ]
FORATTINI, O. P. 2002. Culicidologia médica: identificação, biologia e epidemiologia, vol. II, EDUSP, São Paulo. 864 p. [ Links ]
GÓES-RIVILLAS, Y.; TABORDA, N.; DÍAZ, F.; GÓNGORA, A.; RODAS, J. D.; RUIZ-SÁENZ, J.; OSORIO, J. E. 2010. Antibodies to west Nile virus in equines of Antioquia and Meta, Colombia, 2005-2008. Revista Colombiana de Ciencias Pecuarias 23: 462-470. [ Links ]
GOLDING, N.; NUNN, M.; MEDLOCK, J.; PURSE, B.; VAUX, A.; SCHAFER, S. 2012. West Nile virus vector Culex modestus established in southern England. Parasites and Vectors 5: 32. [ Links ]
GONZÁLEZ, M.; MATTAR, S. 2015. Chikungunya: few months after attack. Revista MVZ Córdoba 19 (2): 4045-4046. [ Links ]
GROOT, H.; MORALES, A.; ROMERO, M.; FERRO, C.; PRÍAZ, E.; VIDALES, H.; BUITRAGO, B.; OLANO, V.; CALVACHE, D.; MÁRQUEZ, G.; DE LA VEGA, P.; RODRÍGUEZ, G. 1996. Estudios de arbovirosis en Colombia en la década de 1970. Biomédica 16: 331-344. [ Links ]
GUBLER, D. 2002. The global emergence/resurgence of arboviral diseases as public health problems. Archives of Medical Research 33: 330-342. [ Links ]
GUIMARAES, A. E.; GENTILE, C.; LOPES, C.; SANT´ANNA, A.; JOVITA, A. 2000. Ecology of mosquitoes (Diptera: Culicidae) in areas of Serra da Bocaina National Park, Brazil. I - Habitat distribution. Revista de Saúde Pública 34: 243-250. [ Links ]
GÚTIERREZ, M.; ALMEIDA, O.; BARRIOS, H.; HERRERA, J.; RAMÍREZ, M.; RONDON, L. 2011. Hallazgo de Aedes albopictus (Diptera: Culicidae) en el municipio de Barrancabermeja, Colombia. Biomédica 31 (suppl3): 23-25. [ Links ]
HALL, T. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series 41: 95-98. [ Links ]
HARBACH, R. E. 2007. The Culicidae (Diptera): a review of taxonomy, classification and phylogeny. Zootaxa 1668: 591-638. [ Links ]
HARBACH, R. E.; HOWARD, T. M. 2007. Index of currently recognized mosquito species (Diptera: Culicidae). European Mosquito Bulletin 23: 1-66. [ Links ]
HARBACH, R. E.; KITCHING, I. J. 1998. Phylogeny and classification of the Culicidae (Diptera). Systematic Entomology 23: 327-370. [ Links ]
HARBACH, R. E.; PEYTON, E. L. 1992. A new subgenus of Culex in the neotropical region (Diptera: Culicidae). Mosquito Systematics 24 (3): 242-252. [ Links ]
HARRINGTON, L.; EDMAN, J.; SCOTT, T. 2001. Why do female Aedes aegypti (Diptera: Culicidae) feed preferentially and frequently on human blood?. Journal of Medical Entomology 38: 411-422. [ Links ]
HASSAN, A. U.; SETSUO, S.; UDDIN, S. M.; FUJIMOTO, C.; HURADA, M.; RAHMAN, S. M. 2009. Molecular phylogeography of Culex quinquefasciatus mosquitoes in central Bangladesh. Acta Tropica 112: 106-114. [ Links ]
HEBERT, P. D.; CYWINSKA, A.; BALL, S. L.; DE WAARD, J. R. 2003. Biological identifications through DNA barcodes. Proceedings of the Royal Society B: Biological Sciences 270: 313-321. [ Links ]
HOYOS, R.; URIBE, S.; VELEZ, I. 2012a. Typification of Colombian specimens of Lutzomyia longipalpis (Diptera: Psychodidae) by "Barcoding". Revista Colombiana de Entomología 38: 134-140. [ Links ]
HOYOS, R.; USME, J.; GALLEGO-GOMEZ, J. C. 2012b. Viral evolutionary ecology: conceptual basis of a new scientific approach for understanding viral emergence. pp. 119-130. En: Nuno, L. (Ed.); Epidemiology - Current Perspectives on Research and Practice. InTech editorial, Rijeka, Croatia. 208 p. [ Links ]
HUDSON, R. R.; SLATKIN, M.; MADISON, W. P. 1992. Estimation of levels of gene flow from DNA sequence data. Genetics 132 (2): 583-589. [ Links ]
JARAMILLO, M.; PEÑA, J.; BERROCAL, L.; KOMAR, N.; GONZÁLEZ, M.; PONCE, C.; ARIZA, K.; MATTAR, S. 2005. Vigilancia centinela para el virus del Oeste del Nilo en culícidos y aves domésticas en el Departamento de Córdoba. Revista MVZ Córdoba 10: 633-638. [ Links ]
JINBO, U.; KATO, T.; ITO, M. 2011. Current progress in DNA bar- coding and future implications for entomology. Entomological Science 14: 107-124. [ Links ]
KEESING, F.; BELDEN, L. K.; DASZAK, P.; DOBSON, A.; HAR- VELL, D. C.; HOLT, R. D.; HUDSON, P.; JOLLES, A.; JONES, K. E.; MITCHELL, C. E.; MYERS, S. S.; BOGICH, T.; OSTFELD, R. S. 2010. Impacts of biodiversity on the emergence and transmission of infectious diseases. Nature 468: 647-652. [ Links ]
KIMURA, M. 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. Journal Molecular Evolution 16: 111-120. [ Links ]
KRZYWINSKI, J.; BESANSKY, N. J. 2003. Molecular systematics of Anopheles: From subgenera to subpopulations. Annual Review of Entomology 48: 111-139. [ Links ]
KUMAR, N. P.; KRISHNAMOORTHY, N.; SAHU, S.; RAJAVEL, A.; SABESAN, S.; JAMBULINGAM, P. 2013. DNA barcodes indicate members of the Anopheles fluviatilis (Diptera: Culicidae) species complex to be conspecific in India. Molecular Ecology Resources 13: 354-361. [ Links ]
KUMAR, N. P.; RAJAVEL, A. R.; NATARAJAN, R.; JAMBULINGAM, P. 2007. DNA barcodes can distinguish species of Indian mosquitoes (Diptera: Culicidae). Journal of Medical Entomology 44: 1-7. [ Links ]
KUMAR, N. P.; SRINIVASAN, R.; JAMBULIGAM, P. 2012. DNA barcoding for identification of sand flies (Diptera: Psychodidae) in India. Molecular Ecology Resources 3: 414-420. [ Links ]
LANE, J. 1953. Neotropical Culicidae - Vol 1. University of São Paulo, São Paulo, 548 p. [ Links ]
LARKIN, M. A.; BLACKSHIELDS, G.; BROWN, N. P.; CHEN- NA, R.; MCGETTIGAN, P. A.; MCWILLIAM, H.; VALEN- TIN, F.; WALLACE, I. M.; WILM, A.; LOPEZ, R.; THOMPSON, J. D.; GIBSON, T. J.; HIGGINS, D. G. 2007. Clustal W and CLUSTALX version 2.0. Bioinformatics 23: 2947-2948. [ Links ]
LAURITO, M.; DE OLIVEIRA, T.; ALMIRON, W.; SALLUM, M. A. 2013. COI barcode versus morphological identification of Culex (Culex) (Diptera: Culicidae) species: a case study using samples from Argentina and Brazil. Memórias do Instituto Oswaldo Cruz 108: 110-122. [ Links ]
LIBRADO, P.; ROZAS, J. 2009. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics 25: 1451-1452. [ Links ]
LINTON, Y. M.; PECOR, J.; PORTER, C.; MITCHELL, L.; GARZÓN-MORENO, A.; FOLEY, D.; PECOR, D.; WILKERSON, R. 2013. Mosquitoes of eastern Amazonian Ecuador: biodiversity, bionomics and barcodes. Memórias do Instituto Oswaldo Cruz 108: 100-109. [ Links ]
MATTAR, S.; EDWARDS, E.; LAGUADO, J.; GONZÁLEZ, M.; ALVAREZ, J.; KOMAR, N. 2005. West Nile virus infection in Colombian horses. Emerging Infectious Diseases 11: 14971498. [ Links ]
MATTAR, S.; KOMAR, N.; YOUNG, G.; ALVAREZ, J.; GONZALEZ, M. 2011. Seroconversion for West Nile and St. Louis encephalitis viruses among sentinel horses in Colombia. Memórias do Instituto Oswaldo Cruz 106: 976-979. [ Links ]
MORENO, M.; BICKERSMITH, S.; HARLOW, W.; HILDEBRANDT, J.; MCKEON, S.N.; SILVA-DO-NASCIMENTO, T. F.; LOAIZA, J. R.; RUIZ, F.; LOURENÇO-DE-OLIVEIRA, R.; SALLUM, M. A.; BERGO, E. S.; FRITZ, G. N.; WILKERSON, R. C.; LINTON, Y. M.; JURI, M. J.; RANGEL, Y.; PÓVOA, M. M.; GUTIÉRREZ, L.; BUILES, L. A.; CORREA, M. M.; CONN, J. E. 2013. Phylogeography of the neotropical Anopheles triannulatus complex (Diptera: Culicidae) supports deep structure and complex patterns. Parasites and Vectors 6: 47. [ Links ]
NIELSEN, R. 2005. Molecular signatures of natural selection. Annual Review of Genetics 39: 197-218. [ Links ]
OLANO, V.; TINKER, M. 1993. Ecología del Aedes aegypti en un pueblo de Colombia, Suramérica. Biomédica 13: 5-14. [ Links ]
ONDREJICKA, D.; LOCKE, S.; MOREY, K.; BORISENKO, A.; HANNER, R. 2014. Status and prospects of DNA barcoding in medically important parasites and vectors. Trends in Parasitology 30 (12): 582-591. [ Links ]
OSORIO, J.; CIUODERIS, K.; LOPERA, J.; PIEDRAHITA, L.; MURPHY, D.; LEVASSEUR, J.; CARRILLO, L.; OCAMPO, M.; HOFMEISTER, E. 2012. Characterization of West Nile viruses isolated from captive American flamingoes (Phoenicopterous ruber) in Medellin, Colombia. The American Journal of Tropical Medicine and Hygiene 87: 565-572. [ Links ]
PARRA, G.; SUÁREZ, L. 2012. Mosquitos (Diptera: Culicidae) vectores potenciales de arbovirus en la región de Urabá, noroccidente de Colombia. Biomédica 32: 252-262. [ Links ]
QUINTERO, L.; NAVARRO, J. 2012. Filogenia intra-específica y variabilidad genética de Culex quinquefasciatus Say (Diptera: Culicidae) con los genes mitocondriales ND5 y COI. Boletín de Malariología y Salud Ambiental LII (2): 45-65. [ Links ]
RIVAS, F.; DIAZ, A.; CARDENAS, M.; DAZA, E.; BRUZON, L.; ALCALA, A.; DE LA HOZ, O.; CACERES, M.; ARISTIZABAL, G.; MARTINEZ, J.; REVELO, D.; DE LA HOZ, F.; BOSHELL, J.; CAMACHO, T.; CALDERON, L.; OLANO, V.; VILLAREAL, L.; ROSELLI, D.; ALVAREZ, G.; LUDWING, G.; TSAI, T. 1995. Epidemic venezuelan equine encephalitis in La Guajira, Colombia, 1995. Journal Infectious Diseases 175: 828-832. [ Links ]
RIVERA, J.; CURRIE, D. 2009. Identification of neartic black flies using DNA barcodes (Diptera: Simuliidae). Molecular Ecology Resources 9: 224-236. [ Links ]
RODRÍGUEZ, G.; BOSHELL, J. 1996. Encefalitis equina venezolana. Biomédica 15: 172-182. [ Links ]
ROSERO, D.; JARAMILLO, L.; CONN, J.; CORREA, M. 2012. Genetic diversity of Anopheles triannulatus s.l. (Diptera: Culicidae) from northwestern and southeastern Colombia. The American Journal of Tropical Medicine and Hygiene 87: 910-920. [ Links ]
RÚA-URIBE, G.; SUÁREZ-ACOSTA, C.; LONDOÑO, V.; SÁNCHEZ, J.; ROJO, R.; BELLO-NOVOA, B. 2011. Detección de Aedes albopictus (Skuse) en la ciudad de Medellín, Colombia. Biomédica 31 (sppl3): 243-244. [ Links ]
RÚA-URIBE, G.; SUÁREZ-ACOSTA, C.; ROJO, R. 2012. Implicaciones epidemiológicas de Aedes albopictus (Skuse) en Colombia. Revista Facultad Nacional de Salud Pública 30 (3): 328-337. [ Links ]
RUIZ, F.; LINTON, Y.; PONSOBY, D.; CONN, J.; HERRERA, M.; QUIÑONES, M.; VELEZ, I. D.; WILKERSON, R. 2010. Molecular comparison of topotypic specimens confirms Anopheles (Nyssorhynchus) dunhami Causey (Diptera: Culicidae) in the Colombian Amazon. Memórias do Instituto Oswaldo Cruz 105: 899-903. [ Links ]
RUIZ, F.; WILKERSON, R.; CONN, J.; MCKEON, S.; LEVIN, D.; QUIÑONES, M.; POVOA, M.; LINTON, Y. 2012. DNA barcoding reveals both known and novel taxa in the Albitarsis Group (Anopheles: Nyssorhynchus) of Neotropical malaria vectors. Parasites and Vectors 5: 44. [ Links ]
RUTLEDGE, C. R.; DAY, J. F.; LORD, C. C.; STARK, L. M.; TABACHNICK, W. J. 2003. West Nile virus infection rates in Culex nigripalpus (Diptera: Culicidae) do not reflect transmission rates in Florida. Journal of Medical Entomology 40: 253-258. [ Links ]
SAITOU, N.; NEI, M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution 4: 406-425. [ Links ]
SCHLICK-STEINER, B. C.; STEINER, F. M.; SEIFERT, B.; STAUFFER, C.; CHRISTIAN, E.; CROZIER, R. H. 2010. Integrative taxonomy: a multisource approach to exploring biodiversity. Annual Review of Entomology 55: 421-438. [ Links ]
SHIN, D.; CIVANA, A.; ACEVEDO, C.; SMARTT, C. T. 2014. Transcriptomics of differential vector competence: West Nile virus infection in two populations of Culex pipiens quinquefasciatus linked to ovary development. BMC Genomics 15: 513. [ Links ]
STRICKMAN, D.; PRATT, J. 1989. Redescription of Cx. corniger Theobald and Elevation of Culex (Culex) lactator Dyar and Knab from synonymy based on specimens from Central Ameri-ca (Diptera: Culicidae). Proceedings of the Entomological Society of Washington 91 (4): 551-574. [ Links ]
SUAREZ, M. 2001. Aedes albopictus (Skuse) (Diptera: Culicidae) en Buenaventura, Colombia. Informe Quincenal Epidemiológico Nacional 6: 221-224. [ Links ]
TAIRA, K.; TOMA, T.; TAMASHIRO, M.; MIYAGI, I. 2012. DNA barcoding for identification of mosquitoes (Diptera: Culicidae) from the Ryukyu Archipelago, Japan. Medical Entomology and Zoology 63 (4): 289-306. [ Links ]
TAJIMA, F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics 123: 585–595. [ Links ]
TAMURA, K.; STECHER, G.; PETERSON, D.; FILIPSKI, A.; KUMAR, S. 2013. MEGA6: Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution 30: 2725-2729. [ Links ]
TAUTZ, D.; ARCTANDER, P.; MINELLI, A.; THOMAS, R. H.; VOGLER, A. 2003. A plea for DNA taxonomy. Trends in Ecology and Evolution 18 (2): 70-74. [ Links ]
TINKER, M.; OLANO, V. A. 1993. Ecología del Aedes aegypti en un pueblo de Colombia, Suramérica. Biomédica 13 (1): 5-14. [ Links ]
TURELL, M. J. 2012. Members of the Culex pipiens complex as vectors of viruses. Journal of the American Mosquito Control Association 28 (4): 123-126. [ Links ]
VALENTINI, A.; POMPANOM, F.; TABERLET, P. 2008. DNA Barcode for ecologist. Trends in Ecology and Evolution 24: 110-117. [ Links ]
VÉLEZ, I. D.; QUIÑONES, M. L.; SUAREZ, M.; OLANO, V.; MURCIA, L.; CORREA, E. 1998. Presencia de Aedes albopictus en Leticia, Amazonas, Colombia. Biomédica 18: 192-198. [ Links ]
VERSTEIRT, V.; NAGY, Z. T.; ROELANTS, P.; DENIS, L.; BRE- MAN, F. C.; DAMIENS, D.; DEKONINCK, W.; BACKELJAU, T.; COOSEMANS, M.; VAN BORTEL, W. 2015. Identification of Belgian mosquito species (Diptera: Culicidae) by DNA barcoding. Molecular Ecology Resources 15 (2): 449-457. [ Links ]
WANG, G.; LI, C.; GUO, X.; XING, D.; DONG, Y.; WANG, Z.; ZHANG, Y.; LIU, M.; ZHENG, Z.; ZHANG, H.; ZHU, X.; WU, Z.; ZHAO, T. 2012. Identifying the main mosquito species in China bases on DNA barcoding. PLoS ONE 7 (10): e47051. [ Links ]
YANOVIAK, S. P.; PAREDES, J. E.; LOUNIBOS, L. P.; WEAVER, S. C. 2006. Deforestation alters phytotelm habitat availability and mosquito production in the Peruvian Amazon. Ecological Applications 16: 1854-1864. [ Links ]
Recibido: 6-mar-2015
Aceptado: 21-dic-2015
Citación sugerida:
HOYOS-LÓPEZ, R.; ROMAN PARDO, S.; CASTAÑO, J. C.; GALLEGO-GÓMEZ, J. C. 2015. Código de barras para la tipificación de culícidos inmaduros de Armenia y Circasia (Quindío, Colombia). Revista Colombiana de Entomología 41 (2): 218227. Julio - Diciembre 2015. ISSN 0120-0488.