Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Ciencias Pecuarias
Print version ISSN 0120-0690On-line version ISSN 2256-2958
Rev Colom Cienc Pecua vol.18 no.3 Medellín July/Sept. 2005
ARTÍCULOS ORIGINALES
Estimación de polimorfismos del gen de leptina bovino en poblaciones de las razas criollas Hartón del Valle, Blanco Orejinegro (BON) y en la raza Brahman
María T Guerra1, Biol; Esperanza Trujillo B1, Biol, MSc; Mario Cerón-Muñoz1,2, Zoot., PhD.
1Grupo de investigación en Genética y Mejoramiento Animal, Instituto de Biología.
2 Grupo GRICA, Facultad de Ciencias Agrarias. Universidad de Antioquia, A.A. 1226, Medellín, Colombia
(Recibido: 28 febrero, 2005; aceptado: 23 agosto, 2005)
Resumen
En investigaciones recientes se han identificado algunas regiones entre y adyacentes al gen de leptina, asociadas con diferentes niveles de grasa de la carne en canal, y cuyas frecuencias varían grandemente de una raza a otra, y de una población a otra. En este trabajo, fueron determinados los polimorfismos para la región 5 UTR’ (región que no traduce) y región flanqueante al extremo 3’ del gen leptina, utilizando dos microsatélites ST y WD (G18586 y U50365). Fueron evaluados 261 animales de las razas Hartón del Valle (100), Brahman (121) y BON (40). Se encontraron 7 alelos diferentes para el marcador ST y 15 para WD, considerando la población total analizada. Las poblaciones de las razas criollas evaluadas para los marcadores ST y WD, no se encuentran en equilibrio de Hardy- Weinberg. El mayor polimorfismo para el microsatélites ST se encontró en la raza Brahman, que presentó 6 alelos. Para WD fue más polimorfica Hartón del Valle con 15 alelos diferentes. La menos polimórfica fue la raza BON, con 4 alelos para ST y 6 para WD.
Palabras clave: microsatélite, gen obeso, ST, WD.
Introducción
Leptina es el producto del gen obeso ob, también llamado leptina (23), un péptido de 167 aminoácidos, altamente conservado. Presenta entre 84 y 97 % de homología en ratones, ratas, humanos y bovinos (5,23), es secretada por los adipositos a la sangre y luego transportada al cerebro (hipotálamo), donde actúa para estimular factores que afectan el consumo de alimento, la composición del cuerpo, el gasto energético, el aumento de la actividad física y la regulación del peso corporal (6). La leptina inhibe su propia expresión por retroalimentación (8) y entre mayor sea el número de adipositos, mayor es la cantidad de RNAm de leptina presente (1,13).
También tiene un efecto directo sobre otros tejidos como músculo esquelético (9), donde desempeña un importante papel en la síntesis del glicógeno y transporte de glucosa (11), pequeñas cantidades de leptina también son secretadas por células en el epitelio del estómago y de la placenta (9,11). El gen de leptina bovino fue clonado por Ji et al (10) y mapeado en el cromosoma 4 región q32 (15, 17). Presenta 3 exones y 2 intrones, con las regiones codificantes ubicadas en los exones 2 y 3 (3). Con respecto al gen leptina varios grupos de investigación, han determinado en razas taurinas la asociación de concentraciones de leptina en sangre con variaciones en el apetito y composición de la carne en canal (6, 20) y de regiones variables en el gen con promedio de grasa, espesor de grasa de cobertura, marmoreo y porcentaje de grasa en costilla (2, 7, 12).
En ratón, el gen mutado ob en forma homocigótica (ob/ob) produce una leptina biológicamente inactiva determinando hiperfagia, reducción del gasto de
energía y obesidad genética, que resulta en un adulto estéril con el 50% del cuerpo como grasa (18). El defecto específico del gen ob en ratón fue reportado
por Zhang et al (23). El descubrimiento del gen de la leptina en ratón y otras especies y sus efectos sobre la grasa del cuerpo, lo convierten en un candidato ideal para evaluación del metabolismo.
El presente trabajo pretende estimar las frecuencias de los polimorfismos de dos microsatélites ligados al gen leptina para su posterior utilización como marcadores de la calidad de carne.
Materiales y métodos
Muestra poblacional
Fueron evaluados 100 animales (60 hembras, 40 machos) de ganado criollo Hartón del Valle (Hv), hato ubicado en Cerrito —Valle del Cauca; 40 de BON (7 machos y 33 hembras) provenientes de la finca Hato Viejo — Risaralda y 121 de Brahman ( 100 machos y 21 hembras), de Puerto Boyacá. Los animales fueron escogidos teniendo en cuenta los registros de las fincas, con la idea de muestrear los menos emparentados posibles.
Extracción de ADN
De cada animal fueron colectados aproximadamente 7ml de sangre de la vena mamaria o arteria coccígea media y almacenados a 4°C hasta su procesamiento. El ADN fue extraído por la metodología de “Salting out” (14) y posterior precipitación con 2- propanol. La concentración de DNA se midió por espectrofotometría a 260 nm.
Genotipificación
Las reacciones de PCR se llevaron a cabo en un volumen total de 25mL, que contenían: 0,7 mg/mL de DNA; buffer de reacción 1X (10Mm Tris-HCL pH 9,0; 50mM KCl; 0,1 % Triton® X-100); 3mM de MgCl2; 0,08mM de cada dNTP; 5 pmol de cada primer y 1 unidad de Taq DNA Polimerasa (Invitrogen). La amplificación se efectuó en un termociclador T-Personal 48 (Biometra® GMBH, D- 37079 Goettingen, Germany). Para la amplificación del microsatélite WD ubicado la región 5’UTR del gen de la leptina bovina (cromosoma 4), fueron utilizados los primers: WD-1 5’- TTCTAATGGTCGAATATGTTCTGG-3’ y WD-2 5’-TAAAGACCGGCTACGACTAGAC-3’ (22) y para el microsatélite ST ubicado en la región que flanquea el extremo 3’ del gen de leptina fueron
utilizados los primers: ST-1 5’- CATCGACGACAGGAACTCC-3’ y ST-2 5’-GGAAT17CGTACAAGGGACC-3’ (17), fabricados por IDT (Coralville, IA 52241, USA). El perfil térmico para cada reacción consistió en un ciclo a 94°C por 2 min, 60°C por 30 seg, 72°C por 30 seg, seguido por 29 ciclos a 94°C por 20 seg, 60°C por 20 seg y 72°C por 30 seg y extensión final a 72°C por 7 min. Los productos de la PCR fueron visualizados en geles de agarosa al 2% conteniendo Bromuro de Etidio (0.7 µg/mL). Para la determinación de los genotipos se utilizaron geles de poliacrilamida al 6%.
Métodos estadísticos
Se estimó el equilibrio de Hardy-Weinberg, las frecuencias genotípicas y las frecuencias alélicas con el programa GENEPOP versión 3.3 (16).
Resultados
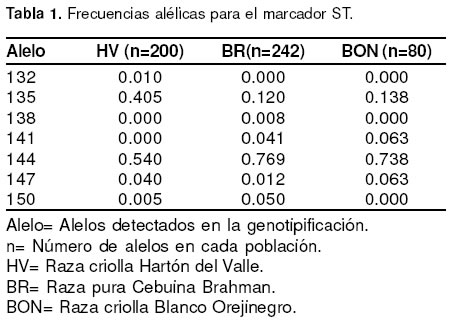
La genotipificación del marcador ST (17), en las poblaciones seleccionadas de las razas criollas Hartón del Valle, BON y de la raza Brahman, dio como resultado la identificación de 7 alelos diferentes para este microsatélite (véase Tabla 1), tres diferentes (132, 141 y 150) de los reportados previamente en investigaciones realizadas por Fitzimmons et al (4) y Tessanne et al (19) quienes encontraron los siguientes alelos 135, 138, 140, 144, 147 y 149. En Hartón del Valle se detectaron 5 alelos para ST, siendo los más frecuentes 135 y 144. En Brahman, 6 alelos en total, siendo el más frecuente el 144. La raza criolla BON presentó 4 alelos diferentes, con la frecuencia más alta para el alelo 144 (véase Tabla 1).
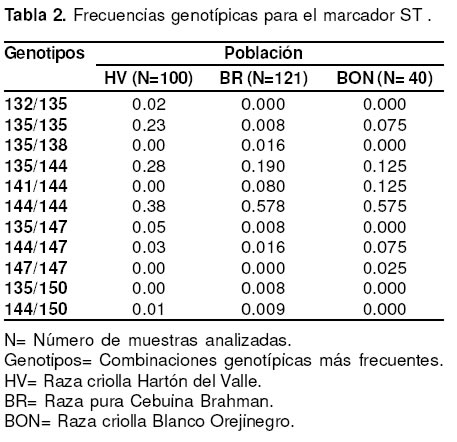
Los alelos 135 y 144 para el marcador ST, representaron para cada una de las tres poblaciones el más alto porcentaje así: Hartón del Valle 94.5%, Brahman 88.9% y BON 87.6%. El alelo 135 mostró una alta frecuencia (0.405) en Hartón del Valle, pero baja en las razas Brahman y BON con valores de 0.12 y 0.14 respectivamente. Los alelos 132 y 138, fueron determinados exclusivamente en animales Hartón del Valle y Brahman respectivamente, con frecuencias de 0.01 (correspondiente a dos animales heterocigóticos) y 0.008 (dos animales heterocigóticos). Los alelos 135, 144 y 147 fueron comunes a las tres poblaciones, (Brahman, Hartón del Valle y BON) (véase Tabla 2).
Se detectaron 7 combinaciones genotípicas para el marcador ST en Hartón del Valle, 6 en BON y 9 en Brahman; el homocigótico 144/144 presentó la más alta frecuencia, en cada una de las tres poblaciones así: Hartón del Valle (0.38), BON (0.57) y Brahman (0.58). Las combinaciones genotípicas que se
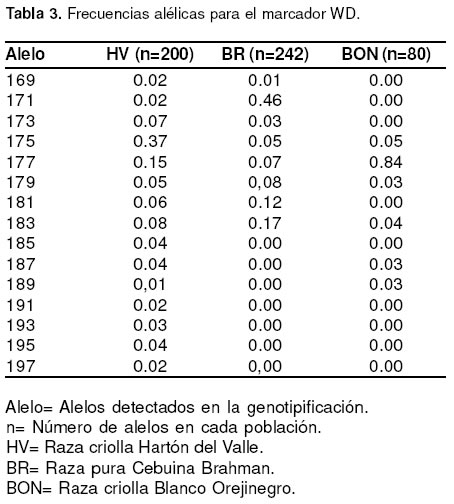
presentaron en menor proporción fueron 147/147 y 135/ 150, en BON y Brahman respectivamente. (véase Tabla 2) El otro microsatélite genotipificado fue WD (22). Wilkins y Davey (22) encontraron 18 alelos diferentes para este marcador en investigaciones con Angus. En la evaluación en el presente estudio para este mismo marcador, se determinaron 15, 8 y 6 alelos para las razas Hartón del Valle, Brahman y BON respectivamente (véase Tabla 3). Los más frecuentes para cada una de las razas analizadas fueron: 171 en Brahman, 175 en Hartón del Valle y 177 en BON, con frecuencias de 0.46, 0.37 y 0.84 respectivamente (véase Tabla 3). En otros resultados en la raza Angus, el alelo más frecuente fue el 177 con 0.51 (19).
Fueron comunes a las tres poblaciones analizadas los alelos 175, 177, 179 y 183 de WD. Los alelos 169, 171, 173 y 181 únicamente se detectaron en las
poblaciones Hartón del Valle y Brahman pero no en BON. 187 y 189 sólo estuvieron presentes en Hartón del Valle y BON y los alelos 185, 191, 193, 195 y 197 solo se encontraron en Hartón del Valle. Ésta última fue la única raza en la que se determinaron la totalidad de los diferentes alelos encontrados en este estudio (véase Tabla 3).
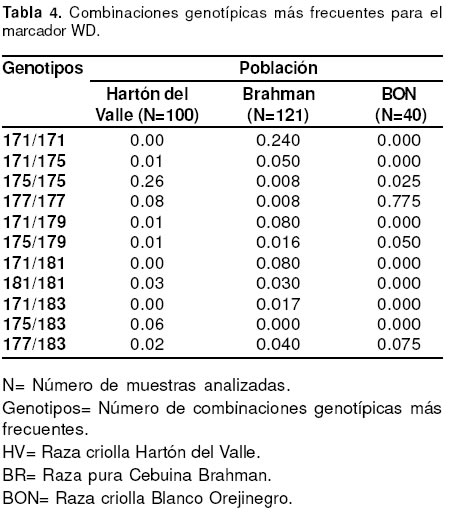
En la genotipificación del marcador WD, se detectaron 6 genotipos diferentes en BON, 40 en Hartón del Valle y 24 en Brahman, siendo la raza criolla
Hartón del Valle, la que más combinaciones genotípicas presentó y cuyo genotipo más frecuente fue el 175/ 175 con un valor de 0.26. En Brahman fue el
homocigótico 171/171, con una frecuencia de 0.24 y en BON el genotipo 177/177 con una frecuencia de 0.77 (véase Tabla 4).
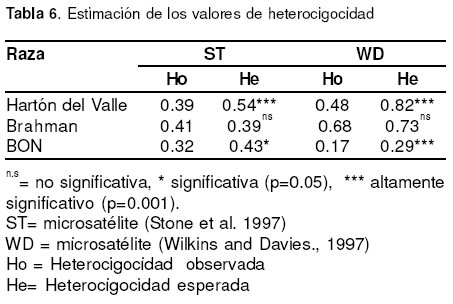
En la tabla 6 se muestran los valores estimados de la heterocigosis observada y esperada para cada uno de los microsatélites (ST y WD) y en cada una de las razas.
Discusión
El gen ob ha sido propuesto como un gen candidato, cuyo producto se cree está involucrado en la regulación y disposición de la grasa en ganado de carne. Varios grupos de investigación (6,12), han evaluado la asociación con variaciones en el apetito, características de la carne en canal y contenido de grasa intramuscular en ganado, de microsatélites ubicados en regiones cercanas y no codificantes al gen leptina tales como ST (17) y WD (22), así como de secuencias internas al gen como el marcador SNP (2).
El microsatélite ST cercano al gen ob fue caracterizado previamente en las razas: Angus, Charolais, Hereford y Simmental por Fitzsimmons et al (4), quienes encontraron cuatro alelos de 138, 140, 147 y 149 pares de bases y una asociación significativa del alelo 138 con un alto espesor de grasa subcutánea en Angus y Hereford y del alelo 147 con un efecto opuesto. El alelo 138 fue reportado con frecuencias de 0.57 en Angus y 0.59 en Hereford (7) y el 147 en
razas como Charolais y Simmental con frecuencias de 0.54 y 0.58 respectivamente (7).
En este estudio, el alelo 138 fue detectado únicamente en la raza Brahman de las tres analizadas y con una muy baja frecuencia (0.008) (un animal), y el 147 presentó frecuencias bajas en las 3 razas analizadas, principalmente en Brahman (véase Tabla 1).
El 132 es una alelo encontrado únicamente en la raza Hartón del Valle, con baja frecuencia (0.10) (véase Tabla 1), no ha sido reportado en investigaciones previas en otras razas tales como: Angus, Charolais, Hereford y Simmental (4,19). Su detección en Hartón del Valle se debe quizás a la presencia de algo de mezcla con otras razas de origen indicus.
En cuanto al alelo 144 del marcador ST, fue el más frecuente en las tres razas analizadas, en concordancia con los resultados de Tessanne et al (19) quienes reportaron para este mismo alelo frecuencias muy altas en la raza Angus (0.51).
El mayor polimorfismo para el microsatélite ST lo presentó la raza Brahman, comparada con lo encontrado en Hartón del Valle y BON. La variabilidad encontrada en Brahman, posiblemente sea el resultado de un mayor pie de cría ancestral y reducidos niveles de endogamia en los hatos. Además, la raza Brahman es el resultado de una mezcla genética entre diferentes razas que contribuyeron en su formación. Entre las tres razas analizadas la menos polimórfica para este microsatélite fue la raza BON ya que presentó únicamente cuatro alelos de los siete determinados en las tres poblaciones.
Para el microsatélite WD, Tessanne et al (19), determinaron en Angus seis alelos diferentes (177, 184, 186, 190, 192 y 209). En esta misma investigación,
este microsatélite fue asociado con calidad de carne, principalmente con relación a mayor área del ojo del lomo, especialmente los genotipos que contienen el alelo 209 (19). Este alelo no fue encontrado en ninguna de las razas evaluadas en este estudio.
Los diferentes alelos encontrados para WD (véase Tabla 3), teniendo en cuenta la población total genotipificada, indican un alto polimorfismo para éste
microsatélite. El alto polimorfismo encontrado, quizás es debido a que es un marcador ubicado en una zona de alta variabilidad en el gen de leptina (región 5’UTR). La población Hartón del Valle fue la raza criolla más polimórfica de las tres y la raza BON la menos polimórfica. El polimorfismo en Hartón del Valle, es debido al número de alelos determinados para WD en la población (aunque con muy bajas frecuencias) (véase Tabla 3) y que denotan variabilidad, se debe
posiblemente, a que en este núcleo empieza a darse la mezcla con parentales de otros orígenes.
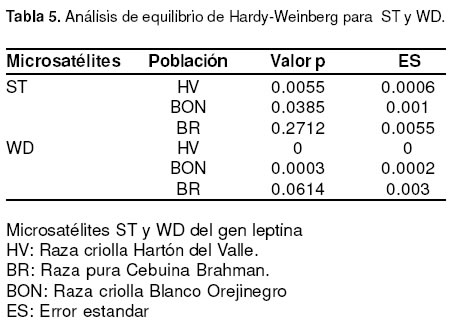
A partir del análisis de equilibrio de Hardy-Weinberg se determinó que, las poblaciones Hartón del Valle y BON no se encontraron en equilibrio para los microsatélites ST y WD (p = 0.05), pero sí la población Brahman (véase Tabla 5).
La pérdida de equilibrio H-W, que se presentó para WD y ST en las razas criollas es lo esperado, ya que es frecuente la utilización de pocos parentales machos en los diferentes hatos, lo que conduce a un alto nivel de endogamia, acorde con la endogamia detectada en los registros históricos que se llevan en estas explotaciones.
Los resultados de la estimación de las diferencias entre la heterocigocidad observada y esperada para ambos microsatélites, mostraron que ambas razas
criollas (Hartón del Valle y BON) tienen un déficit de heterocigóticos (p = 0.05), producido posiblemente porel alto grado de consanguinidad, debido al bajo número de núcleos que conforman cada una de éstas razas, cerrados y aislados, lo que no permite un verdadero intercambio. En Brahman no se presentó un exceso de heterocigóticos (p>0.05) (véase Tabla 6).
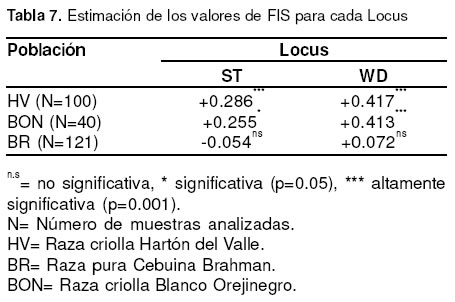
En la estimación de los valores de FIS, se determinó que los resultados para cada locus y para cada población son significativos para el déficit de heterocigóticos en Hartón del Valle y BON. La población Brahman no presentó déficit de heterocigóticos (véanse Tablas 7 y 8).
Se encontró segregación independiente entre los dos loci con un valor p altamente significativo (p<0.05), para cada una de las poblaciones, Hartón del Valle,
Brahman y BON. Este resultado concuerda con el hecho de que ambos marcadores se encuentran alejados entre sí en el mapa genómico, no hay desequilibrio de ligamiento y como consecuencia, no pueden establecerse haplotipos predominantes en la población.
La genotipificación, la determinación de los valores de las frecuencias alélicas y genotípicas encontradas y el análisis de equilibrio de H-W en las razas criollas
y Brahman, con respecto a las regiones 5’UTR y la que flanquea el extremo 3’ del gen leptina, permitió establecer diferencias marcadas entre las razas para
cada uno de los marcadores evaluados. Además, posibilitar la apertura de nuevas investigaciones encaminadas a buscar su asociación con características
de calidad de carne.
Agradecimientos
A la Vicerrectoría de extensión (Universidad de Antioquia. Proyecto № 158.04) y a la Cooperativa Lechera COLANTA.
Summary
The leptin gene polymorphism, in the populations of Hartón del Valle, Blanco-Orejinegro ( BON) and Brahman cattle.
Recent investigations have identified some regions between and adjacent to the leptin gene, associated with different carcass fat levels in bovines in wich requencies vary greatly from one race to another, and from one population to another. This investigation determined the polymorphisms for the region 5 UTR’ and flanking region to the end 3’ of the leptin gene using two markers ST and WD (G18586 and U50365). 261 animals were evaluated of Hartón del Valle (100), Brahman (121) and BON (40) cattle. Were evaluated the result of this analysis where, 7 different alelles for microsatellite ST and 15 for WD, in the total population. The creole populations evaluated for marker ST are not in Hardy-Weinberg (HW) equilibrium. The marker WD only appeared H-W equilibrium for the population Brahman. The Brahman cattle was the most polymorphic cattle for microsatellites ST, where 6 for ST alleles were found. For WD the most polymorphic cattle was Hartón del Valle and less polymorphic cattle was the BON, with 6 for ST and 6 for WD alleles.
Key words: microsatellite, obese gene, ST, WD.
Referencias
1. Auwerx J. and Staels, B. 1998. Leptin. Lancet. 351: 737-742. [ Links ]
2. Buchanan FC, Fitzsimmons CJ, Van Kessel, AG, Thue TD, Windkelman-Sim DC, Schmutz SM. 2002. Association of a missense mutation in the bovine leptin gene with carcass fat content and leptin mRNA levels. Genet. Sci. Evol. 34: 105-116. [ Links ]
3. Coleman DL. 1978. Obese and diabetes: two mutant genes causing diabetes-obesity syndromes in mice. Diabetologia 14: 141-148. [ Links ]
4. Fitzsimmons CJ, Schmutz SM, Bergen RD, and McKinnon JJ. 1998. A potential association between the BM1500 microsatellite and fat deposition in beef cattle. Mamm. Genome 9: 432-434. [ Links ]
5. Funahashi T, Simomura I, Hiraoka H, Arai T, Takahashi M, Nakamura T, Nozaki S Yamashita S, Takemura K, Matsuzawa Y, 1995. Enhanced expression of rat obese (OB) gene in adipose tissues of ventromedial hypothalamus (VMH)- lesioned rats. Biochem Biophys Res Commun. 211: 469-475. [ Links ]
6. Geary TW, McFadin EL, MacNeil MD, Grings EE, Short RE, Funston RN, and Keisler DH. 2003. Leptin as a predictor of carcass composition in beef cattle. J.Anim. Sci. 81: 1-8. [ Links ]
7. Hale CS, Herring WO, Johnson GS, Shibuya H, Lubahn, D. B. and Keisler, DH. 1998. Evaluation of the leptin gene as a possible marker of carcass traits in Angus cattle. UMC Animal Sciences Departmental Report, 25-27. [ Links ]
8. Houseknecht KL, Baile CA, Matteri RL, Spurlock ME. 1998. The Biology of Leptin: A Review. J. Anim. Sci. 76: 1405- 1420. [ Links ]
9. Hossner KL. 1998. Cellular, molecular and physiological aspects of leptin: Potential application in animal production. Can. J. Anim. Sci. 78:463-472. [ Links ]
10. Ji S, Willis GM, Scott RR, Spurlock ME, 1998. Partial cloning and expression of the bovine leptin gene. Anim. Biotechnol. 9: 1-14. [ Links ]
11. Margetic S, Gazzola C, Pegg GG, and Hill R A. 2002. Leptin: a review of its peripheral actions and interactions. Inter. J. Obes. 26: 1407-1433. [ Links ]
12. Marquess L. 2001. The leptin gene as a candidate for fat deposition assessment. Mimeo. [ Links ]
13. Masuzaki H, Ogawa Y, Isse N, Satoh N, Okasaki TM, Shigemoto M, Mori K, Tamura N, Hosada K, Yoshimasa Y, Jingami H, Kawada T, and Nakao K. 1995. Human obese expression and regional differences in the adipose tissue. Diabetes. 44: 855-858 [ Links ]
14. Miller SA, Dykes DD, Poletsky HF. 1988 A simple salting out procedure for extracting DNA from human nucleated cells. Nucl Acids Res 16: 1215. [ Links ]
15. Pomp D, Zou T, Clutter AC., and Barendse, W. 1997. Rapid communication: Mapping of leptin to bovine chromosome 4 by linkage analysis of a PCR- based polymorphism. J. Anim. Sci. Vol. 75, p. 1427. [ Links ]
16. Raymond M, Rousset F. 1995. GENEPOP (Versión 3.3): population genetics software for exact test and ecumenicism. J Heredity 86:248-249. [ Links ]
17. Stone RT, Kappes S. M. and Beattie C. (1997) Two polymorphic microsatellites within an 18 kb genomic clone containing the bovine ob gene. Animal Genetics. 27 (Suppl. 2), 64. [ Links ]
18. Tartaglia LA, Dembski M, Weng X, Deng N, Culpepper J, Devos R, Richards JG, Campfield LA, Clark FT, Deeds J, Muir C, Sanker S, Moriarty A, Moore KJ, Smutko JS, Mays GG, Woolf EA, Monroe CA, and Tepper RI. 1995. Identification and expression cloning of a leptin receptor, OB-R. Cell 83: 1263-1271. [ Links ]
19. Tessanne K, Hines HC, and Davis ME, 1999. Relationships of Polymorphisms in the Bovine Leptin Gene with Differences in Beef Carcass Traits. esearch and Reviews: Beef and Sheep. Special Circular.170. [ Links ]
20. Wegner J, Huff P, Xie CP, Schneider F, Teuscher F, Mire PS, Mir Z, Kazala, EC, Weselake RJ and Ender K. 2001. Relationship of plasma leptin concentration to intramuscular fat content in beef from crossbred Wagyu cattle. Can. J. Anim. Sci. 81:451-457. [ Links ]
21. Wheeler TL, Cundiff LV and Koch RM. 1994. Effect of marbling degree on beef palatability in Bos taurus and Bos indicus cattle. J. Anim. Sci. 72:3145-3151. [ Links ]
22. Wilkins RJ and Davey H. 1997. A polymorphic microsatellite in the bovine leptin gene. Animal Genetics 38: 370-383. [ Links ]
23. Zhang Y, Proenca R, Maffel M, Barone M, Leopoldo L and Friedman JM. 1994. Positional Cloning of the mouse obese gene and its human homologue. Nature (Lond.) 372: 425- 432. [ Links ]