Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Ciencias Pecuarias
Print version ISSN 0120-0690On-line version ISSN 2256-2958
Rev Colom Cienc Pecua vol.19 no.3 Medellín July/Sept. 2006
Polimorfismos de BoLA-DRB3 en el bovino sintético colombiano Lucerna y asociación con conteo de células somáticas y mastitis. Santiago Gómez-Castro1 , Biol; Esperanza Trujillo-Bravo1 , Biol, Msc; Carlos-Vicente Duran2 , Ing Agron, Msc
1Grupo Genética y Mejoramiento Animal, Instituto de Biología, Universidad de Antioquia, A.A. 1226, Medellín, Colombia. 2 Falcultad de Ciencias Agropecuarias, Universidad Nacional de Colombia sede Palmira. Valle del Cauca.
etbravo@epm.net.co
(Recibido: 10 octubre, 2005; aceptado: 28 julio, 2006)
Resumen
En bovinos se han determinado asociaciones de BoLA-DRB3 con enfermedades infecciosas y con el conteo de células somáticas. En este estudio fue analizada la raza sintética colombiana Lucerna, para el microsatélite DRB3 intrón 2. Fueron identificados 24 alelos por amplificación con PCR y geles de poliacrilamida, encontrando frecuencias alélicas desde 0.02 hasta 0.26. El alelo mas frecuente en la población analizada fue el 161, y el menos frecuente 159. Ambas subpoblaciones afectadas y no afectadas por mastitis, presentaron frecuencias similares para la mayoría de los alelos. La población no se encuentra en equilibrio H.W (Hardy- Weinberg). (p <0.05), y presentó déficit de heterocigóticos. El valor medio de recuento de células somáticas transformado (log2 (CCS / 100000 ) + 3), para la población total fue de 4.8 (+/- 2.89) y para cada subpoblación de afectadas y no afectadas fue de 5.76 y 3.86, respectivamente. Se estudiaron las asociaciones potenciales entre los alelos BoLA-DRB3, el recuento de células somáticas y la mastitis clínica, y se encontrando una asociación significativa (p<0.05) entre el alelo 191 con un alto conteo de células somáticas. No se encontró asociación significativa entre el alelo 191 y la enfermedad, pero si hubo significancia estadística (p<0.05) para la asociación de la mastitis con las variables hato y número de partos.
Palabras clave: BoLA, células somáticas, gen DRB3, mastitis.
Introducción
Las enfermedades infecciosas son parte de los problemas que afectan frecuentemente la crianza de animales, representando un factor limitante en la producción en bovinos. Esto se refleja en un aumento en los niveles de mortalidad o en bajos índices de crecimiento y fertilidad, así como en la disminución en la cantidad y calidad de carne y leche (24).
En los mecanismos involucrados en la resistencia o susceptibilidad genética a infecciones, se han identificado varios niveles de respuesta a agentes infecciosos, en los que actúan diversos genes y sus variantes alelicas (1, 2). Actualmente se consideran los genes del complejo mayor de histocompatibilidad (MHC), importantes en la modulación de diferentes aspectos de la respuesta inmune (27).
El complejo mayor de histocompatibilidad (MHC) en bovinos se conoce como BoLA "Bovine Lymphocyte Antigen" (Antígeno Linfocitario Bovino), el cual ha sido mapeado en el brazo corto del cromosoma 23 y esta conformado por las clases I, II y III, de acuerdo con diferencias en la distribución celular y la función (5).
Los genes clase II están distribuidos en dos diferentes regiones IIa y IIb, codifican glicoproteinas que se unen a péptidos exógenos (8) y son normalmente expresadas por células del sistema inmune como macrófagos, células dendríticas y linfocitos T y B. En la región IIa están ubicados los genes DRA, DRB, DQA y DQB, y la región IIb, incluye a los genes DOB, DYB, DYA y DIB (6, 11). El locus DRB presenta tres loci: DRBP1, DRB2 y DRB3 (5); el exón 2 del DRB3 (DRB3.2) codifica un sitio de unión al antígeno y recientemente se han investigado sus características, así como el efecto biológico de sus variaciones (4, 31). Se ha encontrado en investigaciones en ganado Shorthorn (37), Jersey (18) y Ayrshire (38), que este sitio de unión presenta una alta tasa de polimorfismo.
Mediante técnicas moleculares de reacción en cadena de la polimerasa y polimorfismos de longitud (PCR-RFLP), desarrollado por Van Eikj et al (39), se han detectado alrededor de 30 alelos en el segundo exón del gen DRB3.2 bovino y se han identificado por técnicas de secuenciamiento más de 63 alelos diferentes (33). Takeshima et al (36) utilizando reacción en cadena de la polimerasa y tipificación de secuencias de bases (PCR-SBT) identificaron 90 alelos diferentes.
El intrón 2 del gen DRB3 presenta un microsatélite, compuesto de 10 dinucleótidos (TG)n, una larga repetición de dinucleótidos (GA)n interrumpidos y un corto número de tetranucleótidos (CAGA)n. La evolución de este microsatélite involucra mutaciones de longitud en las repeticiones de dinucleótidos (16).
Este microsatélite se encuentra estrechamente ligado a las secuencias del exón 2 de DRB3, por lo cual existe una equivalencia entre los alelos del microsatélite y cada uno de los alelos determinados por PCR-RFLP en el exón 2 (11), permitiendo realizar análisis de DRB3 utilizando cualquiera de los dos grupos de alelos.
Los alelos BoLA-DRB3, afectan potencialmente muchas características relacionadas con la inmunidad, se le han determinado asociaciones con enfermedades infecciosas en ganado vacuno (29), y en estudios en Holstein se ha encontrado relación de los alelos DRB3.2 con resistencia a mastitis por staphylococcus aureus.
Recientemente los alelos DRB3.2*8, *16, *22 y *28, fueron asociados con un elevado conteo de células somáticas (CCS) (13,14) y en reportes realizados por Sharif et al (30), en la raza Holstein, DRB3.2*16 y DRB3.2*23 se asociaron con un bajo CCS y con mastitis coliforme severa, respectivamente.
El propósito de este trabajo fue determinar los polimorfismosdelmicrosatélitedelintrón2deDRB3, en la raza sintética colombiana Lucerna y establecer su asociación con el conteo de células somáticas y susceptibilidad o resistencia a mastitis.
Materiales y métodos
Población
Fueron muestreadas 308 hembras de la raza sintética Lucerna provenientes de 2 hatos, ubicados geográficamente en el departamento del Valle del Cauca-Colombia. Esta raza sintética es derivada de cruzar las razas Holstein (40%), Shorthorn (30%) y la criolla Hartón del Valle (30%), para formar un grupo racial adaptado a condiciones tropicales (35).
En los hatos seleccionados la colección de muestras se realizó en forma pareada. Ambos hatos presentan características comunes tales como: temperatura promedio 24ºC, bosque seco tropical, topografía plana, silvopastoreo, pasto estrella y ordeño mecánico; las muestras de ambos hatos fueron reunidas para esta evaluación, teniendo en cuenta que ambos grupos fueron derivados de un mismo hato ancestral inicial. El número total de animales seleccionados fue de 308 y fueron utilizados para determinar los polimorfismos de DRB3 y sus frecuencias.
Se colectaron los registros históricos de mastitis clínica en las bases de datos para 212 animales del total muestreados, correspondientes a las hembras que se encontraban en producción, en el momento del muestreo, considerando la realización de análisis posteriores. El hato uno presentó registros históricos para 74 hembras afectadas y 23 no afectadas.
En el hato dos se encontraron registros para 59 afectadas y 56 no afectadas. Los registros históricos permitieron agrupar la población en dos subpoblaciones así: 133 animales con diagnóstico previo recurrente de mastitis clínica (afectado = A) y 79 con diagnóstico negativo de mastitis clínica (no afectado = NA). Los animales que habían presentado cuadros de leche anormal, fiebre y anorexia conformaron la subpoblación A y los que no los habían presentado subpoblación NA.
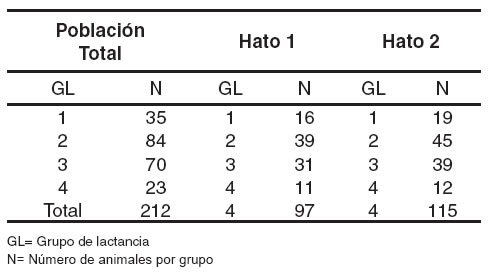
Para realizar el conteo de células somáticas y los análisis estadísticos de asociación, se tuvieron en cuenta los grupos NA y A. En estos dos grupos se determinaron animales con tres o más partos (véase Tabla 1) y se establecieron cuatro grupos de lactancia (< 90, 90-180, 181 -270 y > 90 días), en el momento de la selección (véase Tabla 2).
Tabla 1. Grupo de partos en ganado Lucerna.
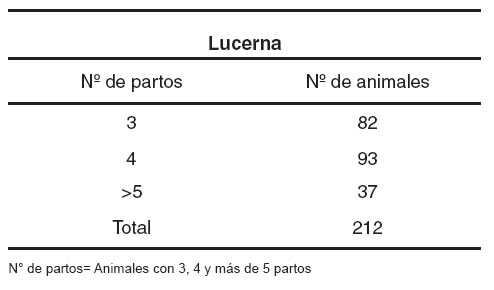
Tabla 2. Grupo de lactancia en ganado Lucerna, se establecieron 4 grupos de lactancia : primero < 90 días, segundo de 90-180, tercero de 181 -270 y cuarto > 90 días.
Conteo de células somáticas (CCS)
Se tomaron muestras de 15ml de leche (3.5 ml por cuarto) del ordeño individual, se fijaron con dicromato de potasio y se conservaron a 4°C hasta realizar el recuento (CCS). El proceso de cuantificación se llevó a cabo en un contador automático de células somáticas, FOSSOMATIC 215 (Foss Electric, 3400 Hillerod, DeNAark). Los valores obtenidos fueron trasformados posteriormente a SCS “somatic cell score” (calificación de células somáticas) con el log2 (CCS / 100000 ) + 3 (3), con el fin de facilitar el análisis.
Extracción de ADN
Aproximadamente 7 ml de sangre fueron colectados de la vena mamaria o arteria coccígea media de cada animal y la sangre completa fue almacenada a 4°C hasta su procesamiento.
El ADN fue extraído por la metodología de “Salting Out” (23) y precipitación con 2-propanol. La concentración del ADN fue medida por espectrofotometría a 260nm.
Genotipificación
El microsatélite del intrón dos del gen DRB3 se genotipificó, utilizando los oligonucleótidos específicos:5`-GCGAATTCCCGAGTGAGTGAA GTATCT-3` y 5’-GAGAGTTTCACTGTGCAG-3`, (IDT, Coraville, Iowa 52241, USA), que amplifican una secuencia que varía entre 155pb y 215pb (16).
Se estandarizaron las condiciones de PCR, en un termociclador T-personal 48 (Biometra® GmbH, D-37079 Goettingen, Germany), usando 20μl de reacción que contenía: 100 ng de DNA, 1μl de buffer 10x (750mM Tris-HCl pH:8.8, 200mM (NH4)2SO4, y 0.1% Tween 20), 1.25mM de MgCl2, 0.15U de Taq DNA polimerasa (recombinante) (MBI Fermentas, Hanover, MD 21076, USA) 2mM de dNTPs, 5pmol de cada oligonucleótido. Para la determinación de los genotipos se utilizaron geles de poliacrilamida al 6%.
Métodos estadísticos
Las frecuencias génicas fueron estimadas teniendo en cuenta las 308 hembras Lucerna, con el paquete estadístico GDA y GENEPOP versión 3.3 (34, 40). Para detectar desviaciones significativas de las proporciones alélicas observadas y esperadas, se llevó a cabo la prueba de Equilibrio de Hardy-Weinberg (H-W) (19).
Se utilizó el test de diferenciación génica para establecer las diferencias de las frecuencias obtenidas entre los grupos seleccionados en cada uno de los hatos.
El análisis de asociación de los alelos con el recuento de células somáticas, se realizó con el procedimiento “General Linear model” GLM de SAS (28), similar a estudios previos (30) con el modelo: Yijklm = μ + hatoi+ partosj+Glactk +Σlbl BoLAijkl+ eijklm donde Yijklm es la variable dependiente SCS, μ es la media de la población, hatoi = sitos del muestreo j = 1-2, partosk = los animales fueron agrupados en 3,4,5 y 6 partos k = 1 - 4, Glactl = efecto del grupo de lactancia,l = 1,2,3 y 4, bl = coeficiente de regresión en el número de alelos, BoLAijkl = efecto fijo del número de copias del los alelos BoLA l (l = 1 – 9 ) (se utilizaron los alelos con las más altas frecuencias, los alelos de menor frecuencia fueron agrupados en una sola categoría), eijlklm= error.
Para determinar la asociación de cada uno de los alelos con susceptibilidad o resistencia a mastitis se realizó el procedimiento CATMOD de SAS (28), con el modelo: Yijlk =μ +hatoi+partosj+Glactk+ΣlblBoLAijkl+ eijkl donde Yijkl es la variable dependiente (mastitis clínica), medida teniendo en cuenta los registros históricos en los hatos, para la presencia o no de mastitis clínica en cada animal muestreado, μ es la media de la población, hatoi = sitos del muestreo i = 1-2, partosj = número de partos en los animales j = 1 -4, Glactk, = efecto del grupo de lactancia, k = 1, 2, 3 y 4, bl = coeficiente de regresión en el número de alelos, BoLAijlm = efecto fijo del número de copias del los alelos BoLA l (l = 1 – 9 ) presentes en los animales, eijlklm = error.
Para confirmar la asociación de los alelos BoLADRB3 con las dos variables consideradas (mastitis clínica y conteo de células somáticas), se calculó el Odd ratio o relación de diferencia (OR) y su intervalo de confianza, según Lemeshow et al (22). Si el OR es mayor que 1, el factor es considerado como de alto riesgo según sea la variable analizada, presencia de la enfermedad o incremento en el recuento de células somáticas. Si OR es menor que 1, el factor puede interpretarse sin efecto sobre mastitis clínica o sobre el recuento de células somáticas.
Resultados
Frecuencias alélicas y equilibrio de Hardy-Weinberg
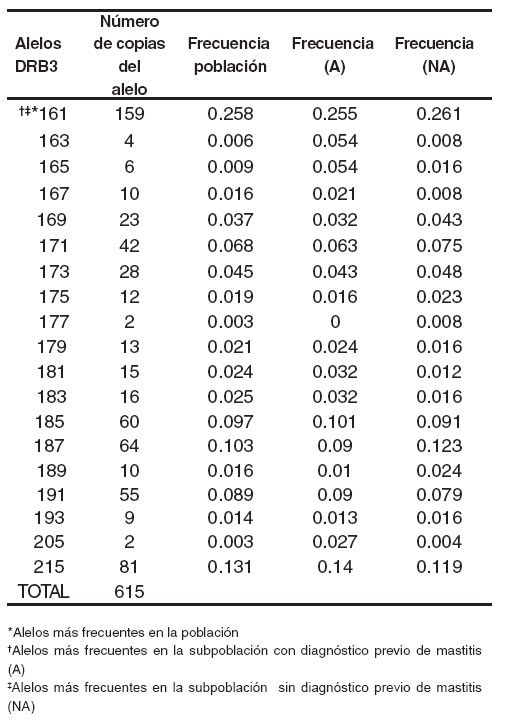
En la población bovina Lucerna, fueron identificados 19 alelos diferentes para BoLA-DRB3 (véase Tabla 3), indicativo de un alto polimorfismo para ésta raza sintética colombiana, comparado con resultados obtenidos para DRB3.2 en Gyr brasilero (10), Holstein Iraní (25), criollo argentino Saavedreno (26), Holstein canadiense (30) y Shorton Japonés (37). Además los tamaños en pares de bases presentados por los alelos encontrados, estuvieron en el rango descrito previamente por Ellegren et al (16).
Las frecuencias alélicas en la población total se ubicaron entre 0.02 hasta 0.26, siendo los alelos 161, 187 y 215 los más frecuentes considerando toda la población. La subpoblación afectada (A) presentó con más frecuencia los alelos 161 y 215 y la subpoblación no afectada (NA) 161 y 187. Los alelos con menor frecuencia en la población total fueron 159, 195, 203 y 207 (véase Tabla 3).
No se encontraron los alelos 159, 177 y 217 en la subpoblación A y 195, 203 y 207 en NA. Ambas subpoblaciones fueron muy similares para la mayoría de los alelos más frecuentes (véase Tabla 3).
Tabla 3. Frecuencias de los alelos BoLA -DRB3 en 308 hembras de la raza Colombiana Lucerna, tipificadas por PCR y geles de poliacrilamida.
La población Lucerna no presentó equilibrio de Hardy-Weinberg y muestró un déficit de heterocigóticos significativo (p < 0.05).
Conteo de células somáticas
El valor medio del conteo de células somáticas transformado SCS (3), para la población total fue de (+/- 2.89 DE), los resultados entre las subpoblaciones A y NA muestran una diferencia significativa (p < 0.01) en los valores de SCS. Para NA la media fue de 3.86 (+/- 2.48 DE) y para A de (+/- 2.88 DE) (véase Tabla 4).
Cuando se analizaron las muestras de acuerdo con su lugar de origen, los hatos uno y dos presentaron diferencias en cuanto al recuento de células somáticas para cada una de las subpoblaciones (A y NA). En el hato uno el promedio de SCS para NA fue de 4.11 (2.40 DE) y en A de 5.12 (2.87 DE), en el hato dos, las NA tuvieron un promedio de 3.61 (2.49 DE) y las A de 6.40 (2.74 DE). El promedio total de SCS en el hato uno fue de 4.61 y en el hato dos de 5.0 (véase Tabla 4).
En el cálculo de las medias de SCS en animales con 0, 1 y 2 copias de cada uno de los alelos más frecuentes, el mayor promedio de SCS encontrado fue para animales con los alelos 171, 191 y 215, (teniendo en cuenta el número de alelos) la media mas alta de SCS fue para el alelo 191 (6.07 +/- 2.75 DE) (véase Tabla 5).
Tabla 4. Registros históricos de mastitis clínica y promedio de SCS por Hato.
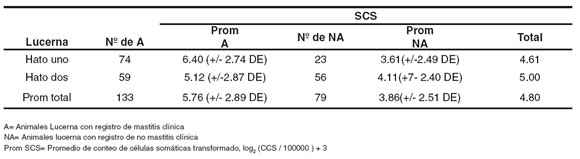
Tabla 5. Medias (+/-DS) de SCS, para cada uno de los alelos mas frecuentes en la población de ganado Colombiano Lucerna.
Análisis de varianza
Con el procedimiento GLM para análisis de varianza con datos desbalanceados, se determinó un efecto significativo (p < 0.05) del alelo 191 (equivale a DRB3.2*22, identificado con PCR-RFLP) (11), sobre un alto recuento de células somáticas. Las demás variables consideradas (nº de parto, hato de origen y grupo de lactancia) no presentaron efectos significativos (p > 0.05) sobre el SCS en esta evaluación. El OR calculado para una copia del alelo 191 fue de 3.35, con un intervalo de confianza al 95% de 1.22 a 9.14 (véase Tabla 6), confirmando la asociación del alelo 191 con alto recuento de células somáticas.
Tabla 6. Odd ratio o relación de diferencia, e intervalo de confianza (IC) 95%, para el alelo BoLA-191 y asociación con SCS.

Con el procedimiento CATMOD para el análisis de datos categóricos, las variables hato y número de partos, se encontraron asociadas significativamente con susceptibilidad a mastitis (p < 0.05).
Discusión
Los resultados del estudio de DRB3 en la raza sintética colombiana Lucerna y su asociación con SCS y mastitis clínica, contribuirán a evaluar la respuesta en bovinos a la susceptibilidad o resistencia a enfermedades infecciosas, así como también la influencia de los polimorfismos de este marcador sobre el recuento de células somáticas.
En estudios previos en bovinos, sobre infecciones y otras enfermedades, se han investigado potenciales asociaciones entre alelos de BoLA-DRB3, con la presencia de enfermedades infecciosas y un elevado conteo de células somáticas (9, 30). El CCS ha sido considerado como indicador de salud de la ubre y de la incidencia de mastitis (20).
La mastitis epidemiológicamente es considerada la enfermedad más común y costosa del ganado lechero en la mayor parte del mundo (15, 17); de origen multifactorial, es el resultado del efecto de condiciones ambientales, grado de exposición a patógenos y susceptibilidad individual (7). Una forma de disminuir su impacto en la población es incrementando la capacidad de resistencia natural de los individuos, por lo que actualmente la investigación se ha enfocado hacia la potenciación de los mecanismos de defensa naturales, durante los períodos de susceptibilidad a la enfermedad (12), aunque hay que considerar el importante papel que pueden desempeñar las condiciones ambientales (15).
En el análisis de equilibrio de H-W (Hardy- Weinberg), los resultados encontrados para la población Lucerna en esta evaluación, podrían ser explicados como una consecuencia de que estos hatos fueron mantenidos en aislamiento, y los machos que son utilizados como parentales sean hijos de hembras del mismo hato, presentando valores de cría superiores para características de interés económico como la producción de leche, favoreciendo en gran medida los cruces consanguíneos.
Los resultados del test de diferenciación génica, indican que ambas poblaciones (hato uno y hato dos), no difieren significativamente (p > 0,05). Con relación a los análisis derivados del cálculo de SCS, los promedios para cada uno de los alelos deterinados mostraron que, los alelos DRB3-171, 191 y 215, presentaron un alto SCS, según el número de copias presentes por alelo.
Los valores de SCS encontrados en las subpoblaciones A y NA y la población total de lucerna que fue muestreada, son bastante altos (véase Tabla 4).
En estos resultados puede estar influyendo además, el aspecto ambiental particular de cada hato, considerado como uno de los factores que incrementa en gran medida el recuento de células somáticas por animal (15). Además las situaciones estresantes de diversa naturaleza que pueden encontrarse en cada hato, pueden agravar el proceso inflamatorio de vacas infectadas y tener poco efecto sobre el CCS de las no infectadas (21). Y de otro lado el ordeño mecánico puede llevar a incrementar el valor de recuento de células somáticas, teniendo en cuenta que puede producir maltrato directo sobre el esfínter del pezón.
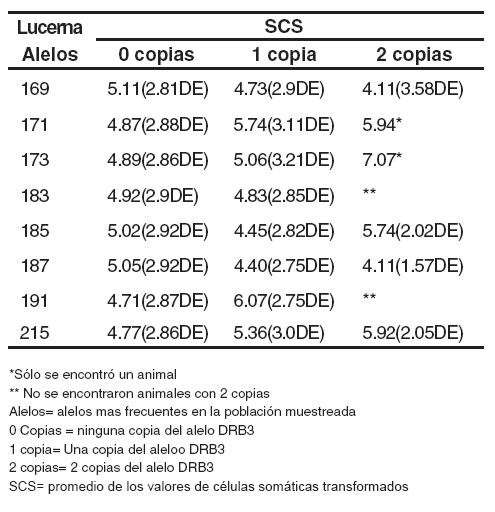
Teniendo en cuenta los resultados del análisis de varianza, y el efecto significativo (p < 0.05) del alelo 191 sobre un alto SCS, se analizó el efecto de la presencia o ausencia del alelo 191 sobre el promedio de SCS, en la población total y en cada una de las subpoblaciones (A y NA). Se pudo observar en los resultados presentados en la tabla 7, que en animales con una copia del alelo 191, el conteo de células somáticas se incrementó en relación con las hembras que no lo presentaron en cada uno de los 3 grupos. Para el alelo 191, el promedio de SCS se redujo a 4.71 (+/-2.87DE), en animales que no presentaron ninguna copia del alelo (véase Tabla 5), siendo significativa (p < 0.05) la diferencia con el promedio de SCS, presentado por animales con un solo alelo. No se encontraron muestras con dos copias de 191.
Tabla 7. Promedios de SCS para el alelo 191, en la población total y en las subpoblaciones afectada y no afectada.
El efecto significativo del alelo 191 sobre el alto recuento de células somáticas, también fue encontrado en estudios realizados por Dietz et al (13) en la raza Holstein, quienes determinaron la asociación de DRB3.2*22 con un alto conteo de células somáticas y susceptibilidad a mastitis. En otras investigaciones, DRB3.2*22 también fue asociado con un bajo riesgo de presentar ovario quístico (30).
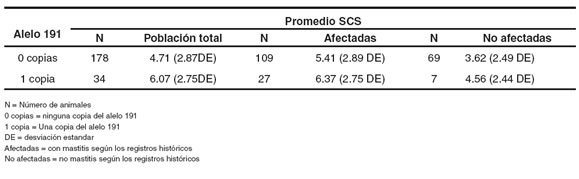
La comparación entre los hatos muestreados con respecto al alelo 191, permitió establecer que este alelo es menos frecuente en el hato uno que en el hato dos (véase Figura 1). La menor frecuencia de este alelo en el hato uno coincide con un menor promedio de SCS en este mismo hato (véase Tabla 4).
En investigaciones sobre susceptibilidad a mastitis, algunos alelos de la región BoLA han sido asociados con la enfermedad. Sharif et al (28), encontraron asociación del alelo DRB3.2*23 con mastitis severa.
En otros estudios, los alelos 207 y DRB3.2*24 fueron significativamente asociados con susceptibilidad a esta enfermedad (9, 11).
En este estudio, el análisis de asociación de la mastitis clínica reconocida por registros históricos, con cada uno de los alelos DRB3 determinados en la población Lucerna, utilizando el procedimiento CATMOD de SAS (28), no se encontró asociación significativa (p>0.05), para ninguno de los alelos determinados.
Resultado que puede ser consecuencia de considerar los registros históricos de mastitis clínica. Estos registros históricos pueden presentar errores de apreciación de los evaluadores de la enfermedad.
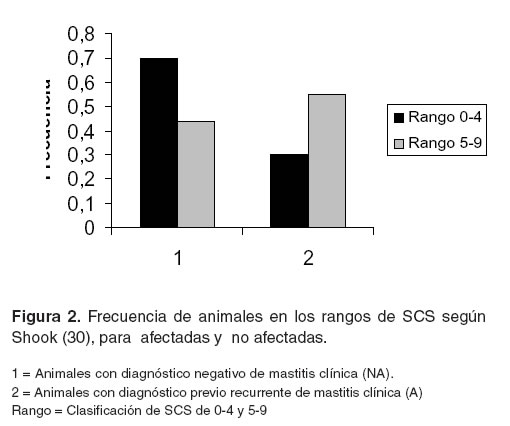
Cuando se analizan conjuntamente el SCS y mastitis clínica, considerando la clasificación por rangos para el conteo de células somáticas establecido por Shook (32) y la clasificación de animales con respecto a mastitis clínica en afectados y no afectados según los registros históricos, los resultados indican que en la población total, un 70% de animales del grupo NA se encuentran ubicados en los cuatro primeros rangos (< 300000 cel/ml) (véase Figura 2). Esto quiere decir que en la subpoblación con diagnóstico negativo de mastitis clínica histórica, la mayor proporción de animales presentó bajo conteo de células somáticas y una mayor tendencia a salud en la ubre. Para el grupo A una mayor proporción de animales se encuentra entre los rangos 5 – 9 (véase Figura 2), es decir, un conteo de células somáticas superior a 300000 cel/ml. Este resultado corrobora lo encontrado en otros estudios donde un alto recuento de células somáticas es indicativo de la incidencia de mastitis (20).
Agradecimientos
A Mario Cerón Muñoz por su asesoría estadística. A la hacienda Lucerna y a la reserva natural del Hatico por su participación en la toma de muestras. A la Cooperativa lechera COLANTA por el apoyo económico en esta investigación.
Summary
BoLA-DRB3 Polymorphism in Colombian synthetic cattle Lucerna and association with somatic cell counts and mastitis.
Previous studies have demostrated an association between BoLA-DRB3, infectious diseases and somatic cells counts in cattle. Polymorphism at the level of the microsatellite intron 2 of synthetic Bovine Colombian Lucerna was investigated. Twentyfour alleles were identified using PCR and poliacrylamide gels; their frequencies ranged from 0.02 to 0.26 among 308 individuals studied. Allele 161 was the most frequent and 159 was the least prevalent. Both subgroups mastatis affected and non affected presented similar frequencies for most alleles, indicating that the population is not in hardy Weinberg equilibrium (p< 0.05). The mean somatic cells score (log2 (CCS / 100000) + 3) for the population was 4.8(+/- 2.89). There was found a significant association between allele 191 and high somatic cell scores but not with clinical mastitis. But mastitis did present a significant association with the variables herd and number of deliveries.
Key Words: BoLA, DRB3, mastitis, somatic cells.
Referencias [ Links ]
2. Adams L.G, Templeton J W. Genetic resistance to bacterial diseases of animals. Rev. Sci. Tech. Off. Int. Epiz 1998; 200- 219[ [ Links ]STANDARDIZEDENDPARAG]
3. Ali A.K.A and Shook G.E. An optimum transformation for somatic cell concentration in milk. J. Dairy Sci; 1980. 63: 487[ [ Links ]STANDARDIZEDENDPARAG]
4. Alizadeh Z, Karrow N, Mallard B.A. Biological effect of varying peptide binding affinity to the BoLA-DRB3*2703 allele. Genet Sel Evol 2003; 35 suppl 1: 51-65. [ Links ]
5. Andersson L, Davis C.J. The major histocompatibility complex. In: Cell-mediated Immunity in ruminants (ed. by B. M. L. Goddeeris & W.I. Morrison ) CRC Press, Boca Ratón, FL. 1994; p 37-57[ [ Links ]STANDARDIZEDENDPARAG]
6. Andersson, L. Bohme, J. Peterson P. A. et al. Genomic hibridization of bovine class II major histocompatibility complex genes: 2. Polymorphism of DR genes and likage disequilibrium in the DQ-DR region. Animal genetic 1986; 17 : 295-304. [ Links ]
7. Barajas R, Jaramillo A, Velásquez O. Factores de riesgo asociados a infecciones subclínicas producidas por biotopos humano y bovino de Staphylococcus aureus en la glándula mamaria de vacas en lactancia. Revista de Veterinaria y Zootecnia de Caldas 1999; 11: 30-33. [ Links ]
8. Banchereau R, Steinman R. M. Dendritic cell and the control of immunity. Nature 1998; 392 : 245-252. [ Links ]
9. Berryere TG, Muggli-Cockett N, Robins JW, Schmutz SM. Molecular studies of DRB relative to Staphylococcus aureus mastitis. Proceedings of the 5th World Congress on Genetic Applied to Livestock Production of Guelph Canada 1994; 21: 187-190. [ Links ]
10. Da Mota AF, Gabriel JE, Martínez ML, Coutinho LL. Distribution of bovine lymphocyte antigen(BoLA-DRB3) alleles in Brazilian dairy Gir cattle (Bos Indicus). European Journal Immunogenetics 2002; Vol 29: 223. [ Links ]
11. Davies C, Joosten I, Andersson L, Arriens M, Bernoco, D, et al. Polymorphism of bovine MHC class II genes. Joint report of the fifth international bovine lymphocyte antigen (BoLA) Workshop, Interlaken, switzerland, 1 August 1992. European Journal of immunogenetic 1994; 21: 259-289. [ Links ]
12. DeRosa D, Shafer-Weaver K, Sordillo L. Immunobiology of the mammary gland. Journal of Dairy Science 1997; 80: 1851. [ Links ]
13. Dietz AB, Cohen ND, Timms L, Kehrli ME. Bovine lymphocyte antigen class II alleles as risk factors for high somatic cell counts in milk of lactating dairy cows. Journal of Dairy Science 1997a ; 80: 406-412. [ Links ]
14. Dietz AB, Detilleux JC, Freeman AE, Kelley DH, Stabel JR, et al. Genetic association of bovine lymphocyte antigen DRB3 alleles with immunological traits of Holstein cattle. Journal of Dairy Science 1997b; 80: 400-405. [ Links ]
15. Dohoo IR, Meek AH. Somatic cell counts in bovine milk. Canadian Veterinary Journal 1982; 23: 119. [ Links ]
16. Ellegren H, Davies CJ, Andersson L. Strong association between polymorphisms in an intronic microsatellite and in the coding sequence of the BoLA-DRB3 gene - implications for microsatellite stability and PCR-based DRB3 typing. Animal Genetic 1993; 24: 269-275. [ Links ]
17. Eberhart RJ. Coliform mastitis. JAm Vet Med Assoc. 1977; 170: 1160. [ Links ]
18. Gillespie B E, B M. Jayarao, H H. Dowlen, et al. Analysis and frequency of bovine lymphocyte antigen DRB3.2 alleles in Jersey cow. Journal Dairy science 1999; 82: 2049-2053. [ Links ]
19. Guo SW, Thompson EA. Performing the exact test of Hardy- Weinberg proportion of multiple alleles. Biometrics 1992; 48:361-372. [ Links ]
20. Harmon RJ. Symposium: Mastitis and genetic evaluation for somatic cell count. J dairy sci. 1994; 77: 2103-2112. [ Links ]
21. Hortet P, Seegers H. Calculated milk production losses associated with elevated somatic cell counts in dairy cows. Review and critical discussion. Vet Res 1998; 29 (6): 497- 510. [ Links ]
22. Lemeshow S, Hosmer DW. Estimating odds ratio with categorically scaled covariates in multiple logistic regression analysis. American Journal Epidemiology 1984; 119: 147-151. [ Links ]
23. Miller SA, Dykes D, Polesky H F. A simple Salting out procedure for extracting DNA from human nucleated cell. Nucleic. Acids. Res 1998; 16: 3p. [ Links ]
24. Morales-Alonso, RA. Genética de la resistencia o susceptibilidad a mastitis bovina. III Congreso Nacional de control de mastitis y calidad de leche, León, Gto. México, 2001; 21- 23 junio. [ Links ]
25. Nassiry MR, Shahroodi FE, Mosafer J, Mohammadi A, et al. Analysis and frequency of bovine lymphocyte antigen (BoLA-DRB3) alleles in Iranian Holstein cattle. Genetika 2005; 41(6): 817-822. [ Links ]
26. Ripoli MV, Liron JP, De Luca JC, Rojas F, et al. Giovambattista, G. Gene frequency distribution of the BoLA-DRB3 locus in Saavedreno Creole dairy cattle. Biochem Genet 2004; 42: 231-240. [ Links ]
27. Rothschild MF, Skow L, Lamont SJ. The major histocompatibility complex and its role in disease resistance and immune responsiveness. En: Breeding for disease resistance in farm animals. 2nd edition. Ed Axford RFE, Bishop SC, Nichols FW Owen JB. CABI publishing 2000; 73p. [ Links ]
28. SAS institute. SAS/STAT User’s Guide: Statistics. Version 8 4th edn. Cary . North Carolina, NC. 1988. [ Links ]
29. Schmutz SM, Berryere TG, Robbins JW, Carrruthers TD. Resistance to Staphylococcus aureus mastitis detected by DNA marker: In proc. 31st Annu. Mtg.Natñl. Mastitis counc., Arlintong , VA Natl Mastitis counc., Inc., Madison, WI. 1992; Pages: 124-123. [ Links ]
30. Sharif S, Mallard BA, Wilkie BN, Sargeant JM, Scott H.M, et al. Associations of the bovine major histocompatibility complex DRB3 (BoLA-DRB3) alleles with occurrence of disease and milk somatic cell score in Canadian dairy cattle. Animal Genetic 1998; 29: 185-193[ [ Links ]STANDARDIZEDENDPARAG]
31. Sharif S, Mallard BA, Wilkie BN. Characterization of naturally processed and presented peptides associated with bovine major histocompatibility complex (BoLA) class II DR molecules. Animal genetic 2003; 34 : 16-123. [ Links ]
32. Shook GE. Approaches to summarizing somatic cell count which improve interpretability. In; 21st. ANN. MTG. NAT. MAST. COUNCIL (1982: Pennsylvania) Procedings Madison: Nat. Mast Council 1982; 150-166. [ Links ]
33. Sigurdardóttir S, Borsch C, Gustafsson K, Andersson L. Cloning and sequence-analysis of 14 DRB alleles of the bovine major histocompatibility complex by using the polymerase chain reaction. Animal Genetic 1991; 22: 199-209[ [ Links ]STANDARDIZEDENDPARAG]
34 Statkin M, and Excoffier L. Testing for linkange dis equilibrium in genotypic data using the expectationmaximization algorithm. Heredity 1996; 76: 377-383. [ Links ]
35. Stonaker HH, Durán-Castro C, Durán-Castro CV. The fundation in the Colombia subtropics of a new dual purpose breeds the Lucerna. Instituto Colombiano Agropecuario Bogotá. 1972; 20p. [ Links ]
36. Takeshima S, Ikegami M, Morita M, et al. Identification of new cattle BoLA- DRB3 allele by sequence based typing. Immunogenetic 2001; 53: 71-81[ [ Links ]STANDARDIZEDENDPARAG]
37. Takeshima S, Nakai Y, Ohta M, et al. Short communication: Characterization of DRB3 alleles in the MHC of japanese shorthorn cattle by polymerase chain reaction-sequencebased typing. Journal Dairy Science 2002; 85: 1630- 1632. [ Links ]
38. Udina IG, Karamysheva EE, Sulimova GE, Pavlenko SP, et al. Comparative analysis of Ayrshire and Black Pied cattle breeds by histocompatibility markers. Genetika 1998; 34: 1668-674. [ Links ]
39. Van Eijk MJT, Stewart- Haynes JA, Lewin, HA. Extensive polymorphism of the BoLA-DRB3 gene distinguished by PCR-RFLP. Animal Genetic 1992; 23: 483. [ Links ]
40. Weir C, and Cockerham R. Covariance of relatives stemming from a population undergoing mixed self and random mating. Biometric 1984; 40 (1): 157-164. [ Links ]














