Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Ciencias Pecuarias
Print version ISSN 0120-0690On-line version ISSN 2256-2958
Rev Colom Cienc Pecua vol.19 no.4 Medellín Oct./Dec. 2006
Genotipificación de la región 3´UTR del gen Nramp1, en ganado Holstein y en criollo Hartón del Valle.
Esperanza Trujillo Bravo1, Biol. MSc; Yasbleydy Valderrama Llanos2, Biol.
1Grupo de Genética y Mejoramiento Animal , Instituto de Biología, Universidad de Antioquia, Apartado Aéreo 1226, Medellín, Colombia.
2Facultad de Ciencias Básicas, Universidad del Tolima.
etbravo@epm.net.co
(Recibido: 29 agosto, 2005; aceptado: 19 septiembre, 2006)
Resumen
En investigaciones sobre respuesta a infecciones por diferentes patógenos intracelulares en humanos y en ratones, el gen Nramp1 se ha descrito como un gen controlador de la resistencia y de la susceptibilidad a algunasenfermedades. En el presente trabajo se genotipificaron 151 bovinos criollosHartón del Valle, y 128 Holstein, para un microsatélite de la región 3’ UTR(que no transcribe), en la posición 1745-1955 del gen Nramp1. Sedeterminaron variaciones de tamaño para éste microsatélite en ambos grupos y se encontró que los más frecuentes son el de 209 y el de207 pares de bases. Los resultados de secuenciación mostraron modificaciones en el número de GTs y se encontraron los polimorfismos (GT)12, (GT)11, previamente reportados por Feng et al, y Horín et al; además se encontraron (GT)14 y (GT)10.
Palabras clave: GTs, patógenos, 3’UTR, repetición, secuenciación, susceptibilidad.
Introducción
La identificación y caracterización de factores genéticos que influyen en la resistencia natural en modelos como el ratón, han reportado información en relación con el mecanismo básico de defensa del huésped frente a las infecciones (8). En el ratón la susceptibilidad o resistencia a microorganismos intracelulares como Mycobacterium bovis (10), Leishmania donovani (4), Salmonella typhimurium (18) y otras micobacterias, se encuentra bajo el control del gen conocido como proteína del macrófago asociada a resistencia natural (Nramp1), localizado en el cromosoma 1 en humanos y en el 2 en bovinos y el cual influye en la tasa de replicación intracelular de estos parásitos en el macrófago. El gen Nramp1 es el mejor candidato en estudios de infecciones con patógenos intracelulares en especies tales como humanos, bovinos y ratones, en los cuales se ha identificado con bastante homología (21, 24).
El gen Nramp1 bovino se encuentra mapeado en el cromosoma 2q43-q44 (6) y tiene un tamaño genómico de aproximadamente 16 kb (5). Presenta polimorfismos debido a la variación en el número de repeticiones GT ubicadas en la región 3’ UTR del gen, que origina los patrones polimórficos designados como Nramp1.1 (6), y Nramp1.2 (12), originados no sólo por cambios en el número de GT repetidos, sino también por variaciones en el número de Gs 5’ adyacentes, generalmente debidos a errores durante la replicación del ADN (7).
Nramp1, codifica una proteína integral de membrana, altamente hidrofóbica, con una masa aproximada de 60 KD. Esta proteína presenta 12 dominios transmembrana, una lupa extracelular glicosilada, varios sitios de fosforilación y un dominio encontrado en muchas proteínas de transporte eucarióticas y procarióticas, esto sugiere que la proteína participa en los procesos de transporte, en los compartimentos endosomal/lisosomal de los macrófagos donde se encuentra expresada (5, 13).
Por el importante papel que parece tener la proteína Nramp1 en la inhibición de la proliferación de microorganismos dentro del macrófago mediante el transporte de cationes y la producción de radicales tóxicos (2,16,20); actualmente se han estudiado formas de relacionar el gen Nramp1 con susceptibilidad o resistencia a patógenos intracelulares.
Es necesario considerar que la resistencia y susceptibilidad a las enfermedades en general, es un rasgo muy complejo, en el cual pueden participar varios genes y diversos factores. Por tal razón, el objetivo de este estudio fue la caracterización del gen Nramp1, región 3’ UTR, en bovinos como Hartón del Valle y Holstein, y además, contribuir a determinar los polimorfismos de este gen en ambas razas.
Materiales y métodos
Población
Fueron seleccionados 151 animales de ganado criollo Hartón del valle de dos núcleos de la zonas centro y norte del Valle del Cauca y 128 de la raza Holstein, muestreadas en cinco localidades del departamento de Antioquia, correspondientes a San Pedro de los Milagros, Belmira, Urrao, Santa Rosa de Osos y Entrerríos, para realizar la genotipificaciones del gen Nramp1, región 3’UTR. Para la extracción de ADN sen utilizaron 7 ml de sangre de cada animal, en tubos vacutainer con EDTA, mediante el método de salting out y precipitación con isopropanol (15).
Genotipificación
La amplificación del microsatélite de la región 3’ UTR de Nramp1 bovino, se estandarizó a un volumen final de 25μl, con 0.7 μg/ml de ADN bovino, buffer de reacción 1 X (50 mM KCl, 10 mM Tris-HCl, pH 9.0, y 0.1% Triton X-100), 0.2 mM de cada deoxinucleótido, 10 mM MgCl2, 0.5 μM de cada cebador y 1U/ul de Taq DNA polimerasa. Los cebadores utilizados, NRAMPF: 5’-GTGGAATGAGTGGGCACAGT-3’ y NRAMPR: 5’-CTCTCCGTCTTGCTGTGCAT-3’ (IDT, Coralville, Iowa, USA), fueron descritos previamente por Feng et al. (6) y amplifican una secuencia ubicada en posición la 1745-1955 de la región 3’ UTR. El perfil térmico de amplificación consistió en un ciclo a 94°C por 2 min, seguido por 30 ciclos a 94°C por 30 seg, 63°C por 30 seg, 72°C por 30 seg, y una extensión final a 72°C por 5 min.
Los productos de PCR fueron verificados en geles de agarosa al 2% por 20 minutos a 72v. Los amplificados fueron visualizados en un gel de poliacrilamida en condiciones desnaturalizantes al 6% durante 90 min a 1900 voltios y teñidos con plata (protocolo de PROMEGA Silver sequence DNA staining reagents).
Los controles positivos se basaron en los resultados obtenidos con la utilización de marcadores de peso molecular, que indicaron el rango de ubicación para cada uno de los tamaños encontrados y posterior comparación con los tamaños reportados en publicaciones previas (6).
Secuenciación
Los productos de PCR de 10 muestras de Hartón del Valle, fueron purificados con el protocolo de extracción QIAquik de Quiagen®, cuantificados y posteriormente secuenciados con un analizador ABI3730XL. “Macrogen Inc”.
Análisis estadístico
La estimación de las frecuencias, alélicas y genotípicas así como la evaluación del equilibrio de Hardy-Weinberg (H-W) se realizó con los programas GENEPOP versión 3.3 (19) y GDA (22, 25).
Resultados
En la genotipificación de los 151 animales de la población Hartón del Valle, se determinaron siete diferentes alelos para Nramp1 (205, 207, 209, 211, 213, 215, 217). Los alelos 211 y 209 fueron reportados previamente por Feng et al (6), y los alelos 205, 215 y 217 que se detectaron solamente en un muy bajo número de animales (2, 1 y 1, respectivamente). Los alelos que más frecuentemente se encontraron en ambas poblaciones fueron 207 y 209 y conforman el 94.6% del total de alelos encontrados (véase Tabla 1).
Tabla 1. Frecuencia de los alelos de Nramp1, región 3´UTR en ganados Holstein y Hartón del Valle.
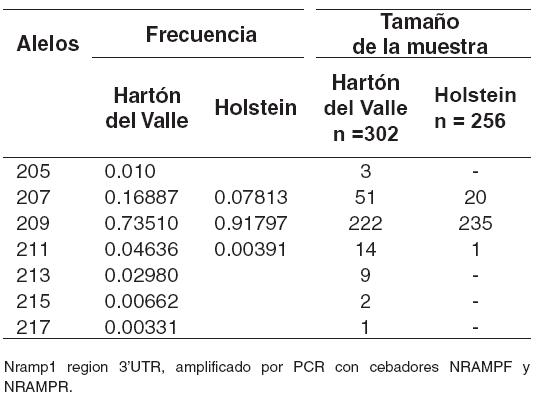
En los 128 animales Holstein que se evaluaron, fueron determinados únicamente tres alelos de Nramp 1: 207, 209, 211, de los cuales el alelo 209 presentó la más alta frecuencia (91.8%) y el 211 la más baja frecuencia (0.003). La frecuencia encontrada para el alelo 207 en esta población, fue de 0.078, que comparada con la encontrada en Hartón del Valle, muestra una notable diferencia (véase Tabla 1).
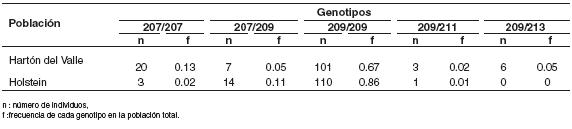
Sólo se presentó homocigosis para los alelos 207 y 209 en ambas poblaciones (véase Tabla 2). Se encontró que, el genotipo homocigótico 209/209, presentó una frecuencia de 0.67 para Hartón del Valle y 0.86 para Holstein, siendo la más alta considerando los demás genotipos. Sin embargo, el homocigótico 207/207 sólo mostró una frecuencia alta en Hartón del valle ya que, en la población Holstein, se encontró con una frecuencia de 0.023 (véase Tabla 2),
Tabla 2. Estimación de las frecuencias de los genotipos más comúnes para Holstein y Hartón del Valle.
En la tabla 3 se muestran los valores estimados de la heterocigosis observada y esperada para el microsatélite de la región 3´UTR evaluado en cada una de las razas.
Tabla 3. Estimación de los valores de heterocigocidad para polimorfismos de la región 3´UTR.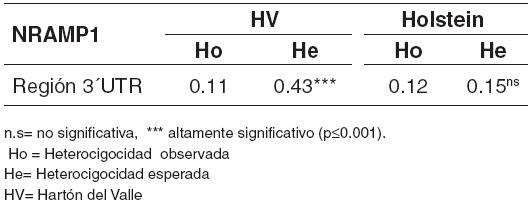
En el análisis de los valores de FIS (índice de fijación), se determinó que para este locus y para cada población son significativos para el déficit de heterocigóticos en la raza criolla Hartón del Valle (véase Tabla 4).
Tabla 4. Valores de FIS para el locus NRAMP1 en cada población.

Los resultados de secuenciación en la raza Hartón del valle, mostraron modificaciones de tamaño de la secuencia de dinucleótidos repetidos GT en la región 3´UTR, diferentes a los encontrados por Feng et al (6) y Horín et al (12). Estas diferencias fueron determinadas en muestras que presentaban los genotipos 209/209, 213/209, 211/209, 209/205 y 207/205 (véase Tabla 5)
Tabla 5. Repeticiones del dinucleótido GT en la región 3´UTR del gen NRAMP.

Discusión
Actualmente se conoce que algunos polimorfismos de la región 3´ UTR del gen Nramp, pueden influenciar fuertemente los niveles de acumulación de RNAm, en respuesta a infecciones por B. abortus y Salmonella (1). Además, se ha encontrado asociación entre los fenotipos de resistencia y susceptibilidad natural y otros polimorfismos del gen Nramp1, que determinan en parte las variaciones en la capacidad funcional de las células fagocíticas (9) por la baja expresión de algunos alelos frente a infecciones (3). Con la genotipificación, determinación de los polimorfismos y posterior estimación de las frecuencias alélicas y genotípicas en las razas Hartón del valle y Holstein se contribuye notablemente al inicio de estudios relacionados con el efecto del gen Nramp particularmente en la raza criolla HV. En la genotipificación de las dos poblaciones analizadas se detectaron siete alelos para la región 3´UTR seleccionada. El polimorfismo encontrado principalmente en HV es mayor que los resultados obtenidos por otros investigadores (6, 12). Sin embargo, la mayor frecuencia corresponde a los alelos 207, 209 y 211, los demás alelos se presentaron en frecuencias demasiado bajas.
En HV se encontraron los alelos 217, 215 y 207 de Nramp1 en un bajo número de animales; estos alelos no han sido reportados en otros trabajos sobre determinación de polimorfismos en la región 3’ UTR de Nramp1 (6, 12). y podrían ser consecuencia de modificaciones en la secuencia en la región 3´UTR, producto de adiciones/deleciones que varían el tamaño de la secuencia (17).
Los resultados de la estimación de las diferencias entre la heterocigocidad observada y esperada para el microsatélite de Nramp 1, en cada una de las razas seleccionadas para este estudio, mostró que la raza Hartón del Valle presentó un déficit de heterocigóticos (p ≤ 0.01), y no presentó equilibrio de Hardy-Weinberg (véase Tabla 3), este resultado debido posiblemente a un alto grado de consanguinidad, quizás por el manejo que se da a esta raza criolla, que implica la utilización de un bajo número de parentales machos, todos con un mismo origen, como efecto de los pocos núcleos que conforman esta raza. Estos núcleos además, son cerrados y aislados, lo que no permite un verdadero intercambio, incrementando la frecuencia de homocigosis en esta población. En la población Holstein no se presentó un déficit de heterocigóticos (p > 0.05) (véase Tabla 3) y se encuentra en equilibrio Hardy-Weinberg.
Feng et al (6) estudiando modificaciones en la región 3’UTR en posición 1781-1804 del gen Nramp1 determinaron el polimorfismo (GT)12. Posteriormente en estudios recientes de Paixao et al, (17) se encontraron variaciones en el tamaño de estas repeticiones y una mayor frecuencia de (GT)13 en animales Cebú considerados genotípicamente resistentes a Brucella y de (GT)14 en animales Holstein considerados susceptibles. Las 10 muestras de Hartón del Valle analizadas por secuenciación, presentaron modificaciones en esta misma región 3’UTR del gen NRAMP1. La repetición (GT)10 fue encontrada en dos de las diez muestras secuenciadas, este polimorfismo fue determinado en investigaciones de Horín et al (12), quienes lo llamaron NRAMP 1.2. También se detectó el polimorfismo (GT)12 (NRAMP 1.1), en uno de los diez animales analizados (véase Tabla 5). Este polimorfismo fue publicado originalmente por Feng et al (6).
En el resto de muestras secuenciadas, se presentaron las repeticiones (GT)14 y (GT)11. Estas modificaciones en el número de repeticiones del dinucleótido GT, pudieron deberse a mutaciones puntuales ocurridas en esta secuencia, como sustitución de bases, determinando una interrupción en la serie de GTs (véase Tabla 5). Otras modificaciones en el número de repeticiones con respecto a lo determinado en bovinos, han sidoencontradas en ovejas (GT)21 (14) y Búfalos (GT)15 (11).
Los resultados de este estudio contribuirán a posteriores estudios en relación con el papel de Nramp1 es la susceptibilidad o resistencia a enfermedades producidas por patógenos intracelulares en bovinos, principalmente en lo que se refiere al ganado criollo colombiano. Indican además, que existen diferencias entre las poblaciones Holstein y Hartón del Valle evaluadas, con respecto a los polimorfismos que presentan para Nramp1 y a las frecuencias de los alelos encontrados. Además es significativo encontrar en HV modificaciones en el número de repeticiones, que aún no han sido reportadas en la literatura y que pueden dar inicio a nuevas investigaciones en esta área en el ganado criollo.
Agradecimientos
A la Cooperativa lechera COLANTA por el apoyo técnico y económico en el desarrollo de esta investigación.
Summary
NRAMP1: Genetic characterization at the 3´UTR region in Holstein and creole Hartón del Valle cattle Summary In studies of natural resistance or susceptibility to different intracellular pathogens the Nramp1 gene has been described as controlling this characteristic in mouse and in the humans. In this project we genotyped 151 Harton of the Valle bovine individuals and 128 Holstein, for a microsatellite of the 3’ untranslated region of Nramp1 gene, in position 1745-1955 and found that the most frequency size of microsatellite were 207 and 211 bp. The sequencing results indicate that there is polymorphim in the number of GT repetitions and (GT)12 and (GT)11 were present as reported previously, as well as (GT)14 y (GT)10.
Key words: GTs, pathogens, repeats, sequencing, susceptibility, 3’UTR.
Referencias
1. Barthel R, Feng J, Piedrahita JA, Mcmurray DN, et al. Stable transfection of the bovine Nramp1 gene into murine RAW264.7 cells: effect on Brucella abortus survival. Infect Immun 2001; 69:3110-3119. [ Links ]
2. Biozzi G, Mounton D, Stiffel C, Bouthillier Y. A mayor role of the macrophage in quantitative genetic regulation of immuno resposiveness and anti-infectious immunity. Adv Immnunol 1984; 36:189-234. [ Links ]
3. Blackwell JM, Searle S, Goswami T, Miller Nancy. Understanding the multiple functions of Nramp 1. Microbes Infect. 2000; 2: 317-321. [ Links ]
4. Bradley DJ. . Genetic control of Leishmania populations within the host. II. Genetic control of acute susceptibility of mice to L. donovani infection. Clin Exp Immnunol 1977; 30:130-140. [ Links ]
5. Céllier M, Govoni G, Vidal S, Kwan T, et al. Human natural resistance-associated macrophage protein: cDNA cloning, chromosomal mapping, genomic organization, and tissue-specific expression. J Exp Med 1994 ; 180:1741-1752. [ Links ]
6. Feng J, Yujing L, Mahmound H, et al. Bovine Natural Resistance Associated Macrophage Protein 1 (Nramp-1) Gene. Genome res 1996; 956-964. [ Links ]
7. Goldstein DB, Pollock DD. Launching microsatellites: a review of mutation processes and methods of phylogenetic inference. Journal of Heredity. 1997; 88:335. [ Links ]
8. Gonovi G, Gros P. Macrophage NRAMP1 and its role in resistance to microbial infections. Department of biochemistry,Mc Gill University, Inflamm Res 1998; 47; 277-284. [ Links ]
9. Gowen JW. Genetic effects in non-specific resistance to infectious disease. Bacteriol Rev 1960; 24:192-200. [ Links ]
10. Gros P. Skamene E. Forget A. Cellular mechanismof genetically controlled host resistence to mycobacterium bovis (BCG). J Immunol 1983; 131:1966-72. [ Links ]
11. Hashad M, FENA J, Refai M, Templeton J W. Cloning, sequencing, and characterization of water buffalo NRAMP cDNA. The EMBLG/Genebank /DDBJ 1996. [ Links ]
12. Horín P, Rychlík I, Templeton JW, Adams LG. A complex pattern of microsatellite polymorphism within the bovine NRAMP1 gene. Eur J Immunogenet 1999; 26: 311-313. [ Links ]
13. Kerpola RE, Ames GFL. Topology of the hydrophobic membrane-bound components of the histidine periplasmic permease. Comparison with other members of the family. J Bio Chem 1992; 267: 2329-36. [ Links ]
14. Mathews G, Crawford AM. Cloning, sequencing and linkage mapping of the NRAMP1 gene of sheep and deer. Animal Genetics 1998; 29: 1. [ Links ]
15. Miller SA, Dykes D, Polesky H F. A simple Salting out procedure for extracting DNA from human nucleated cell. Nucleic Acids Res 1988; 16: 3. [ Links ]
16. Mims CA, White DO. Viral pathogenesis and immunology. Blackwell, Oxford. 1984; 123-167. [ Links ]
17. Paixao TA, Ferreira C, Borges AM, Oliveira DA, Lage AP, Santos RL. Frequency of bovine NRAMP1 (SLc11a1) alleles in Holstein and zebu Breeds. Vet Immunol Immunopathol. 2005http://www.ncbi.nlm.nih.gov/entrez/query.fcgi? CMD=search&DB=pubmed [ Links ]
18. Plant JE, Glynn A.. Genetics of resistance to infection with Salmonella Thyphimurium in mice. J Infect 1976; 133:72-78. [ Links ]
19. Raymond, Rousset F. GENEPOP (version 3.3). updated version of GENEPOP (v. 1.2) described in: population genetics software for exact tests and ecumenicism. J Heredity 2001; 86:248-249. [ Links ]
20. Russell DG. Of microbes and macrophages: entry, survival and persistence. Curr Opin Immunol 1995; 7:479-484. [ Links ]
21. Skamene E, Sur EF, Gros P. 1998. Infection genomics: Nramp1 as a major determinant of natural resistance to intracellular infections. Annual Reviews of Medicine. 49:275. [ Links ]
22. Statkin M, Excoffier L. Testing for linkange disequilibrium in genotypic data using the expectation-maximization algorithm. Heredity. 1996; 76: 377-383. [ Links ]
23. Takahashi K, Satoh J, Kojima Y, Negorok IM, Hinokio Y, Suzuki S, Matsuura N, Shimosegawa T, Oka Y, Promoter polymorphism of SLC11A1 (formerly Nramp1) confers susceptibility to autoinmune type 1 diabetes mellitus in Japanese. Tissue Antigens 2004; 63: 231-236. [ Links ]
24. Vidal SM, Malo D, Vogan K, Skamene E, Gros P. Natural Resistance to Infection with Intracellular Parasites: Isolation of a Candidate for Bcg. Cell 1993: 73: 469. [ Links ]
25. Weir C, Cockerham R. Covariance of relatives stemming from a population undergoing mixed self and random mating. Biometric.1984; 40: 157-164. [ Links ]
Esperanza Trujillo Bravo1, Biol. MSc; Yasbleydy Valderrama Llanos2, Biol.
1Grupo de Genética y Mejoramiento Animal , Instituto de Biología, Universidad de Antioquia, Apartado Aéreo 1226, Medellín, Colombia.
2Facultad de Ciencias Básicas, Universidad del Tolima.
etbravo@epm.net.co
(Recibido: 29 agosto, 2005; aceptado: 19 septiembre, 2006)
Resumen
En investigaciones sobre respuesta a infecciones por diferentes patógenos intracelulares en humanos y en ratones, el gen Nramp1 se ha descrito como un gen controlador de la resistencia y de la susceptibilidad a algunasenfermedades. En el presente trabajo se genotipificaron 151 bovinos criollosHartón del Valle, y 128 Holstein, para un microsatélite de la región 3’ UTR(que no transcribe), en la posición 1745-1955 del gen Nramp1. Sedeterminaron variaciones de tamaño para éste microsatélite en ambos grupos y se encontró que los más frecuentes son el de 209 y el de207 pares de bases. Los resultados de secuenciación mostraron modificaciones en el número de GTs y se encontraron los polimorfismos (GT)12, (GT)11, previamente reportados por Feng et al, y Horín et al; además se encontraron (GT)14 y (GT)10.
Palabras clave: GTs, patógenos, 3’UTR, repetición, secuenciación, susceptibilidad.
Introducción
La identificación y caracterización de factores genéticos que influyen en la resistencia natural en modelos como el ratón, han reportado información en relación con el mecanismo básico de defensa del huésped frente a las infecciones (8). En el ratón la susceptibilidad o resistencia a microorganismos intracelulares como Mycobacterium bovis (10), Leishmania donovani (4), Salmonella typhimurium (18) y otras micobacterias, se encuentra bajo el control del gen conocido como proteína del macrófago asociada a resistencia natural (Nramp1), localizado en el cromosoma 1 en humanos y en el 2 en bovinos y el cual influye en la tasa de replicación intracelular de estos parásitos en el macrófago. El gen Nramp1 es el mejor candidato en estudios de infecciones con patógenos intracelulares en especies tales como humanos, bovinos y ratones, en los cuales se ha identificado con bastante homología (21, 24).
El gen Nramp1 bovino se encuentra mapeado en el cromosoma 2q43-q44 (6) y tiene un tamaño genómico de aproximadamente 16 kb (5). Presenta polimorfismos debido a la variación en el número de repeticiones GT ubicadas en la región 3’ UTR del gen, que origina los patrones polimórficos designados como Nramp1.1 (6), y Nramp1.2 (12), originados no sólo por cambios en el número de GT repetidos, sino también por variaciones en el número de Gs 5’ adyacentes, generalmente debidos a errores durante la replicación del ADN (7).
Nramp1, codifica una proteína integral de membrana, altamente hidrofóbica, con una masa aproximada de 60 KD. Esta proteína presenta 12 dominios transmembrana, una lupa extracelular glicosilada, varios sitios de fosforilación y un dominio encontrado en muchas proteínas de transporte eucarióticas y procarióticas, esto sugiere que la proteína participa en los procesos de transporte, en los compartimentos endosomal/lisosomal de los macrófagos donde se encuentra expresada (5, 13).
Por el importante papel que parece tener la proteína Nramp1 en la inhibición de la proliferación de microorganismos dentro del macrófago mediante el transporte de cationes y la producción de radicales tóxicos (2,16,20); actualmente se han estudiado formas de relacionar el gen Nramp1 con susceptibilidad o resistencia a patógenos intracelulares.
Es necesario considerar que la resistencia y susceptibilidad a las enfermedades en general, es un rasgo muy complejo, en el cual pueden participar varios genes y diversos factores. Por tal razón, el objetivo de este estudio fue la caracterización del gen Nramp1, región 3’ UTR, en bovinos como Hartón del Valle y Holstein, y además, contribuir a determinar los polimorfismos de este gen en ambas razas.
Materiales y métodos
Población
Fueron seleccionados 151 animales de ganado criollo Hartón del valle de dos núcleos de la zonas centro y norte del Valle del Cauca y 128 de la raza Holstein, muestreadas en cinco localidades del departamento de Antioquia, correspondientes a San Pedro de los Milagros, Belmira, Urrao, Santa Rosa de Osos y Entrerríos, para realizar la genotipificaciones del gen Nramp1, región 3’UTR. Para la extracción de ADN sen utilizaron 7 ml de sangre de cada animal, en tubos vacutainer con EDTA, mediante el método de salting out y precipitación con isopropanol (15).
Genotipificación
La amplificación del microsatélite de la región 3’ UTR de Nramp1 bovino, se estandarizó a un volumen final de 25μl, con 0.7 μg/ml de ADN bovino, buffer de reacción 1 X (50 mM KCl, 10 mM Tris-HCl, pH 9.0, y 0.1% Triton X-100), 0.2 mM de cada deoxinucleótido, 10 mM MgCl2, 0.5 μM de cada cebador y 1U/ul de Taq DNA polimerasa. Los cebadores utilizados, NRAMPF: 5’-GTGGAATGAGTGGGCACAGT-3’ y NRAMPR: 5’-CTCTCCGTCTTGCTGTGCAT-3’ (IDT, Coralville, Iowa, USA), fueron descritos previamente por Feng et al. (6) y amplifican una secuencia ubicada en posición la 1745-1955 de la región 3’ UTR. El perfil térmico de amplificación consistió en un ciclo a 94°C por 2 min, seguido por 30 ciclos a 94°C por 30 seg, 63°C por 30 seg, 72°C por 30 seg, y una extensión final a 72°C por 5 min.
Los productos de PCR fueron verificados en geles de agarosa al 2% por 20 minutos a 72v. Los amplificados fueron visualizados en un gel de poliacrilamida en condiciones desnaturalizantes al 6% durante 90 min a 1900 voltios y teñidos con plata (protocolo de PROMEGA Silver sequence DNA staining reagents).
Los controles positivos se basaron en los resultados obtenidos con la utilización de marcadores de peso molecular, que indicaron el rango de ubicación para cada uno de los tamaños encontrados y posterior comparación con los tamaños reportados en publicaciones previas (6).
Secuenciación
Los productos de PCR de 10 muestras de Hartón del Valle, fueron purificados con el protocolo de extracción QIAquik de Quiagen®, cuantificados y posteriormente secuenciados con un analizador ABI3730XL. “Macrogen Inc”.
Análisis estadístico
La estimación de las frecuencias, alélicas y genotípicas así como la evaluación del equilibrio de Hardy-Weinberg (H-W) se realizó con los programas GENEPOP versión 3.3 (19) y GDA (22, 25).
Resultados
En la genotipificación de los 151 animales de la población Hartón del Valle, se determinaron siete diferentes alelos para Nramp1 (205, 207, 209, 211, 213, 215, 217). Los alelos 211 y 209 fueron reportados previamente por Feng et al (6), y los alelos 205, 215 y 217 que se detectaron solamente en un muy bajo número de animales (2, 1 y 1, respectivamente). Los alelos que más frecuentemente se encontraron en ambas poblaciones fueron 207 y 209 y conforman el 94.6% del total de alelos encontrados (véase Tabla 1).
Tabla 1. Frecuencia de los alelos de Nramp1, región 3´UTR en ganados Holstein y Hartón del Valle.

En los 128 animales Holstein que se evaluaron, fueron determinados únicamente tres alelos de Nramp 1: 207, 209, 211, de los cuales el alelo 209 presentó la más alta frecuencia (91.8%) y el 211 la más baja frecuencia (0.003). La frecuencia encontrada para el alelo 207 en esta población, fue de 0.078, que comparada con la encontrada en Hartón del Valle, muestra una notable diferencia (véase Tabla 1).
Sólo se presentó homocigosis para los alelos 207 y 209 en ambas poblaciones (véase Tabla 2). Se encontró que, el genotipo homocigótico 209/209, presentó una frecuencia de 0.67 para Hartón del Valle y 0.86 para Holstein, siendo la más alta considerando los demás genotipos. Sin embargo, el homocigótico 207/207 sólo mostró una frecuencia alta en Hartón del valle ya que, en la población Holstein, se encontró con una frecuencia de 0.023 (véase Tabla 2),
Tabla 2. Estimación de las frecuencias de los genotipos más comúnes para Holstein y Hartón del Valle.

En la tabla 3 se muestran los valores estimados de la heterocigosis observada y esperada para el microsatélite de la región 3´UTR evaluado en cada una de las razas.
Tabla 3. Estimación de los valores de heterocigocidad para polimorfismos de la región 3´UTR.

En el análisis de los valores de FIS (índice de fijación), se determinó que para este locus y para cada población son significativos para el déficit de heterocigóticos en la raza criolla Hartón del Valle (véase Tabla 4).
Tabla 4. Valores de FIS para el locus NRAMP1 en cada población.

Los resultados de secuenciación en la raza Hartón del valle, mostraron modificaciones de tamaño de la secuencia de dinucleótidos repetidos GT en la región 3´UTR, diferentes a los encontrados por Feng et al (6) y Horín et al (12). Estas diferencias fueron determinadas en muestras que presentaban los genotipos 209/209, 213/209, 211/209, 209/205 y 207/205 (véase Tabla 5)
Tabla 5. Repeticiones del dinucleótido GT en la región 3´UTR del gen NRAMP.

Discusión
Actualmente se conoce que algunos polimorfismos de la región 3´ UTR del gen Nramp, pueden influenciar fuertemente los niveles de acumulación de RNAm, en respuesta a infecciones por B. abortus y Salmonella (1). Además, se ha encontrado asociación entre los fenotipos de resistencia y susceptibilidad natural y otros polimorfismos del gen Nramp1, que determinan en parte las variaciones en la capacidad funcional de las células fagocíticas (9) por la baja expresión de algunos alelos frente a infecciones (3). Con la genotipificación, determinación de los polimorfismos y posterior estimación de las frecuencias alélicas y genotípicas en las razas Hartón del valle y Holstein se contribuye notablemente al inicio de estudios relacionados con el efecto del gen Nramp particularmente en la raza criolla HV. En la genotipificación de las dos poblaciones analizadas se detectaron siete alelos para la región 3´UTR seleccionada. El polimorfismo encontrado principalmente en HV es mayor que los resultados obtenidos por otros investigadores (6, 12). Sin embargo, la mayor frecuencia corresponde a los alelos 207, 209 y 211, los demás alelos se presentaron en frecuencias demasiado bajas.
En HV se encontraron los alelos 217, 215 y 207 de Nramp1 en un bajo número de animales; estos alelos no han sido reportados en otros trabajos sobre determinación de polimorfismos en la región 3’ UTR de Nramp1 (6, 12). y podrían ser consecuencia de modificaciones en la secuencia en la región 3´UTR, producto de adiciones/deleciones que varían el tamaño de la secuencia (17).
Los resultados de la estimación de las diferencias entre la heterocigocidad observada y esperada para el microsatélite de Nramp 1, en cada una de las razas seleccionadas para este estudio, mostró que la raza Hartón del Valle presentó un déficit de heterocigóticos (p ≤ 0.01), y no presentó equilibrio de Hardy-Weinberg (véase Tabla 3), este resultado debido posiblemente a un alto grado de consanguinidad, quizás por el manejo que se da a esta raza criolla, que implica la utilización de un bajo número de parentales machos, todos con un mismo origen, como efecto de los pocos núcleos que conforman esta raza. Estos núcleos además, son cerrados y aislados, lo que no permite un verdadero intercambio, incrementando la frecuencia de homocigosis en esta población. En la población Holstein no se presentó un déficit de heterocigóticos (p > 0.05) (véase Tabla 3) y se encuentra en equilibrio Hardy-Weinberg.
Feng et al (6) estudiando modificaciones en la región 3’UTR en posición 1781-1804 del gen Nramp1 determinaron el polimorfismo (GT)12. Posteriormente en estudios recientes de Paixao et al, (17) se encontraron variaciones en el tamaño de estas repeticiones y una mayor frecuencia de (GT)13 en animales Cebú considerados genotípicamente resistentes a Brucella y de (GT)14 en animales Holstein considerados susceptibles. Las 10 muestras de Hartón del Valle analizadas por secuenciación, presentaron modificaciones en esta misma región 3’UTR del gen NRAMP1. La repetición (GT)10 fue encontrada en dos de las diez muestras secuenciadas, este polimorfismo fue determinado en investigaciones de Horín et al (12), quienes lo llamaron NRAMP 1.2. También se detectó el polimorfismo (GT)12 (NRAMP 1.1), en uno de los diez animales analizados (véase Tabla 5). Este polimorfismo fue publicado originalmente por Feng et al (6).
En el resto de muestras secuenciadas, se presentaron las repeticiones (GT)14 y (GT)11. Estas modificaciones en el número de repeticiones del dinucleótido GT, pudieron deberse a mutaciones puntuales ocurridas en esta secuencia, como sustitución de bases, determinando una interrupción en la serie de GTs (véase Tabla 5). Otras modificaciones en el número de repeticiones con respecto a lo determinado en bovinos, han sidoencontradas en ovejas (GT)21 (14) y Búfalos (GT)15 (11).
Los resultados de este estudio contribuirán a posteriores estudios en relación con el papel de Nramp1 es la susceptibilidad o resistencia a enfermedades producidas por patógenos intracelulares en bovinos, principalmente en lo que se refiere al ganado criollo colombiano. Indican además, que existen diferencias entre las poblaciones Holstein y Hartón del Valle evaluadas, con respecto a los polimorfismos que presentan para Nramp1 y a las frecuencias de los alelos encontrados. Además es significativo encontrar en HV modificaciones en el número de repeticiones, que aún no han sido reportadas en la literatura y que pueden dar inicio a nuevas investigaciones en esta área en el ganado criollo.
Agradecimientos
A la Cooperativa lechera COLANTA por el apoyo técnico y económico en el desarrollo de esta investigación.
Summary
NRAMP1: Genetic characterization at the 3´UTR region in Holstein and creole Hartón del Valle cattle Summary In studies of natural resistance or susceptibility to different intracellular pathogens the Nramp1 gene has been described as controlling this characteristic in mouse and in the humans. In this project we genotyped 151 Harton of the Valle bovine individuals and 128 Holstein, for a microsatellite of the 3’ untranslated region of Nramp1 gene, in position 1745-1955 and found that the most frequency size of microsatellite were 207 and 211 bp. The sequencing results indicate that there is polymorphim in the number of GT repetitions and (GT)12 and (GT)11 were present as reported previously, as well as (GT)14 y (GT)10.
Key words: GTs, pathogens, repeats, sequencing, susceptibility, 3’UTR.
Referencias
1. Barthel R, Feng J, Piedrahita JA, Mcmurray DN, et al. Stable transfection of the bovine Nramp1 gene into murine RAW264.7 cells: effect on Brucella abortus survival. Infect Immun 2001; 69:3110-3119. [ Links ]
2. Biozzi G, Mounton D, Stiffel C, Bouthillier Y. A mayor role of the macrophage in quantitative genetic regulation of immuno resposiveness and anti-infectious immunity. Adv Immnunol 1984; 36:189-234. [ Links ]
3. Blackwell JM, Searle S, Goswami T, Miller Nancy. Understanding the multiple functions of Nramp 1. Microbes Infect. 2000; 2: 317-321. [ Links ]
4. Bradley DJ. . Genetic control of Leishmania populations within the host. II. Genetic control of acute susceptibility of mice to L. donovani infection. Clin Exp Immnunol 1977; 30:130-140. [ Links ]
5. Céllier M, Govoni G, Vidal S, Kwan T, et al. Human natural resistance-associated macrophage protein: cDNA cloning, chromosomal mapping, genomic organization, and tissue-specific expression. J Exp Med 1994 ; 180:1741-1752. [ Links ]
6. Feng J, Yujing L, Mahmound H, et al. Bovine Natural Resistance Associated Macrophage Protein 1 (Nramp-1) Gene. Genome res 1996; 956-964. [ Links ]
7. Goldstein DB, Pollock DD. Launching microsatellites: a review of mutation processes and methods of phylogenetic inference. Journal of Heredity. 1997; 88:335. [ Links ]
8. Gonovi G, Gros P. Macrophage NRAMP1 and its role in resistance to microbial infections. Department of biochemistry,Mc Gill University, Inflamm Res 1998; 47; 277-284. [ Links ]
9. Gowen JW. Genetic effects in non-specific resistance to infectious disease. Bacteriol Rev 1960; 24:192-200. [ Links ]
10. Gros P. Skamene E. Forget A. Cellular mechanismof genetically controlled host resistence to mycobacterium bovis (BCG). J Immunol 1983; 131:1966-72. [ Links ]
11. Hashad M, FENA J, Refai M, Templeton J W. Cloning, sequencing, and characterization of water buffalo NRAMP cDNA. The EMBLG/Genebank /DDBJ 1996. [ Links ]
12. Horín P, Rychlík I, Templeton JW, Adams LG. A complex pattern of microsatellite polymorphism within the bovine NRAMP1 gene. Eur J Immunogenet 1999; 26: 311-313. [ Links ]
13. Kerpola RE, Ames GFL. Topology of the hydrophobic membrane-bound components of the histidine periplasmic permease. Comparison with other members of the family. J Bio Chem 1992; 267: 2329-36. [ Links ]
14. Mathews G, Crawford AM. Cloning, sequencing and linkage mapping of the NRAMP1 gene of sheep and deer. Animal Genetics 1998; 29: 1. [ Links ]
15. Miller SA, Dykes D, Polesky H F. A simple Salting out procedure for extracting DNA from human nucleated cell. Nucleic Acids Res 1988; 16: 3. [ Links ]
16. Mims CA, White DO. Viral pathogenesis and immunology. Blackwell, Oxford. 1984; 123-167. [ Links ]
17. Paixao TA, Ferreira C, Borges AM, Oliveira DA, Lage AP, Santos RL. Frequency of bovine NRAMP1 (SLc11a1) alleles in Holstein and zebu Breeds. Vet Immunol Immunopathol. 2005http://www.ncbi.nlm.nih.gov/entrez/query.fcgi? CMD=search&DB=pubmed [ Links ]
18. Plant JE, Glynn A.. Genetics of resistance to infection with Salmonella Thyphimurium in mice. J Infect 1976; 133:72-78. [ Links ]
19. Raymond, Rousset F. GENEPOP (version 3.3). updated version of GENEPOP (v. 1.2) described in: population genetics software for exact tests and ecumenicism. J Heredity 2001; 86:248-249. [ Links ]
20. Russell DG. Of microbes and macrophages: entry, survival and persistence. Curr Opin Immunol 1995; 7:479-484. [ Links ]
21. Skamene E, Sur EF, Gros P. 1998. Infection genomics: Nramp1 as a major determinant of natural resistance to intracellular infections. Annual Reviews of Medicine. 49:275. [ Links ]
22. Statkin M, Excoffier L. Testing for linkange disequilibrium in genotypic data using the expectation-maximization algorithm. Heredity. 1996; 76: 377-383. [ Links ]
23. Takahashi K, Satoh J, Kojima Y, Negorok IM, Hinokio Y, Suzuki S, Matsuura N, Shimosegawa T, Oka Y, Promoter polymorphism of SLC11A1 (formerly Nramp1) confers susceptibility to autoinmune type 1 diabetes mellitus in Japanese. Tissue Antigens 2004; 63: 231-236. [ Links ]
24. Vidal SM, Malo D, Vogan K, Skamene E, Gros P. Natural Resistance to Infection with Intracellular Parasites: Isolation of a Candidate for Bcg. Cell 1993: 73: 469. [ Links ]
25. Weir C, Cockerham R. Covariance of relatives stemming from a population undergoing mixed self and random mating. Biometric.1984; 40: 157-164. [ Links ]














