Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Ciencias Pecuarias
Print version ISSN 0120-0690On-line version ISSN 2256-2958
Rev Colom Cienc Pecua vol.20 no.1 Medellín Mar./Jan. 2007
Caracterización molecular de tres líneas de Cavia porcellus mediante la aplicación de AFLP
Carlos Solarte-Portilla 1, Zoot, PhD; Heiber Cárdenas-Henao2, Biol, MSc; Carol Rosero-Galindo2, Biol, MSc; William Burgos-Paz, Zoot.
1Grupo Producción y Sanidad Animal, Facultad de Ciencias Pecuarias, Programa de Zootecnia, Universidad de Nariño, Ciudad Universitaria Torobajo, A. A. 1175, Pasto, Colombia; 2Laboratorio de Biología Molecular, Departamento de Biología, Facultad de Ciencias, Universidad del Valle, A.A. 25360, Cali, Colombia.
csolarte@udenar.edu.co
(Recibido: 20 febrero, 2006; aceptado: 5 febrero, 2007)
Resumen
Los marcadores moleculares son una herramienta eficaz para determinar variabilidad genética entre y dentro de poblaciones, pero en el caso de Cavia porcellus, no existen reportes referentes al uso de estas técnicas. Con los marcadores moleculares AFLP´s (Amplified Fragment Length Polimorphism), se analizaron tres poblaciones, dos criollas y una mejorada genéticamente, sometida a selección durante varias generaciones y obtenida a partir del cruzamiento entre animales peruanos y nativos de Nariño. Para obtener los marcadores moleculares AFLP´s (Amplified Fragment Lenght Polimorphism), se utilizaron en total cinco combinaciones de cebadores, tres combinaciones recomendadas para el orden Rodenthia y dos por la casa fabricante del Kit, de las cuales sólo una de ellas, con 116 loci, permitió establecer diferencias entre las poblaciones estudiadas, de acuerdo con el valor de distancia genética insesgada de Nei (p<0.01). Las dos poblaciones criollas constituyeron un grupo estrechamente relacionado y distante de la población mejorada genéticamente, lo que indica que los animales importados absorbieron al criollo. De acuerdo con los valores de heterocigosidad promedio, que variaron entre 0.48% y 14.48%, y el porcentaje de polimorfismo que osciló entre 0.00% y 39.65%, se deduce una baja variabilidad intrapoblacional, siendo la población mejorada genéticamente la más polimórfica. La baja variabilidad entre los animales mejorados, se explica por la intensa selección a la que han sido sometidos, mientras que en los núcleos criollos este fenómeno puede atribuirse al bajo tamaño efectivo en las dos poblaciones. Los resultados de esta investigación sugieren un replanteamiento de los programas de mejoramiento genético y conservación de los recursos genéticos autóctonos en la región.
Palabras clave: AFLP´s, Cavia porcellus, variabilidad genética.
Introducción
Diversos documentos reseñados por varios autores como Ortegón y Morales (17), Caycedo (3) y Solarte et al (21) permiten concluir que el cuy Cavia porcellus es originario de Sudamérica y que este mamífero roedor se encontraba completamente domesticado antes de la llegada de los españoles.
Actualmente, en los países andinos existe una población estable de más o menos 35 millones de cuyes. Perú es el país con la mayor población, consumo y distribución en la casi totalidad del territorio, mientras que en Colombia y Bolivia su distribución es regional y con poblaciones menores. Por su capacidad de adaptación a diversas condiciones climáticas, los cuyes pueden encontrarse desde la costa o el llano hasta alturas de 4500 metros sobre el nivel del mar y en zonas tanto frías como cálidas (4).
La carne de cuy se consumía habitualmente en toda la zona Andina de Colombia, hasta casi la mitad del siglo XX, pero debido a creencias erróneas sobre posibles efectos negativos derivados de este consumo y a problemas de índole sanitario en los animales, la producción se fue extinguiendo poco a poco y quedó limitada al departamento de Nariño y a otras pocas regiones, principalmente, los departamentos de Cauca y Putumayo (2).
En el departamento de Nariño, el cuy es un elemento importante de la seguridad alimentaria y de la economía campesina. En las últimas décadas, la producción se ha tecnificado y uno de los aspectos que ha contribuido al logro de mayores niveles de productividad en la cuyicultura es el uso de animales más precoces y eficientes obtenidos por selección artificial. El proceso de mejoramiento genético en la región se inició con la importación de ejemplares peruanos, los cuales presentaban rendimientos notoriamente superiores a los denominados “criollos”, animales ancestralmente utilizados en la zona, caracterizados por su tamaño pequeño y bajos índices productivos (2).
En 1975 se inició un programa de mejoramiento por cruzamientos entre cuyes peruanos y criollos, se logró el efecto favorable de heterosis y por ende el incremento de los rendimientos productivos y reproductivos. Sin embargo, el uso intensivo de animales importados y el corto ciclo reproductivo de la especie condujeron a que el animal criollo fuera rápidamente absorbido, con lo que se puso en riesgo de extinción un patrimonio genético local que jamás fue sometido a procesos de selección, a pesar de mostrar superioridad con respecto a los peruanos, en aspectos como resistencia a enfermedades y aprovechamiento de forrajes de bajo valor nutritivo.
Posteriormente, con los núcleos de ejemplares mestizos, se inició un proceso de selección genética por los métodos clásicos (20) y en los últimos cinco años, tanto la estimación de parámetros como la valoración genética y la selección de reproductores se lleva a cabo con modelos mixtos, concretamente el Modelo Animal -BLUP (21, 22), adaptando a la especie las técnicas y procedimientos indicados por Henderson (8), Boldman y Van Vleck (1) y Meyer (13).
Actualmente, en el departamento de Nariño ninguna explotación cuyícola, con algún nivel de tecnificación, utiliza reproductores criollos, los cuales sólo se encuentran en núcleos conservados como los que posee la Universidad de Nariño y algunas explotaciones tradicionales; por lo tanto, se presume pérdida de variabilidad genética en los animales mejorados por efecto de la alta endogamia, desaprovechamiento de un recurso genético autóctono y riesgo de extinción del mismo.
En el presente estudio se evaluaron tres líneas de Cavia porcellus, dos criollas y una mejorada genéticamente, sometida a selección durante varias generaciones con el fin de establecer la variabilidad genética en la especie, dentro y entre poblaciones, mediante la utilización de marcadores moleculares AFLP`s (Amplified Fragment Length Polimorphism).
Materiales y métodos
Localización
Los ejemplares utilizados para este trabajo provinieron de la Granja “Botana”, propiedad de la Universidad de Nariño, situada a nueve kilómetros al sur de la ciudad de Pasto, en la república de Colombia, a una altura de 2820 m.s.n.m., temperatura promedio de 12.7 ºC, 967 mm. de precipitación pluvial media anual y humedad relativa del 73% (9).
Muestras de sangre
Se utilizaron muestras de sangre procedentes de 36 animales, 12 de cada población, cuyas principales características fenotípicas se describen a continuación:
Población 1. Animales criollos Línea 1. Ejemplares de forma angulosa, cuerpo de poca profundidad y desarrollo muscular escaso, cabeza triangular y alargada con separación evidente del cuerpo, pelo corto y lacio, colores variados desde el negro hasta el albino, baja velocidad de crecimiento y mala conversión alimenticia pero más resistente a enfermedades con respecto a los mejorados. Animal explotado intensivamente en la zona del departamento de Nariño antes de la introducción de los peruanos y que en buena parte corresponde a la descripción de los animales tipo B indicada por la FAO (4).
Población 2. Animales criollos Línea 2. Cuerpo pequeño, pelo largo y encrespado, colores variados, pero no se encuentra el fenotipo de pelaje totalmente negro, rendimientos productivos y reproductivos menores a los de la línea 1. Animal que nunca se ha explotado con fines alimenticios.
Población 3. Animales mejorados genéticamente. Cuyes con una conformación enmarcada dentro de un paralelepípedo clásico en las líneas productoras de carne, buena longitud, profundidad y ancho, mayor desarrollo muscular fijado en una buena base ósea, cabeza pequeña en la que no se distingue la separación con el cuerpo, pelo corto de colores claros, mayor velocidad de crecimiento y buena conversión alimenticia. Actualmente, se utilizan como pie de cría en las explotaciones tecnificadas y semitecnificadas del departamento de Nariño y es comúnmente denominado “peruano”. La descripción anterior coincide con la indicada por la FAO (4) para los animales tipo A.
A cada animal se le tomó una muestra de sangre por punción cardiaca; la cantidad extraída varió entre 0.5 y 2.5 c.c. y fueron aplicadas dos gotas directamente a la tarjeta de FTA® (Whatman Bioscience, Cambridge, United Kingdom), para luego obtener el ADN el cual fue amplificado para realizar los análisis correspondientes. La amplificación del ADN se llevó a cabo utilizando el kit, AFLP®Analysis System I de Invitrogen, acorde con el protocolo de Vos et al (23), y las recomendaciones de la casa fabricante (10).
Combinaciones de cebadores para la amplificación del ADN
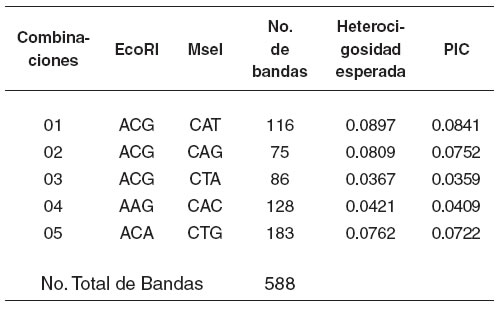
Para el estudio de variabilidad genética, se seleccionaron las combinaciones más polimórficas teniendo en cuenta el valor PIC o Contenido Informativo Polimórfico (véase Tabla 1). Las combinaciones seleccionadas en este estudio son coincidentes con las tres reportadas por Dragoo et al (5) para la familia Muridae (Rodentia): Combinacion1, EcoRI-ACG/MseI-CAT; combinación 2, EcoRI-ACG/ MseI-CAG; combinación 3, EcoRI-ACG/ MseI-CTA, y con las dos reportadas por la casa fabricante (10); combinación 4, EcoRI-AAG/ MseI-CAC; combinación 5, EcoRI-ACA/ MseI-CTG, como combinaciones que son altamente reproducibles en el laboratorio para la estimación de la variabilidad genética en el genoma de roedores y plantas.
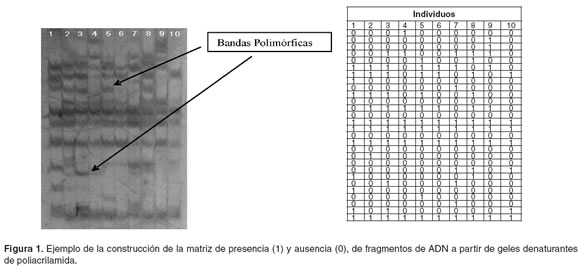
La detección de fragmentos de longitud variable en Cavia porcellus se realizó en un gel desnaturalizante de poliacrilamida al 6% y 3 M de úrea, utilizando una cámara vertical de electroforesis (Sequi-Gen GT Sequencing Cell) de BIO-RAD y tinción con nitrato de plata. Para estimar la variabilidad genética se construyó una matriz de presencia (1) y ausencia (0), de los fragmentos observados en el gel desnaturalizante (véase Figura 1).
Tabla 1. Contenido Informativo Polimórfico (PIC) de cinco combinaciones de cebadores de AFLPs.
Análisis estadístico de polimorfismos, heterocigosidad y distancias genéticas
Para estimar el porcentaje de polimorfismo obtenido con cada combinación de cebadores se utilizó el método de estimación no sesgada de Lynch y Milligan (11). También se determinó el porcentaje de polimorfismo obtenido dentro de cada población mediante el paquete estadístico TFPGA (14).
Las frecuencias alélicas se estimaron teniendo en cuenta la dominancia del marcador AFLP; para ello se usó el método convencional, es decir la raíz cuadrada de la frecuencia del genotipo recesivo y el método de estimación no sesgada de Lynch y Milligan (11), además por tratarse de un marcador dominante, se asumió equilibrio Hardy - Weinberg. Para estimar la heterocigosidad promedio esperada dentro y entre poblaciones, se utilizo la fórmula:
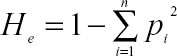 , donde р es la frecuencia del alelo i.
, donde р es la frecuencia del alelo i.
Para generar la matriz de distancia entre pares de poblaciones se utilizó el coeficiente de apareamiento simple a partir de los datos de AFLP`s, con el programa AMOVA-PREP (14). Posteriormente, esta matriz fue procesada con el programa WINAMOVA 1.55 (6) y se llevó a cabo el correspondiente análisis de varianza. Con este último programa, se estableció el nivel de significancia de los componentes de varianza y se calcularon los valores φST, análogo al estadístico F de Wright (24).
El análisis de variabilidad genética entre y dentro de poblaciones, se llevó a cabo utilizando el coeficiente de similitud de Nei (15, 16). El programa TFPGA permitió obtener valores de similitud entre pares de individuos y construir el correspondiente dendrograma con el método de agrupamiento UPGMA.
Adicionalmente, se realizó la prueba exacta de diferenciación poblacional de Raymond y Rousset (18). En este análisis se usaron tablas de contingencia de r filas (poblaciones) por c columnas (alelos o marcadores en un locus) para establecer la existencia de diferencias significativas en las frecuencias alélicas entre grupos de individuos; así fue posible analizar cada locus en la serie de datos y determinar la existencia de diferencias estadísticas en las frecuencias alélicas. Adicionalmente, se empleó la probabilidad combinada de Fisher (7, 12, 19) como una prueba global sobre todos los loci para determinar la significancia total. Para el uso de este procedimiento se asumió que los datos, a partir de cada locus, eran independientes unos de otros.
Resultados
La heterocigosidad promedio insesgada de Nei (16) y el porcentaje de loci polimórficos con nivel de confianza del 95% en cada una de las poblaciones estudiadas y para la población total se pueden apreciar en la tabla 1. La diversidad genética varió entre 0.0048% para la combinación 3 y 14.08% para la combinación 1; ambos valores se observaron en la población 3 de animales mejorados (véase Tabla 2).
Como puede apreciarse en la tabla 3, con los datos obtenidos a partir del “bootstrapping”, el valor de theta promedio más alto lo mostró la combinación 1 con una cifra de 0.2154 y el más bajo la combinación 3 con 0.0136.
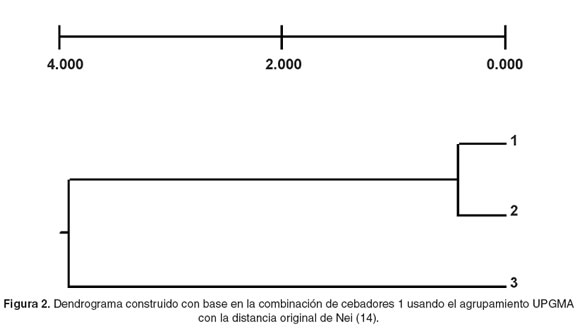
Al calcular las distancias genéticas de Nei (14, 15), entre las parejas de poblaciones comparadas (véase Tabla 4), se confirmó que la combinación 1 fue la que mejor representó la diferencia de la población mejorada, con respecto a las dos líneas criollas, que esquemáticamente se representa en la figura 2.
Los agrupamientos UPGMA (véase Tabla 5) se estimaron usando la distancia original de Nei (14) para cada una de las cinco combinaciones de cebadores y para las cinco combinaciones consideradas conjuntamente.
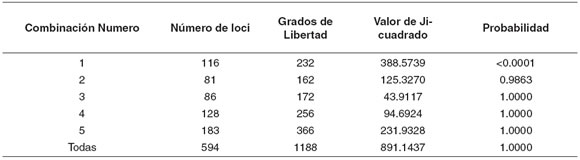
Los resultados de la prueba para diferenciación poblacional de Raymond y Rousset (17) (véase Tabla 6), indican que sólo en la combinación 1 se detectaron diferencias poblacionales altamente significativas (p<0.01), mientras que las combinaciones restantes mostraron homogeneidad total.
Tabla 2. Estadística descriptiva de cada una de las �cinco combinaciones de primers, reportadas por Dragoo (5).
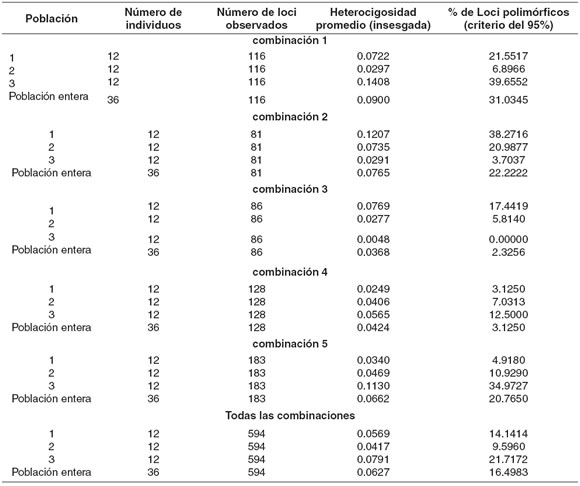
Tabla 3. Valor de theta promedio con su desviación estándar e intervalo de confianza obtenido a partir del “bootstrapping” sobre todos los loci.
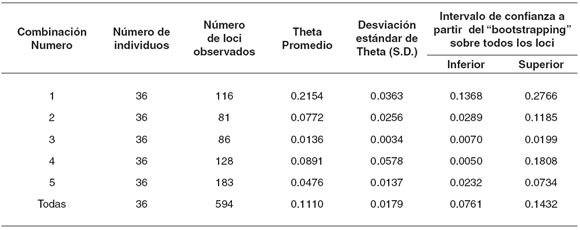
Tabla 4. Distancias genéticas de Nei (14, 15) entre las tres combinaciones posibles de parejas de poblaciones comparadas.
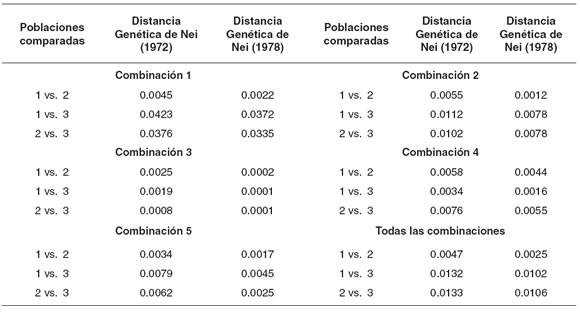
Tabla 5. Agrupamiento UPGMA usando la Distancia original de Nei (14) para cada una de las cinco combinaciones de primer y las cinco combinaciones conjuntamente.
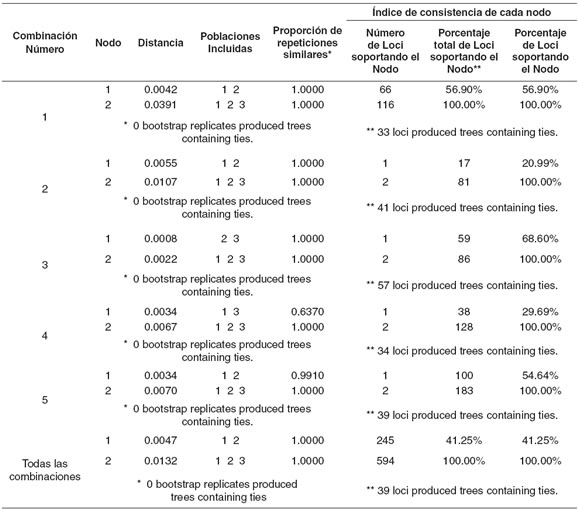
Tabla 6. Prueba de diferenciación poblacional de Raymond and Rousset (17) para cada una de las cinco combinaciones analizadas.
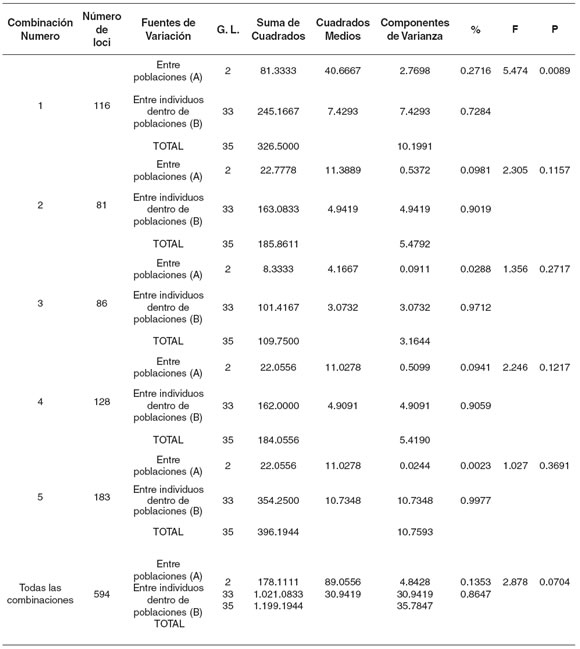
En la tabla 7, están consignados los resultados de los AMOVA realizados para cada una de las combinaciones de cebadores utilizadas y para las combinaciones consideradas como un conjunto. El valor de φST 0.2716 (p<0.01) mostró una alta significancia para la combinación 1, mientras que ninguno de los otros mostró significancia al 95%. Este valor se considera alto, ya que en la estructura poblacional valores de diferenciación superiores al 0.25 se consideran elevados.
Tabla 7. Análisis de Varianza Molecular (AMOVA) entre poblaciones por combinación de primer usada y todas las combinaciones conjuntamente.
Discusión
Los porcentajes de diversidad genética calculados en esta investigación sugieren una notable pérdida de variabilidad en las tres poblaciones estudiadas. Estos valores bajos, en la población 3, se explican por efecto de la selección continua de genotipos para fines comerciales lo que ha conducido a homogenización genética. En el caso de la población 1, explotada intensamente en la zona de Nariño antes de la introducción de los cuyes peruanos, también se observó erosión genética similar a la de la población 3 y confirma la existencia de un núcleo cerrado, correspondiente a los animales conservados en la granja, sin la posibilidad de llevar a cabo procesos de refrescamiento, principalmente por la dificultad de encontrar animales nativos en la zona, para utilizarlos como reproductores. En la población 2, los valores bajos de diversidad revelan pérdida de variabilidad causada por un pie de cría fundador bajo y altamente endogámico. Como ya se indicó, este tipo de animales corresponde a un grupo que no se ha explotado para la alimentación humana por su bajo rendimiento, pero ofrece un potencial económico para lo productores locales, ya que podría comercializarse como mascota, por su apariencia y temperamento.
Todos los resultados analizados en esta investigación son consistentes, ya que indican pérdida de la diversidad genética natural de Cavia porcellus en la región, además, los valores de distancia genética y los nodos del dendrograma (véase Figura 2), permiten asegurar que la población 3 es la línea genéticamente diferente, hecho que confirma la absorción del material genético autóctono por el peruano, luego de 15 generaciones de selección (21) y que los núcleos de animales criollos preservados en la granja de la Universidad de Nariño se conservan como recurso genético local, pero con una baja variabilidad dentro de cada una de las poblaciones, ya que los pie de cría fundadores de las líneas criollas fueron altamente endogámicos, por lo que se recomienda un refrescamiento de las líneas 1 y 2 con el fin de recuperar la variabilidad perdida y conservarlas como reservorio genético.
Por otra parte, el marcador AFLP resultó útil para los estudios de diversidad genética y de diferenciación entre poblaciones altamente endogámicas; sin embargo, de las cinco combinaciones de cebadores que se emplearon para la amplificación selectiva, reportadas por Dragoo et al (5) para la familia Muridae (Rodentia), sólo la combinación 1, con 116 loci, mostró suficiente diversidad genética y diferenciación entre poblaciones.
Para futuros análisis, es necesario la evaluación de las poblaciones de Cavia porcellus con mas combinaciones de cebadores y otros tipos de marcadores moleculares, de tal modo que se compare la estructura poblacional y se programen nuevos planes de mejoramiento genético y conservación.
Agradecimientos
A la Vicerrectoría de Investigaciones, Posgrados y Relaciones Internacionales de la Universidad de Nariño y al Departamento de Biología de la Universidad del Valle por haber facilitado los recursos necesarios para la culminación exitosa de esta investigación.
Summary
Molecular characterization of three populations of Cavia porcellus with AFLP markers
Molecular markers are a powerful tool to determine genetic variability within and among populations, but for the Cavia porcellus there are no reports on the use of these techniques. Three populations, two native and another one, genetically improved which was obtained by crossing native and Peruvian animals and submitted to genetic selection through several generations were analyzed by means of AFLP markers. Five primer’s combinations recommended for Rodenthia were used, but only one allowed to establish significant differences (p<0.01) according to unbiased Nei´s distance Value. Both native populations were grouped in a cluster genetically distant from the genetically improved animals. This showed that foreign animals absorbed the native populations. The average heterosigosity between 0.48% and 14.48% and the percentage of polymorphisms between 0.00% and 39.65% allow to conclude that there was a low variability between the populations, but the population genetically improved was the most polymorphic. The low variability within the improved animals it can be explained because of the intensive selection procedures use with them, whereas within the native populations can be explained because of their very low populations effective size. These results suggest that there is a need to restate the genetic improvement and preservation programs of the native Cavia porcellus in the southwest region of Colombia.
Key words: AFLP, Cavia porcellus, genetic variability.
Referencias
1. Boldman KG, Van Vleck LD. Derivative - Free Restricted Maximum Likelihood estimation in animal models with a sparse matrix solver. J Dairy Sci 1991; 74:4337. [ Links ]
2. Caycedo A. Línea de investigación en cuyes y susalcances en la tecnificación de la explotación. 1 ed. Pasto-Colombia: Universidad de Nariño; 1993. 24 p. [ Links ]
3. Caycedo A. Experiencias investigativas en la producción de cuyes. Contribución al desarrollo tecnológico de la explotación. 1 ed. Pasto-Colombia; Facultad de Ciencias Pecuarias, Universidad de Nariño; 1999. 509 p. [ Links ]
4. Chauca L. Producción de cuyes Cavia porcellus. FAO INIA 1997; [20 Septiembre 2005] URL: http://www.fao.org. [ Links ]
5. Dragoo JW, Salazar-Bravo J, Layne LJ, Yates TL. Relationships within the Calomys callosus species group based on amplified fragment length polymorphisms. Bioch Sys Ecol 2003; 31: 703-713. [ Links ]
6. Excoffier L, Smouse PE, Quattro JM. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA data. Gene 1992; 131:479-491. [ Links ]
7. Fisher RA. Statistical methods for research workers. 10th ed. Edinburgh: Oliver and Boyd;1954. [ Links ]
8. Henderson CR. Theoretical basis and computational methods for a number of different models. Proc. Of animal model Workshop. Edmonton, Alberta: American Dairy Science; 1988. 125 p. [ Links ]
9. Instituto Nacional de Estudios Ambientales (IDEAM). 1999. Reporte de climatología de la estación Botana. Pasto-Colombia: El instituto; 1999. [ Links ]
10. Invitrogen. Instruction manual AFLP Analysis System I, Starter Primer Kit version B 2003; URL http://www.invitrogen.com/content/sfs/manuals/AFLPi.pdf [ Links ]
11. Lynch M, Milligan BG. Analysis of population genetic structure with RAPD markers. Mol Ecol 1994; 3:91-99. [ Links ]
12. Manly BFJ. Randomization and Montecarlo methods in biology. New York: Chapman and Hall; 1991. [ Links ]
13. Meyer K. Estimating variance and covariances for multivariate animal model by restricted maximum Likelihood. Gen Sel Evol 1991; 23:67. [ Links ]
14. Miller MP. Tools for Populations Genetic Analysis (TFPGA) version 1.3. A Windows Program for the Analysis of allozyme and Molecular Population Genetic Data 1997; URL http://herb.bio.nau.edu/~miller/tfpga.htm. [ Links ]
15. Nei M. Genetic distance between populations. Amer Nat 1972; 106:283-290. [ Links ]
16. Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Gene 1978; 89:583-590. [ Links ]
17. Ortegón M, Morales F. El Cuy. 1 ed. Pasto-Colombia: Facultad de Zootecnia Universidad de Nariño; 1987. 295 p. [ Links ]
18. Raymond ML, Rousset F. An exact test for population differentiation. Evol 1995; 49:1280-1283. [ Links ]
19. Sokal R, Rohlf FJ. Biometry. 3rd ed. New York: W.H. Freeman and Co; 1995. [ Links ]
20. Solarte C, Viteri L. Indice de selección, prueba de progenie y prueba de comportamiento en cuyes Cavia porcellus. Zoot 2001; 4:35-44. [ Links ]
21. Solarte C, Soto F, Pérez T. Modelo animal multicarácter para la estimación de parámetros genéticos del Cavia porcellus en Colombia. Rev Cuba Cienc Agríc. 2000; 36:19-24. [ Links ]
22. Solarte C, Imuez M, Pérez T. Modelo animal multicarácter para la selección de reproductores Cavia porcellus en Colombia. Rev Cuba Cienc Agríc. 2000; 36:25-30. [ Links ]
23. Vos P, Hogers R, Bleeker M, Reijans M, Van de Lee T, et al AFLP: a new technique for DNA fingerprinting. Nucl Acid Res 1995;23:4407-4414. [ Links ]
24. Wright S, Evolution in Mendelian populations. Gene 1931; 16: 97-159. [ Links ]














