Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Química
Print version ISSN 0120-2804On-line version ISSN 2357-3791
Rev.Colomb.Quim. vol.39 no.3 Bogotá Sept./Dec. 2010
ANÁLISIS PROTEÓMICO DE CULTIVOS PRIMARIOS DE TIROIDES
PROTEOMIC ANALYSIS OF PRIMARY CULTURES FROM THYROID
ANÁLISE PROTEÓMICO DE CULTURAS PRELIMINARES DO THYROID
Nancy P. Echeverri1, Blanca L. Ortiz1,2, Jorge E. Caminos3
1 Departamento de Química, Facultad de Ciencias. Universidad Nacional de Colombia, sede Bogotá. Bogotá, Colombia.
3 Departamento de Fisiología, Facultad de Medicina. Universidad Nacional de Colombia, sede Bogotá. Bogotá, Colombia.
Recibido: 09/09/09 - Aceptado: 08/11/10
RESUMEN
En el presente estudio se identificaron proteínas de expresión constitutiva, como vimentina, actina, tubulina, proteína de choque térmico de 60 kDa, peroxirredoxina y la ATP sintasa mitocondrial, en cultivos primarios de tiroides normales y de carcinoma papilar de tiroides. Se establecieron las condiciones de extracción, solubilización, análisis cuantitativo y cualitativo de dichas proteínas, y se buscaron las mejores condiciones de isoelectroenfoque (IEF) en la electroforesis en dos dimensiones (2D).
En la extracción y solubilización de las proteínas se evaluó la presencia o ausencia de anfolitos y sales, se obtuvo un mejor resultado empleando en el amortiguador de extracción sales como Tris-HCl y acetato de magnesio que incrementan la solubilidad de las proteínas. Para la cuantificación se recomienda el uso conjunto de técnicas colorimétricas con la electroforesis SDS-PAGE tiñendo con azul de Coomassie y corroborando los resultados mediante western blot, lo cual permite, además, verificar la integridad de las proteínas.
Respecto a la electroforesis en dos dimensiones, se obtuvieron geles con un mayor número de manchas (spots),resueltos, enfocados y reproducibles empleando en el IEF gradientes inmovilizados de pH de 4-7 y voltaje final de 8.000 V. Las proteínas se identificaron mediante el análisis bioinformático de los geles 2D con el programa PDQuest (PDQuest 7.2, Bio-Rad®) y MALDI-TOF.
Palabras clave: proteómica, isoelectroenfoque (IEF), electroforesis 2D.
ABSTRACT
In this paper, proteins of constitutive expression such as vimentin, actin, tubulin, heat shock protein of 60 kDa, peroxiredoxin and the mitochondrial ATP sintase were identified in primary cultures of normal thyroid and papillary carcinoma. The extraction conditions, solubilization, quantitative and qualitative analysis of such proteins were established. In addition, the best conditions for isoelectrofocusing (IEF) in the two-dimensional electrophoresis (2D) were founded.
In the extraction and solubilization of proteins, the presence or absence ofampholytes or salts was evaluated. The best result in the extraction was obtained using buffer salts such as Tris-HCl and magnesium acetate, which increase the solubility of these proteins. For quantification, the recommendation is to combine colorimetric techniques with SDS-PAGE electrophoresis stained with Coomasie blue and Western blot to confirm the results, which allows verifying the integrity of these proteins.
In the two-dimensional electrophoresis, resolved, focused and reproducible gels with greater number of spots were obtained using in the IEF immobilized pH gradients of 4-7 and final voltage of 8000 V. The proteins were identified by the bioinformatic analysis of the 2DE gels with the PDQuest program (PDQuest 7.2 (BioRad®) and MALDI-TOF.
Key words: proteomics, isoelectrofocousing (IEF), 2D electrophoresis.
RESUMO
Neste estudo, se identificaram proteínas da expressão constitutiva, como vimentina, actina, tubulina, proteína de choque do calor do kDa 60, peroxiredoxinaeasintase mitocondrial do ATP, em cultivos primários de tireóide normal e de carcinoma papilar de tireóide. Se estabeleceram as condições de extração e de solubilization, assim como a análise quantitativaeaqualitativa de tais proteínas, e procuraram-se asmelhores condições para isoeletroenfoque (IEF) na eletroforese bidimensional (2D).
Na extração e no solubilization das proteínas, se avaliou a presença ou a ausência de anfolitos ou de sais. O melhor resultado foi obtido usando sais como Tris-HCl e acetato do magnésio no amortecedor da extração, as quais aumentam a solubilida-de das proteínas. Na quantificação, se recomenda o uso de técnicas colorimétricas com eletroforese SDS-PAGE, tingindo com azul de Coomasie. Os resultados se confirmam com Western blot, o qual permite também verificar a integridade das proteínas.
Quanto à eletroforese bidimensional, se obtiveram géis com um maior número de pontos (spots) determinados, enfocados e reprodutíveis no IEF usando gradientes imobilizados de pH 4-7 e tensão final de 8000 V. As proteínas foram identificadas por análise bioinformática de géis 2D com o programa PDQuest (PDQuest 7.2 (Bio-Rad®) e MALDITOF.
Palavras-chave: proteómica, isoeletroenfoque (IEE), eletroforese 2D.
INTOODUCCIÓN
La proteómica estudia el proteoma,entendido como el conjunto de proteínas que se expresa a partir de un genoma, así como sus modificaciones (fosforilación, acetilación, ubiquitinación, entre otras), y provee información acerca de la abundancia, localización e interacciones entre proteínas (1, 2).
En muchos casos no son los cambios en los genes los que dan lugar a la enfermedad, sino alteraciones en los niveles de traducción y modificaciones postraduccionales de las proteínas que en últimas determinan la función biológica. Por esta razón, en los últimos años se han venido desarrollando técnicas que permitan la búsqueda de biomarcadores para la detección de las enfermedades en estadios tempranos, basadas en la identificación de proteínas que están sobreexpresadas o modificadas como consecuencia del propio proceso de la enfermedad.
Existe gran variedad de técnicas para identificar y comparar la abundancia de las proteínas, así como también para determinar modificaciones postraduccionales, localización subcelular e interacciones moleculares, como son la cromatografía líquida, la electroforesis en dos dimensiones (2D), acoplada a espectrometría de masas, y ensayos con anticuerpos, entre otros (3). Dentro de las aplicaciones más ampliamente extendidas de la electroforesis en 2D destacan la identificación a gran escala de proteínas (proteómica global) y el estudio de las diferencialmente expresadas (proteómica diferencial) (4).
El presente trabajo se realizó con el propósito de encontrar las mejores condiciones para el análisis proteómico de cultivos primarios de tiroides, teniendo en cuenta que las enfermedades proliferativas de la tiroides son, aún hoy, muy abundantes.
El potencial analítico de la electroforesis 2D es dependiente de la preparación y cuantificación de la muestra, para poder obtener reproducibilidad, resolución y un gran número de manchas (spots)enel mapa proteómico. En cualquier caso, las proteínas necesitan ser solubilizadas, desagregadas, desnaturalizadas y reducidas. Sin embargo, los procesos de solubilización y cuantificación de proteínas son los pasos más susceptibles de errores, lo cual afecta la reproducibilidad, dificultando el análisis comparativo entre diferentes líneas celulares o tejidos (5). Para la solubilización de proteínas se emplean diferentes mezclas de compuestos caotrópicos, detergentes o surfactantes, agentes reductores y anfolitos. El papel de los caotropos, tales como urea y tiourea, es romper los puentes de hidrógeno, conduciendo a un desplegamiento de la proteína y a su desnaturalización; los surfactantes tales como el ácido 3[(3-colamidopropil)dimetilamonio]-propanosulfónico (CHAPS), sulfobetaina 3-10 (SB 3-10), amidosulfobetaina-14 (ASB-14) y dodecilsulfatodesodio(SDS), actúan sinérgicamente con los caotropos. Los agentes reductores, tales como ditiotreitol (DTT) y tributilfosfina (TBP), son usados para romper las interacciones intra e intermoleculares de los puentes disulfuro (6).
Por lo anterior, en este estudio, empleando muestras de cultivos primarios de tiroides normal (TN) y de cáncer papilar de tiroides (T-CP2), se evaluaron diferentes condiciones de extracción y solubilización de proteínas que incluyeron la adición de anfolitos, los cuales, como se sugiere en algunos estudios, podrían potenciar la solubilidad, ya que minimizan la agregación de las proteínas debido a la interacción carga-carga (5, 6).También se evaluó el método de cuantificación de proteína, ya que la urea, la tiourea, los detergentes y posiblemente los anfolitos interfieren y afectan el resultado de la concentración de proteína (4, 7), se emplearon los métodos colorimétricos, Bradford y Lowry modificado y la electroforesis, validando los resultados por western blot contra tubulina. Se estandarizaron las condiciones de la electroforesis 2D y se realizó el análisis de algunas proteínas de expresión constitutiva mediante el programa bioinformático PD Quest (PDQuest 7.2, Bio-Rad®). Mediante el método matrix-assisted laser desorption/ionisation-time-of-flight (MALDI-TOF), se identificaron las proteínas vimentina, actina, tubulina, proteína de choque térmico de 60 kDa, peroxirredoxina y la ATP sintasa mitocondrial.
MATERIALES Y MÉTODOS
Cultivo celular
Los cultivos primarios de tiroides normales (TN) y de carcinoma papilar (T-CP2) se obtuvieron del banco de tumores tiroideos humanos en cultivo (BANTTIC) de la Universidad de Santiago de Compostela, España. Este banco está establecido a partir de células en cultivo obtenidas directamente de las piezas quirúrgicas de tiroides de pacientes operados de hiperplasias, bocios, adenomas o cáncer. En estos cultivos se estudian de forma funcional mecanismos proliferativos y efecto de mutaciones sobre la fisiología de la célula folicular tiroidea, con la característica importante de que las células mantienen las mismas mutaciones que existen en los pacientes (8, 9).
Las alícuotas de las células congeladas en nitrógeno líquido se descongelaron y se cultivaron a 37 °C en el medio definido Hams F-12 modificado por Coon (10), suplementado con suero fetal bovino al 5% (SFB) (Gibco, BRL), glutamina 2 mM, una mezcla de 5 hormonas: 5H (hormona estimulante de tiroides (TSH) 1 mUI/mL, somatostatina 10 ng/mL, insulina 10 µg/mL, hidrocortisona 1 nM y transferrina 5 µg/mL), penicilina 100 mg/mL, estreptomicina 100 mg/mL y anfotericina B 2,5 ng/mL (8, 9).
Extracción de proteínas
Para la extracción de proteína se sembraron 2x106 células por placa de 90 mm y se incubaron 48 h, en las condiciones descritas previamente (8, 9). Las células adherentes se transfirieron con ayuda de un raspador a 1 mL de medio de cultivo, y se centrifugaron a 400 xg durante 5 minutos a 4 °C. Se realizaron tres lavados con el amortiguador: acetato de magnesio 5 mM y Tris-HCl, 10 mM pH 8,0 (11), y se centrifugaron a 400 xg durante 5 minutos a 4 °C. Una vez finalizado el lavado el residuo de células se resuspendió en 500 µL del amortiguador de lisis correspondiente según la Tabla 1. Se homogenizó en sonicador durante 5 minutos con ciclos de 15 segundos (7, 12). La sonicación debe hacerse siempre en hielo para evitar el calentamiento de la muestra. Se incubó 30 min a temperatura ambiente. Se centrifugó a 15.000 xg durante 5 minutos a 4 °C y el sobrenadante se almacenó a -20 °C.
Para realizar el western blot y la tinción con azul de Coomassie del gel unidimensional, se usó como control la proteína proveniente de 1x106 células cultivadas en placas de 60 mm, extraída luego de incubación a 4 °C durante 20 minutos con 100 µL de amortiguador de lisis Schlessinger (sal sódica del ácido 4-(2-hidroxietil) piperazina-1-etanosulfónico (Hepes) 50 mM, pH 7,5, NaCl 150 mM, glicerol al 10%, tritón al 1%, ácido etilenglicol-O, O'-bis(2-aminoetil)-tetraacético (EGTA) 5 mM, MgCl21,5 mM), al cual se agregó una mezcla de inhibidores de fosfatasas y proteasas (pirofosfato sódico 20 mM, aprotinina 20 µg/mL, fluoruro de fenilmetilsulfonilo (PMSF) 4 mM , inhibidor de proteasas de serina y ortovanadato sódico (Na3VO4)93 µg/mL). Después de centrifugar a 15.000 xg durante 5 minutos a 4 °C, se separó el sobrenadante y se realizó la cuantificación de las proteínas por el método de Bradford (13), ya que este no tiene interferencias con el buffer Schlessinger por la ausencia de urea y tiourea.
Integridad de las proteínas
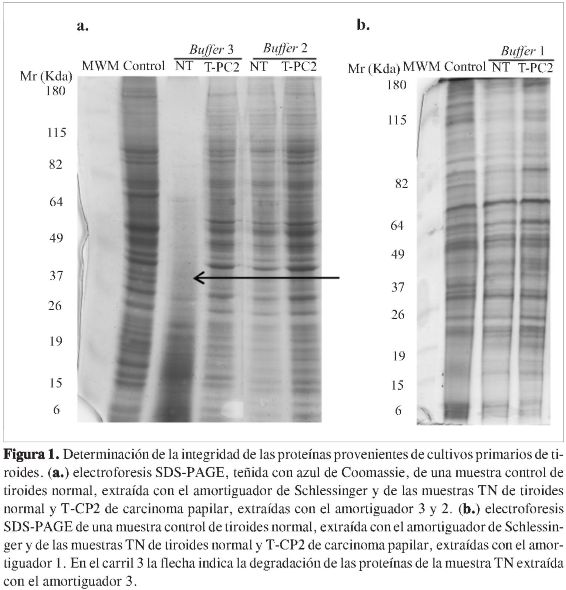
Para determinar la integridad de las proteínas extraídas se empleó la electroforesis 1D de SDS-PAGE (12%), seguida de tinción con azul de Coomassie y western blot.
Electroforesis 1-D de SDS-PAGE (12%): se realizó a 30 mA durante 1% h, con una muestra patrón extraída con el amortiguador de lisis de Schlessinger, y el resto de las muestras con los diferentes amortiguadores (Tabla 1). La concentración teórica de proteína por carril fue de 50µg en un volumen total de 35 µL; el gel se tiñó con azul de Coomassie y se comparó la intensidad de bandas respecto a la muestra control (método cualitativo).
Western blot: las muestras separadas en el paso anterior de electroforesis se transfirieron a una membrana de polidifluoruro de vinilideno (PVDF), la cual se incubó durante la noche a 4 °C en amortiguador de bloqueo (amortiguador de fosfatos 0,05 M, NaCl 0,8%, pH 7,4 (PBS) con caseína al 5%). Se incubó con el primer anticuerpo, anti a-tubulina de ratón (Sigma), diluido 1:2.500, durante 1 hora a temperatura ambiente. La membrana se sometió a cinco lavados de 10 min cada uno con PBS y luego se incubó 1 hora en amortiguador de bloqueo con proteína G marcada con peroxidasa de rábano (dilución 1:10.000), la cual presenta una alta interacción con la región constante del primer anticuerpo. La señal se observó por quimioluminiscencia (ECL, Pierce Biotechnology, USA).
Cuantificación de proteína
Se evaluaron los siguientes métodos de cuantificación de proteínas:
RC-DC BioRad: se empleó el estuche de reactivos RC-DC Bio-Rad (catálogo 500-0119), el cual se basa en el método de Lowry, según las instrucciones del fabricante. Se usó albúmina de suero bovino (ASB) como proteína patrón y se midió la absorbancia a 750 nm.
Bradford: se realizó según Bradford (13), procesando las muestras por triplicado, empleando ASB como proteína patrón y midiendo la absorbancia a 595 nm.
ELECTROFORESIS EN DOS DIMENSIONES Y ANÁLISIS
Primera dimensión
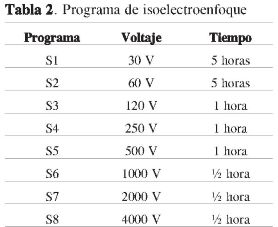
En la primera dimensión se separan las muestras de proteínas de acuerdo con su punto isoeléctrico en las tiras de gradiente inmobilizado de pH (GE Healthcare-Amersham), que varían en cuanto a rango de pH. Para este estudio se usaron tiras de 11 cm y rangos de pH de 4-7 y de 3-10. La cantidad máxima de proteína por cargar en tiras de este tamaño es de 50 µg. Las muestras se rehidrataron con el amortiguador: urea 7 M, tiourea 2 M, Chaps al 4% y azul de bromofenol al 0,05%, además, se añadieron anfolitos al 0,5%, DTT 50 mM o destreak® al 1,2% (a las correspondientes al rango de pH 3-10) en un volumen de 200 µL. Se dejó en agitación mínimo 4 horas a temperatura ambiente. Se centrifugó a 12.000 xg, a temperatura ambiente por 30 minutos, y se procedió a realizar el isoelectroenfoque según el siguiente programa (Tabla 2).
El voltaje total fue inicialmente de 4.000 V y en los experimentos finales fue de 8.000 V en un equipo de IPGphor 3 de Healthcare.
Segunda dimensión
En la segunda dimensión las proteínas se separaron por peso molecular, por lo que debían tener la misma cantidad de cargas. Para ello se equilibraron las tiras provenientes del isoelectroenfoque en la solución: Tris-HCl 50 mM, pH 8,8, urea 6 M, glicerol al 30%, SDS al 2%, azul de bromofenol al 0,05%. Se dejaron en agitación a temperatura ambiente durante 15 minutos en dicha solucion, más DTT al 1%, y luego otros 15 minutos en la solución más iodoacetamida (el DTT reduce los puentes disulfuro y la iodoacetamida alquila los átomos de azufre impidiendo su renaturalización). Las tiras se pusieron, luego, en contacto con el gel SDS-PAGE (12%), y se realizó la electroforesis. Se usó el siguiente programa: 30 V por 15 minutos, 60 V por 15 minutos y 120 V durante 1 hora. La visualización se realizó por tinción con plata empleando el kit Plws One Silver Staning (GE Healthcare Cat 17-1150-01).
Análisis bioinformático de los geles 2D
Este análisis se realizó con la ayuda del programa bioinformático PDQuest (PDQuest 7.2 User Guide, Bio Rad) un software para análisis de imagen, que analiza y almacena la información proveniente de los geles 2D teñidos con plata. Este programa permite el análisis de la imagen en tres formas: la primera es la imagen escaneada, la segunda en un formato Raw-2 y la tercera es la imagen gaussiana, a partir de esta última el programa analiza los spots. Este programa permite seleccionar el tamaño e intensidad de los spots, los cuales pueden ser editados. También permite tomar los replicados de cada gel y normalizar los datos, por ese motivo se recomienda la realización de mínimo 3 geles por muestra. Además, se puede obtener la densidad de los spots, lo cual permite determinar la expresión relativa de las proteínas entre las muestras (los spots diferencialmente expresados).
Una vez realizada la edición y normalización de los geles 2DE, se compararon los diferentes spots de cultivos celulares mediante el programa PDQuest (TN y T-CP).
Digestión de las bandas
Los spots aislados a partir de los geles 2D fueron enviados al Centro Nacional de Biotecnología (CNB) de Madrid, donde se realizó la digestión y obtención de las huellas peptídicas. Los spots fueron tratados con bicarbonato amónico 25 mM antes de su reducción con DDT 10 mM y alquilación con iodoacetamida 100 Mm en bicarbonato amónico 50 mM. Las porciones de este gel 2D fueron después tratadas con bicarbonato amónico 50 mM y acetonitrilo, y secadas en corriente de nitrógeno. Luego se realizó el tratamiento a las proteínas de los spots previamente aislados, con tripsina porcina modificada (sequencing grade, Promega, Madison WI), a una concentración final de 16 ng/µL en bicarbonato amónico 25 mM durante 6 horas a 37 °C. Finalmente, los péptidos resultantes de la digestión proteica se eluyeron del gel con ácido trifluoroacético 0,1% y bicarbonato amónico 25 mM.
Obtención de la huella peptídica
Para mejorar la calidad de los espectros obtenidos se creó una capa fina de matriz de ácido 2,5-dihidroxibenzoico (DHB), la cual se depositó sobre un portamuestras AnchorChip™ MALDI de 400 mm, y se dejó secar para posteriormente añadir la muestra de proteínas digeridas.
La adquisición de la huella peptídica se realizó en un espectrómetro de masas MALDI-TOF Reflex™ IV Bruker (Bruker-Franzen Analytic GmbH, Bremen, Germany). Su tratamiento y análisis con distintos programas informáticos permite enviar los resultados de los espectros de masas de los péptidos trípticos a una base de datos (SwissProt) mediante un software específico en el que se concretan varios parámetros críticos a la hora de la identificación de cada proteína.
RESULTADOS Y DISCUSIÓN
Los estudios en patología de tiroides se basan principalmente en análisis genómicos y proteómicos. En el análisis proteómico, la extracción, solubilización y cuantificación de las proteínas son factores claves para un resultado fiable y reproducible, en especial cuando lo que se desea es determinar las proteínas expresadas constitutivamente y las diferencialmente expresadas. Existen varias técnicas y amortiguadores para la lisis celular. La lisis física ha sido el método de elección por preferencia (14), adicionando detergentes debido a su bajo costo y eficiencia, aunque existen protocolos que tratan de evitarlos debido a las interferencias que presentan con algunos métodos de cuantificación de proteínas (15).
En el presente estudio se evaluaron tres amortiguadores para la solubilización de las proteínas presentes en cultivos primarios de tiroides (Tabla 1), que contienen urea y tiourea como agentes caotrópicos, las cuales desnaturalizan las proteínas inactivando muchas enzimas, entre estas las proteasas, por tal razón, se usaron concentraciones relativamente altas (urea 7 M y tiourea 2 M). La tiourea es un reactivo que rompe las interacciones de carácter hidrofóbico y es insoluble en solución acuosa, sin embargo, su solubilidad se incrementa a medida que incrementa la concentración de urea. El uso de estas sustancias requiere trabajar a temperaturas inferiores a 37 °C, para evitar posibles degradaciones de la urea (la cual forma isocianato que induce carbamilación en las proteínas, modificando su carga y formación de complejos con algunos aminoácidos como la lisina) (16).
El amortiguador 1 contiene bajas concentraciones de sales como Tris-HCl y acetato de magnesio, las cuales incrementan la solubilidad de las proteínas. La principal diferencia entre los amortiguadores utilizados consistió en la presencia o ausencia de anfolitos, ya que estos, según algunos estudios, incrementan la solubilidad de las proteínas, reduciendo la interacción carga-carga, lo cual da como resultado una mayor cantidad y variedad de proteínas en la muestra (5, 6). Por lo tanto, se modificó el protocolo usual de extracción/solubilización (amortiguador 1), adicionando anfolitos de rangos de pH 4-7 y 3-10 (amortiguadores 2y 3, respectivamente).
Una vez realizada la extracción y solubilización de las proteínas, el siguiente paso fue evaluar su integridad con el propósito de garantizar un buen mapa proteico para asegurar que no hubiesen sufrido degradación, y lograr reproducibilidad en los perfiles de expresión. Para ello se empleó electroforesis SDS-PAGE (Figura 1) y los resultados se corroboraron mediante western blot. Se observó que el amortiguador 1, que contenía sales, fue el que mejor solubilizó las proteínas de cultivos primarios de tiroides, ya que en la electroforesis se obtiene mejor resolución y mayor número de bandas en comparación con los amortiguadores 2y3que contenían anfolitos, contrario a lo que varios autores sugieren (4, 7). Además, con el amortiguador 3 se observó alguna degradación de las proteínas provenientes de las muestras de tiroides normal (TN) (Figura 1a).
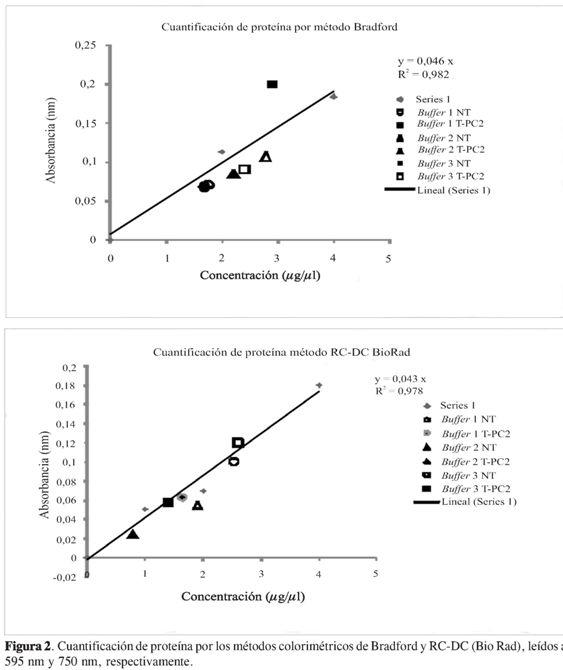
La cuantificación de las proteínas se realizó mediante los métodos de Bradford y Lowry modificado (kit RC-DC de Bio-Rad). Las proteínas deben ser cuantificadas con la mayor precisión posible, sobre todo cuando se pretenden evaluar las proteínas diferencialmente expresadas, para asegurar que las diferencias de expresión de proteínas sean debidas a la patología y no a diferencias de carga de la muestra en la electroforesis.
El método de Bradford es un método que se basa en el uso del colorante azul brillante de Coomassie (Coomassie Brilliant Blue), el cual se une a las proteínas en una solución ácida causando un cambio en la longitud de onda de absorción máxima del colorante de 465 nm a 595 nm. Para la determinación de la concentración de proteínas se realiza la lectura a 595 nm. Uno de los principales inconvenientes del método es que tiene interferencias con la urea y los detergentes utilizados generalmente en los amortiguadores de extracción de muestras para técnicas proteómicas.
El método de Lowry modificado (RC-DC de Bio Rad) es compatible con los agentes reductores y detergentes, y la lectura se realiza a 750 nm. Para la cuantificación se debe reducir el cobre, y en los amortiguadores de solubilización y extracción hay agentes reductores, lo cual podría generar interferencia, pero el método RC-DC se caracteriza por hacer solubilización diferencial de los componentes del amortiguador y posterior precipitación y resuspensión de las proteínas en la solución de cobre.
Al comparar estos métodos de cuantificación, se pudo observar que entre los dos métodos colorimétricos hay concordancia (Figura 2), pero en la mayoría de los casos los valores obtenidos por el método de Bradford fueron mayores que los dados por el RC-DC, lo que puede atribuirse a interferencias con urea, tiourea y detergentes que están en altas concentraciones en los amortiguadores de solubilización y extracción, así que por razones de las características de los diferentes procedimientos se decidió cuantificar las proteínas con el método RC-DC.
Sin embargo, se decidió corroborar los datos obtenidos por RC-DC mediante un western blot, comparándolos mediante la intensidad de las bandas (datos no mostrados). Se encontró que hay concordancia en los resultados y, por lo tanto, se realizó la cuantificación por el método RC-DC confirmado mediante western blot, lo cual permitió, además de la cuantificación de las proteínas, la verificación de su integridad después del proceso de extracción.
En la Figura 3 se observa que el amortiguador 1 fue el que evidenció una mejor reproducibilidad y número de spots, pero en este caso el voltaje final usado, 4.000 V, no fue adecuado para un correcto enfoque y migración de las proteínas (tal como se indica con las flechas en la Figura 3), también se encontró que la mayoría de las proteínas se encuentran enfocadas en un rango de pH de 4-7, en este rango de pH es donde se encuentra el mayor número de proteínas distribuidas de forma homogénea por todo el gel (Figura 3, panel A). Por el contrario, como puede verse en dicha Figura, en los geles de pH de 3-10 la mayoría de las proteínas quedan acumuladas en un rango de pH de 4-7 (Figura 3, panel B), aunque existe un grupo pequeño de proteínas que se enfocan después de pH 7, por lo cual, se recomienda para futuros trabajos no descartar el análisis de proteínas que estén en rangos por encima de pH 7.
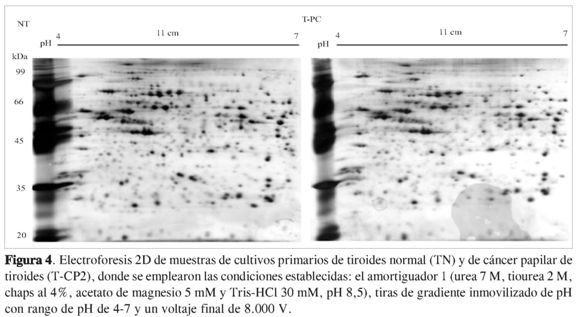
Las condiciones seleccionadas fueron: extracción de las proteínas con el amortiguador 1 (Tabla 1), métodos de cuantificación de proteínas: el colorimétrico de RC-DC confirmado mediante un western blot contra tubulina, isoelectroenfoque en gradiente inmovilizado de pH de 4-7 y voltaje final de 8.000 V. En estas condiciones se procesaron las proteínas provenientes de los cultivos de tiroides normales (TN) y de cáncer papilar de tiroides (T-CP2), ya que así se obtuvieron los geles con un mayor número de spots, resueltos, enfocados y reproducibles (Figura 4).
Para llevar a cabo el análisis bioinformático de los geles 2D con el programa PDQuest (PDQuest 7.2, Bio-Rad®), fue necesaria la realización de mínimo 3 geles 2D del mismo tipo de muestra en diferentes ensayos con las mismas condiciones.
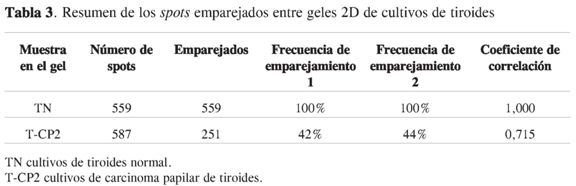
En el programa es necesaria la elección de una de las muestras como patrón, de forma que este realiza el análisis de todos los spots que hay en la muestra patrón, y a partir de esta, intenta ver las coincidencias en el resto de los geles 2D. Por tal motivo, en este estudio los geles se compararon con base en las muestras TN, puesto que estas provienen de los cultivos primarios de tiroides normal. El programa identificó 559 spots en los geles de las muestras TN, mientras que en los de las muestras T-CP2 encontró 587 spots (Tabla 3). En la Tabla 3 se resume la información obtenida con el programa de análisis sobre el número total de spots yel número de spots emparejados. La columna de frecuencia de emparejamiento 1 muestra el porcentaje de spots emparejados relativos al número total de spots en el gel. La columna de frecuencia de emparejamiento 2 muestra la frecuencia de los spots emparejados relativos al numero total de spots en el gel de las muestras TN sobre el cual se comparó; se observa que entre las muestras TN y T-CP2 solo hay 42% de spots idénticos, el resto son las proteínas que se expresan diferencialmente.
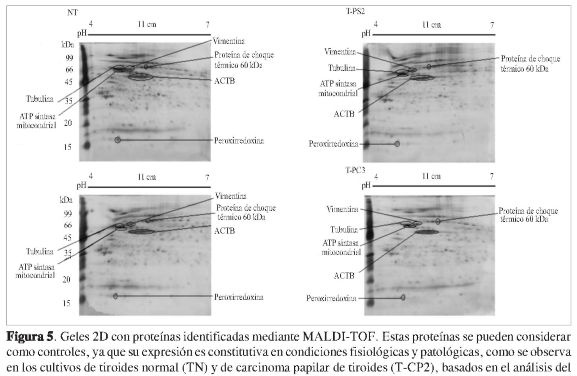
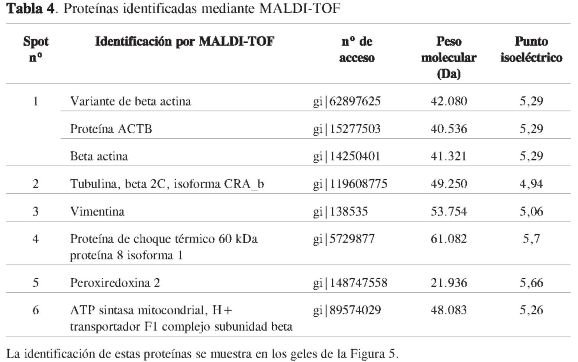
El análisis posterior mediante MALDI-TOF de ese porcentaje de proteínas idénticas permitió la identificación de proteínas control, estas proteínas generalmente se expresan de manera constitutiva y no están relacionadas con la patología. Se identificaron las proteínas que se muestran en la Figura 5 y la Tabla 4, la importancia de estas proteínas radica en que en estudios posteriores servirán para localizarlas en el gel y confirmar que los geles tienen la misma cantidad de proteína y que se han enfocado correctamente. También son importantes para asegurar que la muestra no ha sufrido degradación extra (diferente a la realizada por la digestión con las proteasas) y que la digestión con tripsina ha sido satisfactoria para ser analizada por MALDI-TOF.
Lo ideal en la selección de las proteínas control es distribuirlas por alto, medio y bajo peso molecular (como la HSP60, ACTB y peroxirredoxina, respectivamente), además de contar con algunas con pH 7. En este trabajo la mayoría de las proteínas se encuentran en un rango de pH de 4-6, indicando la preva-lencia de proteínas de carácter ácido en los cultivos celulares estudiados.
Las condiciones encontradas en este trabajo para el análisis proteómico en cultivos primarios de tiroides normales y de carcinoma papilar de tiroides, permitieron la identificación, mediante MALDI-TOF, de algunas proteínas de expresión constitutiva como vimentina, tubulina, proteína de choque térmico de 60 kDa, peroxirredoxina y la ATP sintasa mitocondrial. En estudios posteriores en la línea de investigación en cáncer de tiroides, la localización y análisis de dichas proteínas servirá como control de calidad del proceso.
AGRADECIMIENTOS
Los autores agradecen a la doctora Susana Bravo del Departamento de Fisiología, Facultad de Medicina, Universidad Santiago de Compostela, España, por su asesoría en el desarrollo de este trabajo.
REFERENCIAS BIBLIOGRÁFICAS
1. Petricoin, E. F.; Zoon, K. C.; Kohn, E. C.; Barrett, J. C.; Liotta, L. A. Clinical proteomics: translating benchside promise into bedside reality. Nat. Rev. Drug Discov. 2002. 1 (9): 683-695. [ Links ]
2. Hanash, S. Disease proteomics. Nature. 2003. 422 (6928): 226-232. [ Links ]
3. Arthur, J. M. Proteomics. Curr Opin Nephrol. Hypertens. 2003. 12 (4): 423-430. [ Links ]
4. Canas, B.; Pineiro, C.; Calvo, E.; López-Ferrer, D.; Gallardo, J. M. Trends in sample preparation for classical and second generation proteomics. J. Chromatogr. A. 2007. 1153 (1-2): 235-258. [ Links ]
5. Shaw, M. M.; Riederer, B. M. Sample preparation for two-dimensional gel electrophoresis. Proteomics. 2003. 3 (8): 1408-1417. [ Links ]
6. Molloy, M. P. Two-dimensional electrophoresis of membrane proteins using immobilized pH gradients. Anal. Biochem. 2000. 280 (1): 1-10. [ Links ]
7. De Marqui, A. B.; Vidotto, A.; Polachini, G. M.; Bellato, C. de M.; Cabral, H.; et al. Solubilization of proteins from human lymph node tissue and two-dimensional gel storage. J. Biochem Mol. Biol. 2006. 39 (2): 216-222. [ Links ]
8. Bravo, S. B.; Pampin, S.; Cameselle-Teijeiro, J.; Carneiro, C.; Domínguez, F.; et al. TGF-beta-induced apoptosis in human thyrocytes is mediated by p27kip1 reduction and is overridden in neoplastic thyrocytes by NF-kappaB activation. Oncogene. 2003. 22 (49): 7819-7830. [ Links ]
9. Bravo, S. B.; García-Rendueles, M. E.; Seoane, R.; Dosil, V.; Cameselle-Teijeiro, J.; et al. Plitidepsin has a cytostatic effect in human undifferentiated (anaplastic) thyroid carcinoma. Clin. Cancer Res. 2005. 11 (21): 7664-7673. [ Links ]
10. Coon, H.; Curcio, F.; Sakaguchi, K.; Brandi, M.; Swerdlow, R. Cell cultures of neuroblasts from rat olfactory epithelium that show odorant responses. Proc. Natl. Acad. Sci. U.S.A. 1989. 86: 1703-1707. [ Links ]
11. Li, N.; Guo, R.; Li, W.; Shao, J.; Li, S.; Zhao, K.; et al. A proteomic investigation into a human gastric cancer cell line BGC823 treated with diallyl trisulfide. Carcinogenesis. 2006. 27(6): 1222-1231. [ Links ]
12. Li, C.;Hong, Y.; Tan, Y. X.; Zhou, H.; Ai, J. H.; et al. Accurate qualitative and quantitative proteomic analysis of clinical hepatocellular carcinoma using laser capture microdissection coupled with isotope-coded affinity tag and two-dimensional liquid chromatography mass spectrometry. Mol. Cell. Proteomics. 2004. 3 (4): 399-409. [ Links ]
13. Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal. Biochem. 1976. 72: 248-254. [ Links ]
14. De Duve, C.; Beaufay, H. A short history of tissue fractionation. J. Cell. Biol. 1981. 91 (3 Pt 2): 293s-299s. [ Links ]
15. Wang, H.; Hanash, S. Intact-protein based sample preparation strategies for proteome analysis in combination with mass spectrometry. Mass SpectromRev. 2005. 24 (3): 413-426. [ Links ]
16. Wilkins, M. R.; Appel, R. D.; Van Eyk, J. E.; Chung, M. C.; Gorg. A.; et al. Guidelines for the next 10 years of proteomics. Proteomics. 2006. 6 (1): 4-8. [ Links ]