Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Química
Print version ISSN 0120-2804On-line version ISSN 2357-3791
Rev.Colomb.Quim. vol.40 no.1 Bogotá Jan./Apr. 2011
REGULACIÓN ESPACIO-TEMPORAL DE FENILALANLNA AMONIO LIASA EN CLAVEL (Dianthus caryophyllus L.) DURANTE SU INTERACCIÓN CON EL PATÓGENO Fusarium oxysporum f. sp. dianthi
SPATIO-TEMPORAL REGULATION OF PHENYLALANINE AMMONIA LYASE ENZYME IN CARNATION (Dianthus caryophyllus L.) DURING ITS INTERACTION WITH THE PATHOGEN Fusarium oxysporum f. sp. dianthi
REGULAÇÃO ESPÁCIO-TEMPORAL DA ENZIMA FENILALANINA AMÔNIO LIASA (PAL) EM CRAVO (Dianthus caryophyllus L.) DURANTE A SUA INTERACÇÃO COM O PATOGÉNICO Fusarium oxysporum f. sp. dianthi
Harold D. Ardila1,2, Sixta T. Martínez1, Blanca L. Higuera1
1 Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia, sede Bogotá. Bogotá, Colombia.
Recibido: 15/02/11 - Aceptado: 29/04/11
RESUMEN
Se estudió la regulación espacio-temporal de la actividad enzimática y de los niveles transcripcionales de la enzima fenilalanina amonio liasa (PAL por sus siglas en inglés) en clavel (Dianthus caryophyllus L.) inoculado con el patógeno causal del marchitamiento vascular. Se inocularon con Fusarium oxysporum f. sp. dianthi esquejes de dos variedades con diferencias en los niveles de tolerancia a la enfermedad -Kiss (tolerante) y Uconn (susceptible)- y se realizaron muestreos posinoculación a diferentes horas, tanto en el tallo como en la raíz, con el fin de evaluar los parámetros en estudio. Se determinó que durante la infección con el patógeno, la variedad tolerante presentó inducción de la actividad PAL en la raíz a las 48 horas de la inoculación. Esto indica que en este órgano de la planta se estimula la ruta fenilpropanoide, responsable de la generación de metabolitos fenólicos presentes en una alta diversidad de fenómenos asociados a resistencia vegetal. Considerando que durante la evaluación de los niveles de transcripción en este órgano de la planta mediante la técnica semicuantitativa de transcripción reversa y la posterior reacción en cadena de la polimerasa (RT-PCR por su nombre en inglés) no se presentó aumento importante en los niveles de mRNA para esta enzima, se evidencia la participación de mecanismos de regulación postranscripcional que determinan la generación de la enzima activa. En el tallo no se presentaron cambios en la actividad enzimática ni en los niveles transcripcionales, lo cual indica que la regulación de la respuesta de defensa de la planta está determinada por el órgano involucrado en el proceso de infección.
Palabras clave: Fenilalanina amonio liasa, clavel, Fusarium oxysporum f. sp. dianthi, defensa, Dianthus caryophyllus.
ABSTRACT
The spatio-temporal regulation of enzymatic activity and that of transcriptional levels of phenylalanine ammonia lyase enzyme in cuttings of carnation infected with the pathogen responsible of vascular wilting were studied. Carnation cuttings of two varieties which differ in disease tolerance-Kiss (tolerant) and Uconn (sus-ceptible)-were inoculated with Fusarium oxysporum f sp. dianthi and samplings were taken from root and stem at different times after inoculate them, to evaluate the parameters of interest. During infection, the tolerant variety showed an induction of PAL at 48 hours post-inoculation in roots, showing in this organ that the phenylpropanid pathway is stimulated. This pathway is responsible for producing phenolic metabolites that participate in phenomena associated with plant resistance. Having in account that during the evaluation of transcription levels in root, measured with semiquantitative technique RT-PCR, the levels of PAL mRNA were not increased, the participation of post-transcriptional regulation mechanisms for this active enzyme is evident.
Taking in consideration that stem not presented differential induction in the enzymatic activity nor in levels of mRNA, we evidenced that regulation of responses of plant defense in this model is determined by the organ of the plant involved in the infection process.
Key words: Phenylalanine ammonia lyase, carnation, Fusarium oxysporum f. sp. dianthi, defense, Dianthus caryophyllus.
RESUMO
com o fungo patogênico que causa o murchamente vascular. Inocularam-se estacas de cravo de duas variedades com diferentes niveles de tolerância à doença, Kiss (tolerante) e Uconn (susceptível) com uma Nesta investigação foi estudada a regulação espaço-temporal da atividade enzimática e dos níveis transcripcionais da enzima fenilalanina amônio liasa em estacas de cravo inoculadas cepa de Fusarium oxysporum f. sp. dianthi, e realizaram-se amostragens no caule e na raiz em diferentes horas pós-inoculação com a finalidade de avaliar os parâmetros em estudo. Foi determinado que durante a infecção com o fungo patogénico, a variedade tolerante Kiss apresenta uma indução da atividade PAL às 48 horas pós-inoculação na raiz, indicando que neste órgão da planta se estimula a via fenilpropanoide, responsável da geração de metabolitos de tipo fenólico que participam numa alta diversidade de fenómenos associados à resistência vegetal. Da mesma forma, considerando que durante a avaliação dos níveis de transcrição neste órgão da planta usando a técnica semi-quantitativa de RT-PCR não se apresentou aumento importante nos níveis de mRNA para esta enzima, se evidencia a participação de mecanismos de regulação postranscripcional que determinam a geração da enzima ativa. No caule não se apresentaram modificações na actividade enzimática, nem nos níveis de transcrição, indicando que a regulação da resposta de defesa da planta está determinada pelo órgão implicado no processo de infecção.
Palavras-chave: fenilalanina amônio liasa, cravo, Fusarium oxysporum f. sp. dianthi, defesa, Dianthus caryophyllus.
INTRODUCCIÓN
La floricultura es una de las actividades de mayor importancia económica en Colombia. En este sector, el clavel (Dianthus caryophyllus L.) es uno de los productos de mayor impacto económico y social (1). Uno de los principales factores que afectan la producción de esta flor es el marchitamiento vascular causado por el hongo Fusarium oxysporum f. sp. dianthi (Fod), patógeno ampliamente distribuido en las zonas donde se cultivan claveles en el mundo. Aunque diversos grupos de investigación han enfocado sus estudios en comprender cuáles son los fenómenos histológicos y bioquímicos involucrados en la interacción entre estos dos organismos, dichos fenómenos no han sido totalmente dilucidados y no se conocen por completo las bases bioquímicas y moleculares en que se fundamenta la defensa del clavel contra este patógeno. Diversos estudios realizados al respecto permiten proponer que la ruta fenilpropanoide, causante de la generación de bloques de construcción de estructuras de defensa (2), lignificación (3) y producción de metabolites con actividad antifúngica (4-6), puede representar un papel importante en la activación de la respuesta de defensa del clavel. En estudios con otras plantas se ha encontrado que la enzima PAL (E.C.4.3.1.5) cataliza la primera reacción en esa ruta biosintética y regula, por tanto, la generación de los metabolitos que, potencialmente, pueden participar en defensa de microorganismos patógenos (7). Esta enzima ha sido objeto de estudio en el modelo Clavel- Fod, considerando el potencial efecto regulatorio que puede presentar sobre la ruta fenilpropanoide. Se ha encontrado que, mientras elicitores de origen fúngico estimulan la inducción de actividad enzimática en el tallo, la infección directa con el patógeno no tiene ningún efecto sobre la actividad en este órgano de la planta (8). La estimulación de la ruta fenilpropanoide durante la infección es de particular interés ya que, a pesar de estas evidencias, otros estudios han demostrado que, debido a la interacción con el patógeno, se presentan cambios en la acumulación de metabolitos, productos de dicha ruta (5), sobre todo en la raíz, órgano en donde se presenta el primer contacto entre la planta y el patógeno (6). En el presente estudio se analizó la participación de la PAL con el fin de profundizar en el estudio de los mecanismos bioquímicos y moleculares de la interacción clavel-Fusarium, evaluando su actividad enzimática en cuanto a proteína y las variaciones en el mRNA en el aspecto transcripcional, tanto en el tallo como en la raíz, para dos variedades de clavel con diferencias en la tolerancia a la enfermedad durante la infección con el patógeno. De esta manera, se obtuvo información sobre la regulación espacio-temporal de las respuestas de defensa que se activan en las plantas de clavel, específicamente en lo que tiene que ver con la ruta fenilpropanoide durante la interacción con el patógeno causal del marchitamiento vascular.
MATERIALES Y MÉTODOS Material biológico
Para el presente estudio se utilizó un aislamiento de Fusarium oxysporum f. sp. dianthi -obtenido de plantas de clavel con la sintomatología típica de marchitamiento vascular-, transferido y mantenido en medio PDA (papa, dextrosa, agar). El inóculo para el ensayo in vivo se preparó a partir de este aislamiento, transfiriéndolo a medio líquido Czapek-Dox-Broth a 25 °C con agitación de 80 rpm por 3 días. Después de filtrar con gasa estéril, se preparó una suspensión de 1,0 x 106 conidias/mL. Se usaron esquejes de clavel con 15 días de enraizamiento, de las variedades Uconn (susceptible al marchitamiento vascular causado por este patógeno) y Kiss (tolerante). Este material biológico fue suministrado por la empresa América Flor S. A.
Extracción de PAL y cuantificación de actividad enzimática
La extracción y la cuantificación de la actividad PAL presente en tallos y raíces de clavel se realizó de acuerdo con estudios reportados para esta especie vegetal (8). La extracción se llevó a cabo utilizando de 0,3 a 0,4 g de material vegetal previamente lavado y secado, el cual se maceró con nitrógeno líquido y se le adicionó acetona en una relación 1:2. Se realizaron dos lavados adicionales con este solvente, para eliminar compuestos fenólicos que pueden ser potenciales inhibidores de esta enzima (9). Se adicionó amortiguador borato 100mM pH 8,8, ácido etilendiaminotetraacético (EDTA por sus siglas en inglés) 20mM y β-mercaptoetanol 18mM; posteriormente se centrifugó a 11000 x g durante 30 min. El sobrenadante se retiró cuidadosamente y se guardó a -15 oCpara someterlo a los análisis posteriores de actividad enzimática y de cuantificación de proteína por el método de Bradford (10) y la linealización reportada por Zor y Sellinger (11).
La actividad se determinó en los extractos crudos mediante cuantificación del ácido cinámico, generado del sustrato L-fenilalanina por la enzima, a 270 nm (8). Considerando que algunos estudios reportan la posible presencia de isoenzimas con patrones de expresión diferenciales en tallos y raíces (12,13), las condiciones de cuantificación variaron en función del órgano de la planta que iba a analizarse (8). Para el caso de los extractos obtenidos de tallos de clavel, se usó un ensayo discontinuo en el que 100 µLde extracto enzimático se mezclaron con 400 µL de L- fenilalanina en amortiguador borato de sodio-HCl, pH 8,0. La concentración final del sustrato en la mezcla de reacción fue 35 mM. Luego de 20 min a 37oC, la reacción se detuvo por calentamiento a 80oC durante 10 min y se adicionó 1 mL de H2O desionizada. Se realizó la correspondiente lectura a 270 nm y se calcularon los moles de ácido cinámico generados, usando una curva de calibración realizada bajo las mismas condiciones. Para el caso de los extractos obtenidos de raíces de clavel se realizaron variaciones en cuanto a la concentración final del sustrato en la mezcla de reacción (20 mM), el amortiguador para la determinación de la actividad enzimática (Na2HPO4-NaH2PO4100 mm pH 6,0) y el tiempo de reacción (20 min). Los resultados se expresaron como actividad específica (n moles ácido cinámico/min*mg proteína).
Puesta a punto para la determinación de los niveles transcripcionales de PAL por RT-PCR
Para la determinación de los niveles transcripcionales de los genes codificantes de la enzima PAL, se recurrió a la técnica semicuantitativa de transcripción reversa y posterior reacción en cadena de la polimerasa, la cual permite establecer dicha determinación mediante la comparación rápida con un gen casero, usando transcripción reversa, PCR, electroforesis y densitometría (14-16). Con el fin de realizar este análisis, se llevaron a cabo diversos ensayos que buscaban asegurar una determinación confiable y específica de los niveles de mRNA para estos genes. En primer lugar, se extrajo RNA total de tallos y raíces de clavel usando el reactivo comercial trizol® de la casa comercial Invitrogen®. Para la extracción, el material vegetal fue lavado, secado, macerado con nitrógeno líquido en morteros previamente lavados con agua tratada con una solución de pirocarbonato de dietilo (DEPC por sus siglas en inglés) y almacenado a -70 oC. Se pesaron 0,2g de talloso 0,4 g de raíz para la extracción, según la necesidad, y se siguió el protocolo recomendado por la casa comercial. La pureza y la integridad del RNA extraído fue verificada usando la relación espectrofotométrica A260/A280 nm y electroforesis en gel de agarosa en buffer de ácido 3-[N-morfolino]-propano sulfónico (MOPS por sus siglas en inglés) 1X y formaldehido, como se recomienda para RNA total (17).
Posteriormente, se llevó a cabo transcripción reversa usando extractos de RNA total provenientes de tallos y de raíces de clavel. Aproximadamente 2 /µg de RNA se sometieron a la reacción de retro-transcripción con la transcriptasa reversa de Moloney Murine Leukemia Virus (MMLV por sus siglas en inglés) de la casa comercial Invitrogen®. En este procedimiento, al RNA se le adicionaron 40 pmol de oligodT, 10 nmol de cada dNTP y se completó con H2O a 12 µL. Luego de calentar por 5 min a 65 oC y enfriar en hielo, se adicionaron 4 µL de buffer de reacción 5X (250 mM tris-HCl pH 8.3, 375 mM KCl, 15 mM MgCl2) y 2 µLde ditiotreitol 0,1 M. Después de mezclar e incubar por 2 min a 37 oC, se adicionaron 200 U de la enzima. La mezcla de reacción se mantuvo a 37 oC por 50 min y luego a 70 °C por 15 min. El cDNA se almacenó a -20 oC.
La amplificación del cDNA codificante para las enzimas PAL en clavel fue realizada usando cebadores diseñados específicamente para tal fin, con la secuencia de nucleótidos del cDNA reportada para la enzima PAL-1 del clavel (GI 11761145). El diseño tuvo en cuenta alineamientos de la secuencia de proteína PAL-1 (GI:11761146) con proteínas provenientes de otras fuentes vegetales de las familias PAL1, PAL2, PAL3 y PAL4 (12,13). Así se seleccionó un dominio que, al estar conservado en todas las PAL vegetales, permita amplificar todas las secuencias codificantes para la enzima PAL en el clavel. Es importante resaltar que los cebadores diseñados (Tabla 1) flanquean una secuencia de un intrón permitiendo realizar una selección de las muestras que presentaban contaminación con DNA. Asimismo, se diseñaron los cebadores sobre la secuencia codificante de la β-actina en clavel (GI:10121896), para amplificar un fragmento que sirvió como gen casero o gen de control para la evaluación relativa de los niveles transcripcionales de la enzima PAL (Tabla 1).
En ambos casos, los cebadores se diseñaron con el programa Primer3 (18). La especificidad de estos se verificó mediante la comparación en el programa BLAST del National Center for Biotechnology Information (NCBI por sus siglas en inglés); la no formación de dímeros se evaluó mediante el programa VectorNTI. La amplificación se verificó de manera independiente y simultánea (dúplex) para ambos fragmentos de interés, mediante PCR, usando DNA genómico del clavel y cDNA de tallos y de raíz.
La amplificación por PCR se llevó a cabo usando Taq DNA polimerasa, marca Invitrogen®, de acuerdo con las recomendaciones del fabricante, con algunas modificaciones según ensayos previos que buscaban, además de poner a punto las condiciones de amplificación, verificar que la técnica permitía evaluar de manera confiable los niveles de transcripción para PAL. Se evaluaron, por ejemplo, la concentración de MgCl2 (0,8, 1,6, 2,4 y 3,2 mM), la cantidad de cebadores (20 pmol de cada uno, 40 pmol de cada uno y la combinación 20 pmol-40 pmol para cada par de cebador) y el número de ciclos (27, 32, 37, 41 y 45). Además, se verificó que la intensidad de los amplificados después de la electroforesis estuviera relacionada con la cantidad de RNA usado en la reacción de retrotranscripción. Para ello se evaluaron diferentes concentraciones de RNA durante la realización de la RT (50 ng, 100 ng, 250 ng, 500 ng, 1 µg, 2 µg y 4 µg) y se llevó a cabo la reacción de PCR correspondiente a cada uno de los casos. Las condiciones para realizar la amplificación fueron las siguientes: 1 µL de cDNA se mezcló con 2,5 µL del buffer de reacción 10X (200 mM Tris HCl, pH 8,4 y 500 mM KCl), 2 µL de la mezcla de dNTP 10 mM, 0,8 µL de MgCl2 50 mM, 0,5 µL de cada cebador 10 µM y 0,5 µL de Taq polimerasa. Se adicionó H2O destilada estéril con el fin de completar al volumen final de 25 µL. Los tubos fueron introducidos al termociclador (Bio-rad, MJmin), donde se usó el siguiente programa de temperaturas en la amplificación del cDNA de la enzima PAL: 1 ciclo inicial de 94 oC por 3 min, 40 ciclos de (94 oC por 1 min, 60 oC por 1 min y 72 oC por 1 min) y 1 ciclo final de 72 oC por 10 min. Los productos de PCR fueron sometidos a electroforesis en gel de agarosa al 1% y tinción con bromuro de etidio, finalmente se realizó la captura de imagen y posterior densitometría. Con estos datos se realizó por duplicado la evaluación de los niveles de transcripción, expresados como intensidad fragmento PAL/intensidad fragmento actina, en cada uno de los tiempos de muestreo.
Niveles de actividad enzimática y de transcripción de la PAL determinados durante el ensayo in vivo
Con el fin de evaluar el efecto de la infección del clavel por Fod raza 2 en los niveles de actividad y transcripcionales de la enzima PAL, en el tallo y la raíz, esquejes de clavel de una variedad tolerante (Kiss) y de una variedad susceptible (Uconn) fueron inoculados por inmersión de las raíces durante 20 s en una suspensión de conidias del patógeno de 1 x 106 conidias/mL (19) y sometidos a los análisis propuestos en los diferentes tiempos posinoculación. Se dispuso un diseño experimental con cuatro tratamientos (T1: variedad susceptible control, T2: variedad susceptible inoculada, T3: variedad tolerante control y T4: variedad tolerante inoculada) y muestreos por triplicado de cada tratamiento a las 0, 6, 12, 24, 48, 96 y 192 h. Los controles fueron esquejes sometidos al mismo tratamiento, pero usando agua estéril en vez de la suspensión de conidias. Una vez inoculados, los esquejes se mantuvieron a las condiciones ambientales (18-20 °C y 70-85% de humedad relativa promedio). En cada uno de los tiempos de muestreo, para cada tratamiento, se hicieron triplicados en los procesos de extracción y duplicado en las actividades enzimáticas. El análisis estadístico de los resultados se llevó a cabo por análisis de varianza (ANOVA) con posterior asignación de grupos estadísticamente diferentes usando la prueba de Tuckey, como se ha llevado a cabo en estudios previos (8).
RESULTADOS Y DISCUSIÓN
El objetivo central de la presente investigación fue evaluar la regulación espacio-temporal de los niveles de transcripción y de actividad enzimática para PAL, enzima que regula la ruta biosintética fenilpropanoide, durante el evento de la interacción clavel-Fod. Con este propósito, se llevó a cabo un ensayo in vivo en donde se evaluaron, en una variedad tolerante y una susceptible, los niveles de mRNA y la actividad enzimática en el tallo y la raíz durante la infección con este patógeno. Se usaron los resultados obtenidos en investigaciones previas en las que se pusieron a punto las condiciones para cuantificar la actividad de esta enzima mediante métodos espectrofotométricos, tanto en tallos como en raíces de clavel (8). Es importante resaltar que a la fecha no se habían realizado estudios para evaluar niveles transcripcionales en esta planta durante la interacción con organismos patógenos en tallos y raíces simultáneamente. Se recurrió a la técnica por RT-PCR, usada recientemente en diversos estudios considerando que permite una evaluación rápida de los niveles de transcripción en interacciones planta-patógeno (14-16).
Puesta a punto para la determinación de los niveles transcripcionales de PAL por RT-PCR
Con el fin de realizar una evaluación confiable de los niveles de transcripción de los genes codificantes para la enzima PAL en clavel, considerando la ya documentada presencia de isoenzimas que se pueden expresar de manera diferencial en cada órgano del clavel (8), que de acuerdo con Yoshimoto et al. (20) pueden ser al menos cinco, fue necesario recurrir a condiciones particulares que permitieran evaluar la transcripción de todos los genes que codifican para esta enzima en la planta. Realizando la revisión correspondiente en la base de datos del NCBI (National Center for Biotechnology Information), se contaba solamente con la secuencia de proteína (GI:11761146) correspondiente a la PAL1 en clavel y la correspondiente secuencia de nucleótidos codificante (GI 11761145). Por tanto, fue necesario, para el diseño de cebadores, realizar alineamientos de la secuencia de aminoácidos de PAL1 (GI: 11761146) con secuencias de PAL reportadas en otras plantas con el fin de encontrar sobre esta una secuencia de aminoácidos que fuese conservada en todas las PAL vegetales, que permitiera, a su vez, el diseño de cebadores para realizar la evaluación simultánea de los demás genes reportados en esta planta (20). Con dichos alineamientos se encontró una secuencia altamente conservada que, al comparar con la base de datos CDD (Conserved Domains Database) del NCBI, corresponde a los dominios de tipo PAL-HAL (Phenylalanine Ammonia Lyase-Histidine Ammonia Lyase), Lyase-I-Like (Liasas del tipo I) y el dominio PAL.
Teniendo estos dominios funcionales plenamente identificados sobre la secuencia de aminoácidos PAL1, dentro de la secuencia de mRNA codificante para esta proteína (GI:11761145), se seleccionó la secuencia de nucleótidos que codificaba para estos dominios funcionales conservados. La identificación de esta permitió un diseño de cebadores que no solo amplificara un fragmento para PAL1, sino para todas las secuencias de cDNA que presenten la secuencia que codifica este dominio funcional.
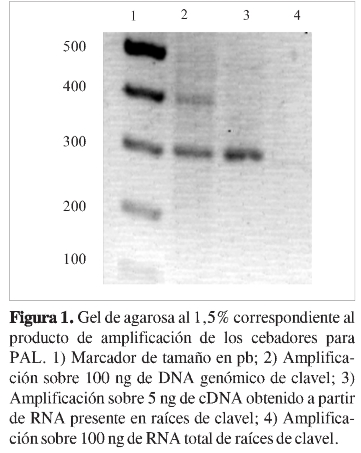
Al realizar alineamientos usando diferentes herramientas bioinformáticas, como el programa Multalin (21), de la secuencia de mRNA (GI 11761145) con otras secuencias de nucleótidos reportadas, como los genes que codifican para esta enzima en Arabidopsis thaliana (NC_003071, NC_ 003074, NC_003076) y sus correspondientes cDNA, (NM_129260.2, NM_ 115186.3, NM_ 120505.3), se pudo identificar varios miembros de las PAL (PAL3 y PAL4) que presentan un intrón conservado de aproximadamente 100 pbs, ausente en otras familias como PAL1 y PAL2. Con esta consideración, el diseño de cebadores se realizó buscando que el amplímero se encontrara en la zona conservada objetivo, hallada con los alineamientos de aminoácidos, buscando flanquear la zona en donde probablemente se encontraría dicha secuencia intrónica, en la única reportada previamente en clavel (GI 11761145). Este diseño permitió tener una herramienta para seleccionar las muestras que presentaban contaminación con DNA genómico. La presencia del intrón conservado es evidente en la Figura 1.
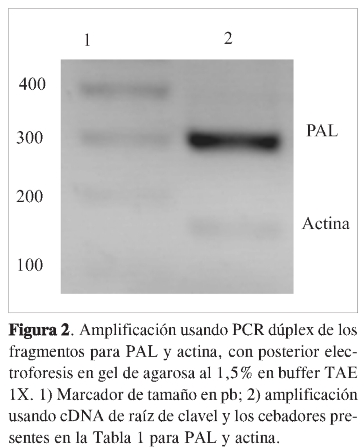
La evaluación por RT-PCR de los niveles de mRNA de la enzima objeto de estudio se realizó comparando con la expresión de un gen control, la cual no debe variar durante el proceso infectivo. Por consiguiente, era necesario tener condiciones que permitieran evaluar de manera simultánea, mediante una PCR dúplex, tanto el amplímero correspondiente a los genes de interés como el correspondiente a dicho gen de referencia. Considerando diferentes estudios que han usado el gen de la beta-actina como gen control en estudios de interacción planta-patógeno (16) y los resultados obtenidos en ensayos preliminares que indicaban la baja variación de la expresión de este gen durante la interacción clavel-Fusarium, fue empleado como gen de referencia o control para el estudio. La amplificación simultánea de estos dos fragmentos con las condiciones de PCR descritas y la posterior visualización por electroforesis fue realizada en tallos y raíces de clavel, presentando resultados iguales. Un ejemplo de dicha amplificación se presenta en la Figura 2.
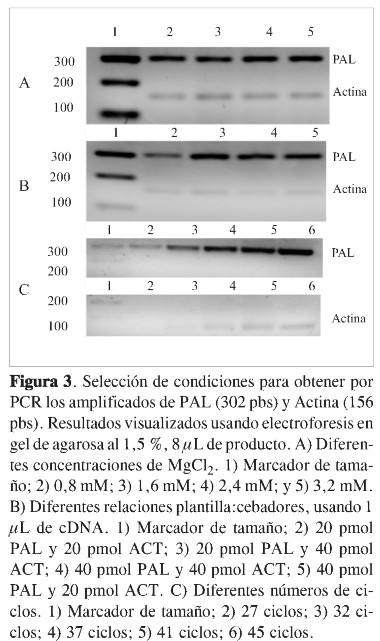
Los resultados correspondientes a la evaluación de variables como relación plantilla:cebadores, concentración de MgCl2 y número de ciclos en la PCR se presentan en la Figura 3.
Se determinó que las condiciones que permiten obtener los mejores resultados durante la amplificación corresponden a 40 pmol de cada cebador por 1 µL de cDNA, 1,6 mM de MgCl2 y 40 ciclos. En estas condiciones se determinó si existía una relación entre la intensidad de las bandas y la concentración de mRNA total usada durante la RT y la posterior PCR (Figura 4). Se determinó que efectivamente dicha relación existe desde 1 µg de RNA total inicial, lo cual indica que la técnica usada permite realizar una cuantificación semicuantitativa confiable para los niveles de transcripción de los genes codificantes de la enzima en estudio.
Niveles de actividad enzimática y de transcripción de la PAL, determinados durante el ensayo in vivo
La regulación espacio-temporal de las respuestas de defensa vegetales ha sido documentada como uno de los fenómenos determinantes de resistencia o susceptibilidad ante enfermedades causadas por fi-topatógenos. Con el fin de conocer cómo se presenta dicha regulación en el caso de los niveles de actividad y transcripcionales de la enzima PAL, se realizó un ensayo in vivo en el que las variedades de clavel Uconn y Kiss -susceptible y tolerante, respectivamente al marchitamiento vascular- fueron sometidas a inoculación con el patógeno. Estos parámetros bioquímicos y moleculares se evaluaron a diferentes horas posinoculación tanto en tallos como en raíces. Es importante citar que, con el fin de relacionar estos parámetros en estudio con tolerancia o susceptibilidad, se evaluó la incidencia de la enfermedad a los 25 días posinoculación, siendo del 88% para la variedad susceptible inoculada y del 20% para la variedad tolerante inoculada. Los controles no presentaron la sintomatología del marchitamiento vascular. Estos resultados indicaron que existían diferencias en la tolerancia a la enfermedad entre las variedades estudiadas, así como en las variables en estudio, debidas quizá a los fenotipos propios de los genotipos determinantes de susceptibilidad o tolerancia. Esta evaluación de incidencia es un punto central en este tipo de estudios, ya que permite soportar los resultados de la investigación (22).
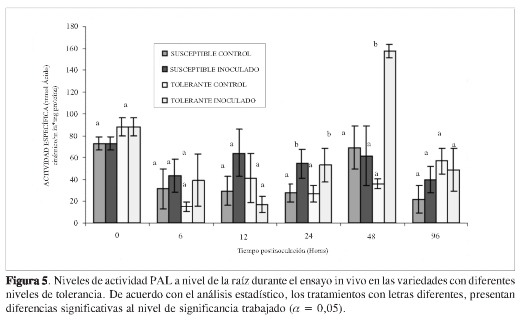
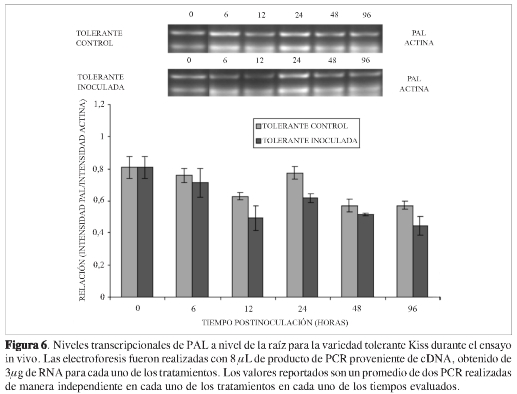
Los resultados encontrados en la raíz para los niveles de actividad de la enzima PAL se presentan en la Figura 5. Se puede observar, para la variedad susceptible, que la actividad de las raíces inoculadas y de los controles permanece igual durante los tiempos evaluados en este ensayo. Lo contrario sucede para el caso de la variedad tolerante, en donde se presenta una inducción bastante importante de la actividad de esta enzima a las 48 horas posinoculación. Los niveles de actividad en este tratamiento son estadísticamente mayores que los presentados por los otros tratamientos en el nivel de significancia trabajado (α = 0,05). Estos resultados indican que entre las respuestas metabólicas que se activan en la raíz durante el ataque del patógeno está el aumento de la actividad fenilalanina amonio liasa. Este ocurre exclusivamente en las plantas de la variedad tolerante inoculada, quizá como una respuesta temprana de los mecanismos de defensa en este órgano de la planta. Estos resultados coinciden con los obtenidos en otros estudios en los que se ha presentado también inducción en la raíz de variedades tolerantes a patógenos del género Fusarium, como es el caso de palma (23) y banana (24,25). La actividad enzimática PAL es un indicador extremadamente sensible de las condiciones de estrés y desafío del patógeno a la planta (24). Por tanto, un tratamiento en clavel con elicitores del patógeno (8) o con el patógeno directamente, como se presenta en este estudio, permite la activación de esta enzima para generar metabolitos secundarios fenilpropanoides que pueden ser precursores de moléculas de lignina o de fitoalexinas, compuestos de bajo peso molecular que presentan actividad anti-fúngica.
Con el fin de verificar si dicho aumento en la actividad enzimática tiene relación con un aumento en el nivel de mRNA, se evaluaron los niveles de transcripción para esta enzima en la raíz de la variedad tolerante (Figura 6). Se determinó que no existen variaciones significativas entre los niveles de mRNA para esta variedad en los tiempos evaluados, lo cual indica que deben existir otros mecanismos de regulación que sustenten el aumento de actividad enzimática observado en esta variedad a las 48 horas posinoculación. De hecho, se evidenció una reducción en los niveles de mRNA, la cual, considerando que se presenta tanto en controles como en inoculados, se debe muy probablemente a procesos fisiológicos propios de la planta sometida a las condiciones del ensayo. Estos resultados indican que mecanismos de regulación postranscripcional comunes en las plantas (26), como el procesamiento en el núcleo, la exportación al citoplasma, la unión al ribosoma o la estabilidad propia del mRNA, actúan modulando los niveles de enzima activa que finalmente se presentan en los tejidos de la planta. En este caso, estos mecanismos de control postranscripcional actuarían en la planta con el fin de generar la enzima activa a una velocidad superior a la que se alcanza cuando la regulación es transcripcional (27).
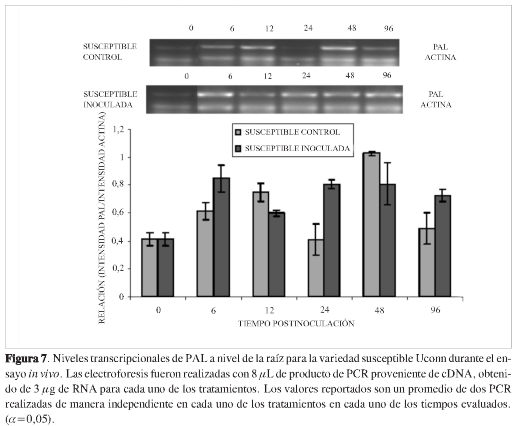
Son interesantes los resultados obtenidos para el caso de la variedad susceptible (Figura 7), considerando que existe un aumento importante en los niveles de mRNA para las plantas inoculadas a las 6, 24 y 96 horas posinoculación, el cual no está relacionado con los niveles de actividad enzimática. Estos pueden indicar que la infección con el patógeno estimula la transcripción de los genes codificantes para esta enzima, pero que otros mecanismos postranscripcionales (26-28) o supresores de mecanismos de defensa del patógeno (29) afectan la síntesis de la enzima activa. La presencia de supresores de mecanismos de defensa vegetales se ha descrito en diferentes modelos planta-patógeno; el patógeno los usa para infectar y colonizar exitosamente los tejidos vegetales susceptibles.
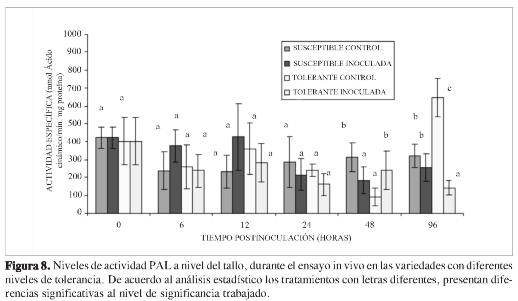
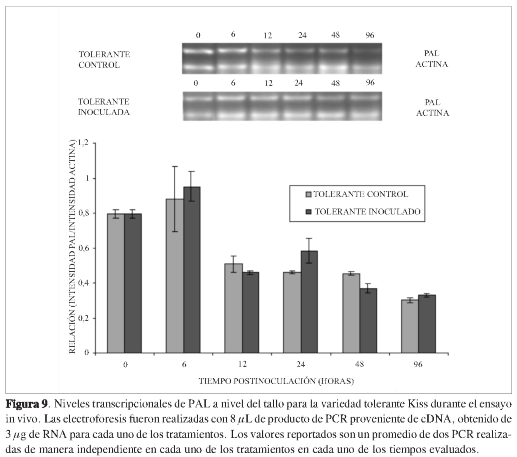
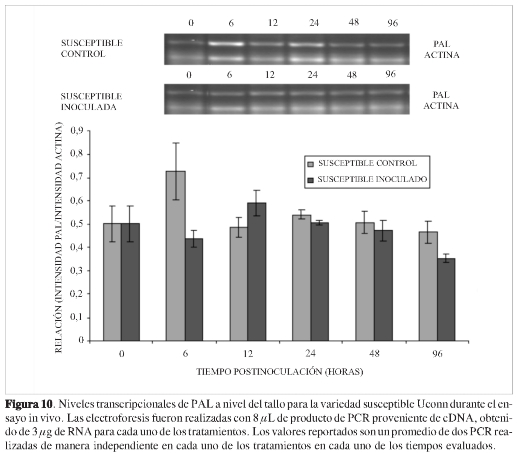
Los resultados a nivel del tallo, indican un comportamiento que difiere de lo encontrado a nivel de la raíz. Es importante notar que la evaluación de los niveles de actividad enzimática muestra que la enzima en estudio no se induce en ninguna de las variedades estudiadas (Figura 8). Esto coincide con lo encontrado en estudios anteriores en los que los niveles de actividad presentes en tallos de clavel durante la infección con el patógeno permanecen constantes durante los tiempos tempranos en los cuales se presentan los aspectos más relevantes de la respuesta de defensa vegetal (8). Al evaluar los niveles de transcripción en este órgano, tanto para la variedad tolerante (Figura 9) como para la variedad susceptible (Figura 10), permanecieron relativamente constantes durante los tiempos evaluados. Estos resultados indican que no hay estimulación de la transcripción temprana para esta enzima en este órgano de la planta durante la infección con el patógeno. Considerando que estudios previos realizados en el tallo han demostrado que se activan procesos que involucran metabolitos de la ruta fenilpropanoide, es probable que la planta use sus metabolitos constitutivos, presentes desde antes del proceso infectivo, o que activen otras rutas metabólicas alternas que puedan suministrarle los metabolitos que requiere, como se ha reportado en otros modelos (30).
En conclusión, la regulación espacio-temporal de la actividad de la enzima fe nilalanina amonio liasa durante la interacción clavel-Fusarium oxysporum f. sp. dianthi es importante dentro de los mecanismos que presenta la planta a nivel de la raíz, primer punto de contacto con el patógeno, en donde se espera que se activen algunos mecanismos determinantes de resistencia a la enfermedad. En este mismo órgano de la planta se determinó la presencia de mecanismos de regulación postranscripcional que, eventualmente, pueden actuar en la planta para generar la enzima activa y ser determinantes, por tanto, en la defensa del clavel. Asimismo, se propone la posible presencia de supresores de la respuesta vegetal, que al ser usados por el patógeno durante el proceso infectivo le permitirían colonizar los tejidos de la planta susceptible. Finalmente, se pudo determinar que con la técnica usada y a los tiempos de muestreos evaluados, no existe inducción de la PAL a nivel del tallo. Este conjunto de evidencias aportan aspectos fundamentales en el conocimiento actual de esta interacción planta-patógeno, teniendo en cuenta que la evaluación simultánea en dos órganos de la planta a diferentes horas posinoculación permiten proponer una regulación espacio-temporal de la respuesta vegetal, la cual, así como en otros modelos planta-patógeno, debe ser determinante de resistencia o susceptibilidad del clavel ante este patógeno, agente causal del marchitamiento vascular.
REFERENCIAS BIBLIOGRÁFICAS
1. Ministerio de Agricultura y Desarrollo Rural. Agenda prospectiva de investigación y desarrollo tecnológico para la cadena productiva de flores y follajes en Colombia con énfasis en clavel. 2010. Bogotá. [ Links ]
2. Baayen, R. P.; Ouellette, G. B.; Rioux, D. Compartimentalization of decay in carnations resistant to Fusarium oxysporum f. sp. dianthi. Phytopathology. 1996. 86: 1018-1031. [ Links ]
3. Baayen, R. P. Responses related to lignification and intravascular epiderm formation on carnations resistant to Fusarium wilt. Canadian Journal of Botany. 1988. 66: 784-792. [ Links ]
4. Niemann, G. J.; Van der Bij, A.; Brand-De-Boer, B.; Boon. J., Baa-yen, R. Differential response offour carnation cultivars to races 1 and 2 of Fusarium oxysporum f. sp. dianthi and to Phialophora cinerescens. Physiological and molecular Plant Pathology. 1991.38: 117-1636. [ Links ]
5. Niemann, G. J.; Van Der Kerk, A.; Niessen, W. M. A.; Versluis, K. Free and wall-bound phenolics and other constituents from healthy and fungus-infected carnations Dianthus caryophyllus L. stems. Physiological and molecular Plant Pathology. 1991.38:417-432 [ Links ]
6. Higuera, B. L. Contribución al estudio de la participación de los compuestos fenólicos en los mecanismos de la interacción Clavel Dianthus caryophyllus L. Fusarium oxysporum f. sp. dianthi. Tesis de doctorado. Universidad Nacional de Colombia. Bogotá. 2001. [ Links ]
7. Bate, N. J.; Orr, J.; Ni, W.; Meromi, A.; Nadler-Hassar, T.; Doerner, P, W.; Dixon, R. A.; Lamb, C. J.; Elkind, Y. Quantitative relationship between phenylalanine ammonia-lyase levels and phenylpropanoid accumulation in transgenic tobacco identifies a rate-determining step in natural product synthesis. Proc. Natl. Acad. Sci. USA. 1994 . 91: 7608-7612. [ Links ]
8. Ardila, H. D.; Baquero, B.; Martínez, S. T. Inducción de la actividad Fenilalanina Amonio Liasa en clavel (Dianthus caryophyllus L) por elicitores del hongo Fusarium oxysporum f. sp. dianthi raza 2. Revista Colombiana de Química. 2007. 36: 151-168. [ Links ]
9. Alunni, S.; Cipiciani, A.; Fiorini, G; Ottavi, L. Mechanisms of inhibition of phenylalanine ammonia-lyase by phenol inhibitors and phenol/glycine synergistic inhibitors. Archives ofbiochemistry and biophysics. 2003. 412: 170-175. [ Links ]
10. Bradford, M. A. A rapid and sensitivity method for detection of binding-dye proteins. Anal. Biochem. 1976. 72: 248-254. [ Links ]
11. Zor y Sellinger. Linearization of the Bradford protein assay increases its sensitivity: Theorical and experimental studies. Anal. Bioche. 1996. 236: 302 [ Links ]
12. Kao, Y. Y.; Harding, S. A.; Tsai, C. J. Differential expression of two distinct phenylalanine ammonia-lyase genes in condensed tannin-accumulating and lignifying cells of quaking aspen. Plant physiology. 2002. 130: 796-807. [ Links ]
13. Cochrane, F. C.; Davin, L. B.; Lewis, N. G. The Arabidopsis phenylalanine ammonia lyase gene family: Kinetic characterization of the four PAL isoformas. Phytochemistry. 2004. 65: 1557-1564. [ Links ]
14. Faize, M.; Faize, L.; Ishizaka, M.; Ishii, H. Expression of potential defense responses of Asian and European pears to infection with Venturia nashicola. Physiol. Mol. Plant pathol. 2005. 64: 319-330 [ Links ]
15. Kostekamp, A. Expression analysis ofdefense-related genes ingrapevine leaves after inoculation with a host and a non-host pathogen. Plant physiology and biochemistry. 2006. 44: 58-67 [ Links ]
16. Zhou, X.; Wu, F. Differentially expressed transcripts from cucumber (Cucumis sativus L.) root upon inoculation with Fusarium oxysporum f. sp. Cucumerinum. Physiol. Mol. Plantpathol. 2006. 74: 142-150. [ Links ]
17. Davis, L.; Kuehl, M.; Battey, J. Basic methods in molecular biology. 2a edición. Appleton & Lange. 1994. New Jersey. [ Links ]
18. Rozen, S.; Skaletsky, H. J. Primer3 on the www for general users and for biologist programmers. 200 In: Krawetz, S.; Misener, S. Bioinformatics Methods and protocols: Methods in molecular biology. Humana press, Totowa, NJ, pp. 365-386. [ Links ]
19. Higuera, B. L.; Montes de G, V. Contribution of HPLC to the study of the defence mechanisms acting in carnation (Dianthus caryophyllus L.) roots on infection with Fusarium oxysporum f. sp. dianthi. Journal of High Resolution Chromatography. 1996. 19: 706-708 [ Links ]
20. Yoshimoto, Y.; Higeta, D.; Ito, Y.; Yoshida H.; Hasebe, M. Y.; Oseki, Y. Isolation and Characterization of a cDNA for Phenylalanine Ammonium Liase (PAL) from carnation (Dianthus caryophyllus L.). Plant Biotechnology. 2000. 17: 325-329. [ Links ]
21. Corpet, F. Multiple sequence alignment with hierarchical clustering. Nucleic Acid Res. 1988. 16: 10881-10890. [ Links ]
22. Wise, R. P.; Moscou, M. J.; Bogdanove, A. J.; Whitham, S. A. Transcript profiling in host-pathogen Interactions. Annu. Rev. Phytopathol. 2007. 45: 329-369 [ Links ]
23. El Modafar, C.; Tantaoui, A.; El Boustani, E. Differential induction of phenylalanine ammonia-lyase activity in date palm roots in response to inoculation with Fusarium oxysporum f. sp. albedinis and to elicitation with fungal wall elicitor. Journal of Plant Physiology. 2001. 158: 715-722. [ Links ]
24. De Ascensao, A. R. C. F.; Dubery, I. A. Panama disease: cell wall reinfoncement in Banana roots in response to elicitor from Fusarium oxysporum f. sp. cubense race four. Phytopathology. 2000. 90: 1173-1180. [ Links ]
25. Subramaniam, S.; Maziah, M.; Sariah, M.; Puad, M. P.; Xavier, R. Bioassay method for testing Fusarium wilt disease tolerance in transgenic banana. Sceince Horticulturae. 2006. 108: 378-389. [ Links ]
26. Kuhlemeier, C. Transcriptional and post-transcriptional regulation of gene expression in plants. Plant Molecular Biology. 1992. 19: 1-14 [ Links ]
27. Waterhouse, P. M.; Brown, J. W. S., Browning, K. SThe RNA world in plants: post-transcriptional Control III. Meeting report. The Plant Cell. 2001. 13: 1710-1716. [ Links ]
28. Gutiérrez, R. A.; Maclntosh, G. C.; Green, P. Current perspectives on mRNA stability in plants: Multiple levels and mechanisms of control. Trend in Plant Science. 1991.4(11): 429-438. [ Links ]
29. Shiraishi, T.; Yamada, T.; Sayito, K.; Kato, T.; Toyoda, K.; Yoshioka, H.; Kim, H. M.; Ichinose, Y.; Tahara, M.; Oku, H. Suppressors: determinants of specificity produced by plant pathogens. Plant cell physiol. 1994. 35: 1107-1119. [ Links ]
30. Quiong, Y., Shi, M., Ng, J.,Wu, Y. Elicitor-induced rosmarinic acid accumulation and secondary metabolism enzyme activities in Salvia miltiorrhiza hairy roots. Plant science. 2006. 170: 853-858. [ Links ]