Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Química
Print version ISSN 0120-2804On-line version ISSN 2357-3791
Rev.Colomb.Quim. vol.40 no.2 Bogotá May/Aug. 2011
IMPLEMENTACIÓN DE TÉCNICAS SENCILLAS DE REMOCIÓN DE PROTEÍNAS MAYORITARIAS DE PLASMA SANGUÍNEO PARA ANÁLISIS POR ELECTROFORESIS BIDIMENSIONAL (2D)
IMPLEMENTATION OF SIMPLE TECHNIQUES FOR REMOVAL MAJORITY PROTEINS FROM BLOOD PLASMA AND ITS POSTERIOR ANALYSIS BY BI-DIMENSIONAL (2D)
ELECTROPHORESIS APLICAÇÃO DE TÉCNICAS SIMPLES DE REMOÇÃO DE PROTEÍNAS MAJORITÁRIAS DO PLASMA SANGUÍNEO PARA SUA POSTERIOR ANÁLISE POR ELETROFORESE BIDIMENSIONAL (2D)
Yenny Bueno2,3, Gerardo Muñoz2,3, Rodrigo Torres Sáez1,3,4
1 Escuela de Química, Facultad de Ciencias, Universidad Industrial de Santander, calle 9 carrera 27, Ciudad Universitaria, Bucaramanga, Colombia.
2 Departamento de Ciencias Básicas, Escuela de Medicina, Facultad de Salud, Universidad Industrial de Santander, calle 9 carrera 27, Ciudad Universitaria, Bucaramanga, Colombia.
3 Grupo de Investigación en Bioquímica y Microbiología, Universidad Industrial de Santander, calle 9 carrera 27, Ciudad Universitaria, Bucaramanga, Colombia.
Recibido: 27/05/11 Aceptado: 22/08/11
Resumen
El plasma sanguíneo representa una de las muestras de mayor interés en el diagnóstico- pronóstico de diversas enfermedades. Sin embargo, constituye la muestra de mayor dificultad para establecer su proteoma, debido a la presencia de interferencias y proteínas abundantes que dificultan la detección de proteínas minoritarias. Con el objetivo de mejorar la resolución de las proteínas separadas por técnicas electroforéticas, se realizó una comparación de diferentes métodos de remoción de proteínas mayoritarias del plasma de pacientes con síndrome coronario agudo, usando técnicas de precipitación con sales y solventes orgánicos a diferentes concentraciones. La remoción de proteínas del plasma tratado mediante estas metodologías se verificó por análisis proteómicos usando electroforesis de proteínas (1D-y 2D-SDS-PAGE), análisis de imágenes y técnicas de bioinformática. El análisis mostró que metodologías sencillas como el tratamiento con acetona 75% (v/v) disminuyó la concentración de albúmina, retiró las interferencias que dificultan la detección de proteínas minoritarias, aumentando la intensidad de las manchas proteicas separadas mediante electroforesis 2D, y por consiguiente, mejorando la resolución y la detección de las proteínas separadas del plasma sanguíneo.
Palabras clave: plasma sanguíneo, proteómica, síndrome coronario agudo, electroforesis.
Abstract
The blood plasma samples are one of the most interesting in the diagnosis, prognosis of various diseases. However, the sample is more difficult when setting its proteome, due to the presence of interference and abundant proteins that hinder the detection of minor proteins. To improve the resolution of proteins separated by electrophoretic techniques, we compare different methods of removal of major proteins in plasma of patients with acute coronary syndrome, by using techniques of precipitation with salts and organic solvents at different concentrations. Subsequently, the removal of plasma proteins treated by these methods was verified by proteomic analysis by using protein electrophoresis (1D-and 2D-SDS-PAGE), image analysis and bioinformatics techniques. The analysis showed that simple methods such as treatment with acetone 75% (v/v) decreased the concentration of albumin, removed the interferences that hinder the detection of minor proteins, increasing the intensity of protein spots separated by 2D electrophoresis, and improving the resolution and detection of separated proteins of blood plasma.
Key words: Blood plasma, proteomics, acute coronary syndrome, electrophoresis.
Resumo
O plasma sanguíneo representa uma das amostras de maior interesse no diagnóstico- prognóstico de diversas doenças. No entanto, constitui a amostra de maior dificuldade no momento de estabelecer o seu proteôma devido à s interferências e à s proteínas abundantes que dificultam a detecção de proteínas minoritárias. A fim de melhorar a resolução de proteínas separadas por técnicas eletroforéticas, comparamse diferentes métodos de remoção de proteínas majoritárias do plasma de pacientes com síndrome coronária agudo, usando técnicas de precipitação com sais e solventes orgânicos em diferentes concentrações. Posteriormente, a remoção de proteínas do plasma tratado por meio destas metodologias foi verificada a través da análise proteômica usando eletroforese de proteínas (1D e 2D SDS-PAGE), análise de imagens e técnicas de bioinformática. A análise mostrou que metodologias simples como o tratamento com acetona 75% (v/v) diminuiu a concentração de albumina, retirou as interferências que dificultavam a detecção de proteínas minoritárias, aumentando a intensidade dos spots de proteínas separados por eletroforese 2D, e melhorando a resolução e detecção das proteínas separadas do plasma sanguíneo.
Palavras–chave: plasma sanguíneo, proteômica, síndrome coronária aguda,
INTRODUCCIÓN
El plasma sanguíneo es una mezcla compleja de proteínas, aminoácidos, hidratos de carbono, lípidos y sales en disolución. Es la muestra humana más utilizada en el diagnóstico de enfermedades, y su proteoma es el más grande y complejo que se pueda obtener, ya que no solo se compone de proteínas plasmáticas sino que también reúne una infinidad de biomarcadores provenientes de todos los órganos y tejidos (1). En los últimos años han comenzado a usarse herramientas para descubrir e identificar nuevos biomarcadores en muestras de plasma, que podrían actuar como indicadores de enfermedades o estados fisiológicos de un paciente (2). Dentro de éstas, se destaca la electroforesis bidimensional 2D-PAGE; una herramienta útil en proteómica, ya que permite separar mezclas complejas que contienen miles de proteínas e incluso resolver aquellas que comparten propiedades físico-químicas similares (3). A pesar de la importancia del plasma desde el punto de vista clínico, sobre todo para el diagnóstico y el pronóstico de enfermedades, son pocos los estudios de expresión de proteínas (proteómica diferencial) realizados en el plasma de pacientes con síndrome coronario agudo (SCA). Además, es difícil la detección de proteínas minoritarias en el plasma humano, debido principalmente a las múltiples dificultades asociadas a la determinación de su proteoma, entre las cuales se cuenta la presencia de proteínas como albúmina e inmunoglobulinas. Estas representan aproximadamente el 70% del contenido proteico total del plasma(1, 4). Además, las proteínas plasmáticas como la albúmina pueden actuar como trasportadores o unirse a otras proteínas y moléculas de la sangre de interés diagnóstico y biomédico, tales como citocinas y quimioquinas (5). Para la remoción de estas proteínas mayoritarias, por ejemplo albúmina e inmunoglobulina G, se han utilizado métodos como cromatografía de afinidad (6, 7), precipitación por solventes orgánicos–por ejemplo, acetona, etanol o TCA– o separación por ultrafiltración (4, 8). Sin embargo, algunas de estas metodologías pueden presentar artefactos metodológicos que dificultan la separación de proteínas y generan ruido en los métodos de tinción utilizados en electroforesis. Estas dificultades muestran que una adecuada preparación de la muestra de plasma es uno de los pasos clave en la separación de proteínas por electroforesis 2D-PAGE, lo cual permite una mejor resolución de proteínas en los geles bidimensionales (9). Aunque se han realizado grandes esfuerzos en el desarrollo de diversos métodos para retirar las proteínas abundantes del plasma, los procedimientos disponibles presentan dificultades de disponibilidad y especificidad. En los métodos basados en la afinidad de la albúmina por algunos colorantes, se ha determinado que su baja especificidad permite la unión de diversas proteínas (10). Además, se ha comprobado que la albúmina presenta múltiples sitios de unión a proteínas y metabolitos, lo cual sugiere que, al retirarla del plasma, también se están perdiendo proteínas de baja abundancia (11). Se han utilizado también técnicas basadas en la unión específica de la albúmina a anticuerpos, pero su alto costo, baja capacidad de muestra y la reactividad cruzada con otras proteínas han limitado su uso (9). Por esta razón, es necesario buscar alternativas más económicas para mejorar la resolución de proteínas menos abundantes. Se ha determinado que la albúmina es soluble en solventes orgánicos y que mezclas de acetona al 80% con ácido trifluoroacético (TFA) han permitido separar la albúmina de la globulina en el suero (2). En este trabajo comparamos varios métodos de precipitación de proteínas del plasma con el objetivo de mejorar la resolución y la separación de estas mediante electroforesis bidimensional (2D) con muestras de plasmas de pacientes con SCA.
MATERIALES Y METODOS
Obtención de la muestra Las muestras de sangre fueron tomadas del plasma de un individuo con angina inestable y un paciente sano tomado como control. Los pacientes participantes en el estudio firmaron el consentimiento informado y el estudio contó con la aprobación del comité de ética del Hospital Universitario de Santander. Los pacientes analizados no presentaban antecedentes patológicos y el tipo de sangre en ambos casos fue O Rh positivo. La concentración de proteínas totales en el plasma del paciente con angina inestable y el paciente control fue 88,5 (±0,5) mg/mL y 86,1 (±0,5) mg/mL, respectivamente. La concentración de proteínas totales se cuantificó mediante el método de Bradford (12). Se utilizó 1 mg de proteína en las experiencias de remoción de las proteínas mayoritarias por precipitación por solventes orgánicos, mientras que el método de precipitación con sulfato de amonio fue realizado con 2,7 mg de proteína soluble.
Preparación de la muestra
Con el propósito de retirar proteínas abundantes y compuestos interferentes, se realizó una comparación entre diversos procedimientos: uso de un kit comercial –Aurum serum protein mini-Kit (Biorad laboratories, Hércules, CA, USA)–, precipitación con ácido trifluoroacético-acetonitrilo (TFA-A) (Sigma-Aldrich, ST.
Kit Aurum serum protein mini-kit (ASP): para usarlo, se siguieron las instrucciones de la casa comercial Bio-Rad. Precipitación con ácido trifluoroacético-acetonitrilo (TFA/ACN): 300 µL de plasma se mezclaron con 200 µL de una solución de TFA/ACN (1mL/L). Posteriormente, se centrifugó la mezcla a 10000g durante 10 minutos, se retiró el sobrenadante y se repitió el ciclo de centrifugación hasta obtener un sobrenadante claro. Finalmente, los precipitados obtenidos fueron disueltos en buffer de resuspensión (BR), compuesto por urea 8M, 3-[(3-colamidopropil) dimetilamonio]-1-propano sulfonato (CHAPS) 2%p/v y di-tiotreitol (DTT) 50 mM (Sigma-Aldrich, ST. Louis, MO, USA). Precipitación con acetona: una muestra de plasma que contenía 799,5µg de proteína fue diluida hasta 50 µL en PBS (buffer fosfato salino). Tres volúmenes de acetona fría fueron agregados a la mezcla e incubada toda la noche en hielo. Posteriormente, fue centrifugada a 10000g durante 30 min. El precipitado obtenido se dejó secar al aire y se resuspendió en buffer BR.
Precipitación con acetonitrilo: 50µl de plasma se diluyeron en 350µl de agua desionizada y 100µl de acetonitrilo. Las muestras fueron incubadas por 30 minutos y posteriormente centrifugadas a 14000g durante el mismo periodo. El precipitado obtenido se resuspendió en buffer BR.
Precipitación con sulfato de amonio (30%, 50% y 70% de saturación): se disolvieron 2mg de proteína de plasma en PBS hasta un volumen final de 500 µl, se adicionaron 88mg de sulfato de amonio hasta alcanzar un 30% de saturación. Se mezcló suavemente por 10min, se incubó durante una hora a temperatura ambiente y se centrifugó a 10000g a 20 °C durante 30min. Las proteínas en el sobrenadante fueron fraccionadas al 50 y 70% de saturación al adicionar 72 y 77mg, respectivamente. La mezcla fue tratada como se detalló. Cada precipitado obtenido fue lavado con 300µl de acetona fría al 90% y resuspendidos en 200µl de BR.
electroforesis en geles de poliacrilamida 1D-PAGe.
Las fracciones proteicas obtenidas con los diferentes tratamientos fueron evaluadas mediante electroforesis unidimensional en condiciones desnaturalizantes. El plasma tratado fue mezclado con buffer de carga que contenía Tris-HCl 0,125M pH 6,8, glicerol 20% v/v, 2-mercaptoetanol 10% p/v, SDS 4% p/v y azul de bromofenol 0,05% p/v. Posteriormente, la mezcla fue sometida a ebullición y congelación por cinco minutos. A continuación, la separación de proteínas se llevó a cabo en cámaras de electroforesis Mini-Protean (BIO-RAD, Hércules, CA, USA), usando geles de poliacrilamida SDS-PAGE al 10% p/v, preparados de acuerdo con el método de Laemmli (16). La separación se realizó a 120V durante una hora y diez minutos. Finalmente, los geles obtenidos fueron teñidos con azul de Coomassie R-250.
electroforesis bidimensional 2D-PAGe
Los procedimientos que presentaron los mejores resultados fueron evaluados mediante electroforesis bidimensional con el siguiente procedimiento: Un volumen de plasma que contenía 180µg de proteína fue disuelto en buffer de rehidratación que contenía urea 8M, CHAPS 2% p/v, DTT 50mM, anfolitos (3-10 pH) 0,2% v/v (Biorad laboratories, Hércules, CA, USA) y azul de bromofenol al 0,001% p/v. Esta mezcla fue aplicada a tiras de 7cm en un rango de pH de 4-7, conservadas en una bandeja de rehidratación durante 10 horas. Posteriormente, las muestras fueron enfocadas en un equipo Protean IEF (Biorad Laboratories, Hércules, CA, USA) durante 14 horas a 20 °C, según el siguiente programa: 30’ 300V, 3h 3500V, 1.5h 6000V (en gradiente), hasta 50000V/h totales. Una vez se equilibraron las muestras con agentes alquilantes y reductores, se realizó la separación de las proteínas de acuerdo con su masa, en cámaras de electroforesis Mini-Protean (Biorad laboratories, Hércules, CA, USA), mediante geles de poliacrilamida al 10% p/v preparados según el método de Laemmli (16). La separación se realizó mediante una fuente de voltaje PowerPac 250 (Biorad Laboratories, Hércules, CA, USA) durante 1 hora y 25 minutos a 120V.
tinción de los geles.
Se realizó la tinción de los geles con azul de Coomassie coloidal G-250 mediante el siguiente procedimiento: el gel fue incubado durante tres horas a temperatura ambiente, con agitación constante en una solución fijadora que contenía etanol 30% v/v y ácido fosfórico 2% v/v. Posteriormente, se le realizaron tres lavados de 10 minutos cada uno con agua tipo I. Manteniendo la agitación, fue incubado durante una hora en la solución de tinción compuesta por 17% p/v de sulfato de amonio, 3% v/v de ácido fosfórico y 33% v/v de metanol. A esta solución fue agregado azul de Coomassie G-250 al 0,06% disuelto en metanol. El gel permaneció en esta solución durante 20 minutos. El contraste adecuado para observar las proteínas fue alcanzado al desteñir con agua.
captura de imágenes y análisis de los resultados de electroforesis bidimensional (2D)
Después de decolorar por 14 horas, los geles fueron escaneados mediante un escáner UMAX Powerlook 2100 XL (Umax technologies, Dallas, TX, USA). Posteriormente, fueron enviados al programa PDQuest 2D software de análisis (BIORAD, Philadelphia, PA, USA), donde las imágenes fueron filtradas y normalizadas las diferencias de tinción mediante el modelo de regresión local. Las manchas identificadas fueron detectadas y emparejadas con una revisión manual pormenorizada de los emparejamientos detectados automáticamente. Finalmente, fueron establecidas las diferencias en el número y la intensidad de las proteínas identificadas en cada tratamiento.
Análisis bioinformático
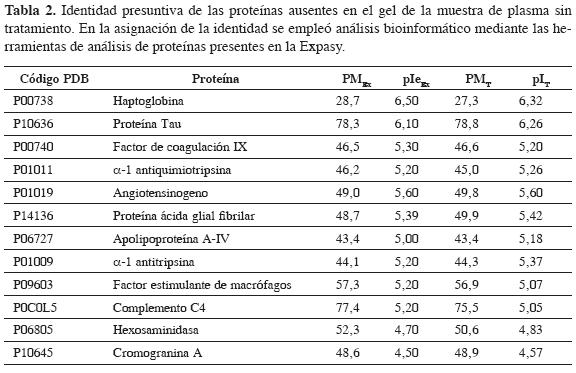
Como una aproximación para encontrar la identidad presuntiva de aquellas manchas ausentes en el gel de la muestra de plasma sin tratamiento, se organizó una base de datos con las proteínas identi ficadas en el plasma mediante análisis proteómicos, relacionadas con enfermedades cardiovasculares. Posteriormente, se determinó el peso molecular (PM) y el punto isoeléctrico (pI) de estas proteínas con la herramienta proteómica de caracterización compute pI/Mw. Estas aplicaciones están disponibles en el sistema experto de análisis de proteínas (expasy) (www.expasy.org). A continuación, se compararon los valores de PM y pI teóricos calculados mediante expasy, con los valores de PM y pI que se asignaron a las proteínas identificadas en el presente estudio. Solo fueron asignadas identidades a las proteínas que presentaban diferencias en sus valores de MP iguales a o menores de ±1,5 kDa, y diferencias en pI iguales a o menores de ±0,2 unidades con respecto a los valores de PM y pI tomados de las bases de datos. El criterio de identificación se determinó teniendo en cuenta las variaciones reportadas en las proteínas identificadas, separadas por 2D-PAGE, que se encuentran en la base de datos SWISS 2D-PAGE (base de datos de proteínas, que contiene los valores de PM y pI, encontrados experimentalmente en geles 2D-PAGE, de las proteínas identificadas por espectrometría de masas (Ms) en el mapa de proteínas del plasma humano).
Resultados Y DiscusiÓn
El plasma humano es la muestra más importante y más usada en el diagnóstico de diversas enfermedades (1). Sin embargo, es una de las muestras más difíciles de analizar por 2D-PAGE. En nuestro estudio, esta situación no fue la excepción, sobre todo debido a la gran proporción de albúmina, el amplio rango dinámico observado de abundancia de otras proteínas, la gran heterogeneidad de sus glicoproteínas predominantes (17) y las interferencias que contiene y que dificultan la separación de proteínas (9).
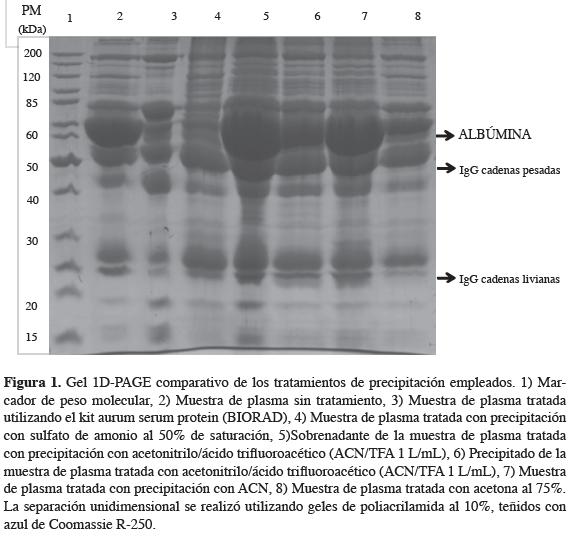
Comparación de los métodos de precipitación utilizando electroforesis unidimensional (1D-PAGE). Con el propósito de monitorear los resultados de los diversos métodos de precipitación empleados, se analizaron inicialmente las proteínas de los plasmas con tratamiento de precipitación y sin este, mediante electroforesis unidimensional (1D-PAGE) (Figura 1). Inicialmente, se realizó la separación de todos los sobrenadantes y precipitados obtenidos. Sin embargo, solo se presentan los procedimientos que mostraron disminución en los niveles de albúmina y presencia de proteínas minoritarias.
Como se observa en la Figura 1, los tratamientos de precipitación con (NH4)2SO4 al 50% p/v de saturación y con acetona al 75% v/v, representados por los patrones de proteínas presentes en los carriles 4 y 8, mostraron patrones electroforéticos similares a los obtenidos con el kit Aurum serum protein (Biorad laboratories, Hércules, CA, USA) (carril 2). Los resultados de estos tratamientos ((NH4)2SO4 50% y acetona al 75%) muestran disminución en la concentración de albúmina, mayor intensidad en las bandas separadas y presencia de algunas proteínas que no fueron detectadas en el patrón electroforético de la muestra sin tratamiento (carril 1). La reducción en los niveles de albúmina por precipitación con acetona (75% v/v) y sulfato de amonio (50% p/ v) mostró resultados satisfactorios en la eliminación de proteínas mayoritarias del plasma. En general, estas técnicas de precipitación con solventes orgánicos y sales son procedimientos simples y poco costosos, y presentan mayor estabilidad, eficiencia y manipulación (18). Sin embargo, el exceso de sales en la muestra de plasma tratada con sulfato de amonio (50% p/v) dificulta la migración de las proteínas, lo cual requeriría un procedimiento adicional de desalinización, que representa un aumento en el tiempo de análisis y en el costo del procedimiento de preparación de las muestras.
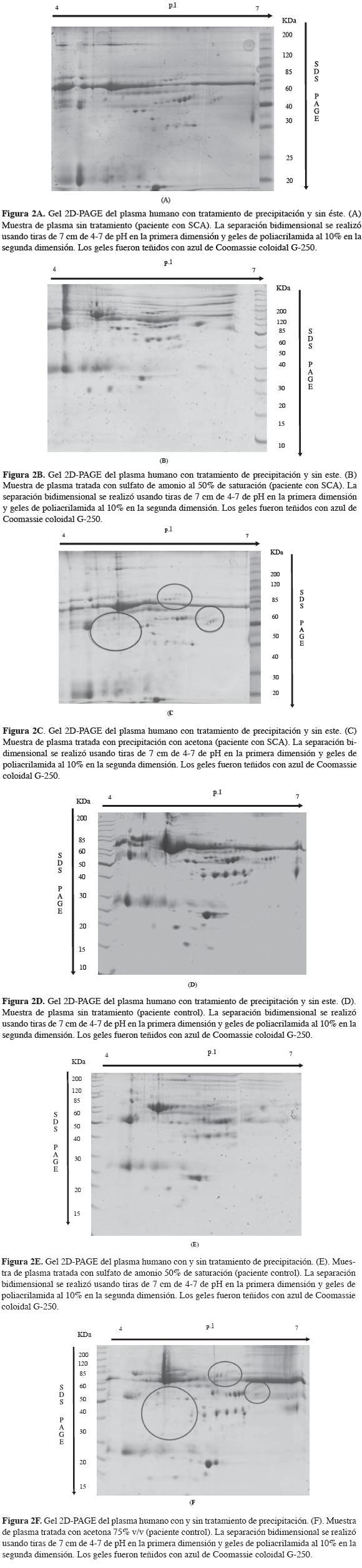
Separación de las proteínas de las muestras de plasma tratadas con los métodos de precipitación seleccionados, utilizando electroforesis bidimensional 2D-PAGE
En las Figura 2 y Figura 3 se observan las imágenes de los geles bidimensionales obtenidos para las muestras de plasma sin tratamiento y las muestras de plasma tratadas con sulfato de amonio al 50% de saturación y acetona (75% v/v), obtenidos para el paciente con SCA y el paciente control. Los geles bidimensionales 2DPAGE de las muestras tratadas con sulfato de amonio presentan aparentes dificultades en la migración de las proteínas. Se observa apiñamiento de las proteínas de pesos moleculares consecutivos, a pesar de que en todas las separaciones realizadas se usaron geles de SDS-PAGE al 10% p/v. Además, se observa distorsión de las manchas separadas y disminución en el número de proteínas detectadas. Esto puede ser producto de sales remanentes después del tratamiento, que podrían dificultar la separación de las proteínas. Se ha establecido que las sales interfieren al enfocar las proteínas en la separación por punto isoeléctrico (9) (Figura 2).
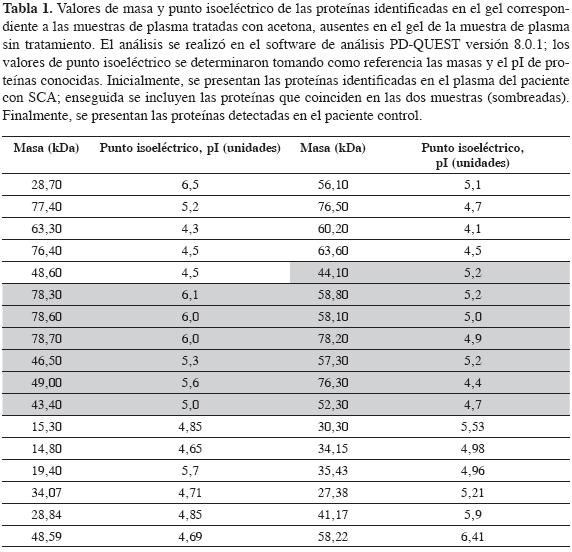
Asimismo, al observar de forma general la tinción de cada gel, los geles correspondientes a las muestras de plasma sin tratar tienen porciones más oscuras, a pesar de la uniformidad en la concentración de la solución de tinción, en los tiempos de corrido, tinción y desteñido aplicados en todos los geles. Del mismo modo, se presenta mayor ruido e interferencias en los geles correspondientes a la muestra de plasma sin tratar comparados con aquellos obtenidos para las muestras tratadas con acetona. Por consiguiente, estos últimos presentan mayor eficiencia al retirar albúmina de la muestra de plasma. Por tanto, aumenta la nitidez en las proteínas separadas y, tal como resalta en los óvalos de las Figura 2C, 2F, se identifican proteínas que no aparecieron en el gel de la muestra de plasma sin tratar. Análisis de los resultados de electroforesis bidimensional (2D) El análisis de las imágenes de la muestra original y la muestra tratada con acetona al 75% v/v del paciente con SCA, mediante el software PD-Quest Advanced (versión 8.0.1), mostró la presencia de 104 manchas, de las cuales 22 no fueron identificadas en el gel sin tratamiento y 44 manchas cuyas intensidades fueron mayores en el gel correspondiente a la muestra tratada con acetona (75% v/v). Se determinó la presencia de 18 manchas que mostraron aumentos mayores del 20% en la intensidad de sus niveles, 19 manchas cuyo aumento sobrepasa el 50% y 7 manchas en las cuales la detección fue mayor del 100%, comparadas con las intensidades detectadas de estas manchas en la muestra sin tratamiento. Al analizar las imágenes del paciente control, se identificaron 119 manchas, de las cuales 38 estaban ausentes en el gel sin tratamiento y 26 manchas cuyas intensidades fueron mayores en el gel correspondiente a la muestra tratada con acetona. Se detectaron 9 manchas que presentaban niveles de expresión aumentados en un 20%, 12 manchas cuyo aumento sobrepasa el 50% y 5 manchas cuyos niveles de expresión exceden el 100%. Estos resultados se deberían a que las proteínas del plasma podrían estar enlazándose a lípidos mediante interacciones hidrofóbicas, las cuales producen artefactos en los geles de 2D-PAGE. Este efecto sería mitigado por la precipitación de las proteínas del plasma con acetona, lo cual disminuiría estas interacciones, permitiendo una mejor separación de las proteínas del plasma. Se ha demostrado que los complejos lípidos-proteína pueden ser completamente insolubles en soluciones acuosas y, por consiguiente, podrían no migrar en la matriz de poliacrilamida (19). En la Tabla 1 se presenta la caracterización por masa y punto isoeléctrico (pI) de las proteínas que no fueron identificadas en los geles correspondientes a la muestras de plasma sin tratamiento (Tabla 1).
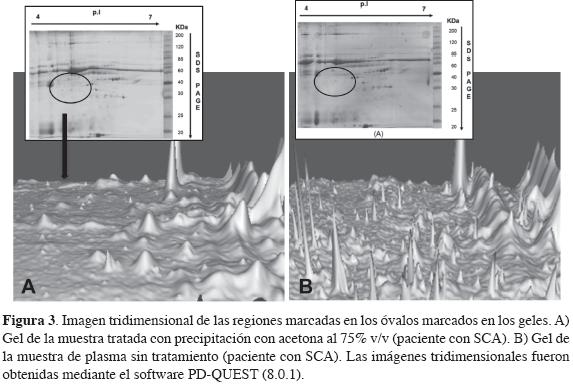
La Figura 3 presenta una imagen tridimensional de las imágenes obtenidas para la muestra precipitada con acetona y la muestra sin tratamiento del paciente con SCA. La apariencia de estas imágenes muestra el gran ruido de fondo presente en la muestra de plasma sin tratamiento, el cual solapa las proteínas de interés; mientras que la imagen obtenida al tratar la muestra con acetona (75% v/v) muestra los picos más definidos y la reducción significativa de las interferencias que producen ruido de fondo con el método de tinción usado. Estas diferencias en la apariencia de la tinción de los geles pueden ser resultado de las interacciones de los lípidos y de las impurezas con las partículas coloidales del colorante. La interacción de impurezas en la solución de tinción podría interferir en el anclaje de las moléculas de colorante a la fracción de proteína y, de esta manera, obstaculizar las interacciones hidrofóbicas de este con los residuos aromáticos del esqueleto polipeptídico (20) (Figura 3).
Análisis bioinformático
La base de datos de proteínas del plasma (HUPO) y los estudios de proteínas plasmáticas basados en herramientas proteómicas (15, 21-24) nos permitieron, mediante la comparación de sus valores de masa molecular (PM) y punto isoeléctrico (pI), con respecto a los valores teóricos establecidos para las proteínas identificadas en el proteoma del plasma, asignar la aparente identidad a las proteínas identificadas en los geles de las muestras de plasma tratadas con acetona (75% v/v), ausentes en los geles de las muestras de plasma sin tratamiento. En la Tabla 2 se muestran las identidades asignadas a aquellas proteínas cuyos valores de PM y pI cumplen con el criterio de comparación establecido en el análisis bioinformático realizado (Tabla 2).
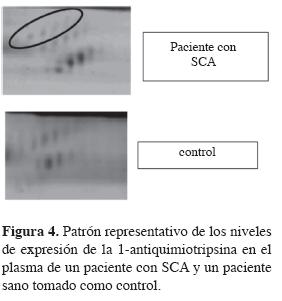
La asignación de la presunta identidad a las proteínas ausentes en los geles de la muestra sin tratamiento muestra la presencia de proteínas relacionadas con procesos como inflamación, coagulación, remodelamiento de los tejidos y metabolismo o transporte de lípidos (24-27). Estos resultados corroboran la importancia de los procedimientos de preparación de la muestra en los análisis de proteínas por 2D-PAGE. Además, se realizó la comparación del patrón de proteínas observado para el paciente con SCA con respecto al observado en el paciente control. Se determinaron diferencias claras en los niveles de expresión de más de veinte proteínas. Entre estas, sobresale un conjunto de isoformas que presentan niveles de expresión aumentados en el paciente con SCA, con respecto al control (Figura 4). El análisis bioinformático reveló que esta proteína podría corresponder a la α-1-antiquimiotripsina, serpina que inhibe la actividad de algunas proteasas (28). Estas proteínas han sido vinculadas al remodelamiento de los tejidos en los sitios de inflamación y lesión posteriores a un SCA. El aumento en los niveles de esta proteína en el paciente con SCA coincide con los resultados de otros autores (29,30). El aumento en los niveles plasmáticos de esta proteína en pacientes con SCA podría indicar su importancia como biomarcador de diagnóstico o pronóstico en los pacientes con SCA.
Los resultados muestran que la precipitación con acetona propuesta en este trabajo reduce la interferencia de la albúmina y el ruido de fondo en la tinción con azul de Comassie coloidal G-250, lo cual mejora la apariencia de las proteínas separadas. Esto permitiría identificar un gran número de candidatos a biomarcadores que podrían ser relevantes en un contexto clínico y que podrían ser validados en el plasma usando una cohorte de pacientes (21).
El tratamiento con acetona representa un procedimiento útil y sencillo en la preparación de las muestras de plasma de pacientes con SCA, previa separación de proteínas con 2D-PAGE. Esta metodología reduce las interferencias causadas por el exceso de albúmina, mejora la resolución de proteínas menos abundantes y reduce las líneas horizontales y verticales en geles teñidos con azul de Coomassie coloidal. Además, el procedimiento es de bajo costo y no requiere equipos tan especializados, lo cual permite que esté al alcance de cualquier centro de investigación.
AGRADECIMIENTOS
El grupo de investigación en Bioquímica y Microbiología (GIBIM) agradece a la Universidad Industrial de Santander y a Colciencias por el apoyo económico brindado para realizar esta investigación a través del proyecto código 1102-408- 20435.
CONFLICTO DE INTERESES
Los autores declaran que no existe ningún conflicto de interés en relación con la originalidad del trabajo y su contenido.
FUENTE DE FINANCIACIÓN
Este trabajo fue financiado por el Instituto Colombiano para el Desarrollo de la Ciencia y la Tecnología Francisco José de Caldas (Colciencias) mediante la aprobación del proyecto "Identificación de marcadores derivados del péptido natriurético pro B como predictores de mortalidad en pacientes con síndrome coronario agudo", código 1102-408-20435.
REFERENCIAS BIBLIOGRÁFICAS
1.Anderson, N.L.; Anderson, N.G. The human plasma proteome: History, character, and diagnostic prospects. Mol. Cell. Proteomics. 2002. 1: 845-867. [ Links ]
2.Chen, Y.; Lin, S.; Yeh, Y.; Hsiao, H.; Wu, Y. A modified protein precipitation procedure for efficient removal of albumin from serum. Electrophoresis. 2005. 26: 2117-2127. [ Links ]
3 Dunn, M.; Corbett, J. Two-dimensional polyacrylamide gel electrophoresis. Methods in Enzymology Academic Press. 1996. 271: 177-203. [ Links ]
4.Georgiou, J.; Har, J.; Gregory, J.; Mark, J. Proteomic analysis of human plasma: Failure of centrifugal ultrafiltration to remove albumin and other high molecular weight proteins. Proteomics. 2001.1503-1506. [ Links ]
5.Burtis, C.A.; Ashwood, E.R. Tietzfundamentals of clinical Chemistry. 5th ed. Philadelphia, PA,: W.B. Saunders Company. 2001. p. 204. [ Links ]
6.Lollo, B.; Harvey, S.; Liao, J.; Stevens, A.; Wagenknecht, R.; Sayen, R.; et al. Improved two-dimensional gel electrophoresis representation of serum proteins by using ProtoClear. Electrophoresis. 1999. 20: 854-859. [ Links ]
7. Gong, Y.; Li, X.; Yang, B.; Ying, W.; Li, D.; Zhang, Y. Different immunoaffinity fractionation strategies to characterize the human plasma. J. Proteome Res. 2006. 5: 1379-1387. [ Links ]
8. Greening, D.; Simpson, R. A centrifugal ultrafiltration strategy for isolating the low molecular weight (=25 K) component of human plasma proteome. J. Proteom. 2010. 73: 637-648. [ Links ]
9. Kim, M.; Kim, C. Human blood plasma preparation for two-dimensional gel electrophoresis. J.Chrom. 2007. 849: 203-210. [ Links ]
10. Stempfer, R.; Kubicek, M.; Lang, I.; Christa, N.; Christopher, G. Quantitative assessment of human serum high abundance protein depletion. Electrophoresis. 2008. 29: 4316- 4323. [ Links ]
11. Curry, S.; Mandelkow, H.; Brick, P.; Franks, N. Crystal structure of human serum albumin complexed with fatty acid reveals an asymmetric distribution of binding sites. Nature structural Biology. 1998. 5(9): 827- 835. [ Links ]
12. Bradford, M. A rapid and sensitive method for the quantification of microgram quantities of protein utilizing the principle of protein dye binding. Anal Biochem. 1976. 72: 248-354. [ Links ]
13. Ala-Kopsala, M.; Magga, J.; Peuhkurinen, K.; et al. Molecular heterogeneity has a major impact on the measurement of circulating N-Terminal fragments of A- and B-Type natriuretic peptides. Clin. Chem. 2004. 50: 1-13. [ Links ]
14. Jiang, L.; He, L.; Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. J. Chrom. 2004. 1023: 317-332. [ Links ]
15. Zhang, Z.; Chen, C.; Lin, L.; Chen, Y. Determination of 61 central nervous system drugs in plasma by protein precipitation-high performance liquid chromatography (Se Pu). Chin. J. Chrom. 2009. 27(6):787-793. [ Links ]
16. Laemmli, UK. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 1974. 227(5259): 680-685. [ Links ]
17. Liumbruno, G.; D Alessandro, A.; Grazzini, G.; Zolla, L. Blood-related proteomics. J. proteomics. 2010. 73: 483-507. [ Links ]
18. Kumar, A.; Yu, I.; Mattiasson, B. Precipitation of proteins: Nonspecific and specific. Lund Sweden. Marcel Dekker. 2003. [ Links ]
19. Lundblad, R. The evolution from protein chemistry to proteomics. Basic science to clinical application. Boca Raton. Taylor & Francis Group. 2006. [ Links ]
20. Candiano, G.; Bruschi, M.; Musante, L.; Santucci, L.; Ghiggeri, G.M.; Carnemolla, B. et al. Blue silver: A very sensitive colloidal Coomassie G-250 staining for proteome analysis. Electrophoresis. 2004. 25: 1327-1333. [ Links ] [ Links ]
22. Arab, S.; Gramolini, A.; Ping, P.; Kislinger, T.; Stanley, B.; Eyk, J.;et al. Cardiovascular proteomics. Tools to develop novel biomarkers and potential applications. J. Am. Coll. Cardiol. 2006. 48: 1733-1741. [ Links ]
23. Vivanco, F.; Martín-Ventura, J.; Durán, M.C.; Barderas, M.G.; Blanco- Colio, L.; Dardé, V.M. Quest of novel cardiovascular biomarkers by proteomic analysis. J. Prot. Res. 2005. 4: 1181-1191. [ Links ]
24. Anderson, L. Candidate-based proteomics in the search for biomarkers of cardiovascular disease. J. Physiol. 2005. 23-60. [ Links ]
25. Dardé, V.; Cuesta, F.; Gil, F.; Álvarez- Llamas, G.; Barderas, M.; Vivanco, F.; et al. Analysis of the plasma proteome associated with acute coronary syndrome: Does a permanent protein signature exist in the plasma of ACS Patients?J. Proteome Res. 2010. 9: 4420-4432. [ Links ]
26. Ping, P.; Vondriska, T.; Creighton, C.; Gandhi, T.; Yang, Z.; Menon,R.; et al. A functional annotation of subproteomes in human plasma. Proteomics. 2005. 5: 3506-3519. [ Links ]
27. Berhane, B.; Zong, Ch.; Liem, D.; et al. Cardiovascular-related proteins identified in human plasma by the HUPO plasma proteome Project pilot phase. Proteomics. 2005. 5: 3520-3530. [ Links ]
28. Kalsheker, N. Alpha 1-antichymotrypsin. J. Biochem. Cell. Biol. 1996.28(9): 961-964. [ Links ]
29. Kazmierczak, E.; Sobieska, M.; Kazmierczak, M.; Mrozikiewicz, A.; Wiktorowicz, K. Intense acute phase response in ischemic patients.Inter. J. Cardiol. 1999. 68(1): 69-73. [ Links ]
30. Murohara, T.; Guo, J.; Lefer, A.M. Cardioprotection by a novel recombinant serine protease inhibitor in myocardial ischemia and reperfusion injury. J. Pharmacol. Exp. Ther. 1995. 274(3): 1246-1253. [ Links ]