Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Química
Print version ISSN 0120-2804
Rev.Colomb.Quim. vol.41 no.2 Bogotá May/Aug. 2012
DISEÑO Y PRODUCCIÓN DE UNA HERRAMIENTA MOLECULAR PARA EL ESTUDIO DEL N-TERMINAL DE LA NICOTINAMIDA MONONUCLEÓTIDO ADENILIL TRANSFERASA (NMNAT) EN Leishmania braziliensis
DESIGN AND PRODUCTION OF A MOLECULAR TOOL TO STUDY OF N-TERMINAL NICOTINAMIDE MONONUCLEOTIDE ADENYLYL TRANSFERASE (NMNAT) IN Leishmania braziliensis
DESIGN E PRODUÇÃO DE UMA FERRAMENTA MOLECULAR PARA O ESTUDO DO TERMINALN DA NICOTINAMIDA CICLASE MONONUCLEÓTIDO TRANSFERASE (NMNAT) EM Leishmania braziliensis
Jesús D. Castaño1, Zuly J. Rivera 1, María H. Ramírez1, 2
1Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia, carrera 30 N.° 45-03, Bogotá, Colombia.
Recibido: 27/06/2012 – Aceptado: 13/08/2012
RESUMEN
Leishmania braziliensis es un parásito protozoario causante de la mayor parte de casos de leishmaniasis cutánea en al menos quince países del continente americano. La Organización Mundial de la Salud (OMS) ha reportado que cerca de doce millones de personas están infectadas en el mundo y que este número aumenta cada año. Debido al delicado problema de salud pública derivado de la prevalencia de esta enfermedad se hace necesario el estudio del metabolismo de este parásito. En tal sentido se ha estudiado la proteína NMNAT de este parásito, la cual es una enzima central del metabolismo de todos los organismos al estar encargada de la síntesis del NAD+, un importante cofactor en reacciones redox de procesos centrales del metabolismo celular. En la NMNAT de L. braziliensis se ha encontrado una secuencia de 44 aminoácidos en el extremo N-terminal carente de homología con la proteína del hospedero. En este estudio se produjeron anticuerpos IgG específicos contra esta secuencia, utilizando como antígenos péptidos que contuvieran la secuencia mencionada. Los anticuerpos obtenidos mostraron un reconocimiento de la NMNAT recombinante de L. braziliensis mediante ensayo por western blot.
Palabras clave: Leishmania braziliensis, NMNAT, extremo N-terminal, péptidos, anticuerpos.
ABSTRACT
Leishmania braziliensis is a protozoan which is cause of the most of the cutaneous leishmaniasis cases in at least 15 countries from America. World Health Organization (WHO) has reported that around 12 millions of people are infected in the world and this number increase every year. Because of the delicate problem of public health due to the prevalence of this disease, it is necessary the metabolism study in this parasite. In this way has been studied NMNAT protein of the parasite, which is a central enzyme of the metabolism of all organisms, since it is in charge of synthesizing NAD+, an important cofactor in oxidation-reduction reactions of central processes in the cellular metabolism. In The NMNAT of L. has been found a 43 amino acids sequence in the N terminal, which does not have homology with the protein in the human host. In this study were produced IgG antibodies against this sequence, using like antigens peptides that had the mentioned sequence. The produced antibodies recognized the recombinant NMNAT of L. braziliensis through western blot assay.Key words:L. braziliensis, NMNAT, N terminal, peptides, antibodies.
RESUMO
Leishmania braziliensis é um parasita protozoário que causa a maioria dos casos de leishmaniose cutânea em pelo menos 15 países das Américas. A Organização Mundial de Saúde (OMS) informou que cerca de 12 milhões de pessoas estão infectadas em todo o mundo e esse número aumenta a cada ano. Devido ao delicado problema de saúde pública decorrentes da prevalência desta doença é necessário estudar o metabolismo do parasita. A este respeito temos estudado a proteína NMNAT deste parasita, que é uma enzima central no metabolismo de todos os organismos de estar envolvido na produção de NAD+, um importante cofator em reações redox de processos centrais de celulares metabolismo. No L. braziliensis NMNAT encontrou uma seqüencia de 43 aminoácidos no terminal N homologia com a proteína faltando host. Este estudo produziu anticorpos IgG específicos para esta seqüência, usando como peptídeos de antígeno contendo a seqüência mencionada. Os anticorpos obtidos mostraram um reconhecimento da NMNAT L. braziliensis recombinantes por meio de julgamento por western blot.Palavras-chave: Leishmania braziliensis, NMNAT, terminal N,peptídeos,anticorpos.
INTRODUCCIÓN
L. braziliensis es un protozoario Trypanosomatida del género Leishmania, endémico del nuevo mundo y responsable de la mayoría de casos de leishmaniasis cutánea en al menos quince países del continente americano (1). Su ciclo de vida comprende una fase en el vector invertebrado y una fase en el hospedero vertebrado, donde se desarrolla la enfermedad. El vector invertebrado es un mosquito del género phlebotomus que transmite el parásito por picadura a seres humanos no infectados (2). Esta enfermedad constituye un grave problema de salud pública por los altos costos que representa a nivel psicológico, sociocultural, y económico (3). Según la Organización Mundial de la Salud (OMS) se cree que hay doce millones de personas infectadas en el mundo, y que cada año se encuentran entre uno y dos millones de nuevos casos. Dada la prevalencia de la enfermedad se hace necesario estudiar más la bioquímica del parásito, para así entender características de su metabolismo. El entendimiento de nuevos procesos puede llevar al desarrollo de blancos farmacológicos para el tratamiento de la enfermedad.
La nicotinamida mononucleótido adenilil transferasa (NMNAT; EC 2.7.7.1) es una enzima esencial en el metabolismo de cualquier organismo, debido a que cataliza el paso final de la biosíntesis de la nicotinamina adenina dinucleótido (NAD+). Las propiedades de transferencia electrónica del NAD+ y sus formas reducidas y fosforiladas (NADH, NADP+, NADPH) le dan una elevada importancia como cofactor en una múltiple variedad de reacciones redox en procesos centrales del metabolismo celular; además, los dinucleótidos participan en diversas vías de señalización celular que involucran la transferencia de electrones e hidrogeniones (4). Numerosas enzimas, particularmente deshidrogenasas, utilizan el NAD+ como cofactor en reacciones catabólicas de óxido-reducción, como la glicólisis, la oxidación de ácidos grasos, el metabolismo del nitrógeno, y en procesos anabólicos como la gluconeogénesis, la síntesis de aminoácidos y colesterol (5).
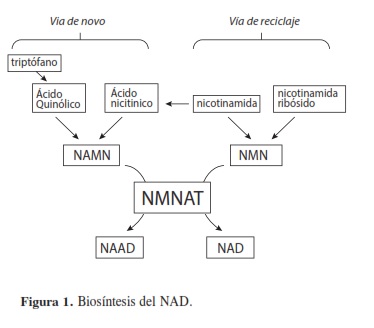
Estudios recientes han dado a conocer funciones diferentes para el NAD+, encontrándose que la molécula se halla involucrada en reacciones que implican modificaciones covalentes de proteínas como mono ADP-ribosilación, poli ADP-ribosilación y de acetilación de proteínas catalizadas por ADPribosiltransferasas (ART), poli-ADPribosa polimerasas (PARP) y sirtuinas respectivamente (6). Estas reacciones afectan de manera importante funciones asociadas con procesos fisiológicos y patológicos como la regulación transcripcional, los mecanismos de reparación del ADN (7), la conservación telomérica, la necrosis, la apoptosis (8) y la regulación del ciclo celular (9). A su vez, el NADP+ está involucrado en procesos de movilización de calcio, al ser precursor de los mensajeros de calcio, ADP-ribosa cíclica y el NAADP. Estas moléculas son importantes para una variedad de eventos de señalización celular como la regulación intracelular de insulina, la activación de células T y la secreción de catecolamina (8, 10). La biosíntesis de la coenzima puede ser llevada a cabo por la vía de novo o por las vías de reciclaje del NAD+ (11) (Figura 1). Cualquiera de las dos vías da productos que convergen a una reacción catalizada por la NMNAT, enzima que cataliza la condensación de ATP con el mononucleótido del ácido nicotínico (NAMN) o con la nicotinamida mononucleótido (NMN). Los productos de la reacción son el NAD+ o el ácido nicotínico adenindinucleótido (NAAD), dependiendo del organismo. La reacción tiene lugar vía un ataque nucleofílico del grupo 5’ fosfato del mononucleótido sobre el α-fosfato del ATP, liberando el dinucleótido y PPi (11, 12, 13) (Figura 2).
Desde que se demostró la existencia de la NMNAT se han realizado diversos estudios con el fin de caracterizar esta enzima; su actividad se ha reportado relacionada con la síntesis del DNA durante el ciclo celular (14, 15). Estudios sobre la NMNAT en humanos han mostrado la presencia de tres isoformas con localizaciones diferenciadas en el núcleo, el aparato de Golgi y la mitocondria, lo que ha sugerido la no redundancia de la proteína y ha resaltado la importancia de los procesos involucrados con el NAD+ en cada uno de los compartimentos (16, 17). Además la enzima exhibe interacciones específicas con la poli (ADP-ribosa) polimerasa (PARP1) y parece estar regulada por fosforilación (18).
En el caso de la NMNAT de L. (LbNMNAT) se han encontrado diferencias relevantes entre la enzima del parásito y la enzima del hospedero humano (19); entre ellas la presencia de un extremo amino terminal con una longitud de 44 aminoácidos carente de homología con proteínas conocidas (20). Se presenta en este caso la oportunidad de emplear esta secuencia para la producción de anticuerpos que específicamente puedan reconocer esta proteína en el parásito y sirvan como una herramienta de estudio de esta secuencia N-terminal, para determinar su posible relevancia en la actividad de la proteína, así como su posible inclusión como blanco farmacológico en el tratamiento de la leishmaniasis. Para tal efecto se propuso la síntesis de péptidos que contuvieran parte de la secuencia de aminoácidos exclusiva de la NMNAT de L. braziliensis.(1MLSSTAAPYALRTDKLKPLEGYAASSPTSTAEAASQVTTPLLQ44P), con el fin de utilizarlos en la producción de anticuerpos. La estrategia empleada para la síntesis de los péptidos fue la síntesis orgánica en fase sólida, usando la estrategia Fmoc/tBu. Los péptidos resultantes fueron caracterizados por cromatografía líquida de alta eficiencia en fase reversa (RP-HPLC) y espectrometría de masas. Los anticuerpos producidos fueron evaluados para determinar los parámetros adecuados de detección, como dilución del anticuerpo, y cantidad de proteína a cargar para la detección.
MATERIALES Y MÉTODOS
Reactivos
La resina Rink amide, los aminoácidos protegidos, N,N‘-diciclohexilcarbodiimida (DCC), 1-hidroxibenzotriazol (HOBt), tetrafluoroborato-o-benzotriazol-il- N,N,N,N-tetrametiluronium (TBTU) y el triisopropilsilano (TIS) fueron adquiridas de AAPPTec (Louisville, USA). Los reactivos N,N-diisopropiletilamina (DIPEA), anhídrido acético, piperidina, piridina, ninhidrina, fenol, KCN, N,N-dimetilformaamida (DMF), Triton X-100, etanoditiol (EDT) y el etanol absoluto, fueron adquiridos de PANREAC (Barcelona, España). El diclorometano (DCM), acetonitrilo (ACN), alcohol isopropílico (IPA) y ácido trifluoroacético (TFA) de Honeywell Burdick & Jackson ®. Las columnas de extracción en fase sólida empleadas fueron las Supelclean ® LC-18 (Supelco, Sigma-Aldrich; St. Louis, MO, USA)
Síntesis, caracterización y purificación
La síntesis química de los péptidos fue realizada manualmente en fase sólida, empleando la estrategia Fmoc/tBu. La metodología fue adaptada de (21) y, en términos generales, se realizaron los siguientes pasos:(i) La resina Rink amide (200 mg, sustitución 0,66 meq/g) se sometió a un proceso de hinchamiento por tratamiento con DMF por 2 h, con agitación constante y a temperatura ambiente. (ii) La remoción del grupo protector Fmoc de la resina se realizó por tratamiento con solución de desprotección (piperidina 25 % (v/v), tritón 1 % (v/v) en DMF), dos veces, con agitación constante a temperatura ambiente por 10 min. Luego la resina fue lavada extensivamente empleando DMF (4×), IPA (4×) y DCM (2×). Después de este tratamiento los grupos amino quedaron libres para permitir la incorporación del primer aminoácido. (iii) Luego se realizó la reacción de acople del aminoácido correspondiente; para tal fin se emplearon cuatro excesos de aminoácido respecto a la resina, que fueron preactivados empleando aminoácido protegido/DCC/ HOBt (1/1/1) disueltos en DMF y se dejaron reaccionar por 15 min a temperatura ambiente. Posteriormente, se trató la resina con la solución de aminoácido activado para permitir la formación del enlace peptídico. La solución de aminoácido fue removida por filtración y la resina lavada extensivamente. La reacción fue monitoreada y, cuando se detectaron grupos amino libres, se procedió a tratar la resina nuevamente con aminoácido activado, esta vez usando dos excesos de aminoácido protegido/TBTU/HOBt/DIPEA (1/1/1/3). La reacción se dejó con agitación constante por 4 h a temperatura ambiente. (iv) La reacción de acople fue monitoreada cualitativamente usando el reactivo de ninhídrina, así a una fracción de la resina-péptido seca se le adicionaron las soluciones A (40 μl) y B (20 μl). Una vez la mezcla fue homogenizada se sometió a calentamiento a 105 °C por 5 min y se observó el color generado por la reacción. Una coloración azul era indicativa de la presencia de grupos aminos libres y la coloración amarilla correspondía a prueba negativa. La solución A está compuesta por 40 g de Fenol en 10 ml de etanol absoluto, 1 ml del stock de KCN (65 mg de KCN/100 ml de agua) y 50 ml de piridina, y la solución B contenía 1,25 g de ninhídrina en 25 ml de etanol absoluto. (v) Luego se desprotegió el grupo α-amino para obtener los grupos alfa aminos libres disponibles para el acople del siguiente aminoácido y se realizó el monitoreo de la reacción de desprotección usando el reactivo de ninhídrina. Esta serie de reacciones (ii-v) se realizaron hasta incorporar todos los aminoácidos. (vi) Se desprotegió el grupo amino terminal de cada péptido y se secó la resinae procedió entonces (vii) a retirar los grupos protectores de las cadenas laterales y desanclar el péptido del soporte sólido, para esto la resina-péptido (1g/10 ml) fue tratada con un coctel, que contenía TFA/TIS/EDT/H2O (93/2/2,5/2,5 % v/v), por 6 h con agitación constante y temperatura ambiente. Después de este tratamiento la resina fue filtrada y el péptido precipitado por adición de éter etílico frío y se lavó cinco veces con el éter y se secó el producto.(viii)Los productos de síntesis fueron analizados en una columna ECLIPSE C18 (Agilent 4,6 × 150 mm, 3,5 μm) usando un cromatógrafo líquido Agilent 1260 (Omaha, Nebraska, USA) con detector UV-vis (210 nm). Los solventes empleados para el análisis fueron agua 0,05 % TFA (solvente A) y acetonitrilo -0,05 % TFA (solvente B); se aplicó un gradiente lineal de 0 % hasta 70 % de B en 45 min a un flujo de 1,0 ml/min.
(ix) Los péptidos fueron purificados por extracción en fase sólida (SPE) usando columnas Supelclean® LC-18 de 1 g de soporte. Las columnas fueron activadas antes de usar con 30 ml de acetonitrilo (0,05 % TFA) y luego equilibradas con 30 ml de agua (0,05 % TFA). El método de purificación consistió en absorber 10 mg de péptido crudo en la columna y eluir con soluciones que tenían diferente concentración de acetonitrilo; cada fracción fue de 3,0 ml. Las fracciones fueron analizadas por RP-HPLC analítica según el método descrito anteriormente. Las fracciones que contenían el péptido puro fueron mezcladas y liofilizadas.(x) Se determinó la masa molecular de los péptidos mediante espectrometría de masas con ionización por desorción asistida por matriz (MALDI) usando un analizador de tiempo de vuelo (TOF); como matriz fue empleado el ácido alfa-ciano- 4-hidroxicinámico (CHCA). El equipo utilizado fue un espectrómetro de masas Bruker Daltonics Ultraflex TOF.
Oxidación de los péptidos
El procedimiento seguido se adaptó de (22). La muestra liofilizada se oxidó para obtener polímeros que contuvieran la secuencia peptídica; así, 1,5 mg de péptido puro se disolvieron en 150 μl de NH4HCO3 20mM (pH 7,6) y 10 μl de DMSO (oxidante). La mezcla se dejó en agitación durante aproximadamente 30 h. Una vez transcurrido este tiempo se adicionaron 4 ml de agua/TFA 0,05 % para detener la reacción. El proceso de formación del polímero se siguió por RP-HPLC, empleando el método antes descrito. Una vez culminada la oxidación, el DMSO de cada muestra fue retirado por extracción en fase sólida. El péptido oxidado fue entonces liofilizado y se disolvió en PBS, para dar una solución de 1 μg/μl.
Obtención de los anticuerpos
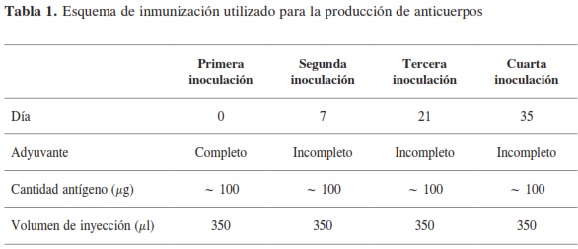
La obtención de anticuerpos policlonales contra las secuencias peptídicas de la proteína LbNMNAT sintetizadas se realizó empleando protocolos estandarizados en trabajos previos en el grupo de investigación. Se emplearon ratones hembra Mus musculus BALB-C de seis semanas de edad para realizar inoculaciones intraperitoneales. Se utilizaron tres individuos por cada péptido. El programa de inoculación empleado se describe en la Tabla 1. Las emulsiones antigénicas se prepararon con adyuvante completo de Freud para la primera inoculación, y con adyuvante incompleto de Freud para las tres inoculaciones restantes como sigue: se tomaron 100 μl de la solución de péptido polimerizado preparada previamente para obtener una cantidad total de 100 μg de péptido en el inóculo, a continuación se completó a 175 μl con PBS; a esta solución se adicionaron ~175 μl del adyuvante correspondiente para obtener una mezcla 1:1 de adyuvante y solución de antígeno. Se emulsificó esta mezcla con vortex durante 15 min. Como control de la inmunización se inoculó PBS mezclado con el adyuvante apropiado, policlonales contra las secuencias Nterminales de la proteína LbNMNAT en ratones
Preparación de los antisueros
Una semana después de la cuarta inoculación se extrajeron 600 μl de sangre del seno orbital de cada individuo. Estas muestras se incubaron a 4 °C durante 3 h para permitir la coagulación, posteriormente se centrifugó a 5000 rpm por 20 min a 4 °C. Se separó el sobrenadante, se suplementó con glicerol al 10 % (v/v) y se alicuotó el suero en viales de 0,6 ml. Estos sueros se almacenaron a -80 °C.Western blot
Se realizó la separación electroforética de la proteína LbNMNAT mediante SDS-PAGE en un gel discontinuo al 10 % deacrilamida, el cual se preparó de la siguiente manera: gel separador, 2,5 ml de una solución de acrilamida 30 % bisacrilamida 0,8 %, 3,12 ml de agua desionizada, 1,88 ml de buffer separador 4x pH 8,8 (1,5 M Tris-HCl pH 8,8, 0,4 % SDS), 25 μl de una solución de persulfato de amonio 10 %, y 5 μl de TEMED. Esta solución se sirvió en el casete y se le adicionaron 200 μl de etanol 70 %, después de lo cual se dejó polimerizar por aproximadamente 1 h; posteriormente, se descartó el etanol. Una vez terminado este proceso se preparó el gel concentrador así: 320 μl de una solución de acrilamida al 30 %, bisacrilamida 0,8 %, 1,5 ml de agua desionizada, 620 μl de buffer concentrador 4x (0,5 M Tris-HCl, pH 6,8, 0,4 % SDS), 12,5 μl de una solución de persulfato de amonio 10 % y 2,5 μl de TEMED. Esta solución se sirvió sobre el gel separador y se colocó un peine para marcar los pozos. Se dejó polimerizar por cerca de 30 min.
El gel polimerizado se colocó sobre una cámara de corrida electroforética, la cual se llenó con buffer para electroforesis (Tris-base, Glicina y SDS). A continuación se sembró en los pozos 2,5 μg de proteína recombínate NMNAT purificada., y se corrió el gel a una diferencia de potencial de 100 V. Luego de la electroforesis, las proteínas se transfirieron hacia una membrana de PVDF, la cual fue previamente activada por inmersión de esta en metanol durante 30 segundos, seguido de inmersión en agua desionizada y buffer de transferencia (192 mM glicina, 44 mM Tris y 10 % (v/v) metanol) por 3 minutos. Se armó el casete que pone en contacto el gel con la membrana de PVDF y se sumergió en una cámara de transferencia llena con este buffer. La transferencia se realizó toda la noche a 20 V. Una vez terminada, esta se verificó ésta haciendo tinción con rojo ponceau durante un minuto. La detección inmunológica se realizó utilizando como anticuerpo primario diferentes diluciones (1:50, 1:100, 1:200, 1:500) del suero obtenido en TBS, el anticuerpo secundario fue un anti-ratón peroxidasa (Anti-mouse IgG, HRP adquirido de Sigma) una dilución (1:1000). La detección cromogénica de los complejos inmunológicos se realizó usando el sustrato de la peroxidasa de rábano en la siguiente preparación: 5 ml de TBS, 1 ml de 4-cloronaftol, y 5μl de H2O2. El suero no relacionado (1:500) y el anticuerpo monoclonal contra la etiqueta de 6 histidinas (mAb to 6x His tag® adquirido de Abcam) (1:2500) se utilizaron como controles negativo y positivo respectivamente. La membrana se bloqueó con TBS-leche 2 % durante 1 h, posteriormente se incubó con el anticuerpo primario durante 3 h, seguido de tres lavados con TBS-leche 2 % de 10 min cada uno. A continuación se incubó 1 h con el anticuerpo secundario, y se lavó con TBS-leche 2 % 3 veces, de 10 min cada lavado. Finalmente se reveló empleando el sustrato de la peroxidasa, el cual se incubó con la membrana durante 10 min en la oscuridad.
RESULTADOS Y DISCUSIÓN
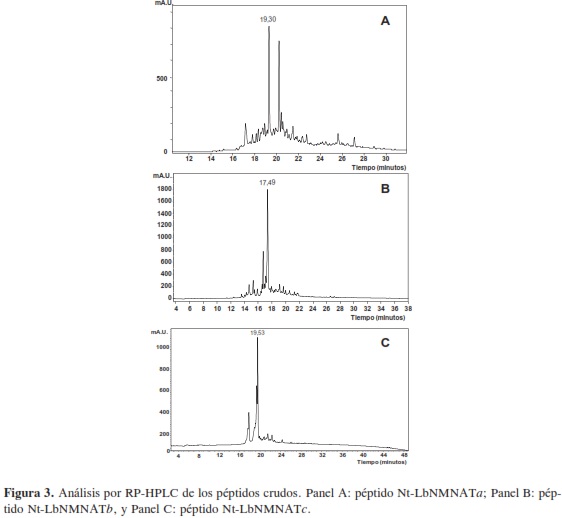
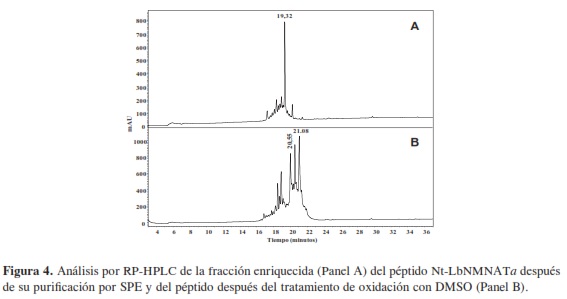
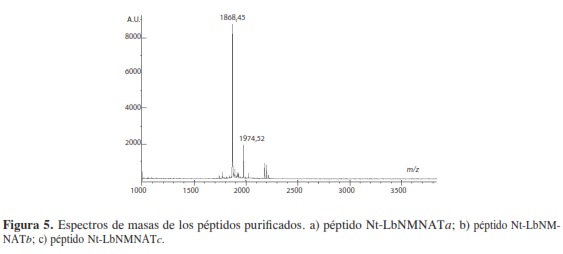
Utilizando la secuencia N-terminal de la proteína NMNAT de L. braziliensis, 1MLSSTAAPYALRTDKLKPLEGYAASSPTSTAEAASQVTTPLLQ44P, se diseñaron tres péptidos, dividiendo toda la secuencia en fragmentos peptídicos de catorce y quince aminoácidos. Para evitar el uso de una proteína transportadora al momento de la inoculación de los péptidos en el modelo animal seleccionado, se sintetizaron las cadenas con la adición de un espaciador en cada extremo, glicina (G), seguido de cisteína (C), con el fin de usar este último para la formación de puentes disulfuro entre cadenas sucesivas, de tal forma que se obtuviera un polímero, capaz de inducir reacción inmunológica por sí mismo. Los péptidos diseñados son listados en la Tabla 2. La síntesis de los péptidos llevada a cabo mediante la estrategia Fmoc/tBu mostró ser eficiente en la producción mayoritaria del péptido de interés, de acuerdo con el análisis de espectrometría de masas realizado al producto principal de cada síntesis. Es importante mencionar que la duración del proceso de síntesis depende directamente de las estrategias de acople empleadas; así se observó que para la adición secuencial de aminoácidos luego del duodécimo residuo a la cadena peptídica, la estrategia que empleaba AA/ DCC/HOBt no resultaba efectiva. En este punto, la extensión de las cadenas y las cercanías de los diferentes puntos de síntesis en la resina pueden permitir que se de interacción intercatenaria, y que exista plegamiento y formación de estructuras secundarias que dificulten la adición de los aminoácidos siguientes a las cadenas (23); aunque es normal que ocurra esto en cadenas altamente hidrofóbicas y en resinas del mismo tipo que no presenten suficiente hinchamiento, la renuencia a la adición del siguiente aminoácido planteó la necesidad de considerarlo como una opción, por tal motivo una estrategia fue la adición de una pequeña cantidad de detergente (Triton X-100) con el fin de romper dichas estructuras, así como el empleo de DCM en la mezcla de reacción, debido a la capacidad de este solvente para hinchar la resina, lo que en principio permitiría una mayor accesibilidad del aminoácido entrante a la cadena anclada a la resina. Durante la síntesis se observó que dichas estrategias tendían a mejorar la eficiencia de los acoples, sin embargo el resultado no fue lo suficientemente bueno, extendiendo demasiado el tiempo de síntesis del péptido. Con el fin de mejorar la reactividad del aminoácido entrante se utilizó la estrategia de acople AA/TBTU/HOBt/DIPEA, en este caso el DIPEA mejoraba la reactividad del grupo carboxilo, desprotonándolo y haciéndolo más reactivo hacia la generación de la forma activada del aminoácido por reacción con el TBTU, pues el medio de reacción que contenía DCM permitía el hinchamiento de la resina, como se mencionó anteriormente. Esta variación en la estrategia de acople permitió adicionar los aminoácidos restantes, mejorando la eficiencia de la síntesis por lo que es recomendable, para estas secuencias en particular, el uso de esta estrategia de acople cuando los aminoácidos excedan los doce residuos.En los perfiles cromatográficos obtenidos para los péptidos crudos se observa la presencia de varias especies, diferenciándose claramente una principal (Figura 3); esta especie fue enriquecida mediante extracción en fase sólida (SPE), usando un gradiente de elución, ; las fracciones recolectadas fueron analizadas por RP-HPLC, reunidas y liofilizadas; a manera de ejemplo en la Figura 4A se muestra el análisis de la fracción enriquecida del péptido Nt-LbNMNATa. En la Figura 3C el cromatograma del péptido Nt-LbNMNATc muestra la presencia de un pico muy cercano al pico principal difícil de resolver, el cual puede corresponder a un producto deshidratado. Ya que los picos eluyen muy cerca se utilizó la fracción que contenía las dos especies para los procedimientos posteriores, obteniéndose buenos resultados como se observa más adelante, justificando la elección.
Las especies poliméricas necesarias para la inmunización de los ratones fueron obtenidas por oxidación con DMSO de las fracciones enriquecidas de cada péptido. La reacción fue monitoreada por RP-HPLC, analizando la reacción después de las primeras 7 h de reacción. En la Figura 4B se muestra a manera de ejemplo y de forma comparativa el perfil cromatográfico del péptido Nt-LbNMNATa después de la reacción de oxidación. Es clara la formación de varias especies correspondientes a las diferentes posibilidades de reacción; estas aparecen a un mayor tiempo de retención, lo que es consecuente con la formación de un polímero que interactuará más con la columna por fuerzas intermoleculares de Van der Wals, que son de mayor intensidad en moléculas de alto peso molecular.
Cabe aclarar que los las fracciones purificadas de los péptidos fueron analizadas antes del proceso de oxidación mediante espectrometría de masas MALDI TOF para verificar que correspondían a la especie deseada. En la Figura 5 se muestra uno de estos análisis y en la Tabla 2 se resumen los datos de los análisis de todos los péptidos.
Análisis de los anticuerpos generados a partir de los péptidos sintetizados
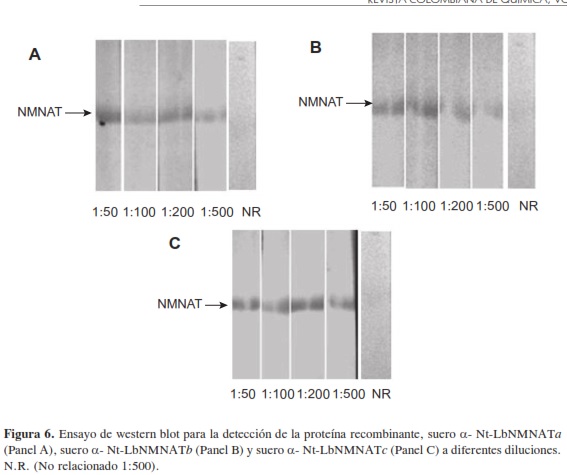
En la Figura 6 se observa el ensayo de inmunodetección realizado con los anticuerpos provenientes del ratón inoculado con los péptidos Nt-LbNMNATa, Nt-LbNMNATb y Nt-LbNMNATc. En los tres casos se tiene un resultado positivo para la detección de la proteína recombinante LbNMNAT. Se analizaron diferentes diluciones del anticuerpo y se encontró que para la dilución 1:500 se observaba una señal claramente identificable, en tanto para el suero no relacionado no se observaba una señal, lo que permite establecer que esta dilución es adecuada para obtener buenos resultados sin la presencia de falsos positivos debidos a interacciones inespecíficas entre proteínas. Los ensayos realizados con diferentes cantidades de la proteína recombinante y diferentes diluciones del anticuerpo permitieron determinar que para observar un reconocimiento de la proteína con el anticuerpo es necesario cargar al menos 2 μg de proteína.
Los resultados obtenidos sugieren que los anticuerpos obtenidos empleando los polímeros de péptidos con secuencias de la proteína NMNAT son inmunógenos adecuados para la producción de anticuerpos contra esta proteína; no obstante, debido a que el epítope de reconocimiento de la proteína es solo una reducida secuencia de la proteína total, es necesario emplear una baja dilución del anticuerpo. A fin de utilizar esta herramienta desarrollada para estudiar la proteína endógena en el parásito se hace necesario estandarizar técnicas de detección de la proteína NMNAT en extractos del parásito, con el fin de concluir sobre la importancia del extremo amino terminal de la proteína en cuestión. Una de las aplicaciones interesantes a evaluar con esta herramienta es la posible afectación de la actividad de la proteína en presencia de los anticuerpos, lo que podría inducir interesantes aproximaciones a la importancia de esta secuencia en el proceso de plegamiento de la proteína.
CONCLUSIONES
Se sintetizaron y analizaron eficazmente péptidos, los cuales contenían parte de la secuencia N-terminal de la proteína NMNAT de L. braziliensis. Se generaron con éxito anticuerpos contra la proteína en estudio utilizando como agentes inmunogénicos secuencias peptídicas polimerizadas, derivadas del extremo Nterminal de la proteína del parásito. Se determinaron asimismo los parámetros adecuados para la inmunodetección de la proteína recombinante, encontrándose que para la detección es necesario cargar 2,5 μg de proteína, con una dilución del anticuerpo de 1:500.AGRADECIMIENTOS
A la Universidad Nacional de Colombia, y a la Dirección de Investigación Sede Bogotá (DIB) por la financiación de los proyectos de investigación 14365 y 14418.REFERENCIAS
1. Grimaldi, Jr. G.; Tesh, R. B. Leishmaniases of the New World: current concepts and implications for future research. Clin Microbiol Rev. 1993. 6: 230-50. http://www.who.int/tdr/pulications/documents/leishmaniasis-life-cycle.swf. Consultado (03/06/2011). [ Links ]
2. Calmet, J. Risk factors and leishmaniasis: possible contributions for control strategies. In: Wijeyaratne, P.; Goodman, T.; Espinal, C. Leishmaniasis Control Strategies: A critical evaluation of IDRC-supported research. Ottawa. 1992. pp. 206-222. [ Links ]
3. Hassa, P. O.; Haenni, S. S.; Elser, M.; Hottiger M. O. Nuclear ADP- ribosylation Reactions in Mammalian Cells: Where Are We Today and Where Are We Going? Microbiol Mol Biol Rev. 2006. 70(3): 789-793. [ Links ]
4. Berger, F.; Ramirez-Hernández, M. H.; Ziegler, M. The New Life of a Centenarian. Trendsin Biochemical Sciences. 2004. 29: 111-118. [ Links ]
5. Berger, F.; Lau, C.; Dahlmann, M.; Ziegler, M. Subcellular Compartmentation and Differential Catalytic Properties of the Three Human Nicotinamide Mononucleotide Adenylyltransferase Isoforms. J. Biol Chem. 2005. 280(43): 36334-46341 [ Links ]
6. Ziegler M. New functions of a longknownmolecule. Emerging roles of NAD in cellular signaling. Eur. J. Biochem. 2000. 267(6): 1550-1564. [ Links ]
7. Dawson, V.; Dawson, T. M.; Hong, S.J. Nuclear and Mithocondrial Conversations in Cell Dearh: PARP-1 and AIF Sidnaling. Trends Pharma. Sci. 2004. 25: 259-264. [ Links ]
8. Corda, D.; Di Girolamo, M. Functional aspects of the mono- ADP-ribosylation. EMBO J. 2003. 22: 1953-1958. [ Links ]
9. Yu, S. W.; Andrabi, S. A.; Wang, H.; Kim, N. S.; Poirier, G. G.; Dawson, T. M.; Dawson; V. L. Apoptosis Inducing Factor Mediates poly (ADP-Ribose) (PAR)polymer- Induced Cell Death. PNAS. 2006. 103: 18314-18319. [ Links ]
10. Magni, G.; Amici, A.; Emanuelli, M.; Raffaelli, N.; Ruggieri, S. Enzymology of NAD+ biosynthesis. Adv. Enzymol. Relat. Areas Mol. Biol. 1999. 73: 135-182. [ Links ] [ Links ]
12. Magni, G.; Amici, A.; Emanuelli, M.; Orsomando, G.; Raffaelli, N.; Ruggieri, S. Enzymology of NAD+ homeostasis un man. Cell. Mol. Life Sci. 2004. 61: 19-34. [ Links ]
13. Jayaram, H. N.; Kusumanchi P.; Yalowitz J. A. NMNAT expression and its relation to NAD metabolism. Curr Med Che. 2011. 18(13): 1962-1972. [ Links ]
14. Solao, P. B.; Shall, S. Control of DNA replication in Physarum polycephalum. I. Specific activity of NAD pyrophosphorilase in isolated nuclei during cell cycle.Exp. Cell Res. 1971. 69: 295-300. [ Links ]
15. Schweiger, M.; Hennig, K.; Lerner, F.; Niere, M.; HirschKauffmann, M.; Specht, T.; Weise, C.; Li Oci, S.; Ziegler, M. Characterization of recombinant human nicotinamide- mononucleotide adenylyl transferase (NMNAT), a nuclear enzyme essential for NAD synthesis. FEBS Letts. 2001. 492(1-2): 95-100. [ Links ]
16. Raffaelli, N.; Sorci, L.; Amici, A.; Emanuelli, M.; Mazzola, F.; Magni, G. Identification of a novel humannicotinamide monucleotide adenylyltransferase. BiochemBiophys Res Commun. 2002. 297(4):835-840. [ Links ]
17. Hening K.; Hirsch-Kauffmann M.; Lerner F.; Niere M.; Schewieger M.; Shiao L.O.; Spetch T.; Ziegler. FEBS Letters. 2001. 492: 95-100. [ Links ]
18. Contreras L. Aproximación al metabolismo del dinucleótido de nicotinamida y adenina (NAD+) en Leishmania. Trabajo de Grado. Departamento de Biología, Universidad Nacional de Colombia, Sede Bogotá. 2009. [ Links ]
19. Neme, R. Aproximación bioinformática y bioquímica al metabolismo del dinucleótido de nicotinamida (NAD+) en Leishmania braziliensis . Trabajo de Grado. Departamento de Biología, Universidad Nacional de Colombia, Sede Bogotá. 2009. [ Links ]
20. Moore J. M. R. Peptide Library synthesis, Guy Labororatory. UCSF. 2004. pp. 7-10. [ Links ]
21. Amblard, M.; Fehrentz, J.; Martinez, J.; Subra G. Methods and protocols of modern solid phase peptide synthesis. Molecular Biotechnology2006. 33(3): 239-254. [ Links ]
22. Arunan, C.; Rajasekharan Pillai, V.Solid-phase synthesis of hydrophobic peptides on 1,6-hexanediol dyacrilate crosslinked poly-styrene resin: Comparison with Merrifield resin. Protein and Peptide Letters. 1999. 6: 391-398. [ Links ]




![INTERACCIÓN DE LA GLUTAMINA SINTETASA (GS) Y EL PÉPTIDO β -AMILOIDE COMO UNA ESTRATEGIA DE PURIFICACIÓN [title language="en"]INTERACTION OF GLUTAMINE SYNTHETASE (GS) AND AMYLOID β-PEPTIDE AS A PURIFICATION STRATEGY](/img/en/prev.gif)