Introducción
La floricultura es una de las actividades de mayor importancia económica en Colombia, nuestro país es el segundo productor de flor fresca cortada a nivel mundial, superado solamente por Holanda. El cultivo del clavel (Dianthus caryophyllus L.) es una de las actividades de mayor impacto dentro del sector, considerando que detrás de las rosas, esta flor es el segundo producto con mayor demanda internacional [1]. Sin embargo, la producción de los claveles en Colombia se ha visto afectada por enfermedades de tipo parasitario, las cuales han impactado negativamente el sector disminuyendo la rentabilidad del cultivo de las empresas floricultoras. Dentro de estas enfermedades se destaca el marchitamiento vascular causado por Fusarium oxysporum f. sp. dianthi (Fod), en la cual este fitopatógeno, al obstruir de manera progresiva los haces vasculares de la planta, genera clorosis, marchitamiento de las hojas basales y posterior amarillamiento completo, provocando en muchos casos la muerte de la planta [2-4].
Actualmente, para el control de la enfermedad, se emplean estrategias de tipo químico y físico, las cuales, además de ser costosas y tener un impacto negativo en el ambiente, no son lo suficientemente eficientes. Por esta razón, es necesario estudiar nuevas alternativas que, además de ser compatibles con los sistemas de producción actuales, sean más eficientes y no tengan un impacto negativo con los ecosistemas. Es bien sabido por la comunidad científica que el desarrollo de dichas alternativas requiere del conocimiento bioquímico y molecular determinantes en la interacción entre estos dos organismos. Dicho conocimiento es el insumo para el desarrollo de nuevas variedades resistentes y de controladores biológicos y para la generación de inductores naturales de resistencia. Por ejemplo, conocer cuáles son los genes que se expresan de manera temprana en las variedades resistentes del clavel durante la interacción con el patógeno es un primer paso en el desarrollo de marcadores moleculares que pueden ser usados para la generación de variedades resistentes. Sin embargo, estos requieren de estudios transcriptómicos comparativos que-evalúen los cambios a nivel del ARNm en los tejidos de la planta, durante la interacción directa con el patógeno [5].
Para el desarrollo de dichos estudios, se ha empleado en los últimos años una variación de la técnica de la reacción en cadena de la polimerasa (PCR, por las siglas en el inglés de Polymerase Chain Reaction), la cual permite la cuantificación de los niveles transcripcionales de un gen o genes de interés, conocida como RT-qPCR (por las siglas en el inglés de Reverse Transcription-quantitative PCR). Esta metodología se ha usado como herramienta para el estudio de respuestas moleculares asociadas con resistencia o susceptibilidad en muchas interacciones planta-patógeno. En estos estudios se han usado dos posibles aproximaciones, en la primera, conocida como cuantificación absoluta, se determinan los niveles transcripcionales para el gen de interés, al comparar con un vector que presenta la secuencia específica a evaluar; este proceso requiere de la elaboración de una curva de calibración para cada gen, lo cual aumenta de manera significativa el tiempo de análisis y los costos de la técnica. En la segunda, conocida como cuantificación relativa o normalización, los niveles transcripcionales del gen o gen de interés se comparan con la expresión de un gen de referencia, el cual presenta expresión constitutiva y no cambia sus niveles durante el proceso biológico objeto de estudio [6,7]. Esta última aproximación permite el análisis transcripcional de varios genes de manera simultánea, disminuyendo el tiempo y los costos de los análisis. No obstante, el uso de esta técnica requiere de un gen de referencia validado, para el cual se haya comprobado, además de su expresión constitutiva, su estabilidad durante el proceso biológico objeto de estudio. En el caso de las interacciones planta-patógeno no deben existir cambios significativos en su expresión en los tejidos de la planta durante la inoculación con el patógeno. En estudios previos, en el modelo Clavel-Fod, se encontró que los genes de α-tubulina y β-actina, usados como genes de referencia en otros modelos [8-10], presentan cambios en sus niveles transcripcionales a horas tempranas posteriores a la inoculación, indicando que no pueden ser usados como genes de referencia. Estos resultados impulsaron los estudios para identificar aquellos genes candidatos de referencia que pueden ser usados para análisis transcripcionales en esta interacción planta-patógeno.
En la presente investigación, se evaluó si genes que han sido usados en estudios transcripcionales en otros patosistemas pueden ser usados como referentes en estudios moleculares que involucren al clavel y al patógeno Fod. Para ello, se evaluó la expresión constitutiva en tejidos de la planta y el efecto de la inoculación con el patógeno en genes codificantes para las proteínas Gliceraldehido 3 fosfato deshidrogenasa (GAPDH por sus siglas en inglés) [11], Histona H3 (HIS) [12], PUX1[13], ubiquitina (UBQ) [5], el factor de elongación 1 (EFαd) [14], la proteína ribosomal 13S PR13S [15] y la subunidad ribosomal18S [16]. Después del diseño de cebadores específicos que flanqueaban secuencias conservadas en los genes de estudio, se determinó la presencia de los mismos en DNA de variedades de clavel comerciales cultivadas en nuestro país usando PCR convencional. Posteriormente, se verificó la expresión constitutiva de dichos genes en tallos, raíces y hojas de clavel, usando la técnica RT-PCR semicuantitativa. Finalmente, usando la técnica RT-qPCR, se evaluó el efecto de la inoculación con el patógeno sobre los niveles de expresión de los diferentes genes objeto de estudio. Los resultados de la presente investigación permiten proponer genes de referencia que pueden ser usados para el estudio de los fenómenos moleculares involucrados en la interacción entre el clavel y el agente causal del marchitamiento vascular, a saber, el patógeno Fusarium oxysporum f. sp. dianthi.
Materiales y métodos
Material biológico
Se utilizaron esquejes de clavel (Dianthus caryophyllus L.) con tres semanas de enraizamiento de dos variedades con diferencias en los niveles de resistencia al marchitamiento vascular: Golem (Resistente) y Solex (Susceptible). Estos esquejes fueron suministrados por la empresa floricultura Florval S.A.S. sede QFC (Gachancipá-Sabana de Bogotá). Se utilizó un aislamiento del hongo Fusarium oxysporum f.sp. dianthi (Fod), obtenido de plantas de clavel de la Sabana de Bogotá, donadas por la empresa América Flor S.A., las cuales presentaban los síntomas típicos descritos para el marchitamiento vascular. Este aislamiento fúngico se propagó en placas de medio Agar Papa Dextrosa (PDA, por sus siglas en inglés Potato Dextrose Agar) a 20 °C en oscuridad hasta la saturación superficial. Se confirmó para este aislamiento el género y la raza con herramientas moleculares por medio de PCR convencional con cebadores específicos tanto para para género y raza [17,18].
Evaluación de la expresión constitutiva de genes candidatos de referencia en tejidos de clavel (Dianthus caryophyllus L.)
Diseño de cebadores
Para verificar la presencia de las secuencias codificantes para las proteínas GADPH, PUX1, HIS, UBQ, EFα1, PR13S y para elARN ribosomalARNr18s en el material genético de variedades de clavel cultivadas en Colombia, se diseñaron cebadores usando las secuencias reportadas en la base de datos del genoma del clavel (http://carnation.kazusa.or.jp/) y en el GeneBank (https://www.ncbi.nlm.nih.gov/). En el caso de las especies proteicas, las secuencias de aminoácidos hipotéticas generadas por traducción conceptual, a partir las secuencias reportadas de ADNc en cada caso, fueron alineadas con al menos 10 secuencias de aminoácidos que se encuentran en la base de datos (https://www.ncbi.nlm.nih.gov/) para proteínas con la misma función presentes otras especies vegetales, usando la interfase (http://multalin.toulouse.inra.fr/multalin/). Esta aproximación tenía como fin verificar la presencia de dominios conservados en las proteínas hipotéticas del clavel y seleccionar las secuencias presentes en cada ADNc, las cuales serían usadas para el correspondiente diseño de cebadores usando el servidor Primer 3 (http://fokker.wi.mit.edu/primer3/input.htm) [19].
Para el caso del gen codificante para el ARNr18s, se alinearon secuencias pertenecientes a plantas del género Dianthus, anotadas en la base de datos GeneBank, con el fin de seleccionar secuencias especificas del género y evitar amplificaciones inespecíficas por presencia de ADN fúngico en el material vegetal sometido a inoculación. Considerando que se esperaba usar estos en ensayos posteriores de RT-qPCR, el diseño se realizó teniendo en cuenta parámetros como un tamaño de los amplímeros de 100- 400 pb, un contenido de G-C de 40-60%, una Tm (temperatura de fusión) muy cercana o igual entre ellos y la menor posibilidad de formación de homodímeros y heterodímeros. En la Tabla 1., se presentan las secuencias de los cebadores diseñados que se usaron en esta primera etapa del estudio, indicando para las secuencias usadas en el diseño, el ID reportado en la correspondiente base de datos del genoma del clavel (http://carnation.kazusa.or.jp/).
Confirmación de la presencia de los genes de interés en el ADN genómico de clavel
Con el fin de realizar la extracción de ADN, los tallos de la variedad Golem fueron lavados y macerados con nitrógeno líquido. Se pesaron 0,4 g del material macerado en un tubo de 2 mL, se añadió 1 mL de solución CTAB 2% (p/v) (acrónimo en inglés de bromuro de cetil trimetil amonio) y se incubó durante 1h a 65 °C con agitación constante en el equipo Thermal Mixer (Thermo Fisher). Posteriormente, se añadió 1 mL de cloroformo, se agitó, se centrifugó por 10 min a 13000 x G a 4 °C y se transfirió la fase acuosa a un tubo de 2 mL. Se repitió el paso anterior y la fase acuosa final se transfirió a un tubo nuevo. A esta solución se le adicionó 1 mL de isopropanol frio y se centrifugó a 12000 x G a 4 ºC durante 10 min. El sobrenadante se descartó y el pellet se lavó primero con etanol al 70% y, posteriormente, con etanol absoluto, se secó durante 30 min a temperatura ambiente. Finalmente, se resuspendió en 70 μL de buffer TE (Tris-EDTA) a 35 °C [20]. La cuantificación del ADN extraído se realizó por métodos espectrofotométricos, midiendo la absorbancia a 260 nm y la pureza se estimó de acuerdo a la relación A260/A280 en el equipo Genesys 10UV (Thermo spectronic). La integridad del ADN obtenido fue evaluada mediante electroforesis horizontal en gel de agarosa al 1% usando Syber-safe (Invitrogen) para la visualización de las bandas en buffer TAE 1x, un voltaje de 120 V con un tiempo de corrida de 1 h y 30 min. La obtención de las imágenes se realizó usando el equipo Criterion Stain FreeTM - Gel Imaging System de Bio-Rad®, USA con bandeja para detección de fluorescencia.
Con el fin de verificar la presencia de los genes de interés en el ADN genómico del clavel, se usó la técnica PCR, con posterior evaluación del tamaño de los amplímeros obtenidos, usando electroforesis en gel de agarosa. Para esto, se usó como plantilla 1 μg del ADN genómico extraído de clavel y los diferentes pares de cebadores especificados en la Tabla 1. Se usó la enzima Taq polimerasa (Invitrogen) de acuerdo con las especificaciones del fabricante en el equipo MJ Mini Personal Thermal Cycler (Biorad). Se usaron dos programas de temperaturas, el primero (Programa 1) incluía 1 ciclo inicial de 95 °C por 3 min, 35 ciclos (95 °C por 1 min, 59 °C por 1 min y 72 °C por 1 min) y, por último, 1 ciclo final de 72 °C por 10 min. El segundo programa (Programa 2) presentó como variación una temperatura de anillamiento de 55 °C. Para la verificación del tamaño de los amplímeros obtenidos se usó electroforesis en gel de agarosa al 1,5% con un marcador de peso molecular Hyperladder™ IV bajo las condiciones previamente descritas en el apartado anterior.
Evaluación de la expresión constitutiva de los genes de interés usando RT-PCR
Con el fin de verificar la transcripción constitutiva de los genes de interés, se usaron tallos, raíces y hojas de dos variedades de clavel: Golem (variedad resistente) y Solex (variedad susceptible). La extracción del ARN se realizó usando la siguiente metodología, los tejidos fueron macerados de manera independiente con nitrógeno líquido en un mortero estéril previamente lavado con una solución al 0,1% (p/v) de dietil pirocarbonato (DEPC, por sus siglas en inglés). El material macerado (0,4 g) fue sometido a extracción del ARN total mediante el método del TRIZOL Reagent (Invitrogen), siguiendo el protocolo sugerido por la casa comercial con algunas adaptaciones. Al macerado se le añadió 1 mL de trizol, se agitó, se incubó durante 5 min a temperatura ambiente y se centrifugó a 12000 x G a 4 °C por 10 min. El sobrenadante se recuperó y se transfirió a un nuevo tubo para adicionar 200 μL de cloroformo. Esta mezcla se agitó durante 15 s, se incubó a temperatura ambiente por 3 min y se centrifugó a 12000 x G a 4 °C por 15 min. La fase acuosa superior rica en ARN se retiró y se transfirió a un nuevo tubo para adicionar 500 μL de alcohol isopropílico. La mezcla obtenida se mantuvo a temperatura ambiente durante 10 min y se centrifugó a 12000 x G a 4 °C por 10 min. Después de eliminar el sobrenadante, se agregó 1 mL de etanol al 75% y se centrifugó a 7500 x G a 4 °C por 5 min. El sobrenadante se descartó sin alterar el pellet rico en ARN, se dejó secar por 10 min a temperatura ambiente, se disolvió en 30 μL de agua DEPC y se incubó a 65 °C por 10 min. La cuantificación y la pureza del ARN se llevó a cabo mediante la medición de la absorbancia a 260 y 280 nm en el equipo Genesys 10UV (Thermo spectronic).
La síntesis de ADNc se realizó mediante transcripción reversa, usando como plantilla el ARN total extraído previamente de cada uno de los tejidos de ambas variedades. Para ello, se usó oligo dT como iniciador y transcriptasa inversa M-MLV (virus de leucemia murina de Moloney, invitrogen®), de acuerdo con el protocolo del fabricante. Para esto, a 2 μg de ARN se le adicionaron 2 μL de oligo dT (20 mM), 1 μL de cada dNTP (10 mM) y se completó con H2O a 12 μL. Esta mezcla se mantuvo a 65 oC por 5 min, se enfrió por 2 min y se adicionaron 4 μL de buffer de reacción 5X (Tris-HCl 250 mM pH 8,3, KCl 375 mM y MgCl2 15 mM) y 2 μL de DTT 0,1 M. Luego de mantener esta mezcla a 37 °C durante 2 min, se adicionó 1 μL de la enzima MMLV-RT para realizar la síntesis del ADNc a esta misma temperatura por 50 min. Luego de inactivar la enzima a 72 oC, se dejó enfriar el sistema hasta alcanzar una temperatura de 4 °C y se almacenaron las muestras a -20 oC para posteriores análisis. La evaluación de la transcripción se realizó a partir del ADNc previamente obtenido de cada uno de los tejidos mediante PCR convencional, usando los pares de cebadores que se habían amplificado sobre ADN genómico, tal y como se describió previamente. Sin embargo, en esta etapa, se usaron las temperaturas de anillaje específicas para cada uno de los pares de cebadores (Tabla 1).
Evaluación de la expresión de genes candidatos de referencia en plantas de clavel (Dianthus caryophyllus L.) durante la inoculación con Fod
Ensayo in vivo
La inoculación de los esquejes se realizó de acuerdo a lo establecido por Higuera [21]. Para ello, la mitad de los esquejes de cada variedad (Golem y Solex) se inocularon por inmersión de las raíces durante 30 s en una suspensión de conidias de Fod de 1*106 conidias/mL. Los esquejes fueron sembrados en recipientes con sustrato libre de patógenos constituido por una mezcla de cascarilla de arroz y suelo en una relación 50/50. Para los tratamientos inoculados, al sustrato se le añadió una solución nutritiva con inóculo a una concentración de 1,0*103 conidias/mL. La otra mitad de los esquejes (tratamiento control) se sometió al mismo procedimiento, sustituyendo la inmersión de las raíces en las condias por una inmersión en agua estéril y adicionando al sustrato agua estéril en lugar de inóculo. Los esquejes control e inoculados fueron sembrados y mantenidos bajo las mismas condiciones de temperatura de 16 °C y humedad relativa del 60%, con riego de agua por aspersión cada día de por medio. Se realizaron muestreos a los 0, 1, 2, 7 y 14 días postinoculación (dpi) por triplicado biológico para cada tratamiento; cada unidad experimental correspondía a 10 plantas completas. Se hizo el correspondiente seguimiento de incidencia y severidad para los diferentes tratamientos durante dos meses con el fin de verificar las diferencias en resistencia a la enfermedad de acuerdo a estudios previos [22].
Evaluación mediante RT-qPCR de la expresión de genes candidatos de referencia en plantas de clavel (Dianthus caryophyllus L.) inoculadas con Fod
Para la evaluación de la expresión de los candidatos a genes de referencia mediante RT-qPCR, se realizó, primero, la extracción de ARN y luego, la síntesis de ADNc siguiendo los procedimientos descritos previamente. El ADNc obtenido se cuantificó, utilizando el Kit de ensayo Qubit dsADN BR (broad range) and HS (high sensitivity) en el equipo de fluorescencia Qubit® 3.0, con el fin de mantener niveles iguales en cada reacción [7]. La amplificación por qPCR se realizó usando el reactivo iQTM SYBR® Green Supermix de Biorad(R), de acuerdo a las especificaciones del fabricante, en un termociclador en tiempo real C1000 Thermalcycle acoplado al sistema de detección CFX96 Biorad. Se emplearon como programas de temperatura 1 ciclo inicial de 95 °C por 3 min, 35 ciclos (95 °C por 10 s, (temperatura adecuada para cada pareja de cebadores por 30 s) y 72 °C por 30 s) y, por último, 1 ciclo final de 65 °C por 5 s. Se realizó la curva de fusión correspondiente después de cada análisis, desde 65 °C hasta 95 °C, con el fin de verificar la especificidad en la amplificación con los diferentes cebadores. Los valores de Ct (Ciclo umbral) obtenidos para los diferentes tratamientos fueron sujetos a los análisis estadísticos de comparación que se describen a continuación.
Validación de genes candidatos de referencia en la interacción clavel (Dianthus caryophyllus L.) - Fod
La comparación estadística del efecto de la inoculación con el patógeno sobre los niveles transcripcionales de los genes candidatos de referencia se realizó para los genes codificantes para la proteína HIS y la subunidad ribosomal ARNr18s, considerando los resultados de especificidad de amplificación obtenidos durante los ensayos de qPCR. Para estos genes se hicieron evaluaciones, usando todos los tiempos postinoculación objeto de muestreo (0, 1, 2, 7 y 14 dpi). Los análisis estadísticos se hicieron determinando las posibles diferencias entre los valores de Ct, usando los programas Statgraphic 5.0, GeNorm v3 [23] y Normfinder [24].
Resultados y discusión
Confirmación de la presencia de los genes de interés en el ADN genómico de clavel (Dianthus caryophyllus L.)
La primera etapa de la presente investigación consistió en el diseño de cebadores que permitieran la amplificación de secuencias parciales de genes candidatos de referencia. Para el caso de las proteínas, se seleccionaron aquellas secuencias de ADNc reportadas en la base de datos del genoma del clavel (Tabla 1), las cuales presentaban dominios conservados de acuerdo a los alineamientos realizados (resultados no mostrados). La selección de genes codificantes para el diseño de cebadores del ARNr18S se realizó mediante alineamientos de secuencias reportadas para el género Dianthus, lo cual permitiría una amplificación específica del ADN de la planta en material inoculado, en donde la posible presencia de material genético del patógeno podría generar amplificaciones inespecíficas. En cada uno de los casos se obtuvo más de un par de cebadores que cumplía con los requisitos planteados previamente para ser usados en la técnica de qPCR. Con estos, se hizo una primera selección, realizando una amplificación por PCR, utilizando como plantilla el ADN total extraído de tallos de clavel de la variedad Golem. El criterio de selección en esta etapa fue la concordancia entre el tamaño del amplificado diseñado y el obtenido, la selectividad en la amplificación (presencia de una única banda) y la intensidad de los amplímeros obtenidos mediante electroforesis horizontal en geles de agarosa. Esta etapa inicial es fundamental en cualquier estudio de selección de genes de referencia, pues permite descartar rápidamente pares de cebadores que no presenten buena selectividad durante la amplificación [8,16].
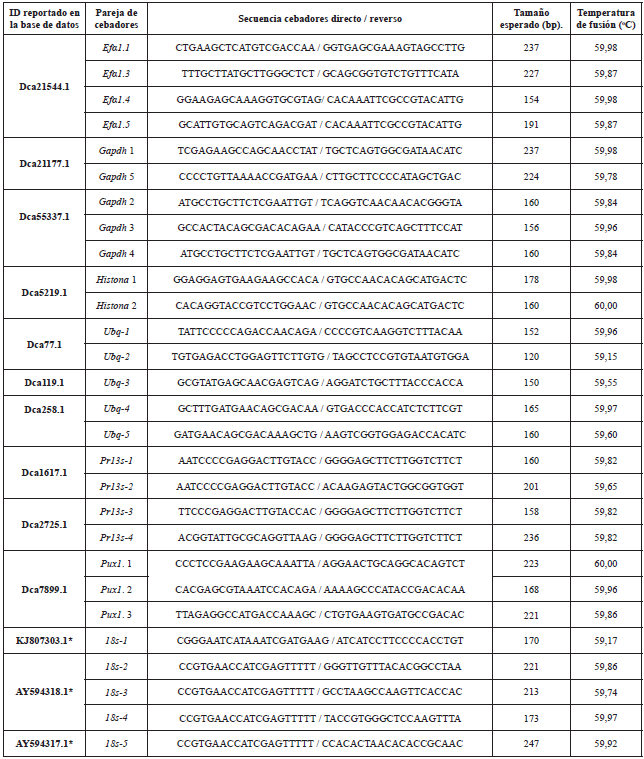
En la figura 1A se observan las bandas correspondientes a los productos de PCR obtenidas usando el programa de amplificación #1 con cebadores diseñados para amplificar secuencias codificantes de las proteínas GADPH, HIS y PUX1. Se observó que, con estas condiciones, solamente se obtienen productos de amplificación, usando los pares de cebadores Histona1 e Histona2. Debido a que no se observaron productos para los otros casos, se utilizó el programa de temperaturas #2, el cual, al presentar una temperatura de anillaje más baja, disminuye la astringencia y favorece la amplificación. Los resultados (Figura 1B), demuestran que, bajo dichas condiciones, sí se presenta amplificación de las secuencias codificantes para las proteínas GADPH (cebadores Gadph3, 4 y 5) y PUX (cebadores Pux1.2), generando amplímeros de tamaños cercanos a los esperados.
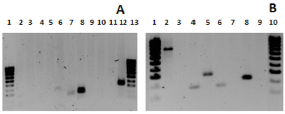
Figura 1 Gel de electroforesis horizontal en agarosa al 1,5% de los amplímeros, usando ADN genómico extraído de tallo de la variedad resistente Golem como plantilla para la evaluación de los cebadores. Gel A Carril: 1 y 13 (marcador de peso molecular hypperlader IV™, 100-1000 bp); 2-6 (gapdh1, gapdh2 gapdh3, gapdh4, gapdh5); 7-8 (histona1, histona 2); 9-11 (pux1.3, pux 1,1, pux 1,2); 12 (control positivo DcPrx02). Gel B Carril: 1 y 10 (marcador de peso molecular hypperlader IV ™); 2-6 (gapdh1, gapdh2 gapdh3, gapdh4, gapdh5); 7-9(pux1.3, pux 1,1, pux 1,2).
La selección de cebadores para la amplificación de secuencias presentes en los genes codificantes para las proteínas Efα1, UBQ (figura 2A) y para el gen pr13s y el ARNr 18s (Figura 2B), se realizó usando el programa de temperatura 1 con los cebadores previamente diseñados (Tabla 1). Se observó que para el caso de los genes codificantes para las proteínas UBQ y Efα1 (figura 2A) existe amplificación de los pares de cebadores Ubq2, Efα1.1, Efα1.3 y Efα1.4. Para el caso de los genes codificantes para la proteína ribosomal 13s PR13s y la subunidad ribosomal 18s (Figura 2B), se observó que para el primero, se presentó amplificación con los pares de cebadores Pr13s-2 y Pr13s-3, mientras que para el segundo, se presentó amplificación con los pares de cebadores 18s-2,18s-3,18s-4,18s-5, aunque con una mayor eficiencia al usar la pareja 18s-5. De acuerdo con los resultados obtenidos hasta este punto, se evidenció que, para todas las proteínas propuestas, existe al menos una pareja de cebadores que permiten la amplificación de la secuencia parcial del gen codificante, confirmando su presencia en el genoma de la variedad objeto de estudio. Esto era de esperarse, considerando el papel transversal que cumplen dichas proteínas y el ARNr 18s en procesos fundamentales para el funcionamiento celular [16]. Es importante tener en cuenta que, en esta primera etapa, en muchos de los casos, se presentaron tamaños de amplímeros algo mayores a los proyectados; esto puede explicarse considerando que la amplificación se realizaba sobre ADN y la posibilidad de la presencia de secuencias intrónicas no se podía descartar, pues el diseño se había realizado sobre secuencias de ADNc. Sin embargo, algunos de los amplímeros, además de presentar el tamaño esperado, presentaron buena especificidad e intensidad de la banda; por lo tanto, los pares de cebadores seleccionados para la siguiente etapa del estudio, en donde se evalúo la expresión constitutiva en diferentes tejidos de la planta, fueron: Gadph-3, Histona-2, Pux1.1 Histona-2, Ubq-2, Efα-1.1, PR13s-1 y 18s-5.
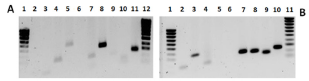
Figura 2 Geles de electroforesis horizontal en agarosa al 1,5% de los amplímeros obtenidos, usando ADN genómico extraído de tallo de la variedad resistente Golem como plantilla para la evaluación de los cebadores diseñados Gel A Carril: 1y 12 (marcador de peso molecular hypperlader IV™, 100-1000 bp); 2-6 (ubq-1, ubq-2, ubq-3, ubq-4, ubq-5); 7-11 (efα1.1, efα1.2, efα1.3, efα1.4, efα1.5) Gel B Carril: 1y11 (marcador de peso molecular hypperlader IV ™);2-5 (pr13s-1, pr13s-2, pr13s-3, pr13s-4); 6-10 (ARN18s-1, ARN18s-2, ARN18s-3, ARN18s-4, ARN18s-5).
Evaluación de la expresión constitutiva
Considerando que uno de los criterios principales para la selección de genes de referencia es su expresión a nivel constitutivo en los tejidos objeto de estudio, se evalúo la transcripción de los genes propuestos mediante la técnica de RT-PCR. Para ello, se usaron los cebadores seleccionados teniendo como plantilla ADNc obtenido del ARN total constitutivo extraído de hojas, tallos y raíces de clavel. Esta técnica permite determinar de manera semicuantitativa la transcripción de genes, siendo ampliamente usada en diferentes estudios de expresión de genes en tejidos vegetales [10,25]. Considerando que en ensayos preliminares no se presentó amplificación cuando se usaron los cebadores efα-1.1 y ADNc obtenido a partir de ARNm de hoja, este gen se descartó para ensayos posteriores (resultados no mostrados). La verificación de la amplificación en todos los tejidos de la planta aseguraría que las secuencias seleccionadas efectivamente hacen parte de genes de expresión constitutiva con funciones basales necesarias para el funcionamiento celular; este es uno de los criterios básicos que se deben cumplir en la selección de un gen de referencia [25]. El estudio se continuó con los genes restantes, los resultados se presentan en las figuras 3 y 4.
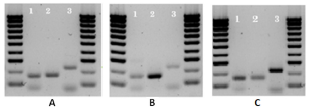
Figura 3 Geles de electroforesis horizontal en agarosa al 1,5% de los amplímeros, usando ADNc obtenido de ARN total extraído de tallo, raíces y hojas como plantilla para la evaluación de la expresión constitutiva y sintetizados para el ensayo de RT-qPCR, marcador de peso molecular hypperlader IV™, 100-1000 bp. Figura A: Hoja. Figura B: Raíz. Figura C: Tallo. Carril: (1) gadph-3, (2) histona-2, (3) pux1.1.
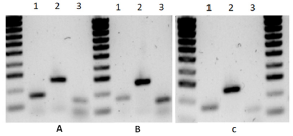
Figura 4 Geles de electroforesis horizontal en agarosa al 1,5% de los amplímeros, usando ADNc obtenidos a partir de ARN total extraído de tallo, raíces y hojas como plantilla para la evaluación de la expresión constitutiva y sintetizados para el ensayo de RT-qPCR, marcador de peso molecular hypperlader IV™, 100-1000 bp. Figura A: Raíz. Figura B: Tallo. Figura C: Hoja. Carril: (1)pr13s, (2) 18s, (3) ubq.
Se evidenció la presencia de ARNm codificante para las proteínas GADPH, HIS, PUX1, UBQ y PR13S y la subunidad ribosomal 18S en los tres tejidos del clavel. Esto demostró que, efectivamente, dichas entidades moleculares se expresan de manera constitutiva en las plantas de clavel, tal y como se esperaba, considerando sus funciones básicas a nivel celular [5,11-15]. Sin embargo, la amplificación de las secuencias parciales de los genes gadph, pux1, pr13sy ubiquitina, presentaron dímeros de cebadores, denotando que su amplificación es menos específica, usando como plantilla ADNc y que, si se querían usar en la siguiente etapa de RT-qPCR, era necesario hacer ensayos adicionales para su uso. Para confirmar estos resultados, se evalúo la formación de dímeros directamente de las curvas de fusión de los amplímeros obtenidos mediante PCR en tiempo real. Estos ensayos demostraron que las temperaturas de fusión (Tm) para los amplímeros obtenidos a partir de las secuencias presentes en los genes histona y ARNr18s (Figura 5) se dieron a temperaturas de 85 y 87 °C respectivamente, sin la presencia de inespecificidades. Estos resultados concuerdan con los obtenidos en los geles de agarosa y los posiciona como los candidatos con mejores condiciones técnicas para aplicar la técnica qPCR considerando su alta especificidad. Es bien sabido que la presencia de amplímeros inespecíficos genera problemas al aplicar esta técnica, sobre todo cuando se usan fluoróforos para la detección, basados en el intercalamiento en las bases nitrogenadas del ADN [26].
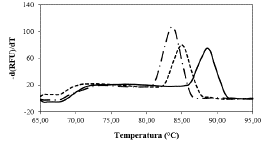
Figura 5 Resultados obtenidos para las curvas de fusión, empleando los cebadores diseñados para la evaluación de los niveles transcripcionales. Las líneas continua, segmentada y punteada corresponden respectivamente a los genes ARNr18S, histona y peroxidasa como control positivo.
Respecto a los amplímeros obtenidos a partir de las secuencias codificantes para PR13s y GADPH, si bien es cierto que sus temperaturas de fusión se encuentran a 83 y 84 °C respectivamente (Figura 6A), la presencia de señales en el rango de temperaturas de 73-78 °C indica que existen inespecificidades que afectarían su uso en qPCR. Esto mismo sucedió durante la amplificación de las secuencias codificantes para PUX1 y UBQ (Figura 6B), en donde junto a los amplimeros con Tm de 83 y 84 °C respectivamente, se presentan productos no esperados en el rango de temperaturas entre 75 y 78 °C. En conjunto, estos resultados indicaron que, tal y como se había evidenciado en geles, existía amplificación inespecífica y que, si se querían evaluar los niveles transcripcionales para los genes pr13s, gadph, ubq y pux1, se requería hacer diferentes ajustes adicionales.
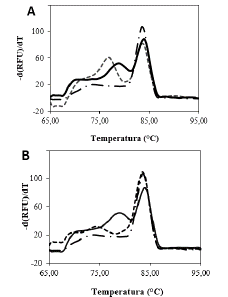
Figura 6 Resultados obtenidos para las curvas de fusión, empleando los cebadores diseñados para la evaluación de los niveles transcripcionales. En la figura A, las líneas continua, segmentada y punteada corresponden respectivamente a los genes pr13s y gadph y peroxidasa como control positivo. En la figura B, las líneas continua, segmentada y punteada corresponden respectivamente a los genes ubq, pux y peroxidasa como control positivo.
Se ha encontrado que modificaciones en las mezclas de reacción como cambios en la relación plantilla/cebadores, la temperatura de anillamiento, adición de sustancias como dimetil sulfóxido (DMSO por sus siglas en inglés), urea y algunos detergentes, pueden disminuir la generación de dímeros [27]. Sin embargo, a pesar de realizar algunos cambios en cuanto a la temperatura de anillamiento y a la relación plantilla/cebadores, dichos dímeros se siguieron presentando (resultados no mostrados). Bajo dichas consideraciones, se seleccionaron los genes codificantes para la proteína Histona H3 (Dca5219.1) y para el ARNr18s, con el fin de evaluar sus niveles de expresión en tejidos de tallos y raíces de clavel, durante su inoculación con el patógeno usando RT-qPCR.
Evaluación de la expresión de genes candidatos de referencia en plantas de clavel (Dianthus caryophyllus L.) durante la inoculación con Fod
Los genes de referencia para el estudio de las interacciones planta-patógeno, no deben presentar variaciones en presencia del estrés biótico objeto de estudio, es por ello que, mediante un ensayo in vivo de inoculación con el patógeno, se determinaron sus niveles de transcripción en tallos y raíces. Para ello, se usaron esquejes de dos variedades de clavel que presentan diferentes niveles de resistencia al marchitamiento vascular (variedad resistente Golem y variedad susceptible Solex). Considerando las diferencias en incidencia y severidad al marchitamiento vascular, que se presentaron durante este ensayo in vivo, dichas características contrastantes de resistencia entre las dos variedades seleccionadas fueron validadas [28].
Se realizó la evaluación en los tiempos 0, 1, 2, 7 y 14 dpi, con el fin de establecer si la expresión de dichos genes era afectada por la presencia del patógeno. En la figura 7 y 8 se presentan los resultados correspondientes al gen codificante para el ARNr18s. Se observó que los niveles de transcripción, para la variedades susceptible y resistente, en ambos tejidos de las plantas, no presentan diferencias significativas por efecto de la inoculación con el patógeno (p<0.05). Estos resultados contrastan con lo encontrado en otros modelos planta-patógeno, como el de uva (Vitis vinífera)-Botrytis Cinerea [ 29] y el de ginseng indio (Withania somnífera (L.) Dunal)- Alternaria alternata [ 30], en donde la expresión del gen codificante para dicho ARNr presenta cambios significativos durante el evento de la interacción. Algunos reportes describen cambios en la expresión de este gen por efecto de las condiciones ambientales [31]; en el presente estudio dichos cambios no se esperan, pues el ensayo in vivo se realiza bajo condiciones ambientales relativamente constantes. De acuerdo al presente estudio, la inoculación con el patógeno no genera cambios en su expresión y permite establecer que el gen codificante para el ARNr18s es un fuerte candidato para ser gen de referencia en este modelo.
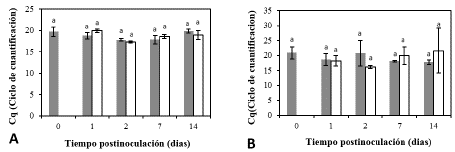
Figura 7 Niveles transcripcionales de ARNr18s en raíz de dos variedades de clavel con diferencias en los niveles de resistencia al marchitamiento vascular. Figura A: Golem (Resistente) y Figura B: Solex (Susceptible) respectivamente a diferentes tiempos posinoculación con Fod. Los colores gris y blanco corresponden a los tratamientos control e inoculado respectivamente. Los grupos estadísticamente diferentes se señalan con letras diferentes (p<0.05) con n igual a 3.
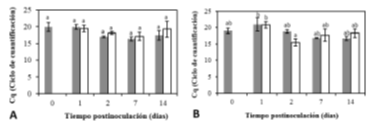
Figura 8 Niveles transcripcionales de ARNr18s en tallo de dos variedades de clavel con diferencias en los niveles de resistencia al marchitamiento vascular: Figura A: Golem (Resistente) y Figura B: Solex (Susceptible) respectivamente a diferentes tiempos posinoculación con Fod. Los colores gris y blanco corresponden a los tratamientos control e inoculado respectivamente. Los grupos estadísticamente diferentes se señalan con letras diferentes (p<0.05) con n igual a 3.
En las figuras 9 y 10, correspondientes a los niveles de expresión del gen codificante para la Histona H3, no se presentaron diferencias significativas entre el tratamiento control y el tratamiento inoculado para ninguna variedad a ningún tiempo evaluado. Sin embargo, sí se presentaron algunas diferencias por efecto del tiempo en raíces de la variedad Golem y tallos de la variedad Solex en algunos de los días evaluados con respecto al tiempo cero; para raíces de Golem a los días 2, 7 y 14 dpi y para tallos de la variedad Golem en el día 14 solamente. Estas diferencias están probablemente asociadas a cambios propios debidos a procesos de diferenciación y crecimiento, los cuales se llevan a cabo en la planta de manera normal. Se ha reportado que existen variaciones durante el crecimiento en la expresión de genes codificantes para histonas en plantas de arroz (Oryza sativa) [ 32].
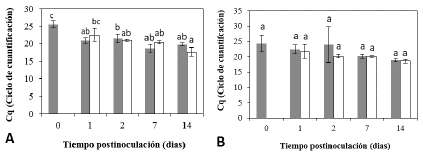
Figura 9 Niveles transcripcionales de histona en raiz de dos variedades de clavel con diferencias en los niveles de resistencia al marchitamiento vascular: Figura A: Golem (Resistente) y Figura B: Solex (Susceptible) respectivamente a diferentes tiempos posinoculación con Fod. Los colores gris y blanco corresponden a los tratamientos control e inoculado respectivamente. Los grupos estadísticamente diferentes se señalan con letras diferentes (p<0.05) con n igual a 3.
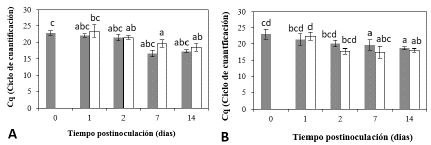
Figura 10 Niveles transcripcionales de histona en tallo de dos variedades de clavel con diferencias en los niveles de resistencia al marchitamiento vascular: Figura A: Golem (Resistente) y Figura B: Solex (Susceptible) respectivamente a diferentes tiempos posinoculación con Fod. Los colores gris y blanco corresponden a los tratamientos control e inoculado respectivamente. Los grupos estadísticamente diferentes se señalan con letras diferentes (p<0.05) con n igual a 3.
No obstante, considerando que estas diferencias se presentan también en el control no inoculado, dichas variaciones no afectan su postulación como gen de referencia en este modelo planta-patógeno, pues no existe efecto de la inoculación sobre su expresión.
Es probable que la estabilidad de la expresión del gen que codifica para la histona en este modelo esté asociado a que este cumple una función básica celular como es el empaquetamiento del material genético, en donde los procesos de transcripción no se afectan por los niveles de las histonas, sino de las modificaciones postraduccionales temporales realizadas sobre estas, como lo son los procesos de acetilación y metilación [12,33]. Estos resultados coinciden con el modelo Sésamo (Sesamum indicum L.) -Fusarium, donde la histona presenta una alta estabilidad frente al estrés biótico [8].
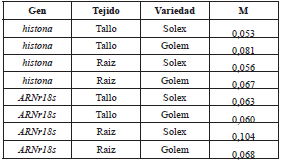
En la tabla 2, se presentan los índices de estabilidad M calculados por el programa de validación GeNorm para los dos genes candidatos en ambas variedades y tejidos objeto de estudio. De acuerdo con esta herramienta, ampliamente usada en la validación de genes de referencia, valores de M menores a 0,2 indican una alta estabilidad de expresión del gen a evaluar [23]. Considerando los resultados obtenidos en esta investigación, en donde en ningún caso el índice M supera un valor de 0.180, la expresión de los genes codificantes para la subunidad ribosomal 18s y la proteína HIS no se afectan durante la interacción con el patógeno y, por tanto, son aptos para ser usados como genes de referencia en el modelo Clavel-Fod. Estos resultados son validados también usando el programa Normfinder (Tabla 3), el cual determina un índice de estabilidad basado en la estimación de la varianza de los valores de Ct [24]. De acuerdo a esta última herramienta, el índice de estabilidad para la expresión de los genes, objeto de estudio en la presente investigación, no supera en ninguno de los casos el valor de 0.105, valores bajos si se comparan con los reportados para genes de referencia encontrados en otros estudios relacionados [8,9]. Por ejemplo, en el modelo sésamo (Sesamum indicum L.) Fusarium [ 8], el gen seleccionado como gen de referencia por su alta estabilidad presentó un valor de 0,119, el cual es superior al encontrado en esta investigación para los genes codificantes para rRNA18s y la proteína HIS. En ese mismo estudio realizado en sésamo, los genes codificantes para estas mismas especies moleculares presentan índices de estabilidad de 0,250 y 0,430 para GeNorm y 0,205 y 0,226 para NormFinder, respectivamente. Estos resultados indican su baja estabilidad en dicho modelo y confirman la necesidad de validar para cada modelo planta-patógeno los genes que pueden ser usados como referentes en estudios transcriptómicos. En general, los resultados de validación obtenidos por estas herramientas, ampliamente usadas en el estudio de las interacciones planta-patógeno [8,9,29,30], coinciden con lo encontrado mediante ANOVA, usando el programa Statgraphics 5.1, lo cual indica que un análisis estadístico preliminar mediante esta última herramienta puede ser usado en una primera etapa, como aproximación en la selección de genes candidatos de referencia, para ser validados posteriormente.
Tabla 2 Índice de estabilidad obtenidos por GeNorm de los genes histona y ARNr18s en tallo y raíz de dos variedades de clavel con diferencias en los niveles de resistencia a la enfermedad: Golem (Resistente) y Solex (Susceptible) evaluados a diferentes tiempos posinoculación (0,1,2,7 y 14 dpi) con Fod.
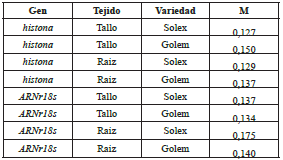
Tabla 3 Índice de estabilidad obtenidos por Normfinder de los genes histona y ARNr18s en tallo y raíz de dos variedades de clavel con diferencias en los niveles de resistencia a la enfermedad: Golem (Resistente) y Solex (Susceptible) evaluados a diferentes tiempos posinoculación (0,1,2,7 y 14 dpi) con Fod.
Conclusiones
De acuerdo a los resultados del presente estudio se evidencia que los genes codificantes para una histona H3 y la subunidad ribosomal 18S pueden ser usados como genes de referencia en estudios transcripcionales en la interacción clavel (Dianthus caryophyllus L)- Fusarium oxysporum f. sp. dianthi. Esto considerando su expresión constitutiva en los diferentes tejidos evaluados, su alta especificidad durante la amplificación usando qPCR y la estabilidad de su expresión durante la inoculación con el patógeno. Los resultados aquí presentados, además de plantear un procedimiento general que puede ser usado para la validación de genes de referencia en otros modelos planta-patógeno, permite validar el uso de estudios transcripcionales con cuantificación relativa en este modelo planta-patógeno; un paso importante en el desarrollo de técnicas que permitan avanzar en el estudio de los mecanismos bioquímicos y moleculares involucrados en el marchitamiento vascular del clavel.