Introducción
Escherichia coli O157:H7 es una bacteria patógena que ocasiona Enfermedades Transmitidas por Alimentos (ETAS) debido a que puede encontrarse contaminando alimentos como carne cruda o poco cocida, leche cruda, hortalizas y semillas germinadas crudas [1]. Entre las enfermedades producidas por E. coli O157:H7 se incluye la colitis hemorrágica, síndrome urémico hemolítico (HUS) y Enfermedad Diarreica Aguda (EDA) [2]. Este tipo de enfermedades se convierten en un problema a nivel mundial de salud pública significativa, ya que se han encontrado cepas de E. coli O157:H7 que muestran resistencia a diferentes antibióticos, como ampicilina, amoxicilina, ácido clavulánico, estreptomicina, gentamicina, kanamicina, neomicina, tetraciclina, cloranfenicol, ciprofloxacina, sulfametoxazol, entre otros [3].
Esta resistencia se ha convertido en una amenaza por la diseminación de bacterias resistentes, por lo cual los péptidos antimicrobianos (AMP, del inglés Anti-Microbial-Peptide) han surgido como una nueva alternativa de antimicrobianos, estos se caracterizan por su bajo peso molecular, gran variedad en su estructura secundaria, composición de aminoácidos variable y polaridad catiónica y anfipática. Debido a que presentan diferentes blancos de acción, dificultan el desarrollo de resistencia bacteriana [4] siendo algunos de sus mecanismos de acción la permeabilización de la membrana, la ruptura estructural de la célula y la interrupción de acciones biológicas de componentes citoplásmicos cruciales como proteínas, enzimas, ARN y ADN [5].
Flórez et al., en el año 2014, obtuvieron sintéticamente la familia de péptidos Ib-M, con modificaciones en la carga neta e hidrofobicidad en el dominio C-terminal con respecto a su análogo Ib-AMP4, péptido antimicrobiano derivado de Impatiens balsamina [6], obteniendo mayor efecto inhibitorio con respecto al péptido nativo en Escherichia coli K-12 [7]. Así mismo, se ha encontrado que de esta familia, el péptido Ib-M1 posee un valor de concentración mínima inhibitoria (CMI) de 4,7 μM y 1,6 μM de concentración mínima bactericida (CMB) contra E. coli O157:H7. Adicionalmente, presenta baja citotoxicidad en células VERO con un valor de concentración citotóxica 50 (CC50) de 395,2 μM [8]; lo que convierte a este compuesto en candidato a nuevos antibióticos para el tratamiento de infecciones ocasionadas por este patógeno. Por otra parte, se ha demostrado que la inmovilización de péptidos de la familia Ib-M en nanopartículas magnéticas de óxido de hierro (IONPs, por sus siglas en inglés) no afecta su actividad contra E. coli O157:H7, lo cual puede ser utilizado para el diseño de nuevos nanotransportadores de la familia Ib-M [9]. A pesar de las ventajas que los péptidos Ib-M presentan, aún no se conoce su mecanismo de acción antibacteriano.
Las respuestas bacterianas a nivel molecular en presencia de algún efector de su crecimiento llevan a cambios en los niveles de expresión de proteínas que son responsables de diversos procesos fisiológicos como la transducción de señales celulares, reacciones bioquímicas de catálisis [10], entre otros [11]. Por lo anterior, el objetivo de este trabajo fue realizar una aproximación inicial al conocimiento en los cambios proteómicos ocasionados por Ib-M1 e IONP@Ib-M1 sobre Escherichia coli O157:H7. De esta manera, se busca dar un primer indicio del posible mecanismo de acción contra este microorganismo.
Materiales y métodos
Compuestos
El péptido Ib-M1 tiene una carga catiónica de +6 y 20 aminoácidos en su estructura [7], este fue fabricado por Biomatik® y la solución patrón fue preparada en tampón Tris-HCl (10 mM pH 7,4) a una concentración final de 2000 μM y almacenado a -80 °C hasta su uso.
La síntesis de las IONPs se llevó a cabo mediante el método de coprecipitación [9] haciendo uso de cloruro de hierro tetrahidrato (II) (Sigma Aldrich, ≥99,0%), cloruro de hierro hexahidrato (III) (ACS, Reag. Ph Eur), hidróxido de amonio (NH4OH de Merck), quitosano (Sigma Aldrich, peso molecular medio), ácido acético glacial (Merck, 100%, Merck), N,N-diisopropiletilamina (DIPEA, Sigma Aldrich, ≥99%), O-(benzotriazol-1-il)-N,N,N',N'-tetrametiluronio tetrafluoroborato (TBTU, Sigma Aldrich, ≥98,0%), nitrógeno gaseoso de grado comercial (Praxair), y agua tipo 1 desionizada (18,2 MΩ ∙cm).
Cepas y condiciones de crecimiento
E. coli O157:H7 (ATCC® 43888TM) se cultivó por primera vez en 5 mL de caldo Luria-Bertani (LB) a 37 °C durante 18 h a 37 °C antes del ensayo; posterior a la incubación, se realizó el ajuste a escala McFarland 3 (OD 625 nm = 0,58/9*108 UFC/mL) en caldo Mueller Hinton (MH), luego se llevó a cabo la inoculación en cada uno de los tratamientos (Ib-M1, IONPs, IONP@ y IONP@Ib-M1) y se obtuvo una concentración celular final de 4,4x108 UFC/mL para cada tratamiento, realizando dos réplicas biológicas para cada ensayo independiente.
Obtención de IONP@Ib-M1
La obtención del bioconjugado IONP@Ib-M1 se realizó siguiendo el procedimiento reportado por Ropero-Vega, et al. [9]. Inicialmente se obtuvieron las IONP por el método de coprecipitación utilizando como precursores sales de Fe+2 y Fe+3 y como base NH4OH. Para la inmovilización de Ib-M1 se realizó un recubrimiento de las IONP utilizando 160 μL de quitosano al 0,5% p/v en ácido acético al 2% v/v. Posteriormente, se realizó la formación de un enlace peptídico entre los grupos amino del quitosano y el grupo carboxilo del residuo Glu del péptido; para esto se mezclaron 70 μL del péptido con 44,9 mg de TBTU y 24,2 μL de DIPEA. Después de 20 min de reacción, la mezcla del péptido fue adicionada a una suspensión de 800 μg de IONPs. Para la determinación de la cantidad de péptido inmovilizado se estableció la concentración del péptido antes del proceso de inmovilización y el péptido en el sobrenadante después de la inmovilización utilizando el método de Bradford [12] con el kit Quick Start™ Bradford Protein Assay de BioRad determinando la absorbancia a 595 nm en un espectrofotómetro de fibra óptica AvaSpec-ULS2048L StarLine fiber-optic (Avantes).
Cinética de crecimiento de E. coli O157:H7
La inhibición del crecimiento de la bacteria se determinó mediante el método de microdilución de acuerdo con los procedimientos descritos por el Instituto de Normas Clínicas y de Laboratorio [13], utilizando placas estériles de 96 pozos de fondo redondo. Se prepararon diluciones seriadas de Ib-M1, IONPs, IONP@, IONP@Ib-M1 y luego se agregó el inóculo del caldo MH obteniendo un volumen final de 200 μL por pocillo. Se realizaron controles de crecimiento negativos y positivos agregando cada una de las suspensiones caldo MH o E. coli O157:H7 con caldo MH a los pozos. La cinética de crecimiento se determinó midiendo la absorbancia de la suspensión a 595 nm utilizando Multiskan Sky (Thermoscientific) a 37 °C durante 24 h y en agitación constante.
Extracción de proteínas intracelulares y cuantificación
Posterior a determinar el crecimiento celular, se llevó a cabo la inoculación de E. coli O157:H7 con los diferentes tratamientos a 37 °C / 140 rpm en un volumen final de 5 mL. Finalizada la incubación, se realizó la centrifugación en tubos eppendorf de 1 mL. Una vez obtenido el pellet celular, se realizó el rompimiento de las células, posteriormente la extracción de proteínas siguiendo lo establecido por el fabricante [14] y después se llevó a cabo la cuantificación de las proteínas siguiendo lo establecido por la casa comercial con un volumen final de 1,5 mL [15].
Determinación del perfil proteico de E. coli O157:H7
Electroforesis de isoelectroenfoque: una vez las proteínas fueron extraídas se separaron de acuerdo con sus puntos isoeléctricos mediante isoelectroenfoque (IEF) utilizando el equipo Protean IEF i12 de Bio-Rad. Para ello, se emplearon tiras IPG de pH 4-7, (rango lineal) de 11 cm, las cuales fueron rehidratadas con una solución de 8 M urea, 2% CHAPS, 50 mM DTT, 0,2% anfolitos, y trazas de azul de bromofenol [16] y con 200 μL de muestra de proteína durante 18 h y posteriormente sometidas a electroforesis IEF teniendo en cuenta la siguiente programación: (1) voltaje 250 V - tiempo 20 min; (2) voltaje 8000 V - tiempo 2 h 30 min; (3) voltaje/hora 20,000.
Electroforesis SDS-PAGE: una vez terminado el corrido IEF, las tiras IPG fueron sometidas a un proceso de equilibrio según lo establecido por la casa comercial [16] y posteriormente se realizó una electroforesis en gel de poliacrilamida SDS vertical con concentraciones finales de acrilamida de 12% y 5% p/v para los geles de separación y apilamiento, respectivamente. La electroforesis se llevó a cabo durante 330 min a 150 V en un sistema de electroforesis Protean II XI Cell (Biorad) utilizando como patrón 20 μL de marcador de peso Chromatein Prestained Protein Ladder® (Vivantis). Finalizada la electroforesis, se realizó la tinción de los geles por medio de nitrato de plata [17]. Se preparó un litro de cada una de las soluciones en recipientes limpios antes de iniciar el procedimiento y utilizando agua de calidad Milli-Q. Cada uno de los geles fueron depositados en solución de fijación (12% ácido acético glacial, 50% etanol, 0,5 mL/L formaldehído grado analítico) durante una hora con agitación constante. Posteriormente, fueron depositados en la solución de lavado (50% etanol) por 5, 10 y 15 min. A continuación, fueron lavados rápidamente con solución de tiosulfato de sodio (0,2% Na2S2O3*5H2O), después, los geles se lavaron con agua Milli-Q tres veces y se sumergieron en la solución de nitrato de plata (2% AgNO3, 0,75 mL/L formaldehído grado analítico) durante un tiempo de 20 min. Transcurrido este tiempo, los geles fueron lavados nuevamente tres veces con agua Milli-Q, seguido de esto, se sumergieron en la solución de revelado (3% Na2CO3, 20 mL/L de solución de tiosulfato de sodio, 0,5 ml/L de formaldehído grado analítico), durante 10 min. Finalmente, los geles se introdujeron durante 5 min en la solución de fijación y se almacenaron en la solución de conservación (25% etanol, 3% glicerol) a temperatura ambiente para su posterior registro mediante imágenes en formato *.TIFF de los patrones visibles de proteínas a través de un sistema Gel-DocTM XR+Imager (Bio Rad).
Determinación de la variabilidad de perfiles proteicos
La determinación de la variabilidad de los perfiles proteicos se realizó mediante análisis bioinformáticos. Para ello, por medio del software Photo-CaptMw versión 10.01, se efectuó la determinación de los puntos isoeléctricos (pI) y pesos moleculares de cada una de las manchas (spot) expresadas en cada perfil comparando la movilidad electroforética del marcador de peso molecular Chromatein Prestained Protein Ladder® (Vivantis). A partir de este análisis se determinó, de acuerdo con estos parámetros, la posible proteína expresada y su función relacionada. Posteriormente se llevó a cabo la determinación de la variabilidad de los perfiles protéicos, haciendo uso de PDQuest 2-D Analysis Software® (Bio Rad), en el cual se detectó el número total de manchas (spots) para cada gel de tratamiento, se identificaron coincidencias entre cada uno de ellos (matching) y se realizó un análisis de acuerdo con los atributos.
Análisis estadístico
Mediante la cuantificación de densidad de pixeles de cada una de las manchas, se llevó a cabo un análisis de varianza (ANOVA) de una vía, el cual permitió determinar si existen diferencias estadísticamente significativas entre las medias de los diferentes tratamientos de una misma proteína teniendo en cuenta las dos réplicas biológicas de cada una de las condiciones control, Ib-M1, IONP@Ib-M1, IONPs y IONP@. La comparación de medias de los tratamientos se realizó con el test de Bonferroni, en donde se determinó que cuando p≤0,05 hay diferencia significativa. Este análisis se realizó haciendo uso del software Stata/MP versión 14.0®.
Resultados y discusión
Obtención de bioconjugado a partir de inmovilización de Ib-M1 en IONPs
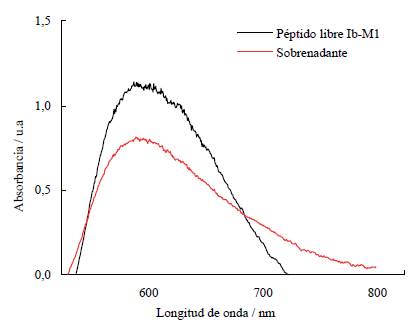
La inmovilización del péptido Ib-M1 en IONP@ se monitoreó por medio de la determinación de la absorbancia a 595 nm antes y después de realizar el lavado. Los resultados se muestran en la figura 1, en donde se observa una disminución en la absorbancia del sobrenadante proveniente de la centrifugación del IONP@Ib-M1, lo que indica que disminuyó la cantidad de péptido en solución y fue, por tanto, inmovilizado después de dos horas de agitación. El porcentaje de inmovilización del péptido Ib-M1 en las IONP@ fue de 45,23%.
Cinética de crecimiento de E. coli O157:H7 bajo diferentes tratamientos con Ib-M1, IONP@Ib-M1, IONPs y IONP@
Los efectos inhibitorios de Ib-M1 se han estudiado en varias ocasiones [7, 8] bajo diferentes condiciones de cultivo. Se ha encontrado que el grado de inhibición depende de diferentes parámetros como la concentración celular. Teniendo en cuenta lo anterior, y con el objetivo de determinar cómo se podría afectar la cinética de crecimiento y a partir de qué momento de incubación se podría realizar el análisis con base en su cambio drástico en crecimiento, se analizó la respuesta de crecimiento celular bajo tratamientos con Ib-M1, IONP@Ib-M1, IONPs y IONP@ usando el método de microdilución. En la figura 2 se muestran los resultados obtenidos de cinética de crecimiento de E. coli O157:H7 bajo los diferentes tratamientos.
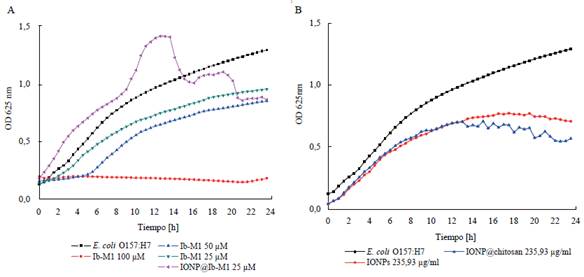
Figura 2 (A) Cinética de crecimiento de E. coli O157:H7 (ATCC 43888) en presencia de péptido libre Ib-M1 a (•) 100 μM, (▲) 50 μM, (▼) 25 μM, (◄) y IONP@Ib-M1 a 25 μM. El crecimiento de E. coli O157:H7 (ATCC 43888) sin la adición de péptido (■) se incluyó con fines comparativos. (B) Cinética de crecimiento de E. coli O157:H7 (ATCC 43888) en presencia de (•) IONPs y (A) IONP@ a 235,93 μg/ml. El crecimiento de E. coli O157:H7 (ATCC 43888) sin la adición de tratamiento (■) se incluyó con fines comparativos.
De acuerdo con la figura 2A, el crecimiento de E. coli O157:H7 en ausencia de los tratamientos, alcanzó una densidad óptica (OD) de 0,8 después de 9 h de tratamiento. Cuando este microorganismo se inocula junto con el péptido Ib-M1, se observa una disminución de 24; 40 y 78% del crecimiento frente a las concentraciones 25, 50 y 100 μM respectivamente después de 9 h de incubación. Bajo estas condiciones se observó que el Ib-M1 a concentración de 50 μM llevó a una fase de latencia extendida 5 h después de la incubación. Para la concentración de 25 μM de Ib-M1, la densidad celular estuvo por debajo del control durante todo el periodo de incubación. El análisis anterior mostró que bajo el tratamiento con Ib-M1 el estrés condujo a una fase de latencia prolongada e inhibió el máximo crecimiento celular a una concentración de 100 μM; mientras que las tasas de crecimiento celular mostraron un aumento a medida que se disminuye la concentración.
Por otro lado, cuando E. coliO157:H7 es puesta en crecimiento junto con IONP@Ib-M1 a una concentración de 25 μM, se observa que el crecimiento celular presenta un cambio significativo en su fase de crecimiento a partir de la hora 9 de incubación, iniciando su fase exponencial. Es posible que este comportamiento esté relacionado con el aumento de la concentración focal del péptido sobre la membrana bacteriana [18]. De acuerdo con los resultados obtenidos por Prada et al. (2020), se observó diferencia en la cinética de crecimiento a lo largo de todo el periodo de incubación [8], esto posiblemente a causa de la diferencia en concentración celular utilizada en esta investigación. Así mismo, en el estudio realizado por Flórez-Castillo et al., se demostró que las IONP y IONP@ no poseen actividad antimicrobiana contra E. coli O157:H7 a una concentración de 120,7 y 60,3 μg/mL respectivamente. En este estudio, se determinó la cinética de crecimiento de este microorganismo en presencia de estas IONPs a concentraciones superiores, resultados que se presentan en la figura 2B, en donde se encontró que con respecto a IONPs y IONP@ a una concentración de 235 μg/mL en E. coli O157:H7 a partir de la sexta hora de incubación, se alcanza la fase estacionaria con respecto al control.
Análisis proteómico de E. coli 0157:H7 bajo tratamiento con Ib-M1y I0NP@Ib-M1
Para determinar los cambios a nivel de expresión de proteínas bajo los diferentes tratamientos transcurridas nueve horas de incubación, se realizó el análisis por medio de electroforesis bidimensional. Para esto, se utilizaron entre 1,2 y 2,6 mg de muestra de proteína. Se realizaron dos réplicas biológicas para el control positivo y para los tratamientos Ib-M1, IONP@ Ib-M1, IONPs y IONP@. Posteriormente se efectuó el análisis como se menciona en el apartado de metodología. Se llevó a cabo la normalización de las intensidades de cada una de las manchas expresadas, utilizando la intensidad total del punto por gel, para compensar las variaciones entre las réplicas. En la figura 3 se muestra el resultado de la exposición de E. coli O157:H7 a Ib-M1 a una concentración de 25 μM. Las manchas de proteínas se encuentran en todo el rango de pH de la tira IPG y los pesos moleculares de las manchas se distribuyen a lo largo de todo el gel, entre 22 y 95 kDa aproximadamente.
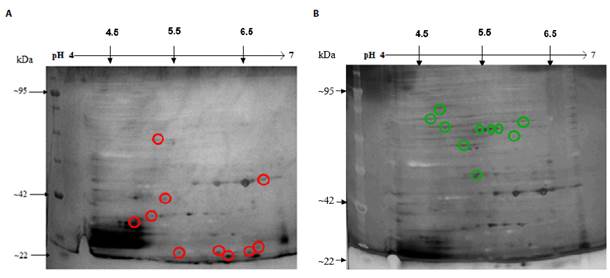
Figura 3 (A) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación. (B) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación con Ib-M1. Los círculos rojos evidencian manchas inhibidas bajo tratamiento y los círculos verdes son manchas expresadas bajo tratamiento.
Se obtuvo la expresión total de 39 puntos de proteínas; 15 se inhibieron con respecto al control, 15 se expresaron con respecto al control, 23 tuvieron una sobreexpresión ≥ 1,5 y siete una inhibición ≤ 1,5. De aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con Ib-M1, seis presentaron diferencias estadísticamente significativas (p ≤0,05), las cuales se muestran en la figura 4.

Figura 4 Mapa bidimensional del proteoma de E. coli 0157:H7 ATCC 43888 después de 9 h de incubación con tratamiento Ib-M1. Se encuentran enmarcadas aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con Ib-M1 y que presentaron diferencias estadísticamente significativas (p ≤0,05).
Después de identificar los patrones de expresión se realizó la aproximación a la identificación de las proteínas expresadas que presentaron diferencias estadísticamente significativas, aquellas que para nuestro conocimiento no han sido reportadas se indicaron con NR. La posible identificación de las proteínas se lista en la tabla 1.
Tabla 1 Proteínas de E. coli O157:H7 expresadas diferencialmente frente al tratamiento Ib-M1 con respecto al perfil de proteínas control.
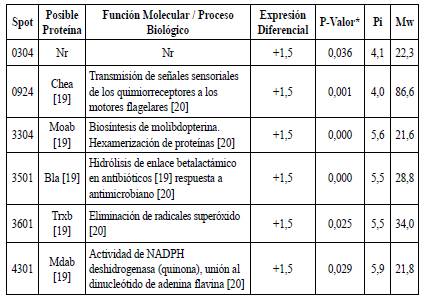
* El p-valor resulta luego de hacer el Anova y test de Bonferroni. NR Proteínas no reportadas de acuerdo con peso molecular (Mw) y punto isoeléctrico (pI).
De acuerdo con lo anterior, se observó la sobreexpresión de proteínas relacionadas con mecanismo de respuesta inmediata para contrarrestar los efectos del péptido Ib-M1 libre. Una de las proteínas con expresión diferencial podría corresponder a cheA (histidina quinasa), la cual es regulada por señales enviadas de quimio aceptores de metiloproteínas (MCP) en respuesta a cambios en el comportamiento flagelar a través de un movimiento conocido como quimiotaxis [21]. La expresión diferencial de la proteína identificada como moaB está involucrada en la biosíntesis del cofactor de molibdeno (MoCo) [22]. La falta MoCo en Escherichia coli causa hipersensibilidad a ciertos análogos de bases N-hidroxiladas, ya que se ha encontrado que E. coli contiene una molibdoenzima capaz de desintoxicar tales análogos (Kozmin & Schaaper, 2007). Así mismo, se ha encontrado que MoCo es el componente esencial de un grupo de enzimas de óxido-reducción que catalizan una amplia variedad de transformaciones de carbono, átomos de azufre y nitrógeno [23]. La proteína identificada como Bla (β-lactamasa) está asociada a la biosíntesis de molibdopterina, hexamerización de proteínas e hidrólisis de enlaces betalactámicos en antibióticos [24]. Se ha encontrado que este tipo de proteína es producida por ciertas bacterias que pueden hidrolizar cefalosporina [25], así mismo, confieren resistencia a la ampicilina, amoxicilina, aztreonam y a la mayoría de las cefalosporinas [26]. La proteína identificada como MdaB (modulador de la actividad del fármaco B/NADPH quinona reductasa) proporciona protección contra la toxicidad de biomoléculas conocidas como quinonoides por sistema de reducción de electrones, además, regula el ciclo redox de la quinona en E. coli [27]. Haciendo uso de la herramienta en línea STRING V11.0, no se obtuvo interacción entre las posibles proteínas identificadas, sin embargo, esto no necesariamente implica que no se trate de proteínas biológicamente significativas, solo que aún no se conocen otras interacciones.
En la figura 5 se muestra el resultado de la exposición de E. coli O157:H7 a IONP@Ib-M1 a una concentración de 25 μM. Las manchas de proteínas se encuentran en todo el rango de pH de la tira IPG y los pesos moleculares de las manchas se distribuyen entre 22 y 42 kDa aproximadamente.

Figura 5 (A) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación. (B) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación con IONP@b-M1. Los círculos rojos evidencian manchas inhibidas bajo tratamiento y los círculos verdes son manchas expresadas bajo tratamiento.
Como resultado de la exposición de E. coli O157:H7 bajo el tratamiento con IONP@Ib-M1, fueron detectadas un total 17 puntos de proteínas expresados; 25 se inhibieron con respecto al control, dos se expresaron con respecto al control, 11 tuvieron una sobreexpresión ≥ 1,5 y dos una inhibición ≤ 1,5. De aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con IONP@Ib-M1, seis presentaron diferencias estadísticamente significativas (p≤0,05) las cuales se muestran en la figura 6.

Figura 6 Mapa bidimensional del proteoma deE. coli O157:H7 ATCC 43888 después de 9 h de incubación con tratamiento IONP@Ib-M1. Se encuentran enmarcadas aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con IONP@Ib-M1 y que presentaron diferencias estadísticamente significativas (p ≤0,05).
Después de identificar los patrones de expresión se realizó la aproximación a la identificación de las proteínas expresadas y aquellas que presentaron diferencias estadísticamente significativas, aquellas que para nuestro conocimiento no han sido reportadas se indicaron con NR. La posible identificación de las proteínas se lista en la tabla 2.
Tabla 2 Proteínas de E. coli O157:H7 expresadas diferencialmente expuestas a tratamiento IONP@Ib-M1 con respecto al control.
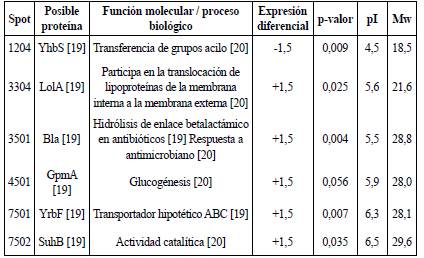
* El p-valor resulta luego de hacer el Anova y test de Bonferroni. NR Proteínas no reportadas de acuerdo con peso molecular (Mw) y punto isoeléctrico (pI).
Al igual que con el tratamiento con Ib-M1, se obtuvo la expresión significativa de la proteína identificada como Bla (β-lactamasa), la cual está asociada a la biosíntesis de molibdopterina, hexamerización de proteínas e hidrólisis de enlaces betalactámicos en antibióticos [24]. Así mismo, se obtuvo expresión significativa de LolA, la cual es una chaperona periplásmica que interviene en la síntesis de lipoproteínas de membrana externa, proceso que es dependiente de ATP [28]. La proteína identificada como GpmA hace parte de proteínas que participan en el transporte de aminoácidos y el metabolismo en respuesta a estrés de tetraciclina y hexabromociclododecano (HBCD) [29]. La proteína identificada como YrbF se conoce que está involucrada en la regulación de fosfolípidos en la membrana externa de bacterias Gram-negativas, con el objetivo de formar y expulsar vesículas al medio externo como factor de virulencia y, así mismo, intercambiar componentes de la superficie celular [30]. Igualmente, la proteína está relacionada con funciones como actividad catalítica, transportador hipotético ABC y respuesta a antimicrobiano [20]. Haciendo uso de la herramienta en línea STRING V11.0, no se obtuvo interacción entre las posibles proteínas identificadas, sin embargo, esto no necesariamente implica que no se trate de proteínas biológicamente significativas, solo que aún no se conocen otras interacciones.
En la figura 7 se muestra el resultado de la exposición de E. coli O157:H7 a IONPs a una concentración de 235,93 μg/mL. Las manchas de proteínas se encuentran en todo el rango de pH de la tira IPG y los pesos moleculares de las manchas se distribuyen a lo largo de todo el gel.
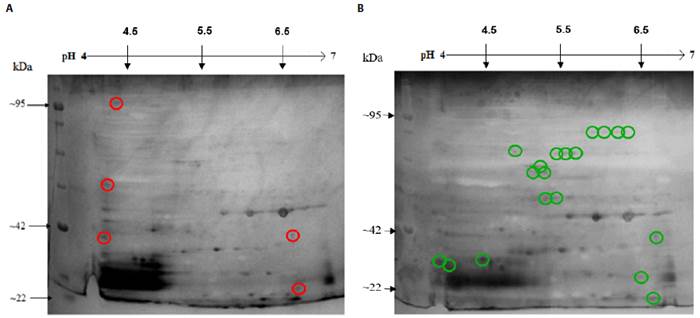
Figura 7 (A) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación. (B) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación con IONPs. Los círculos rojos evidencian manchas inhibidas bajo tratamiento y los círculos verdes son manchas expresadas bajo tratamiento.
Como resultado de la exposición de E. coli O157:H7 bajo el tratamiento con IONPs, se obtuvo un total de 55 puntos de proteínas; cinco se inhibieron con respecto al control, 20 se expresaron en relación con el control, 13 tuvieron una sobreexpresión ≥ 1,5 y nueve una inhibición ≤ 1,5. Este resultado puede deberse a la interacción electrostática entre las nanopartículas catiónicas y las membranas aniónicas del patógeno [31]. De aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con IONPs, 6 presentaron diferencias estadísticamente significativas (p ≤0,05), las cuales se muestran en la figura 8.

Figura 8 Mapa bidimensional del proteoma de E. coli O157:H7 ATCC 43888 después de 9 h de incubación con tratamiento IONPs. Se encuentran enmarcadas aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con IONPs y que presentaron diferencias estadísticamente significativas (p ≤0,05).
Después de identificar los patrones de expresión se realizó la aproximación a la identificación de las proteínas expresadas y aquellas que presentaron diferencias estadísticamente significativas. La posible identificación de las proteínas se lista en la tabla 3.
Tabla 3 Proteínas de E. coli O157:H7 expresadas diferencialmente expuestas a tratamiento IONPs con respecto al control.
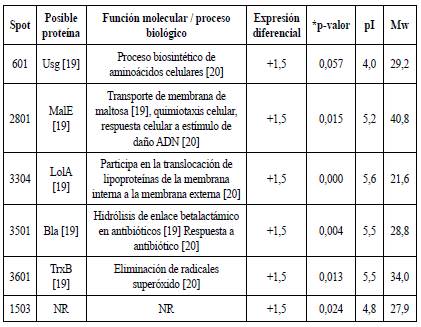
* El p-valor resulta luego de hacer el Anova y test de Bonferroni. NR Proteínas no reportadas de acuerdo con peso molecular (Mw) y punto isoeléctrico (pI).
Una de las proteínas con expresión diferencial es Usg, esta hace parte del complejo acetil coenzima A carboxilasa (ACC), así mismo, está relacionada con el control de la traducción del ARNm uniéndose a una horquilla probable en el extremo 5' del ARNm, inhibiendo la traducción [20]. Además, se obtuvo la sobreexpresión de la proteína identificada como MalE, la cual es una proteína periplásmica de unión a maltosa (MalE o MBP) requerida en bacterias Gram-negativas para el transporte de maltosa y maltodextrinas y para quimiotaxis hacia estos sustratos, también se ha encontrado que esta se expresa a un alto nivel en consecuencia de shock osmótico [32]. Al igual que en el tratamiento con el Ib-M1, se obtuvo la sobreexpresión significativa de proteínas como Bla, TrxB y LolA como lo obtenido en el tratamiento IONP@Ib-M1 cuya función es mencionada anteriormente. Haciendo uso de la herramienta en línea STRING V11.0, se obtuvo que esta red tiene una interacción entre las proteínas LolA y trxB.
En la figura 9 se muestra el resultado de la exposición de E. coli O157:H7 a IONP@ a una concentración de 235,93 μg/mL. Las manchas de proteínas se encuentran en todo el rango de pH de la tira IPG y los pesos moleculares de las manchas se distribuyen a lo largo de todo el gel.
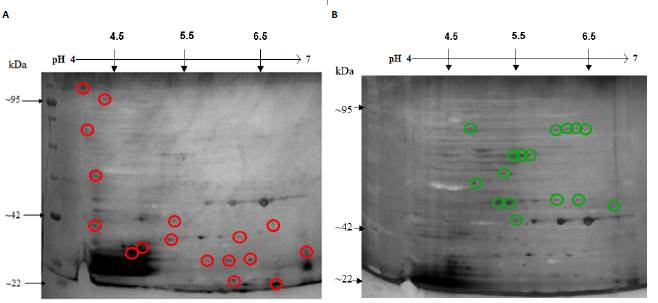
Figura 9 (A) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación. (B) Mapa bidimensional del proteoma control de E. coli O157:H7 ATCC 43888 después de 9 h de incubación con IONP@. Los círculos rojos evidencian manchas inhibidas bajo tratamiento y los círculos verdes son manchas expresadas bajo tratamiento.
Como resultado de la exposición de E. coli O157:H7 bajo el tratamiento con IONP@, se obtuvo un total de 37 puntos de proteínas; 19 se inhibieron con respecto al control, 16 se expresaron en relación con el control, 9 tuvieron una sobreexpresión ≥ 1,5 y 2 una inhibición ≤ 1,5. De aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con IONP@, 4 presentaron diferencias estadísticamente significativas (p ≤0,05) como se muestra en la figura 10.
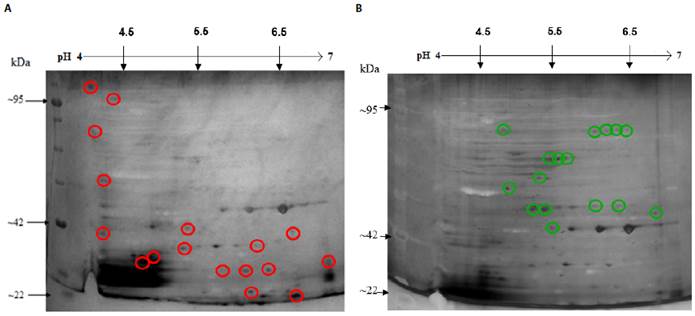
Figura 10 Mapa bidimensional del proteoma de E. coli 0157:H7 ATCC 43888 después de 9 h de incubación con tratamiento I0NP@. Se encuentran enmarcadas aquellas proteínas que estuvieron presentes tanto en el perfil de proteínas control como en el perfil de proteínas del tratamiento con I0NP@ y que presentaron diferencias estadísticamente significativas (p ≤0,05).
Después de identificar los patrones de expresión se realizó la aproximación a la identificación de las proteínas expresadas y aquellas que presentaron diferencias estadísticamente significativas. La posible identificación de las proteínas se lista en la tabla 4.
Tabla 4 Proteínas de E. coli O157:H7 expresadas diferencialmente expuestas a tratamiento IONP@ con respecto al control.
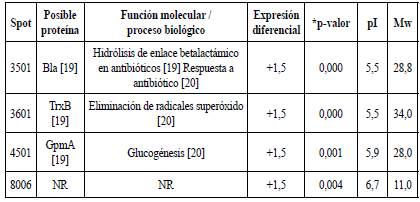
* El p-valor resulta luego de hacer el Anova y test de Bonferroni. NR Proteínas no reportadas de acuerdo con peso molecular (Mw) y punto isoeléctrico (pI).
Al igual que en el tratamiento con el Ib-M1, se obtuvo la sobreexpresión significativa de proteínas como Bla, TrxB y GpmA, cuyas funciones fueron mencionadas anteriormente. Haciendo uso de la herramienta en línea STRING V11.0, no se obtuvo interacción entre las posibles proteínas identificadas, sin embargo, esto no necesariamente indica que no se trate de proteínas biológicamente significativas, solo que aún no se conocen otras interacciones.
Conclusiones
En este estudio se realizó el análisis proteómico comparativo bajo los diferentes tratamientos de antimicrobianos, como lo son Ib-M1, IONP@Ib-M1, IONPs y IONP@; el cual reveló un total de 72 proteínas. La expresión diferencial permitió determinar cambios relacionados con funciones como quimiotaxis, síntesis y mantenimiento de proteínas de membrana externa, procesos de óxido-reducción, crecimiento celular, protección contra toxicidad de biomoléculas, transporte de aminoácidos e inhibición de traducción. Estos hallazgos determinan un primer paso de mecanismos de acción de estos antimicrobianos alternativos, así mismo, se confirman las ventajas de análisis proteómicos sobre estrategias para identificar en futuros estudios proteínas clave en respuesta al estrés.