Introducción
El parásito intracelular Leishmania braziliensis pertenece al orden Tripanosomátida y es el agente causal de la leishmaniasis, enfermedad parasitaria que afecta en promedio un millón de personas anualmente en el mundo y considerada por la OMS como una enfermedad endémica en Colombia. Las medidas de manejo existentes dependen del uso de fármacos tóxicos derivados de antimonio para combatir la infección y del control del vector para reducir la transmisión [1,2].
Debido a ello y a la ausencia de vacunas en humanos disponibles en la actualidad para combatir este parásito, el estudio de las proteínas implicadas en el metabolismo energético, indispensable para la supervivencia de este organismo, es de gran interés. Por esto se desea estudiar la proteína Lb-NMNAT, previamente identificada en el grupo de investigación [3], debido a que sintetiza el dinucleótido de nicotinamida y adenina (NAD), cofactor indispensable en el metabolismo del parásito [4].
El NAD es un cofactor enzimático fundamental, derivado de la vitamina B3 (Niacina), que se relaciona con diversos procesos celulares como su participación en reacciones de óxido-reducción y su utilización como sustrato para la modificación de proteínas (deacetilaciones o poli ADP-ribosilaciones) y como precursor de moléculas movilizadoras de calcio a nivel intracelular [5]. El NAD participa en múltiples rutas metabólicas como el ciclo de los ácidos tricarboxílicos y la cadena transportadora de electrones, con lo cual la síntesis de ATP depende en gran medida de este dinucleótido [5-6]. De igual manera, el NAD exhibe elevada importancia en la modificación y reparación del material genético, ya que puede ser utilizado como sustrato por enzimas del tipo ADP-ribosiltransferasas y deacetilasas tipo Sirtuinas [7].
Adicionalmente, el NAD es el precursor del NADP, importante cofactor enzimático redox, sintetizado por la enzima NAD-quinasa. El NADP está implicado en procesos reductivos como la síntesis de ácidos grasos, colesterol y fotosíntesis, así como en rutas anfibólicas como la vía de las pentosas fosfato [6, 8]. También tiene una elevada incidencia en la regulación de los mecanismos antioxidantes celulares, regenerando compuestos como la tioredoxina y el glutatión [9] o actuando en la reducción de radicales libres de forma directa [10].
Por su parte, la NMNAT (Nicotinamida/Nicotinato Mononucleótido Adenilil Transferasa, EC 2.7.7.1/18) cataliza la adenililación del mononucleótido de nicotinamida (NMN) o del ácido nicotínico (NaMN), generando NAD o el dinucleótido de adenina y ácido nicotínico (NaAD), respectivamente. El NaAD posteriormente es convertido a NAD por la enzima NAD sintasa [11]. La NMNAT es indispensable para la supervivencia del parásito, ya que las rutas de biosíntesis identificadas para la producción del NAD convergen en esta enzima [12].
En consecuencia, la NMNAT es una enzima interesante de estudio, no solo como posible blanco terapéutico, sino también como elemento central de la biología del parásito. La región N-terminal de esta enzima se presenta exclusivamente en tripanosomátidos y está ausente en las isoformas humanas de la NMNAT [13]. Recientemente, nuestro grupo encontró, que esta región amino terminal (1-43) es indispensable para la actividad catalítica de la enzima [3]. Por estas razones, péptidos derivados de esta región podrían interaccionar con la enzima de forma no covalente y afectar su actividad. La interacción de péptidos con proteínas o sus fragmentos pueden conducir a cambios que inciden sobre su estructura tridimensional y afectar su actividad [14]. Dos péptidos representativos y exclusivos de la región N-terminal de la NMNAT de Leishmania braziliensis se sintetizaron químicamente para evaluar su acción sobre la actividad de la enzima.
Materiales y métodos
Síntesis de los péptidos
Se realizó la síntesis de los péptidos en fase sólida, utilizando una estrategia Fmoc (9-fluorenilmetoxicarbonilo) tert-Butilo en una resina Rink amida MBHA, con aminoácidos protegidos Fmoc/tBu [16], en una escala de síntesis de 9 unióles, llevando a cabo la desprotección de cada residuo con una solución de piperidina (4-metil-piperidina) al 25% en N,N’-dimetilformamida (DMF) y 0,1% de Tritón X 100. Como solvente se empleó una solución de diclorometano y DMF, con constante agitación a temperatura ambiente y un tiempo de reacción de 15 h. El desanclaje se realizó usando ácido trifluoroacético (TFA) al 93%, H2O (2,5%), EDT (2,5%) y TIS (2,0%). Luego, se precipitó el péptido con éter etílico frío y se centrifugó para obtener el precipitado. Posteriormente, se lavó con éter etílico (5X) y se dejó secando a temperatura ambiente toda la noche y finalmente se resuspendió en agua.
Purificación y caracterización de los péptidos
Para la purificación de los péptidos se utilizó extracción en fase sólida (SPE) usando una columna pre-empacada con soporte C18 en un sistema de solventes en gradiente ascendente de acetonitrilo en agua. Posteriormente, se liofilizaron las fracciones obtenidas durante 72 h y finalmente se almacenaron a -20 °C. Luego, se realizó la caracterización de los péptidos obtenidos por cromatografía en fase reversa, RP-HPLC, usando una columna analítica C18 en un sistema de solventes en gradiente agua-acetonitrilo.
Expresión y purificación de la proteína recombinante 6xHis Lb-NMNAT
La obtención de la proteína recombinante 6xHis Lb-NMNAT se realizó a partir de un clon de expresión obtenido previamente (6xLbNMNATpQE30) con el cual se transformaron células E. coli M15. Brevemente, el clon se obtuvo por sub-clonación del fragmento lbnmnat amplificado mediante PCR acoplado a retro-transcripción (RT-PCR) en el vector pGEMT-Easy, para posterior digestión y ligación con el vector pQE30 [2]. A partir de las células E. coli M15, se efectuó un inoculo líquido en medio Tryptic Soy Broth suplementado con ampicilina (100 μg/mL), el cual se dejó en crecimiento toda la noche a 28 °C. Posteriormente, se realizó una dilución 1:20 y se incubó hasta una densidad óptica de 0,6 a 600 nm. La expresión se hizo durante toda la noche a 24 °C, utilizando como agente inductor IPTG a una concentración final de 1mM [3]. Las células inducidas se colectaron mediante centrifugación a 8000 rpm, durante 10 min a 4 °C. El precipitado celular se resuspendió en buffer de lisis (300 mM NaCl, 50 mM Na2PO4 pH 8,0/NaOH, 10 mM imidazol) suplementado con lisozima (1 mg/mL), inhibidores de proteasas (Sigma P8340) y b-mercaptoetanol (2 mM), incubando por 10 min a 4 °C. A continuación, las células se sonicaron sobre hielo por 5 min aplicando pulsos de 15 s con una amplitud de 40%, seguido de 15 s de reposo. Posteriormente, se centrifugaron las muestras a 12 000 rpm obteniendo la fracción soluble, la cual se incubó durante una hora con la resina de afinidad a Níquel ProBond (Thermo Fisher Technology) a 4 °C [3]. Luego, se realizó la elución de la proteína con el buffer de lisis variando la concentración de imidazol de 10 a 300 mM; los eluídos obtenidos se monitorearon por SDS-PAGE. Finalmente, los eluídos se dializaron durante 24 h en baño de hielo, utilizando una membrana con tamaño de poro de 6000-8000 Da.
Inmunodetección de la proteína recombinante 6xHis Lb-NMNAT
La proteína recombinante 6xHis Lb-NMNAT se electro-transfirió hacia membranas de nitrocelulosa aplicando una corriente de 20 mA por 12 h en buffer de transferencia (0,2 M glicina, 10 mM Tris/HCl pH 8,0, 10% (v/v) metanol), utilizando cámara de transferencia EBX-700 (CBS SCIENTIFIC) y la fuente de poder POWERPAC HV (BioRad). La efectividad de las transferencias se evaluó con solución de tinción reversible Ponceau S. Posteriormente, las membranas se bloquearon 2 h con solución de leche descremada 5% (p/v) en TBS-Tween (150 mM NaCl, 20 mM Tris/HCl pH 7,5, 0,1% (v/v) Tween 20). La inmunodetección de la proteína recombinante se realizó con el anticuerpo primario anti-6xHis (1:5000) (Abcam) y el anticuerpo secundario antirratón conjugado a biotina (1:5000) (Sigma). La detección cromogénica de los complejos inmunológicos se llevó a cabo utilizando estreptavidina A conjugada a fosfatasa alcalina (1:3000) (Promega) y los sustratos nitro-azul de tetrazolio (NBT) (50 mg/mL) y 5-bromo-4-cloro-3'-indoilfosfato (BCIP) (50 mg/mL) (Promega) en buffer de revelado (150 mM NaCl, 100 mM Tris/HCl pH 9,0, 1 mM MgCl2). La detección inmunológica se detuvo con agua destilada al evidenciar productos coloreados sobre las membranas.
Ensayos directos de actividad enzimática
El ensayo de actividad midió la formación de NAD+ a partir de la reacción entre NMN y ATP después de separar los productos de los sustratos a través de una columna cromatografía RP C18. Para ello, se mezclan 25 μL de buffer HEPES (100 mM, 40 mM de MgCl2, pH 7,4), se adicionan 3 μL de NMN (5 mM en agua), 20 μL de 6xHis Lb-NMNAT (1,25 mM) y el volumen de solución de péptido (para una concentración final de 2,5 mM) en 90 μL de volumen total.
En el caso del control, se preparó la misma mezcla sin péptido. Esta solución se incuba a 37 °C por 15 min, tras lo cual se inicia la reacción agregando 10 μL de ATP (1,25 mM, en buffer HEPES 100 mM pH 7,4). Esta solución se vuelve a incubar por 20 min a 37 °C. Para parar la reacción enzimática a 100 μL de reacción se le adicionan 50 μL de ácido perclórico 1,2 mM. Esta suspensión se deja 10 min a 0 °C y se centrifuga a 6000 rpm a 4 °C. Del sobrenadante resultante se toma una alícuota de 130 μL y se neutraliza con 35 μL de bicarbonato de potasio 1 M. Esta mezcla se deja en hielo 15 min y se centrifuga a 12000 rpm por 5 min. 100 μL del sobrenadante se toma para ser analizado por HPLC. Para el análisis por HPLC se usó un equipo Agilent Technologies de la serie 1260®, equipado con una bomba cuaternaria (Quat Pump VL, G1311C), automuestreador (ALS, G1329B), compartimiento de columna termostatado (TCC, G1316A), detector de onda variable (VWD VL, G1314B), controlado por el programa OpenLAB CDS ChemStation Edition. Para la separación cromatográfica se usó una columna Kinetex® de Phenomenex (5,0 μm C18 100 Å, 100 x 4,6 mm). Como sistema de solventes se usó: A. Buffer fosfatos pH 6,0 B. Metanol. El programa cromatográfico consta de tres etapas: 2 min de 100% de A, 2 min de 0 al 20% de B, 1,5 min de 20 al 25% de B, 1,5 min de 25% de B, 3,5 min 25-0% de B y 2 min de 100% de A.
Estudio bioinformático de la interacción de los péptidos con la proteína LbNMNAT
La secuencia de la proteína se obtuvo a partir del genoma de L. braziliensis (MHOM/BR/75/M2903 LbrM2903_17 de 675,383 a 676,306), cepa referenciada cuyo genoma secuenciado se encuentra bajo el número de acceso PRJNA165955 en el NCBI. Se empleó el programa I-TASSER V 4.4 (Iterative Threading ASSEmbly Refinement) [17]. Para refinar el modelo se usaron los servidores 3D refine [18], FG-MD (Fragment Guided MD simulation) [19] y ModRefiner14 [20]. La calidad de las estructuras evaluadas se realizó empleando Verify3D [21] y su confiabilidad se analizó con ERRAT [22]. El mejor modelo propuesto por este algoritmo fue validado utilizando el servidor SAVES (Structural Analysis and Verification Server, https://services.mbi.ucla.edu/SAVES/), con los programas ERRAT y Verify3D. La evaluación del modelo inicial mostró un factor total de calidad bajo (ERRAT 70,903%). Posteriormente se aplicaron diferentes estrategias de refinado usando los servidores ModRefine, 3D Refine y FG MD refine. La estructura de mejor calidad se encontró después de usar el algoritmo de 3D Refine seguido de una iteración adicional con FG MD, que permitió obtener un modelo con un factor total de calidad del 97,9%.
La inspección visual y edición de imágenes de realizó con el programa UCSF Chimera 1.14 [23]. El sitio de unión de los sustratos (NMN y ATP) a la LbNMNAT se predijo usando el programa COACH [24]. Los cálculos de acoplamiento molecular se efectuaron con AutoDock Vina [25]. La preparación del modelo tridimensional de la proteína y de los posibles ligandos se llevó a cabo usando el paquete AutoDockTools. Los ligandos peptídicos se prepararon con el mismo programa. La estructura de partida de los ligandos se minimizó usando el programa Avogadro 1.1.1 [26], utilizando el campo de fuerzas UFF y como algoritmo de convergencia el método "steepest desent". El acoplamiento molecular de los ligados a la proteína blanco se configuró de forma que tuviera el máximo número de torsiones posibles al mantener todas las cadenas laterales y solo las rotaciones de los enlaces amida en el esqueleto peptídico, empleando una caja centrada alrededor del sitio activo de la enzima y una "exhaustiveness" de 32, debido al tamaño de los compuestos.
Resultados y discusión
Síntesis y purificación de péptidos representativos del extremo N-terminal de la proteína Lb-NMNAT
Los péptidos sintetizados poseen secuencias derivadas del extremo N-terminal de la proteína Lb-NMNAT exclusivas de la enzima de este parásito. Los dos péptidos corresponden a las secuencias PLLQP (SA5), residuos 39 al 43 en la secuencia de la proteína nativa, y CPTSTAEAASQVTPLLQP (SA18), residuos 26 al 43. Estas regiones se seleccionaron, ya que son secuencias que no se encuentran en la NMNATs humanas y que son exclusivas de LbNMNAT, aun comparándolas con otros tripanosomátidos [3]. Las secuencias de los dos péptidos coinciden en el punto de inserción de esta región exclusiva de la NMNAT de Leishmania braziliensis con la región homóloga de otras NMNATs en la región N-terminal [3]. El péptido corto coincide con el extremo C-terminal del péptido largo, con el objetivo de evaluar el efecto de la longitud de la secuencia. Después de la síntesis, los péptidos crudos se caracterizaron por RP-HPLC, aplicando elución en gradiente. Se observaron tres picos mayoritarios en el cromatograma que corresponden a los dos péptidos de interés en los tiempos 3,13 y 4,47 min y una impureza en un tiempo de retención de 4,37 min que puede deberse a un péptido que presenta un menor número de aminoácidos (figura 1A). Posteriormente, se procedió a purificar los péptidos deseados empleando cromatografía en columna, en fase reversa. Después de ello se evaluó la purificación de los péptidos mediante RP-HPLC, con lo cual se obtuvieron los dos péptidos totalmente purificados (figuras 1B y C).
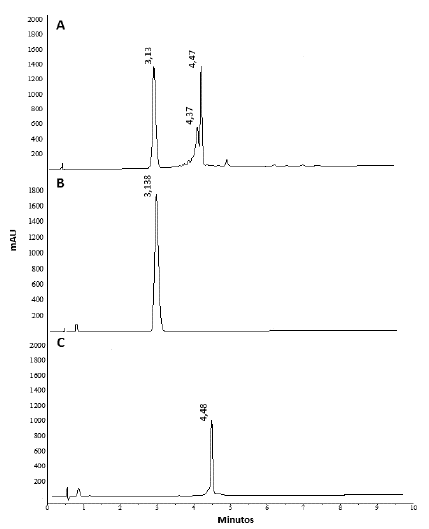
Figura 1 Hidrólisis y purificación de los péptidos. Cromatogramas RP-HPLC correspondientes a (A) Productos crudos del desanclaje de los péptidos sintetizados en la resina Rink amida. (B, C) Péptidos purificados SA5SA5 y SA18, respectivamente, mediante cromatografía en columna preempacada C18, en gradiente ascendente acetonitrilo:agua.
Expresión y purificación de la proteína 6xHis Lb- NMNAT
Para la expresión de la proteína 6xHis Lb-NMNAT se utilizaron células M15 previamente transformadas con el vector pQE30-Lb-NMNAT. Este vector le confiere a la proteína una etiqueta de hexahistidinas en el extremo amino terminal, utilizada para la purificación de esta mediante cromatografía de afinidad a níquel. El peso molecular de la proteína recombinante es de 36 kDa. Después de la inducción de la expresión de la proteína en las células, se procedió a la lisis celular y a la posterior recolección de las fracciones soluble e insoluble. Esta expresión se evaluó mediante SDS-PAGE y se encontró en las fracciones celulares una banda del tamaño esperado para la proteína recombinante, tanto en la fracción soluble como insoluble (figura 2).
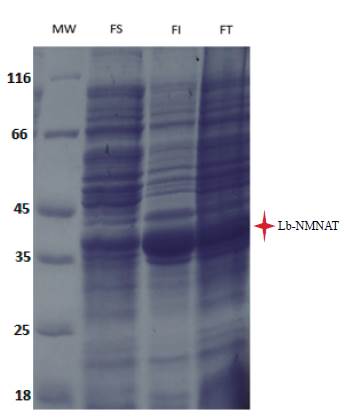
Figura 2 Expresión de la proteína 6xHis Lb-NMNAT en células M15. SDS-PAGE al 12%, teñido con azul de Coomassie. FT: fracciones totales, FI: fracción insoluble, FS: fracción soluble, MW: patrón de peso molecular (kDa). La estrella roja indica la franja del gel que contiene la proteína recombinante.
Se observa una acumulación de la proteína 6xHis Lb-NMNAT en la fracción insoluble, debido a la formación de cuerpos de inclusión. Sin embargo, se observa una cantidad considerable de proteína recombinante en la fracción soluble, por lo cual se procede con su purificación mediante cromatografía de afinidad a níquel. La purificación parcial de la proteína 6xHis Lb-NMNAT se evaluó mediante SDS-PAGE y se encontró para las fracciones 4-8 una banda enriquecida del tamaño esperado (36 kDa) (figura 3). Para demostrar la presencia de la proteína recombinante purificada, se procedió a realizar western-blot con el anticuerpo anti 6xHis (figura 4).
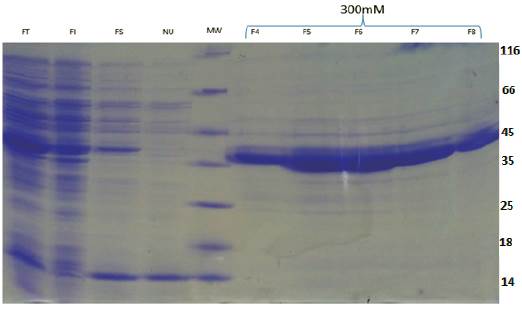
Figura 3 Expresión y purificación parcial de la proteína 6xHis Lb-NMNAT en células M15. SDS-PAGE al 12%, teñido con azul de Coomassie. FT: fracciones totales, FI: fracción insoluble, FS: fracción soluble, UN: proteínas no unidas, MW: patrón de peso molecular (kDa), F4-8): eluidos de la proteína Lb-NMNAT realizados con 300 mM de imidazol.
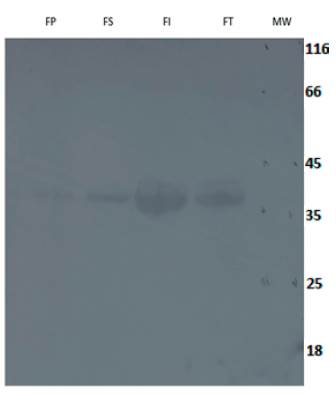
Figura 4 Inmunodetección de la proteína recombinante 6xHis Lb-NMNAT. Westernblot en membrana de nitrocelulosa. Anticuepo primario Anti 6xHis y sistema de revelado con fosfatasa alcalina. FP: proteína purificada, FS: fracción soluble, FI: fracción insoluble, FT: fracción total, MW: patrón de peso molecular (kDa).
Los péptidos representativos dei extremo N-terminal de ia proteína Lb-NMNAT modulan su actividad enzimática
La actividad enzimática de la proteína 6xHis Lb-NMNAT previamente purificada se analizó por RP-HPLC, observándose que la proteína es activa debido a la aparición de un pico en un tiempo de retención de 4,75 min que corresponde al NAD que se produce cuando la enzima cataliza la reacción (figura 5C). Por otra parte, para la evaluación del efecto de los péptidos sintetizados y purificados anteriormente (SA5 y SA18) en la actividad de la enzima, se realizaron los ensayos previamente descritos adicionando 2,5 mM de cada uno de los péptidos al ensayo de actividad. Para el caso del péptido corto SA5, se observa que la proteína aumenta su actividad enzimática cercano al doble, mientras que para el péptido SA18 se encuentra que la proteína disminuye su actividad enzimática (figuras 5A y B).
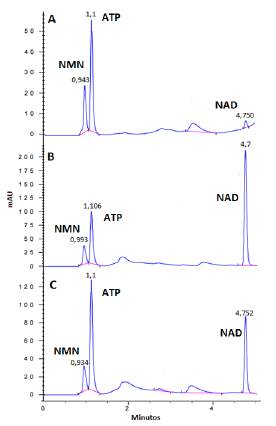
Figura 5 Evaluación del efecto de los péptidos sobre la actividad enzimática de la proteína 6xHis Lb-NMNAT. Cromatogramas correspondientes a (A) Ensayo de actividad de la proteína 6xHis Lb-NMNAT en presencia del péptido SA18 y (B) SA5. (C) Ensayo de actividad control de la proteína 6xHis Lb-NMNAT en ausencia de péptidos.
Este comportamiento se puede explicar como consecuencia de cambios conformacionales que experimenta la proteína cuando interactúa con cada uno de los péptidos, afectando el sitio activo. Con estos péptidos se logró observar la variación de la actividad de la Lb-NMNAT, con lo cual se concluye que existe interacción entre cada uno de los péptidos y la proteína. La NMNAT de Leishmania presenta el plegamiento tipo Rossman, típico de esta familia de enzimas de unión a nucleótidos, no obstante, al eliminar el extremo N-terminal exclusivo del parásito, se evidenció la pérdida de la actividad enzimática, por lo cual este extremo resulta indispensable para el correcto funcionamiento de la proteína [2], lo cual podría asociarse a un papel modulador.
El acercamiento bioinformático podría explicar el efecto inhibidor del péptido SA18 sobre la actividad de la LbNMNAT, debido a que aparentemente, como sugiere el modelo obtenido, el péptido interactúa con la proteína y obstruye el sitio de unión del ATP (figura 6A). De otro lado, el péptido corto SA5 puede interactuar con menor afinidad, debido a que su tamaño posibilita otro tipo de interacción sobre la superficie de la proteína, contribuyendo a estabilizar su estructura y favoreciendo la actividad enzimática (figura 6B). El eventual tratamiento de la leishmaniasis implementando los péptidos reportados en el presente trabajo requiere de estudios adicionales que permitan validar su factibilidad y bioseguridad, analizando, entre otros aspectos, reacciones cruzadas con las NMNATs del humano.
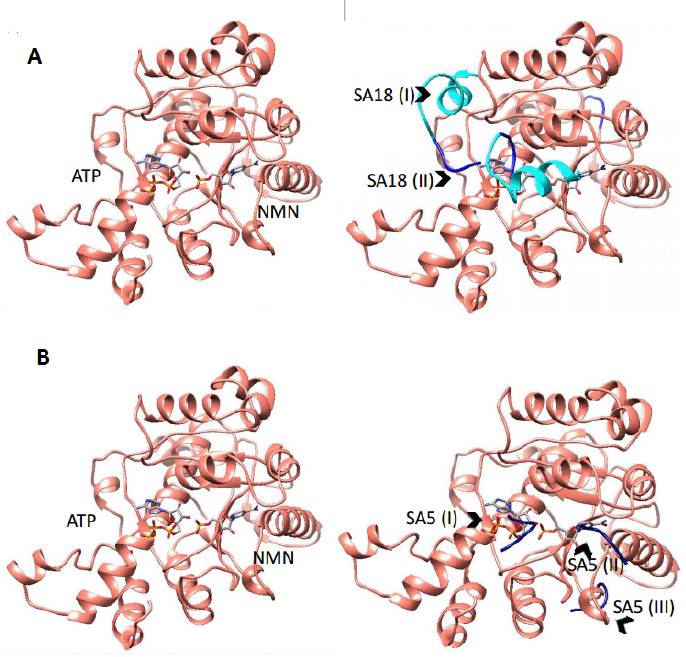
Figura 6 Representación gráfica del complejo de la proteína LbNMNAT con sus sustratos (ATP y NMN) y con los péptidos SA5 y SA18. (A) Proteína LbNMNAT (rosado) y sustratos ATP y NMN (en modelo de barras). Adicionalmente se presenta el complejo entre el péptido SA18 (azul claro) y la proteína, en azul oscuro se resalta la secuencia de 5 residuos de aminoácidos (39-43). SA18 (I) and SA18 (II) corresponden a dos conformaciones de baja energía obtenidos de los cálculos de Docking. (B) Representación de la proteína LbNMNAT y sustratos ATP y NMN. Adicionalmente, se presenta el complejo formado entre el péptido SA5 (azul) y la proteína. SA5 (I), SA5 (II) y SA5 (III) corresponden a tres posibles conformaciones obtenidas por Docking.
Conclusiones
Se evaluó el efecto modulador de dos péptidos sintetizados representativos del extremo N-terminal (1-43) de la proteína LbNMNAT sobre su actividad enzimática. Los péptidos revelaron un efecto contrapuesto, agonista (SA5) y antagonista (SA18), respectivamente, sobre la actividad de la enzima. Lo cual constituye un punto de partida para el diseño de posibles moléculas moduladoras de la actividad de esta enzima.














