Introducción
Los organismos tripanosomátidos del género Leishmania corresponden al agente causal de la Leishmaniasis, enfermedad parasitaria de gran incidencia a nivel mundial. Colombia, debido a sus características biogeográficas, resulta fuertemente afectado por estos parásitos intracelulares, encontrándose 9 de las 21 especies asociadas a la enfermedad [1]. Existen tres formas clínicas principales de esta parasitemia según la especie del parásito involucrado, factores genéticos y la respuesta inmune del hospedero [2]. Dentro de estas formas se destacan lesiones cutáneas que afectan la piel de manera local o difusa provocadas principalmente por L. mayor, L. mexicana, L. amazonensis y L. braziliensis; lesiones desfigurantes o mucocutáneas causadas por L. braziliensis y L. panamensis; o viscerales, afectando el hígado, bazo y/o medula ósea producidas por L. donovani y L. infantum [3], [4].
Estos microorganismos se caracterizan por presentar un ciclo de vida complejo denominado digénico en el cual alterna entre un hospedero invertebrado, con función de vector, y uno vertebrado como residente final. El ciclo comienza cuando el insecto díptero hematófago transmite la forma promastigote al hospedero vertebrado durante la picadura, ingiriendo sangre e inyectando el parásito. Al ingresar al torrente sanguíneo, los protozoarios en estado promastigote son engullidos por macrófagos y células dendríticas del sistema inmune, donde se transforman en amastigotes y se multiplican por fisión binaria (reproducción asexual) dentro del compartimento fagolisosómico hasta la ruptura de las células, aumentando así la propagación e infección [5]. Dentro del vector los amastigotes migran al intestino medio donde se promueve su diferenciación en promastigotes que se ubican finalmente en las glándulas salivales del insecto para ser transmitidos nuevamente [3].
Numerosos acercamientos terapéuticos a esta enfermedad se han desarrollado en todo el mundo; sin embargo, hasta el momento ninguno ha exhibido alta eficiencia, por el contrario, se resalta la presencia de numerosos efectos secundarios tales como mialgia, artralgia, cefalea, fiebre, convulsiones, anorexia, náuseas, nefrotoxicidad y ototoxicidad, entre muchas otras [6]. Adicionalmente, en ocasiones se combinan medicamentos para combatir esta enfermedad, resultando en costos excesivos y aparición de cepas resistentes [4], [7].
Durante la etapa de infección, cuando el promastigote es fagocitado por los macrófagos, la célula hospedera genera especies reactivas de oxígeno (ROS) y/o nitrógeno (RNI) con el fin de establecer condiciones desfavorables para el desarrollo de la infección, estos compuestos resultan tóxicos para los parásitos, cuya capacidad para combatirlos determina el éxito y la persistencia de la enfermedad [8], [9]. Para evitar el daño celular provocado por estas moléculas, los tripanosomátidos poseen peroxidasas con un característico puente disulfuro; estas enzimas usan equivalentes reductores derivados de la tripanotiona, una forma inusual de glutatión que se encuentra exclusivamente en estos protozoos [10]. En la cascada TR-TXN-TXNPx la transferencia de electrones ocurre entre la tripanotiona reductasa (TR), una flavoproteína perteneciente a la familia de las oxidorreductasas dependientes de NADPH y la triparedoxina (TXN) para la reducción del peróxido de hidrógeno (H2O2) y peroxinitrito (ONOO-) por la triparedoxina peroxidasa (TXNPx), siendo la enzima terminal en este sistema antioxidante [11], [13].
La triparedoxina peroxidasa (TXNPx) pertenece a la familia de las 2Cys peroxiredoxinas que poseen dos cisteínas (Cys) distintivas en la posición 52 y 173; estas enzimas exhiben características conservadas en todos los reinos con al menos 30% de identidad de secuencia y pueden agruparse de acuerdo con su compartimentación en el citosol o las mitocondrias [9], [14]. La TXNPx es indispensable para la supervivencia del parásito puesto que se encarga de la destoxificación primaria de H2O2, una amplia gama de hidroperóxidos orgánicos y, en algunos casos, ONOO-, un componente esencial del principal sistema de captación enzimática para ROS y RNI en tripanosomátidos [15]. La acción combinada de la TXNPx con enzimas antioxidantes es crucial para el mantenimiento de una baja concentración de H2O2, regulando el estrés oxidativo y nitrosativo a través del desplazamiento de equivalentes reductores desde el NADPH hacia hidroperóxidos y peroxinitritos [12].
La importancia bioquímica de la TXNPx ha impulsado diversos estudios acerca de su inhibición como estrategia contra la Leishmaniasis. Entre estos estudios se incluyen aproximaciones in silico basados en la construcción de modelos 3D de la enzima de L. braziliensis y su posterior acople molecular con inhibidores de tipo flavonoide como quercitina y taxifolina, los cuales afectan a nivel bioinformático a la enzima [16]. En términos experimentales, la estructura de la TXNPx de L. major se ha resuelto mediante cristalografía de rayos X, permitiendo comprender las características estructurales de la enzima y los mecanismos moleculares de reducción del H2O2. Inclusive, estos estudios revelaron la existencia de epítopes antigénicos que permiten proponer la potencial implementación vacunal de la TXNPx [17].
Por otra parte, estudios genéticos han demostrado la importancia del sistema TR-TXN-TXNPx para el desarrollo y virulencia de estos parásitos [8], convirtiendo a las proteínas involucradas en posibles y potenciales objetivos moleculares para el desarrollo de fármacos de control parasitario. Por esta razón, el objetivo principal del presente trabajo se enfocó en la detección de la enzima triparredoxina peroxidasa citosólica de L. braziliensis (LbTXNPxII) y sus posibles interacciones moleculares con rutas metabólicas de síntesis de NAD+. Tal propósito requirió la clonación, expresión y purificación de la proteína recombinante 6xHis-SUMO-LbTXNPxII para producir anticuerpos policlonales aviares usados en la inmunodetección de la LbTXNPxII.
Materiales y métodos
Construcción del vector recombinante pET SUMO-LbTXNPxII
Apartir del ADN genómico de L. braziliensis (M2904 MHOM/BR/75M2904) previamente obtenido mediante el método de la proteinasa K y utilizando oligonucleótidos directo (5'-TGTCCTGCGGTGACGCCAAAATG-3') y reverso (5'-TTACTGCTTGCTGAAGTAGCCCTCAA-3') (Integrated DNA Technologies), se amplificó la secuencia que codifica la triparedoxina peroxidasa (ID: 5414074) [18] empleando la enzima Taq polimerasa (Thermus aquaticus) producida y purificada "in house" y el termociclador Veriti 96-Well [19]. La región codificante amplificada se clonó en el vector de expresión bacteriano pET SUMO (Thermo Fisher) según especificaciones del manual [20]; los plásmidos extraídos mediante lisis alcalina de clones positivos se evaluaron mediante PCR [21] y corte con enzimas de restricción BamHI y EcoRV (Fermentas) [22].
Producción de la proteína recombinante 6xHis-SUMO-LbTXNPxII en el sistema heterólogo E. coli
Células BL21 (DE3) (Invitrogen) se transformaron mediante choque térmico con el plásmido recombinante pET SUMO-LbTXNPxII [23]. Se permitió su crecimiento en medio Luria Broth (LB) (VWR Life Science) suplementado con kanamicina 50 pg/mL (Merck) a 37 °C en agitación constante (140 rpm) hasta alcanzar O.D.600 nm de 0,6 en incubadora con agitación (LabTech); la expresión se indujo con isopropil P-D-1-tiogalactopiranósido (IPTG) (Sigma-Aldrich) a concentración final de 1,0 mM. Luego de 4 h el cultivo se centrifugó (Thermo Heraeus Megafuge 16R) a 7000 rpm durante 10 min, el pellet se resuspendió en buffer de lisis pET (fosfato de potasio 50 mM, NaCl 400 mM, KCl 100 mM, glicerol 10%, tritón X-100 0,5% e imidazol 10 mM) [20], lisozima (Invitrogen) a concentración final de 1 pg/mL e inhibidor de proteasas (1:400) (Sigma P8340), permitiendo interacción de 1 h a 4 °C en agitación. Finalmente, las células se sonicaron (3 min con amplitud 50% y pulsos de 15 s) (Sonics VibraCell) y las fases se separaron por centrifugación (12000 rpm, 20 min).
La expresión de la proteína recombinante se evaluó mediante electroforesis vertical en condiciones desnaturalizantes (SDS-PAGE) utilizando el sistema MGV-402 (CBS Scientific). Se trató una alícuota de las muestras (totales, solubles e insolubles) resuspendiéndolas con buffer carga 6X (tris HCl 0,5 M pH 6,8, glicerol 30%, SDS 10%, ß-mercaptoetanol 33% y azul de bromofenol 0,4 mg/mL) en relación 1:5 y desnaturalizándolas a 92 °C por 10 min. Se utilizaron geles de poliacrilamida discontinuos de gel separador al 12% (acrilamida 30% Merck: 29 g acrilamida + 1 g bisacrilamida [Promega]) y gel concentrador al 4% (acrilamida 30%: 29 g acrilamida + 1 g bisacrilamida). Las muestras se separaron a 100 V constantemente, siendo visualizadas con azul de Coomassie G250: tiñendo el gel por 45 min y luego decolorando en solución de ácido acético (99%) y 2-propanol (99%) en proporción 1:1. Con el fin de realizar la identificación de la proteína expresada mediante Western blot, y aprovechando la etiqueta 6xHis provista por el vector, las muestras provenientes del cultivo celular se separaron por SDS-PAGE y se electrotransfirieron hacia membrana de nitrocelulosa (Thermo Scientific) en buffer de transferencia (tris base 25 mM, glicina 192 mM y metanol 10%, pH 8,3) a 200 mA durante 2 h en el sistema EBX-700 (CBS Scientific) [24]. La membrana se bloqueó con TBS-T leche 5% (tris-HCl 20 mM pH 7,5, NaCl 150 mM y Tween 20 0,1%) durante 2 h. Seguidamente, se adicionó el anticuerpo primario a6xHis-mouse (1:5000) (Abcam) preparado en TBS-T, permitiendo su interacción con la membrana por 2 h, luego se agregó el anticuerpo secundario amouse-fosfatasa alcalina (1:10000) (Invitrogen) preparado en TBS-T durante 1 h y, finalmente, se reveló con 16,5 μL NBT (S380C Promega) y 16,5 μL NBCIP (S381C Promega) en 5 mL de buffer sustrato fosfatasa alcalina (Tris-HCl 100 mM pH 9,0, NaCl 150 mM y MgCl2 1 mM), la reacción se detuvo con H2O. En cada paso la membrana se lavó 3 veces durante 10 min con TBS-T [25]. Se utilizó marcador de peso molecular (Thermo Scientific 26610) y tanto geles como membranas se foto-documentaron con el sistema Gel Doc XR (BioRad).
Purificación de la proteína recombinante 6xHis-SUMO-LbTXNPxII mediante cromatografía de afinidad a metales inmovilizados (IMAC) y desde cuerpos de inclusión
La proteína 6xHis-SUMO-LbTXNPxII expresada posee una etiqueta de histidinas (6xHis) en su extremo N-terminal que facilita su purificación mediante cromatografía de afinidad utilizando metales inmovilizados (IMAC), los cuales son capaces de unirse específicamente a esta etiqueta [26]. Se utilizó resina de níquel y ácido nitrilotriacético (Ni-NTA) con soporte de agarosa (Quiagen) de la cual se tomaron 100 μL por cada 1 mL de fracción soluble a purificar. La resina se equilibró con buffer de lisis pET (fosfato de potasio 50 mM, NaCl 400 mM, KCl 100 mM, glicerol 10%, Tritón X-100 0,5% e imidazol 10 mM). Luego, se permitió la interacción de esta con la fracción soluble durante 1 h a 4 °C en agitación constante; posteriormente, se cargó una columna de cromatografía recogiendo por gravedad las diferentes fracciones. En primer lugar, se recogieron "las proteínas no unidas"; seguido, se realizaron lavados con 6 volúmenes de buffer de lisis pET suplementado con imidazol (Sigma Aldrich) en concentraciones crecientes (35, 75 y 150 mM). Finalmente, la proteína recombinante unida a la resina se eluyó aumentando la concentración de imidazol a 300 mM [27]. Los eluídos recolectados se cuantificaron por el método de Bradford (0,1 mg/mL Coomassie G-250 en ácido ortofosfórico 8,5%) con albumina de suero bovino (BSA) (Merck) como patrón; se preparó una curva de calibración con seis patrones en concentraciones de 0 a 1 mg/mL. De cada patrón se tomaron 20 μL y se mezclaron con 980 μL de reactivo de Bradford, las muestras se incubaron 5 min a temperatura ambiente en oscuridad, luego se registraron medidas espectrofotométricas a 595 nm (JenWay 7315) [28]; adicionalmente, el proceso y la pureza de las muestras se corroboraron mediante SDS-PAGE 12% y Western blot.
Los cuerpos de inclusión, contenidos en la fracción insoluble de la lisis, se purificaron por SDS-PAGE preparativo al 12%. Inicialmente se homogenizaron las fracciones insolubles con buffer de lavado 1 (tris-HCl 100 mM pH 7,0, EDTA 5 mM, DTT 5 mM, Urea 2 M y Triton X-100 al 2 %), luego se centrifugó (Jouan GR2022) a 12000 rpm durante 30 min, repitiendo este proceso 3 veces. Nuevamente el pellet se homogenizó con buffer de lavado 2 (tris-HCl 100 mM pH 7,0, EDTA 5 mM y DTT 5 mM) y se centrifugó; finalmente se repitió el proceso con el buffer de extracción (tris-HCl 50 mM pH 7,0, EDTA 5 mM, DTT 5 mM y guanidina-HCl 8 M) centrifugando esta vez a 14000 rpm durante 1 h. El sobrenadante se recuperó y dializó a 4 °C durante 16 h en buffer tris-HCl 50 mM y NaCl 150 mM, empleando una membrana de 10 kDa (MWCO, Molecular Weight Cut-Off). Mediante centrifugación a 12000 rpm por 20 min se recuperó la proteína dializada, la cual se desnaturalizó (90 °C) y analizó en un gel preparativo (SDS-PAGE 20x20 cm) utilizándo marcador preteñido (Opti-Protein Marker, ABM) para realizar el corte de la banda desde el gel según el peso molecular esperado para la proteína recombinante (37 kDa). La banda de gel retirada se maceró y se sometió a elución con agua MilliQ a 37 °C en agitación constante durante 12 h. La muestra se centrifugó a 5000 rpm por 10 min, recuperando el sobrenadante. Los eluídos recolectados se cuantificaron por el método de Bradford y el proceso se monitorizó por SDS-PAGE 12% y Western blot.
Producción de anticuerpos policlonales α6xHis-SUMO-LbTXNPxII-IgY en modelo aviar
La proteína 6xHis-SUMO-LbTXNPxII purificada se utilizó para la inmunización de gallinas Hy-Line Brown de 35 semanas en el centro de investigaciones avícolas de la Facultad de medicina veterinaria de la Universidad Nacional de Colombia, sede Bogotá, bajo las condiciones de mantenimiento estándar para el trabajo animal. Monitorizando, de igual manera, un individuo control que se inoculó con PBS 1X (buffer fosfato salino). Las condiciones de inmunización utilizadas han sido reportadas en la literatura y estandarizadas en el Laboratorio de Investigaciones Básicas en Bioquímica - LIBBIQ [29]. La primera inoculación se realizó homogenizando con vortex a velocidad 8 (Vortex Genie 2, VWR Scientific) la proteína antígeno (150 μg) con adyuvante completo de Freund en relación 1:1, esto por vía intramuscular (día 0) y, luego, se realizaron tres posteriores refuerzos: el primero post-15 d y los siguientes con intervalos de 8 d, inoculando el antígeno con adyuvante incompleto (relación 1:1) [30] (Tabla 1). En cada caso se inocularon 300 μL de homogenizado. Igualmente, desde el día cero se recolectaron los huevos y muestras de sangre periférica para analizar sueros preinmunes e inmunes posteriores a cada inoculación [31].
Tabla 1 Esquema de inmunización de gallinas para la obtención de anticuerpos policlonales IgY contra la proteína LbTXNPxII.
| α6xHis-SUMO-LbTXNPxII | Control | |||||||
|---|---|---|---|---|---|---|---|---|
| Día | 0 | 15 | 23 | 30 | 0 | 15 | 23 | 30 |
| Adyuvante de Freund* | C | I | I | I | C | I | I | I |
| Antígeno (ug) | 150 | 150 | 150 | 150 | - | - | - | - |
| PBS 1x (uL) | - | - | - | - | 150 | 150 | 150 | 150 |
*C: Completo / I: Incompleto.
Para obtener los anticuerpos policlonales del suero de cada muestra se permitió la coagulación de la sangre durante 30 min a 37 °C y, luego, mediante centrifugación (Thermo Heraeus Megafuge 16R) (12000 rpm durante 20 min a 4 °C) se separó el suero, almacenándolo con glicerol (JT Baker) (20% v/v) a -80 °C (REVCO). Con el fin de determinar el nivel de reconocimiento de los anticuerpos presentes en los sueros se realizó Western blot, teniendo en cuenta la concentración de anticuerpo producido (α6xHis-SUMO-LbTXNPxII) para realizar diluciones en TBST-T y, adicionalmente, la concentración de antígeno (6xHis-SUMO-LbTXNPxII) a reconocer. Como anticuerpo secundario se utilizó achicken producido en burro (Gallus Inmunitech inc.).
Con el fin de purificar los α6xHis-SUMO-LbTXNPxII-IgY presentes en suero, se utilizó el método de afinidad a antígeno; para esto, la proteína recombinante antigénica purificada (250 pg) se separó mediante SDS-PAGE, transfiriéndola posteriormente a una membrana de PDVF (Millipore) (200 mA por 2 h). La banda con la proteína de interés se visualizó con rojo Ponceau S (Sigma Aldrich) y se cortó (37 kDa), realizando un pre-eluído con glicina 100 mM pH 2,5 por 10 min en agitación constante. Seguidamente, la membrana se lavó con TBS-T 2% durante 10 min y se bloqueó con TBS-T 2% leche 5% por 1 h, retirando el exceso de leche mediante 3 lavados de 10 min con TBS-T 2%. Posteriormente, la banda de membrana se cortó en cuadros de 2x2 mm y se incubó toda la noche (O/N) en agitación constante con 500 μL de sueros sanguíneos. Los anticuerpos no unidos o suero remanente se retiraron mediante pipeteo y se conservaron a -20 °C en congelador convencional. Se realizaron lavados con TBS-T 2% que se monitorizaron por espectrofotometría hasta alcanzar una absorbancia a 280 nm menor a 0,1. Finalmente, para eluir los anticuerpos se adicionaron 150 μL de glicina 100 mM pH 2,5 permitiendo interacción con la membrana durante 10 min aplicando vortex a velocidad 8 (Vortex Genie 2, VWR Scientific); los eluídos se neutralizaron con 15 μL de tris HCl 2 M pH 8,5 y se almacenaron a -20 °C, siendo cuantificados espectrofotométricamente (JenWay 7315) a 280 nm.
Por otro lado, con el fin de extraer los anticuerpos α6xHis-SUMO-LbTXNPxII-IgY presentes en yema de huevo, se procedió a realizar la precipitación de estos con concentraciones crecientes de polietilenglicol (PEG) 6000 (Sigma Aldrich) [32]. En primera instancia, las yemas se separaron cuidadosamente y se solubilizaron en dos volúmenes de buffer PBS 1X pH 7,4, una alícuota de 1 mL de cada huevo se utilizó para una evaluación preliminar de todas las muestras. Se realizaron precipitaciones consecutivas utilizando porcentajes crecientes de PEG6000 (3,5, 8,5 y 12%) con el fin de obtener las inmunoglobulinas enriquecidas. En la primera precipitación se conservó la fase intermedia (eliminando lípidos); en la segunda, el sobrenadante; y en la tercera, el pellet (resuspendido en 1mL de PBS 1X). Para todos los casos las muestras se dejaron en agitación sobre hielo en plataforma de agitación (LabNet) durante 10 min y se centrifugaron a 14000 rpm por 20 min. El precipitado obtenido a un porcentaje de saturación del 12% se resuspendió en 400 μL de PBS 1X, se cuantificó espectrofotométricamente en celda de cuarzo a 280 nm (JenWay 7315) y se tituló mediante ensayo de ELISA indirecto para determinar contenido de IgY. El proceso de purificación se monitorizó por SDS-PAGE 10% [33], [34].
Evaluación y caracterización de anticuerpos policlonales α6xHis-SUMO-LbTXNPxII-IgY
Los anticuerpos α6xHis-SUMO-LbTXNPxII purificados a partir de suero se evaluaron mediante ensayos de Western blot teniendo en cuenta concentraciones del anticuerpo producido (realizando diluciones en TBST-T) y, adicionalmente, la concentración de antígeno (6xHis-SUMO-LbTXNPxII) a reconocer.
Por otro lado, con respecto a los α6xHis-SUMO-LbTXNPxII extraídos de yema de huevo, se realizó su titulación mediante ELISA indirecto en placas de 96 pozos en lector de placas (BioTek ELx8000). Para esto el antígeno (6xHis-SUMO-LbTXNPxII) se preparó en buffer carbonato (carbonato/bicarbonato) pH 9,6 a una concentración de 10 μg/mL, sembrando 100 μL/pozo, esto se incubó durante 1 h a 37 °C (LabTech) y O/N a 4 °C en nevera (cubriendo la placa con papel vinipel y aluminio). Al día siguiente se realizó el bloqueo adicionando 200 μL de leche descremada 5%p/v en PBS 1X durante 2 h a 37 °C; seguido se permitió interacción con el anticuerpo primario α6xHis-SUMO-LbTXNPxII en una dilución 1:500 durante 2 h a 37 °C y, posteriormente, se agregaron 100 μL del anticuerpo secundario αIgY-HRP (Sigma) en una dilución 1:8000 preparado en PBS 1X, incubando durante 2 h a 37 °C. El revelado de la placa se realizó con 2,2'-azino-bis (3-etilbenzotiazolina-6-ácido sulfónico) (ABTS - Sigma) y peróxido de hidrógeno en buffer citrato-fosfato 100 mM pH 5,0 incubando durante 1 h a 37 °C, siendo detenida la reacción con 20 μL de SDS 1%. Entre cada paso la placa se lavó por aspersión abundante con PBS 1X por lo menos 3 veces. Finalmente, se registró la absorbancia a 405 nm.
Tabla 2 Controles utilizados para la titulación de los IgY contra la LbTXNPxII extraídos de yema mediante ELISA indirecto.
| No | Tipo de control | Ag | Ab-1rio | Ab-2rio |
|---|---|---|---|---|
| 1 | Negativo | BSA | α6xHis-SUMO-LbTXNPxII | αIgY-HRP |
| 2 | Negativo | 6xHis-SUMO-LbTXNPxII | α6xHis-SUMO-LbTXNPxII | PBS 1X |
| 3 | Negativo | 6xHis-SUMO-LbTXNPxII | PBS 1X | αIgY-HRP |
| 4 | Negativo | 6xHis-SUMO-LbTXNPxII | PBS 1X | PBS 1X |
| 5 | Negativo | PBS 1X | PBS 1X | PBS 1X |
| 6 | Positivo | αIgY-HRP (1:4000) | PBS 1X | αIgY-HRP |
Inmunodetección de la proteína LbTXNPxII endógena
Con el propósito de reconocer la proteína LbTXNPxII endógena a partir de extractos celulares, aproximadamente 7,5x108 promastigotes de L. braziliensis (M2904 MHOM/BR/75M2904) se colectaron mediante centrifugación a 6500 rpm por 10 min a 4 °C, realizando 2 lavados con 10 mL de PBS 1X pH 7,4 y siendo estos resuspendidos finalmente en 500 μL de buffer de lisis (PBS 0.1X, coctel de inhibidores de proteasas (1:250) (Sigma P8340), Tritón X-100 0.1%), incubando, a su vez, durante 40 min a 4 °C en agitación constante. Seguido, las fracciones se separaron por centrifugación (Thermo Heraeus Megafuge 16R) durante 20 min a 12000 rpm y 4 °C. A la fracción soluble se le adicionó buffer de carga 6X (1:5) para SDS-PAGE y a la insoluble 200 μL de buffer de carga 1X, siendo desnaturalizadas a 90 °C. Adicionalmente, otra muestra de parásitos se lisó directamente con buffer de carga 1X (células totales) en relación 1x105 células/μL. Las muestras se analizaron mediante SDPS-PAGE 12% y Western blot (anticuerpo primario a6xHis-SUMO-LbTXNPxII purificado).
Otra manera de aprovechar los anticuerpos policlonales IgY específicos contra la proteína 6xHis-SUMO-LbTXNPxII, producidos en el modelo aviar en la inmunodetección de la proteína endógena, es por medio de inmunofluorescencia; para ello, promastigotes deL. braziliensis provenientes de 1 mL de cultivo confluente se centrifugaron (5000 rpm por 5 min a 4 °C) y lavaron 3 veces con volúmenes con 1 mL de PBS 1X (homogenizando suavemente), siendo finalmente resuspendidos en 300 μL. Cerca de 2x106 parásitos se aplicaron sobre laminas tratadas con poli-L-lisina fijados con paraformaldehído 4% preparado en PBS 1X (1 h a 4 °C). Posteriormente, se adicionó glicina fría 100 mM durante 15 min y se permeabilizó con acetona fría durante 10 min a 4 °C. El montaje se bloqueó empleando BSA 1% en PBS 1X durante 1 h, luego se permitió interacción con el anticuerpo primario a6xHis-SUMO-LbTXNPxII purificado (1 ha temperatura ambiente), diluido 1:1000 en solución de bloqueo. El anticuerpo secundario αIgY acoplado a Alexa 488 (1:1000 en solución de bloqueo) (Invitrogen) se aplicó sobre las láminas durante 1 h en oscuridad. La marcación del ADN se realizó con DAPI (1 μg/ml en PBS 1X) (Thermo Scientific) durante 5 min. Entre cada paso las láminas se lavaron una vez con PBS 1X durante 10 min. Finalmente, las láminas se cubrieron con medio de montaje (Fluoromont-G- Invitrogen) y se visualizaron empleando el microscopio Nikon Eclipse C1 Plus y el Software EZ-C1.
Co-inmunoprecipitación de la proteína LbNMNAT utilizando α6xHis-SUMO-LbTXNPxII-IgY en extractos proteicos solubles de promastigotes
Se utilizaron 400 μL de resina tiofílica (Pierce® Thiophilic Adsorbent -Thermo Scientific) previamente equilibrada (3 lavados de 300 μL durante 10 min sobre hielo en plataforma de agitación [LabNet]) en buffer IP 2X (tris-HCL 50 mM pH 7,5, NaCl 150 mM, MgCl2 5 mM y glicerol 5% v/v) y distribuida en dos tubos de 1,5 mL con 200 μL de resina cada uno. Se realizó el proceso de aclarado, permitiendo interacción de 2 mg de fracción protéica soluble de promastigotes de L. braziliensis en relación 1:1 con buffer IP 2X, con 200 μL de resina durante 3 h sobre hielo en plataforma de agitación y en presencia de inhibidor de proteasas (Sigma P8340). Posteriormente, se centrifugó (Thermo Heraeus Megafuge 16R) a 2000 rpm durante 3 min a 4 °C, retirando el sobrenadante a un tubo nuevo de 1,5 mL y adicionando 4 mg del anticuerpo α6xHis-SUMO-LbTXNPxII-IgY. Esta mezcla se incubó toda la noche sobre hielo en plataforma de agitación; siendo agregada finalmente a los 200 μL de resina restante. La mezcla se incubó durante 2 h sobre hielo en plataforma de agitación, conservando el sobrenadante como fracción de "proteínas no unidas". Se realizaron 3 lavados con 500 μL de buffer IP 2X de 10 min sobre hielo en plataforma de agitación, conservando el sobrenadante para posterior análisis. Finalmente, las muestras inmunoprecipitadas se resuspendieron en 200 μL de buffer de carga para SDS-PAGE 1X, se incubaron a 96 °C durante 10 min y se centrifugaron a 3000 rpm durante 3 min, el sobrenadante se conservó como "proteínas unidas". El proceso se monitorizó mediante SDS-PAGE 10% con tinción de plata y Western blot, utilizando los anticuerpos primarios α6xHis-SUMO-LbTXNPxII-IgY generados y α6xHis-LbNMNAT-IgG [35], [36].
Procedimientos bioinformáticos
Se realizó un alineamiento múltiple con el programa CLC Main Workbench (versión 7), utilizando secuencias de ortólogos depositadas en NCBI para la TXNPx en los géneros Leishmania y Trypanosoma. La predicción de la localización subcelular del candidato LbTXNPxII se adelantó mediante los servidores Euk-mPLoc 2.0, SignalIP 5.0 y DeepLoc-1.0. Por su parte, la construcción del modelo estructural del candidato se completó utilizando el método de "Threading" con el servidor I-TASSER [37]. El modelo se refinó con el servidor 3Drefine [38], mientras que su visualización se realizó con el programa (UCSF Chimera) [39]. En relación con la predicción de estructuras cuaternarias para el candidato, se emplearon las herramientas SWISS-MODEL [40] y GalaxyHomomer [41].
Resultados
Análisis in silico del candidato a triparedoxina peroxidasa de L. braziliensis (LbTXNPxII)
El alineamiento múltiple realizado a partir de secuencias de ortólogos para la TXNPx en la familia Trypanosomatidae (géneros Leishmania y Trypanosoma) indicó que esta proteína es altamente conservada en dicho grupo de organismos (Figura 1). A excepción de los ortólogos de los parásitos T. conorhini y T. rangeli, el tamaño de la proteína es de 199 aa. Teniendo en cuenta la función fundamental que esta cumple en la neutralización de compuestos prooxidantes, los cuales pueden ser producidos de manera endógena por la cadena respiratoria o exógena como respuesta del hospedero a la infección, dos isoenzimas han sido identificadas en organismos como L. donovani y L. infantum ubicadas en citoplasma y mitocondria [42]. Para el caso de L. braziliensis, de Morais et al, en el 2013 identificaron la proteína mitocondrial designada como LbTXNPx [8]. Continuando con la caracterización de estas enzimas y utilizando servidores como Euk-mPLoc 2.0, SignalIP 5.0 y DeepLoc-1.0, se encontró a nivel in silico un nuevo candidato con posible localización citoplasmática denominado LbTXNPxII [43], [45].
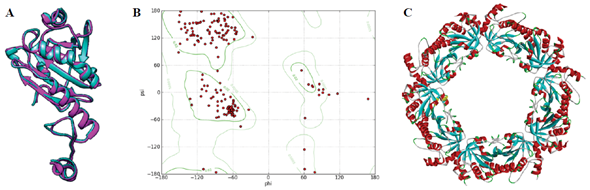
Figura 1 Alineamiento múltiple de los ortólogos TXNPx en la familia Trypanosomatidae (Imagen obtenida con el programa CLC Main Workbench).
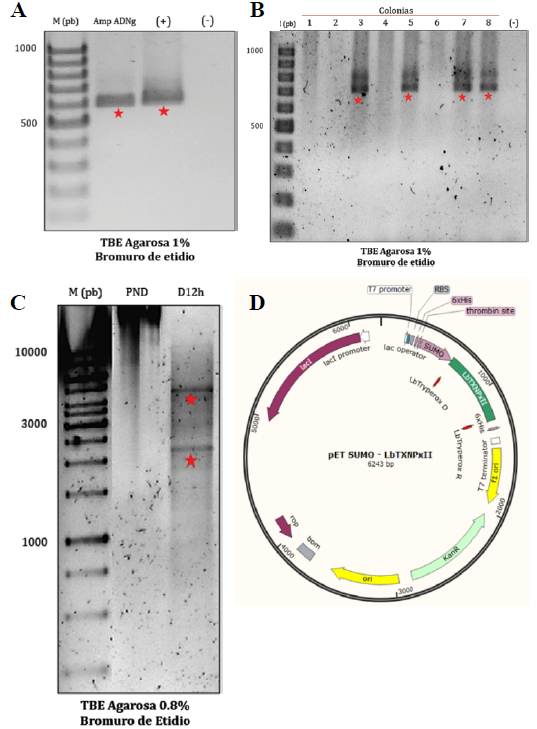
Figura 2 Análisis bioinformatico del candidato LbTXNPxII. A. Superposición estructural (RMSD= 0.425Å) de la TXNPx cristalizada de L. mayor en magenta (PDB 4K1F) con la predicción realizada en el servidor I-TASSER y refinada en el servidor 3Drefine, para el candidato LbTXNPxII en azul (UCSF Chimera). B. Validación del modelo obtenido para el candidato LbTXNPxII mediante gráfico de Ramachandran (Imagen obtenida con el programa Ramachandran Plot [48]). C. Predicción de oligomerización del candidato LbTXNPxII, estructura compuesta por la unión de 5 homodímeros (Swiss-Model y GalaxyHomomer) [37], [39], [46], [47].
Por otro lado, con respecto al análisis in silico del candidato encontrado, el servidor I-TASSER se utilizó para realizar la predicción tridimensional de la estructura de la proteína LbTXNPxII (Figura 2 A.), este emplea el método de "Threading" partiendo de una estructura cristalizada seleccionada mediante homología [37], [39], [46], [47]. Al valorar dicho modelo mediante el gráfico de Ramachandran, se obtuvo el 94,4% de los aminoácidos que componen la proteína en la región favorecida, luego de que el modelo se refinó mediante el servidor 3Drefine [48] (Figura 2.B.). Por último, partiendo de análisis de oligomerización realizados en L. mayor, donde se predice una posible formación de homodecámeros in vivo para llevar a cabo su función, se utilizaron los servidores SWISS-MODEL y GalaxyHomomer a fin de realizar este análisis para el candidato LbTXNPxII, obteniendo la posible formación de un pentámero de homodímeros, conformando finalmente, una estructura cuaternaria de homodecámeros (Figura 2. C.) [41], [49], [50].
Producción de la proteína recombinante 6xHis-SUMO-LbTXNPxII en el sistema heterólogo E. coli
La secuencia LbTXNPxII de 600 pb se amplificó a partir de ADN genómico mediante PCR con Taq polimerasa (Figura 3. A.). Posteriormente, el producto amplificado se clonó en el vector de expresión bacteriano pET-SUMO. Al tratarse de un vector tipo -TA-, la direccionalidad del inserto se corroboró realizando PCR con un oligonucleótido directo ubicado en la región SUMO corriente arriba del sitio de clonación múltiple, junto con el oligonucleótido reverso diseñado para la amplificación del candidato, esperando así un aumento en el tamaño del amplificado de 100 pb; es decir, una banda de alrededor de 700 pb (Figura 3 B.). Finalmente, utilizando el servidor NEBcutter2.0 se seleccionaron las enzimas de restricción (BamHI y EcoRV) para corroborar la identidad del plásmido recombinante mediante un perfil diferencial en comparación con el plásmido vacío (Figura 3 C.) [22]. El mapa del vector recombinante construido se indica en la Figura 3. D.
Figura 3 Construcción del vector recombinante pET SUMO- LbTXNPxII. A. Amplificación de la secuencia de interés de 600pb a partir de ADN genómico. B. Evaluación de las colonias obtenidas tras la transformación, de las cuales 4 resultaron direccionadas en el vector (700pb). C. Evaluación del perfil de digestión del plásmido, obteniendo una banda de ~ 4201pb y otra en ~ 2042pb, corroborando así la presencia del inserto e identidad del plásmido recombinante. D. Representación del vector recombinante pET SUMO- LbTXNPxII construido (Imagen obtenida con el programa SnapGene).
La TXNPx es una enzima clave para la resistencia de los parásitos al estrés oxidativo, desempeñando un importante papel antioxidante. Adicionalmente, posee una alta divergencia a nivel de ortólogos en otros grupos animales, para el caso de las posibles especies hospederas como animales salvajes, domésticos o hasta los propios seres humanos, estas enzimas poseen un homólogo distante que se desvía a otra clase [14]. Por lo tanto, estudiar el candidato desde la aproximación de proteínas recombinantes resulta ser enriquecedor para entender función y localización en L. braziliensis en aras de caracterizar la bioquímica y rutas metabólicas de estos organismos.
Tras la corroboración de la identidad del plásmido pET SUMO-LbTXNPxII, este se utilizó para la transformación de la cepa de expresión BL21(DE3) [51]. Posterior a la inducción con IPTG se encontró que a partir de las 2 h se observa en el SDS-PAGE una evidente expresión de la proteína en el peso esperado (37 kDa) (Figura 4 A.); sin embargo, teniendo en cuenta que a las 4 h se observa una mayor cantidad, este tiempo se implementó para realizar el fraccionamiento celular (Figura 4. B.). Finalmente, al realizar la detección de la 6xHis-SUMO-LbTXNPxII mediante Western blot, se obtuvo señal tanto en la fracción soluble como insoluble del sistema de expresión (Figura 4 C.).
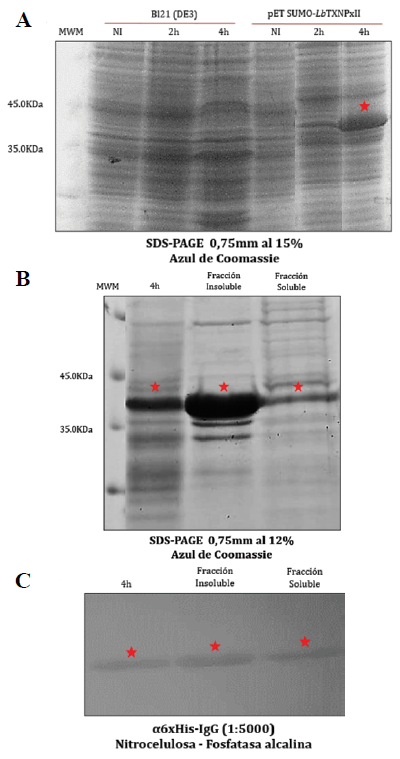
Figura 4 Expresión de la proteína 6xHis-SUMO-LbTXNPxII en la cepa E. coli BL21(DE3). A. Evaluación de horas de inducción (NI: No inducidas, Horas de inducción monitorizadas 2 h y 4 h), teniendo como control negativo células BL21(DE3) no transformadas. La cantidad de muestra cargada se normalizó con base en la OD de las células al momento de su recolección. B. Fraccionamiento celular a partir de la muestra de 4 h de inducción. C. Corroboración de la identidad de la proteína 6xHis-SUMO-LbTXNPxII sobre expresada en proteínas totales y fracciones celulares mediante Western blot.
Purificación de la proteína 6xHis-SUMO-LbTXNPxII mediante cromatografía de afinidad a metales inmovilizados (IMAC) y SDS-PAGE preparativo
Aprovechando la etiqueta 6xHis conferida por el vector, la proteína 6xHis-SUMO-LbTXNPxII se purificó empleando resina de Níquel (Ni), obteniendo la mayor cantidad de proteína purificada en los eluidos 5 - 7, 0,786 mg a partir de 300 mL de inducción (Figura 5 A.). Por otro lado, aprovechando la cantidad abundante de la proteína 6xHis-SUMO-LbTXNPxII en la fracción insoluble, se procedió a realizar su purificación a partir de cuerpos de inclusión utilizando SDS-PAGE preparativo, encontrando que, posterior al paso de diálisis, la proteína permanecía tanto en fracción soluble como insoluble, por lo que se decidió utilizar ambas fracciones para la separación preparativa (Figura 5 B.). Finalmente, se obtuvieron 1,22 mg de proteína eluída desde un pellet de 2,1 g de cuerpos de inclusión.
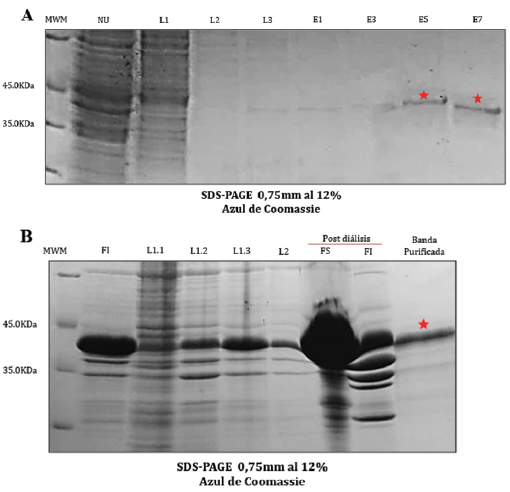
Figura 5 Purificación de la proteína 6xHis-SUMO-LbTXNPxII (37kDa). A. Purificación a partir de la fracción soluble empleando cromatografía de afinidad a metales inmovilizados (IMAC) (NU: No unidas, Lx: Número del lavado, Ex: Número del eluído). B. Purificación a partir de la fracción insoluble (cuerpos de inclusión) empleando SDS-PAGE preparativo (FI: Fracción insoluble, FS: Fracción soluble, Lx: Número del lavado).
Producción, evaluación y caracterización de anticuerpos policlonales aviares «6xHis-SUMO- LbTXNPxII-IgY
Gallinas de la raza Hy-Line Brown de 35 semanas se utilizaron para la producción de anticuerpos policlonales contra el candidato 6xHis-SUMO-LbTXNPxII. Durante el esquema de inmunización se recolectaron cuatro sueros, uno preinmune (SPI) y tres post-inoculación, los cuales muestran una clara señal en la detección de la proteína recombinante purificada (Figura 6 A.). Por otro lado, utilizando el suero 1 (S1) se procedió a realizar la caracterización de los anticuerpos, encontrando que son capaces de reconocer 250 ng de la proteína 6xHis-SUMO-LbTXNPxII en una dilución de hasta 1 en 10000 (Figura 6.B). Adicionalmente, detectando la cantidad mínima de antígeno reconocido en una dilución de 1 en 5000 se encontró que estos anticuerpos son capaces de reconocer hasta 18 ng bajo estas condiciones (Figura 6.C).
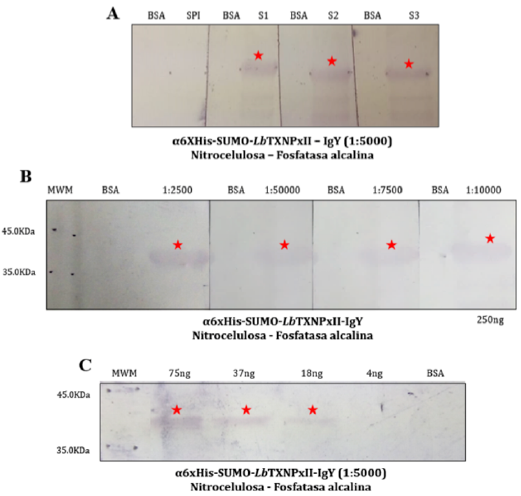
Figura 6 Evaluación de anticuerpos policlonales α6xHis-SUMO-LbTXNPxII-IgY, presentes en sueros sanguíneos. A. Reconocimiento del antígeno (250 ng) por parte de los sueros α6xHis-SUMO-LbTXNPxII-IgY. (BSA: Albumina de suero bovino 250 ng, SPI: Suero pre inmune, SX: Número del suero evaluado del 1-3). B. Caracterización de los anticuerpos en suero, dilución de trabajo sobre 250 ng de antígeno. C. Caracterización de los anticuerpos en suero, cantidad mínima de antígeno reconocida.
El modelo aviar permite la obtención de los anticuerpos policlonales de interés tanto a partir de sangrías del animal como de los huevos que este produce. Durante el esquema de inmunización realizado se recolectaron 36 huevos. Posterior a su procesamiento, las muestras se analizaron por espectrofotometría (280 nm) con el propósito de estimar la concentración aproximada (mg/mL) de proteínas totales presentes en cada una. Seguidamente, se realizó la titulación de estas mediante ELISA indirecto, en el cual se determinaron las muestras con mayor cantidad de anticuerpos específicos contra la 6xHis-SUMO-LbTXNPxII, seleccionadas para la completa purificación. Es evidente que las muestras que presentan mayor concentración de proteínas totales no necesariamente son las que poseen mayor cantidad de los anticuerpos de interés; adicionalmente, existe una clara respuesta del sistema ante la inoculación, observándose los picos más altos (Abs 405 nm) posterior a esta (Figura 7. A). Finalmente, los anticuerpos α6xHis-SUMO-LbTXNPxII-IgY purificados a partir de la yema de los huevos se evaluaron mediante Western blot, obteniendo señales claras y específicas en el reconocimiento de la proteína recombinante (Figura 7 B).
Inmunodetección de la proteína LbTXNPxII endógena (23kDa) en extractos del parásito.
La implementación de los anticuerpos α6xHis-SUMO-LbTXNPxII-IgY purificados anteriormente en ensayos de Western blot sobre extractos totales de L. braziliensis permitió detectar la proteína LbTXNPxII endógena con un peso molecular aproximado de 23 kDa, utilizando como control positivo del ensayo la proteína recombinante 6xHis-SUMO-LbTXNPxII producida y purificada (37 kDa) (Figura 8). La inmunodetección de la proteína endógena resultó específica, dado que la implementación del suero preinmune (SPI) en el ensayo de Western blot no generó señales sobre la membrana del ensayo. Adicionalmente, al cargar BSA como control negativo de detección, no hubo reconocimiento de la misma.
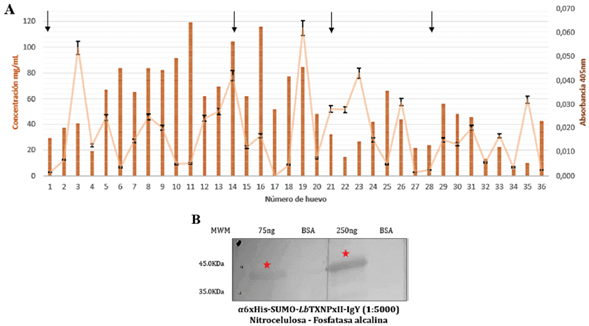
Figura 7 Producción de anticuerpos policlonales α6xHis-SUMO-LbTXNPxII-IgY en huevos. A. Titulación mediante ELISA indirecto de los anticuerpos presentes en yema de huevos recogidos durante el esquema de inmunización (las flechas negras indican días de inoculación). Los datos de absorbancia a 405 nm indican el promedio y la desviación estándar (n= 3). B. Evaluación de los anticuerpos α6xHis-SUMO-LbTXNPxII-IgY provenientes de yema de huevo mediante Western blot.
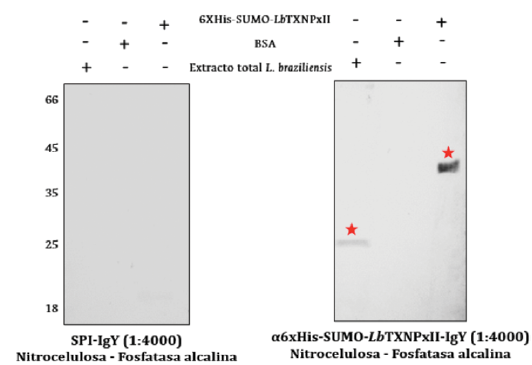
Figura 8 Inmunodetección de la proteína LbTXNPxII endógena sobre extractos proteicos totales de L. braziliensis. Se analizaron extractos proteicos totales del estadio infectivo (promastigote) del parásito implementando el suero preinmune (SPI) o los anticuerpos α6xHis-SUMO-LbTXNPxII-IgY purificados sobre extractos proteicos de promastigotes, BSA y la proteína recombinante a6xHis-SUMO-LbTXNPxII.
Por otro lado, empleando técnicas de inmunofluorescencia indirecta sobre promastigotes de L. braziliensis y con el uso de los anticuerpos α6xHis-SUMO-LbTXNPxII-IgY producidos y caracterizados se encontró que el candidato exhibe un patrón de distribución citoplasmático en la fase móvil del parásito, como sugirió el modelo in silico anteriormente presentado acerca de la localización subcelular de la proteína (Figura 9).
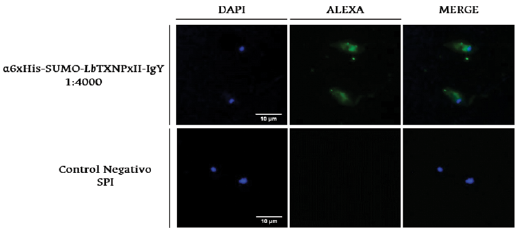
Figura 9 Inmunolocalización celular de la LbTXNPxII endógena sobre promastigotes. Análisis de inmunofluorescencia sobre parásitos de L. braziliensis fijados con paraformaldehído. SPI: Suero preinmune.
Análisis realizados por Joya et al. en el 2019 por Co-IP/MS-MS sugirieron una posible interacción entre la triparedoxina peroxidasa (TXNPxII) y la nicotinamida/nicotinato mononucleótido adenililtransferasa (NMNAT), enzima clave en el metabolismo del dinucleótido de nicotinamida y adenina NAD en L. braziliensis. Para corroborar dicha interacción, los anticuerpos policlonales α6xHis-SUMO-LbTXNPxII-IgY generados se utilizaron en la inmunoprecipitación e inmunodetección cruzada de estas enzimas, observándose para cada caso la detección de estas (Figura 10), confirmando esta conexión molecular.
Discusión
Considerando la importancia de los sistemas de defensa anti-oxidante para la supervivencia de Leishmania dentro del macrófago del hospedero, se detectó la triparredoxina peroxidasa citoplasmática de L. braziliensis (LbTXNPxII), la cual participa en la neutralización de ROS y RNI a expensas de equivalentes reductores derivados de la tripanotiona. Adicionalmente, se exploró su interacción molecular con enzimas propias de la síntesis de NAD, como la nicotinamida/nicotinato mononucleótido adenilil transferasa (LbNMNAT).
La caracterización in silico del candidato LbTXNPxII concluyó que, en general, corresponde a una enzima altamente conservada entre ortólogos, con un tamaño constante de 199 aa (Figura 1) y predominantemente expresada en el estado amastigote como respuesta a la reproducción asexual que se da durante este estadio dentro del compartimento fagolisosómico [52]. Así mismo, el promastigote representa un buen modelo de estudio puesto que se expone a estrés oxidativo tanto en el intestino del vector como durante la interacción con los macrófagos tras la transmisión al hospedero mamífero (15); motivo este por el cual la LbTXNPxII endógena de 23 kDa con distribución citosólica es inmunodetectada también en dicho estado.
La inmunodetección reportada en este trabajo requirió la construcción de un vector de expresión apropiado para el sistema heterólogo E. coli BL21 (DE3), el cual resultó en la producción abundante de la proteína recombinante 6xHis-SUMO-LbTXNPxII. Este sistema representa una fuente biológica para obtener insumos útiles en la producción de herramientas inmunológicas como anticuerpos policlonales en diversos sistemas animales. En este sentido, la proteína en mención se aprovechó como antígeno para la producción aviar de IgYs (Figuras 6 y 7), destacándose su aplicación en la detección de la proteína citoplasmática LbTXNPxII del parásito (Figuras 8 y 9). Adicionalmente, esta herramienta podrá utilizarse en otros estudios de inmunodetección en el estado amastigote, constituyendo un reactivo útil puesto que no se dispone de anticuerpos comerciales para estos fines en Leishmania.
Las enzimas triparredoxina peroxidasas, como la detectada en el presente estudio, poseen cisteínas (Cys) conservadas en las posiciones 52 y 173, involucradas con el mecanismo de acción, la estabilidad de la conformación nativa y la formación de oligómeros propiamente homodecámeros [13]. Adicionalmente, Barr y Gedamu en 2001 determinaron que las peroxirredoxinas de L. chagasi pueden existir como multímeros de alto peso molecular in vivo, fenómeno observado también en ortólogos de L. donovani y T. cruzi [52]. Estas evidencias concuerdan con la predicción de estructura cuaternaria realizada para la enzima LbTXNPxII (Figura 2 C).
Para la contención de diversas especies reactivas en el proceso de infección, el parásito depende de isoenzimas mitocondriales y citoplasmáticas de la TXNPx, las cuales, a su vez, dependen del NADPH para catalizar sus reacciones. Estudios previos han reportado en L. braziliensis la interacción molecular entre proteínas propias de los sistemas antioxidantes con aquellas involucradas en la síntesis del NAD, como la NMNAT [12]. Dicha evidencia se confirmó en el presente estudio (Figura 10), en el cual se hace patente la importancia de profundizar el estudio acerca de la relación existente entre vías metabólicas y de regulación bajo situaciones de estrés REDOX. Aunque se desconoce si el aumento en los niveles de estrés oxidativo podría estimular la síntesis de NAD y subsecuentemente de NAD(P), su estudio será necesario para identificar posibles objetivos moleculares para el desarrollo de fármacos contra el parásito.
Conclusiones
En respuesta a la importancia del sistema TR-TXN-TXNPx para la neutralización de ROS y RNI relacionada con supervivencia y capacidad de infección, así como para el desarrollo y virulencia de parásitos Tripanosomátidos, se detectó la triparredoxina peroxidasa citoplasmática de L. braziliensis (LbTXNPxII), una enzima de 23 kDa altamente conservada. Para ello se produjo y purificó la proteína recombinante 6xHis-SUMO-LbTXNPxII utilizada como antígeno para la producción de anticuerpos policlonales aviares que facilitaron su caracterización en el estadio infectivo del parásito (promastigote) y, adicionalmente, permitió la corroboración de la interacción LbTXNPxII- LbNMNAT conectando el metabolismo REDOX con la síntesis de NAD.















