Introducción
Giardia duodenalis (syn. lamblia e intestinalis) es un eucariote basal de interés médico, ya que es el agente causal de la giardiasis, la enfermedad gastrointestinal de mayor incidencia mundial. Esta enfermedad ataca tanto a animales de interés agropecuario y veterinario como a personas de todas las edades en diferentes regiones del mundo; los principales afectados son niños de países en vías de desarrollo. Actualmente no se cuenta con una vacuna para prevenir esta enfermedad y los tratamientos existentes resultan poco eficientes por los efectos secundarios que producen y por su costo; por lo tanto, urge la búsqueda de nuevas dianas terapéuticas que permitan el desarrollo de estrategias de control efectivas contra esta parasitemia.
Un punto de partida en dicha búsqueda corresponde al estudio del metabolismo energético y sus enzimas, teniendo en cuenta que están encargadas de regular procesos vitales en todos los organismos. Dentro del metabolismo energético pueden destacarse las rutas de biosíntesis y catabolismo del dinucleótido de adenina y nicotinamida (NAD), que involucra enzimas como las sirtuinas, las cuales están asociadas a la regulación de funciones vitales como la reparación de daños en el DNA, la progresión del ciclo celular, la longevidad celular, entre otras.
Las sirtuinas (E.C. 2.4.2) son proteínas globulares que tienen un tamaño muy variable; sin embargo, una de sus características más importantes es la presencia de un dominio catalítico conservado de aproximadamente 250 aminoácidos, conocido como plegamiento sirtuina y que consta de dos regiones: una pequeña y otra grande, que en conjunto permiten que la proteína lleve a cabo su actividad catalítica.
Dada la estructura conservada del dominio catalítico, las sirtuinas presentan también un mecanismo de reacción conservado en todos los organismos en donde han sido caracterizadas. Dicha reacción consiste en la remoción de un grupo acetilo de los residuos lisina de diferentes proteínas usando como cosustrato NAD+ y generando como productos nicotinamida, 2'-O-Acetyl-ADP-Ribosa (2'AADPR) y el péptido deacetilado.
El estudio de las sirtuinas en organismos parásitos, puntualmente protozoarios, es un campo poco explorado, pero promisorio, ya que la evidencia que se tiene a la fecha demuestra que, además de las funciones típicas, estas proteínas les otorgan ventajas adaptativas como la variación antigénica y formas alternativas para la progresión del ciclo celular. Estudios previos del Laboratorio de Investigaciones Básicas en Bioquímica (Libbiq) permitieron la identificación bioinformàtica de cinco genes candidatos a sirtuina en el genoma de G. duodenalis, se ha realizado la evaluación experimental de los dos primeros, GdSir2.1 y GdSir2.2, y se encontró que son proteínas de la familia sirtuina debido a sus características estructurales y sus funciones enzimáticas.
Teniendo en cuenta la posible importancia de las sirtuinas en este organismo, se planteó este trabajo para identificar un nuevo candidato a sirtuina en G. duodenalis, mediante herramientas bioinformáticas y experimentales, con el fin de ampliar el conocimiento sobre la bioquímica de este parásito, para proponer nuevos blancos terapéuticos que permitan desarrollar estrategias de control efectivas contra esta parasitemia.
Materiales y métodos
A partir de la secuencia del candidato a sirtuina (GdSir2.3), identificado previamente en bases de datos, se realizaron dos aproximaciones para su caracterización:
Aproximación bioinformática
Búsqueda de dominios conservados
La secuencia candidato GdSir2.3 (GL50803_16569) fue utilizada para un alineamiento múltiple con las siete sirtuinas humanas (códigos de acceso UniProt: Q96EB6; Q8IXJ6; Q9NTG7; Q9Y6E7; Q9NXA8; Q86T7; Q9NRC8) y los cinco candidatos a sirtuina de Giardia, de los cuales se han identificado experimentalmente dos (códigos de acceso GiardiaDB: GL50803_10708, GL50803_10707, GL50803_16569, GL50803_11676, GL50803_6942.) Dicho alineamiento múltiple se realizó y visualizó con el software CLC MainWorkbench v. 7. 8. Adicionalmente, la secuencia candidato se evaluó en los servidores Conserved Domains Database y Pfam [1] para identificar dominios conservados pertenecientes a la familia sirtuina.
Análisis estructural del candidato
A partir de la secuencia de aminoácidos del candidato (GdSir2.3) se construyeron diferentes modelos de estructura terciaria basados en los algoritmos de homología (Swiss Model) [2], threading (I - Tasser) [3] y ab initio (Robetta) [4]. Los modelos obtenidos fueron validados mediante los puntajes de cada servidor, gráfico de Ramachandran con el servidor Rampage [5] y por superposición con la estructura cristalizada de la SIRT5 de Homo sapiens, la sirtuina con mayor homología con el candidato. Adicionalmente, se realizó la superposición de estos con la sirtuina 1 humana (SIRT1). Los modelos y las superposiciones fueron visualizados en el software UCSF Chimera 1. 11. 2 65.
Predicción de modificaciones postraduccionales del candidato
Se realizó la predicción de fosforilación del candidato empleando los servidores NetPhos 3.1 [6] y GPS 5.0 [7]. Dichas predicciones se realizaron con la secuencia de aminoácidos de la proteína y se evaluaron siguiendo los parámetros establecidos por cada uno de los servidores.
Aproximación experimental
Clonación del gen GdSir2.3
El gen GdSir2.3 se amplificó mediante PCR bajo las siguientes condiciones de reacción: 2 mM de Buffer de PCR 1X (20 mM Tris-HCl pH 8,8, 10 mM KCl, 0,1% (v/v) Triton X-100, 0,1 mg/ml de BSA), 0,2 mM de dNTPs, 0,2 pM de primer directo (5'- CAC CAT GGT ACC CGA TGT AGA TG -3') y reverso (5'- GTC GAC CTA ATC TAT ATT GAG AAT AGA CG -3'), 1U de Pfu polimerasa, 50 ng de DNA genómico de G. duodenalis y H2O DEPC, para un volumen final de 25 μL. El ciclo térmico de PCR consistió en una etapa de desnaturación a 95 °C durante 5 min, seguida por 25 ciclos de 45 s a una temperatura de 56 °C y con una etapa de extensión de 7 min a 72 °C. Los resultados fueron visualizados mediante geles de TBE - Agarosa 1% p/v y documentados en el analizador de imágenes Imager® Gel DocTMXR, con el software Quantity One Basic 4.6.3 de Bio-Rad™. El producto de PCR fue ligado en el vector pET100/D-TOPO y transformado en células químicamente competentes E. coli cepa TOP10 de acuerdo con las instrucciones del fabricante [8]. Los plásmidos recombinantes se extrajeron mediante lisis alcalina [9] y se confirmó su identidad por PCR y digestión con enzimas de restricción [10].
Expresión y purificación de la proteína recombinante 6xHis-GdSir2.3
El plásmido recombinante se usó para la transformación de células competentes E. coli de la cepa SHuffle [11]. Las células se incubaron a 37 °C con agitación constante en medio líquido LB suplementado con glucosa 1% m/v, ZnCl2 2,5 μM y 100 pg/mL de ampicilina hasta alcanzar una OD600 de 0,6. En este punto se adicionó el inductor (isopropil-β-D-l-tiogalactopiramsido, IPTG) a concentración final de 0,1 μM, se incubaron las células durante 4 h y se colectaron mediante centrifugación a 6000 rpm durante 10 min a 4 °C.
Los pellets obtenidos se resuspendieron en buffer de lisis (50 mM buffer fosfatos pH 7.8, NaCl 400 mM, KCl 100 mM, glicerol 10%, Triton X-100 0,5% e imidazol 10 mM) agregando 5 mL de buffer por cada gramo de células y lisozima a una concentración final de 1 mg/mL. Se incubó la mezcla durante media hora en baño de hielo con agitación constante, se lisaron las células en ausencia de inhibidor de proteasas mediante sonicación durante 10 min (15 s de pulso, 15 s de reposo) y se centrifugaron a 14.000 G durante 20 min a 4 °C para separar la fracción soluble (sobrenadante) y la fracción insoluble (pellet). Los resultados de la expresión fueron evaluados mediante SDS - PAGE y western blot [12].
La fracción soluble obtenida en la lisis fue incubada con 5 volúmenes de resina Ni - NTA Agarose empacada durante 4 h con agitación constante a 4 °C. Luego se empacó la mezcla en una columna de cromatografía y se realizaron los siguientes lavados: un primer lavado de 1 mL con buffer de lisis (50 mM buffer fosfatos pH 7,8, NaCl 400 mM, KCl 100 mM, glicerol 10%, Triton X-100 0,5% e imidazol 10 mM), dos lavados de 1 mL cada uno con el buffer de lavado 1 (50 mM buffer fosfatos pH 7.8, NaCl 400 mM, KCl 100 mM, glicerol 10%, Triton X-100 0,5% e imidazol 35 mM) y dos lavados de 1 mL cada uno con el buffer de lavado 2 (50 mM buffer fosfatos pH 7,8, NaCl 400 mM, KCl 100 mM, glicerol 10%, Triton X-100 0,5% e imidazol 75 mM). La proteína recombinante se eluyó de la columna con 500 pL del buffer de elución (50 mM buffer fosfatos pH 7,8, NaCl 400mM, KCl 100 mM, glicerol 10%, Triton X - 100 0,5% e imidazol 300 mM) [13]. El resultado fue monitoreado mediante SDS - PAGE y los eluidos fueron cuantificados por el método colorimétrico de Bradford y densitometría.
Paralelamente, se realizó la purificación de la proteína 6xHis-GdSir2.3 desde los cuerpos de inclusión mediante SDS - PAGE preparativo [14] y por cromatografía de afinidad bajo condiciones desnaturantes [13]. La proteína purificada fue dializada para remover el imidazol siguiendo el protocolo de Herrera y colaboradores (2019) [15].
Producción de anticuerpos policlonales contra la proteína recombinante 6xHis-GdSir2.3 en modelo aviar
Se inocularon dos gallinas de la raza Hyline de 50 semanas bajo el siguiente esquema de inmunización: para la primera inoculación el individuo control fue inyectado con PBS y adyuvante completo de Freund en una relación 1:1 para un volumen final de inyección de 300 μL; por otra parte, el individuo tratamiento fue inyectado con 100 μg de 6xHis-GdSir2.3 (antígeno purificado) y adyuvante completo de Freund para una proporción y volumen final iguales que en el individuo control. El primer refuerzo de inmunización se realizó a los 15 días de la primera inoculación, empleando en esta ocasión adyuvante incompleto de Freund para ambos individuos. Los siguientes dos refuerzos se realizaron cada 8 días [16]. Una vez finalizado el esquema de inmunización, se evaluó la capacidad de reconocimiento de las sangrías mediante western blot con 6xHis-GdSir2.3 y se purificaron los anticuerpos por afinidad a antígeno [17].
Cultivo celular y enquistación de G. duodenalis
Con el objeto de identificar la proteína endógena en el parásito en sus diferentes estadios de vida trofozoítos de G. duodenalis cepa WB fueron cultivados a 37 °C en medio TYI-S-3368 [18] hasta alcanzar la fase logarítmica. Para la recolección de los parásitos se incubaron los tubos de cultivo a 4 °C durante 1 h y luego se centrifugaron a 3000 rpm en el rotor Microliter 30x2 sealed de la centrífuga Heraeus Megafuge 16R de ThermoFisher durante 15 min. Se lavó el pellet hasta 3 veces con PBS 1X para retirar el exceso de medio de cultivo, se resuspendieron los parásitos en a medio TYI - S - 33 y se incubaron los parásitos a 37 °C. Una vez pasadas 24 h, se retiró el medio de cultivo y se incubaron los parásitos en agua para lisar por choque hipotónico las células que no se hubieran enquistado [19]. Los quistes resultantes se lavaron dos veces con PBS 1X filtrado, se resuspendieron en 1 mL de PBS 1x filtrado y se almacenaron a -20 °C.
identificación de GdSir2.3 en extractos celulares e inmunofluorescencia
Con el objetivo de identificar la proteína nativa GdSir2.3 se recolectaron trofozoítos asincrónicos y durante las 24 horas de la enquistación. Los extractos fueron evaluados mediante western blot empleando el anticuerpo IgY - α - 6xHis - GdSir2.3 (1:50) siguiendo el protocolo de Herrera y colaboradores (2019) [15], usando el sistema super signal west substrate (Thermo). Como control de enquistación se reconocieron las proteínas de la pared del quiste CWP1 empleando un anticuerpo comercial contra estas en una dilución de 1:500.
Para establecer la ubicación subcelular de GdSir2.3 se realizaron ensayos de inmunofluorescencia. Se resuspendieron los trofozoítos y quistes en PBS 1X para una concentración final de 1000 parásitos/μL. Se sembraron 10 pL de células sobre una lámina de vidrio y se unieron por incubación a 37 °C durante 1 h. Las células se fijaron con paraformaldehído 4% (m/v) y se bloquearon e impermeabilizaron durante 2 h a 37 °C con solución de bloqueo (Triton X - 100 0,5% (v/v), BSA 3% (m/v) en PBS). Se adicionó el anticuerpo primario α-6xHis-GdSir2.3 IgY a una dilución de 1:100 en PBS y se incubó durante toda la noche a 37 °C. Así mismo, se adicionó el anticuerpo primario a-CWP conjugado a TAMRA (1:250 en PBS) sobre los extractos de quistes bajo el mismo tratamiento del anticuerpo anterior. Como anticuerpo secundario para α-6xHis-GdSir2.3 IgY se adicionó el a-IgY acoplado a Alexa Fluor (ThermoFisher) 488 (1:1000 en PBS). Se uso un anticuerpo comercial contra alfa tubulina acetilada como control (1:500) y se empleó el a-mouse acoplado a Dylight (ThermoFisher) (1:1000 en PBS), los anticuerpos secundarios se incubaron durante 1 h a 37 °C en oscuridad y posteriormente se lavó la lámina dos veces con PBS. Finalmente, se marcaron los núcleos con 4',6-diamidino-2-fenilindol (DAPI, 1 pg/pl) durante 3 min a temperatura ambiente, se retiró el exceso de DAPI con agua MQ y se almacenó la lámina a 4 °C en oscuridad para su posterior registro en el microscopio de fluorescencia Nikon C1 con fluoromount y un objetivo de 40X. Las imágenes obtenidas con el microscopio fueron procesadas con el software ImageJ [20].
Evaluación de la actividad deacetilasa de la proteína recombinante 6xHis-GdSir2.3
Para evaluar la actividad enzimática de 6xHis-GdSir2.3 se usó el kit CycLex SIRT1/Sir2 Deacetylase Fluorometric Assay Kit Ver. 2 siguiendo las indicaciones del fabricante [21], empleando 300 ng de proteína recombinante en presencia y ausencia de 8 mM NAD+ y teniendo como control la SIRT2 de Homo sapiens [21]. Se realizaron tres ensayos independientes de deacetilación en el equipo CFX96 Touch Real-Time PCR Detection System (BioRad) con una longitud de onda de excitación de 488 nm y la lectura de fluorescencia a 521 nm.
Resultados y discusión
El candidato GL50803_16569 (GlSir2.3) presenta dominios conservados de la familia sirtuina
Al analizar la secuencia de aminoácidos correspondiente al candidato GL50803_16569 (381 aminoácidos, 43,9 kDa) mediante alineamientos múltiples con sirtuinas ya caracterizadas se encuentra el motivo G - A - G y los residuos de cisteína característicos de las sirtuinas hacia la región amino terminal que corresponde a un plegamiento tipo Rossman, compuesto por una serie de hélices α y láminas β antiparalelas con características típicas de unión a nucleótidos. En esta región se une el dinucleótido de adenina y nicotinamida (NAD+) como cosustrato, adyacente al sitio de unión de la acetil-lisina [22] (Figura 1).

Figura 1 Alineamiento múltiple de las secuencias de las sirtuinas de G. duodenalis (GdSir2.3, GdSir2.1, GdSir2.2, GdSir2.4 y GdSir2.5, respectivamente) con las sirtuinas humanas (SIRT1 - 7). Se presenta el alineamiento parcial de secuencias en donde se observa señalado en el recuadro rojo el motivo G - A - G y en amarillo las cisteínas conservadas. Realizado con CLC Main Workbench V. 7.8.
La presencia del dominio G-A-G y las cuatro cisteínas en la secuencia candidata podría indicar que se trata de una sirtuina, ya que se ha encontrado que los residuos de cisteína se ubican en la región pequeña del dominio catalítico coordinando un átomo de zinc, que, si bien no participa en la catálisis, resulta indispensable para la actividad enzimática como lo han demostrado diferentes estudios donde al mutar estos residuos o adicionar agentes quelantes se da la pérdida de la actividad deacetilasa de la proteína [23]. Al observar el alineamiento también se encuentran otras regiones de alta homología entre las sirtuinas del parásito y las humanas; no obstante, estas se encuentran en dominio catalítico y permiten su organización estructural.
Para corroborar que el candidato GlSir2.3 presenta características estructurales de una sirtuina, se buscaron dominios conservados en la secuencia con los servidores CDD y Pfam; se encontró que esta secuencia presenta el dominio típico de la superfamilia sirtuina entre los residuos 60 y 270 aproximadamente, lo cual se valida con el E - value obtenido para cada búsqueda (Tabla 1, Figura 2). Lo anterior corresponde a una región de unos 200 aminoácidos que es la longitud promedio del dominio catalítico; además, debido a la ubicación de este a lo largo de la secuencia, es acorde con la presencia de regiones de baja homología hacia los extremos amino y carboxi terminal, las cuales tienen una función reguladora de la actividad enzimàtica, principalmente por ser los blancos de modificaciones postraduccionales [24].
Tabla 1 Dominios conservados encontrados en la secuencia de GdSir2.3 mediante los servidores CDD y Pfam. Se presentan los códigos de acceso para cada base de datos, el intervalo que corresponde al dominio y el E - value para cada uno.
| Servidor | Nombre | Acceso | Intervalo | E-value |
|---|---|---|---|---|
| CDD-NCBI | SIR2 Superfamily | C100195 | 61-274 | 6,43e - 32 |
| Pfam | SIR2 | cl0085 | 66-274 | 7.2e-22 |
Al someter la secuencia del candidato GdSir2.3 al servidor NetPhos 3.1 de ExPASy se observó que esta presenta 62 residuos potenciales para fosforilación como se muestra en la Figura 3. Dichos residuos se ubican en su mayoría hacia los extremos N - Terminal y C - Terminal de la secuencia, fuera de la región que se espera conforme el dominio catalítico. Cabe resaltar que existe también la probabilidad que GdSir2.3 sea fosforilada en su dominio catalítico; esto se ha visto experimentalmente en SIRT1 en donde esta modificación postraduccional actúa como un interruptor de la actividad catalítica que al ser removida promueve la actividad enzimática, de manera que permite la progresión del ciclo celular [25]. Las fosforilaciones en sirtuinas de parásitos protozoarios no han sido estudiadas de forma experimental; no obstante, es un campo promisorio para entender los mecanismos de regulación de estas enzimas que representan funciones vitales en dichos organismos.
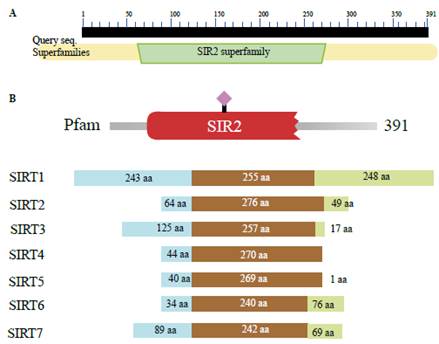
Figura 2 Representación esquemática de la estructura sirtuina. A. Predicción de dominios conservados con el servidor CDD - NCBI. B. Predicción de dominios conservados con el servidor Pfam C. Representación esquemática de las estructuras de las siete sirtuinas humanas. En azul se observa el extremo N - Terminal; en café, el dominio conservado; en verde, el C - Terminal. Tomado de Sacconay y colaboradores (2016).
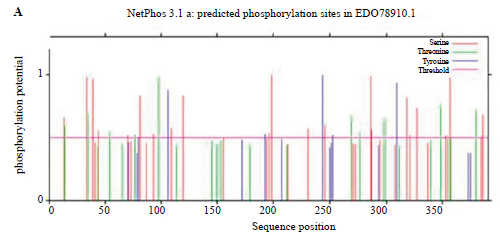
Figura 3 Predicción de fosforilaciones del candidato GdSir2.3. A. Predicción de fosforilaciones sobre el candidato GdSir2.3 mediante el servidor NetPhos 3.1 de Expasy.
La probabilidad de que el candidato GdSir2.3 esté regulado por modificaciones como la fosforilación es importante para su estudio como blanco farmacológico, ya que se ha encontrado que esta modificación actúa como un interruptor de la actividad catalítica de diferentes sirtuinas dentro de las que se encuentran las humanas. La regulación de la actividad catalítica mediada por modificaciones postraduccionales ha sido ampliamente estudiada en las siete sirtuinas humanas y se ha encontrado que para todas ellas es común la fosforilación [24].
En organismos filogenéticamente cercanos a G. duodenalis también se han estudiado las modificaciones postraduccionales de las sirtuinas; tal es el caso de Mycobacterium tuberculosis y su única sirtuina (mDAC) que se fosforila en el residuo Thr - 314, lo que genera la inhibición de la enzima, contrario a lo observado en las sirtuinas de eucariotes superiores [26]. En G. duodenalis la fosforilación juega un papel muy importante en el dimorfismo celular a lo largo de su ciclo de vida. Estudios sobre el kinoma de este parásito han revelado que presenta menos quinasas en comparación con otros eucariotes. Entre las quinasas ausentes en G. duodenalis se encuentran algunas que regulan procesos como la progresión del ciclo celular y la reparación de daños en el DNA, las cuales se encuentran entre las funciones descritas para sirtuinas de otros organismos [27].
Por otra parte, al realizar el modelamiento tridimensional del candidato con diferentes algoritmos, se observó que se mantiene el plegamiento sirtuina en todos los modelos (Figura 4) y, de acuerdo con los parámetros de evaluación de los modelos, se observa un mejoramiento de estos. De este modo, para el modelo obtenido por el algoritmo de homología del servidor SwissModel se tiene una baja identidad con la plantilla empleada, la cual podría corresponder únicamente al modelamiento del dominio sirtuina. En cuanto al siguiente modelo, realizado por el servidor I-Tasser, empleando el algoritmo de threading, se observa una disminución de la calidad de aquel debido a que el servidor intenta modelar aquellas regiones que no presentan homología con la plantilla teniendo en cuenta el contexto molecular de los aminoácidos involucrados. No obstante, se mantiene el tamaño esperado para el dominio catalítico. En el modelo obtenido mediante el algoritmo de ab initio se evidencia una mejoría debido a una reducción del RMSD entre un mayor número de átomos apareados entre ambas plantillas.
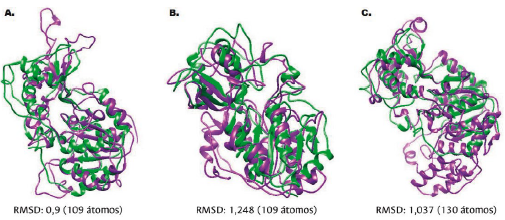
Figura 4 Comparación por superposición de modelos tridimensionales de GdSir2.3 (GL50803 16569), realizados en diferentes servidores, con plantilla cristalizada de SIRT5 (Q9NXA8). En cada superposición se indica el número RMSD que es menor de dos en todos los casos. A. Modelo por homología. B. Modelo por threading. C. Modelo por ab initio.
La sirtuina GdSir2.3 es una deacetilasa dependiente de NAD presente en ambos estadios de vida del parásito
El producto de PCR correspondiente al candidato GL50803_16569 se insertó en el vector de expresión pET100/D-TOPO el cual otorga una etiqueta de polihistidina en el extremo N - Terminal de la proteína recombinante. La identidad del plásmido recombinante se verificó mediante PCR y digestión enzimática (Figura 5). Al realizar la inducción de la expresión de la proteína recombinante con IPTG se observa el aumento de una proteína de aproximadamente 46 kDa, la cual es reconocida de manera específica en extractos celulares totales, fracción soluble y fracción insoluble mediante western blot con anticuerpos que reconocen la etiqueta de polihistidina (Figura 6).
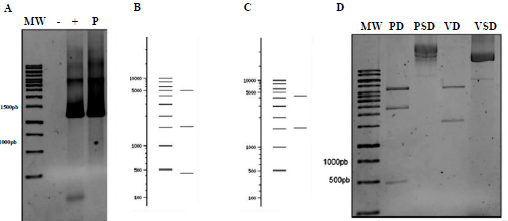
Figura 5 Creación del plásmido recombinante pET-100-GdSir2.3. A. Evaluación de plásmido recombinante por PCR. TBE -Agarosa 1%. MW: marcador de peso molecular 1Kb. P: plásmido pET-100-GdSir2.3; B. Perfil de digestión esperado para el plàsmido recombinante. NebCutter V. 2.0; C. Perfil esperado para el plàsmido vacío NebCutter V; D. Digestión del plásmido con EcoRV, en orden: PD: plásmido recombinante digerido, PSD: plásmido sin digerir, V: plásmido vacío digerido, VSD: plásmido vacío sin digerir 2.0. TBE - Agarosa 0,8%.
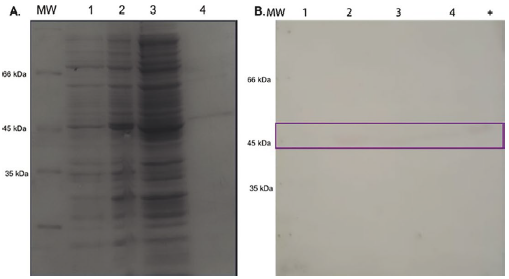
Figura 6 Purificación de 6xHis - GdSir2.3 a partir de la fracción soluble de E. coli cepa Shuffle por cromatografía de afinidad a níquel. A. SDS - PAGE, acrilamida 10%. B. Western blot, membrana de nitrocelulosa. 1°: IgY - α - 6xHis - GdSir2.3 (1:1000), 2°:α - IgY - Fosfatasa alcalina (1:10000). MW: marcador de peso molecular, 1: células totales sin inducir, 2: células totales inducidas, 3: fracción soluble, 4: eluido, +: Fracción insoluble de E coli cepa Shuffle inducidas, -: BSA.
Al evaluar la actividad deacetilasa mediante el kit de deacetilación in vitro CycLex SIRT1/Sir2 Deacetylase Fluorometric Assay Kit Ver. 2 de la proteína soluble purificada se observa emisión de fluorescencia que indica que 6xHis-GdSir2.3 presenta actividad deacetilasa dependiente de NAD (Figura 7). Comparando la actividad deacetilasa de 6xHis-GdSir2.3 con la de SIRT1 humana se observan diferencias tanto en los niveles de deacetilación alcanzados, expresados en URF uy (Unidades Relativas de Fluorescencia) (Figura 6A), como en el comportamiento de ambas enzimas a través del tiempo (Figura 6B). Esto puede explicarse teniendo en cuenta la actividad específica de cada enzima y la afinidad por el sustrato, de tal manera que para SIRT1 se vea favorecida la reacción de deacetilación de manera màs eficiente que para el candidato. Cabe resaltar también que la remoción del imidazol no fue completa y a pesar de que su concentración se haya reducido hasta aproximadamente 10 mM, existe la posibilidad de que siga ejerciendo un efecto inhibitorio en GdSir2.3 Finalmente, se debe tener en cuenta también que el kit de deacetilación fue desarrollado bajo las condiciones óptimas de reacción para la SIRT1 humana, y al ser G. duodenalis un organismo filogenéticamente lejano de los mamíferos, se espera que las condiciones óptimas para la actividad de GdSir2.3 sean muy diferentes.
Por otra parte, se ha encontrado que algunas sirtuinas ya caracterizadas en parásitos protozoarios presentan actividad deacetilasa y ADP -ribosiltransferasa. Tal es el caso de la TbSir2rp1 de T. brucei que está implicada en la reparación de daños en el DNA mediante la ADP -ribosilación [28]. Para llevar a cabo su función, esta proteína presenta dos rutas: la primera, que consiste en la interacción de la 2 - O - acetil - ADP -ribosa, producida en la deacetilación, y las histonas; y la otra, dependiente de la interacción entre el péptido acetilado y la ADP - ribosa, pero que aún no ha sido completamente dilucidada [29]. Además, también se ha encontrado que las sirtuinas presentan la capacidad de remover varios grupos acilo de las lisinas gracias a variaciones estructurales presentes dentro y fuera del dominio catalítico; y junto al contexto que acompaña el aminoácido modificado, la variabilidad de afinidad por diferentes sustratos permite que las sirtuinas tengan diferentes funciones en un mismo organismo [30]. Teniendo en cuenta la posible actividad ADP - ribosiltransferasa y una mayor afinidad por otros sustratos, junto con las condiciones óptimas de reacción, se podría explicar la baja actividad enzimática presentada por GdSir2.3 en el ensayo in vitro.
En cuanto a la ubicación subcelular, se encontró, mediante western blot sobre extractos celulares de parásitos (Figura 8) e inmunofluorescencia en trofozoítos y quistes empleando los anticuerpos policlonales obtenidos en modelo aviar (Figura 9), que GdSir2.3 está presente en ambos estadios del parásito.
Al evaluar por western blot con el anticuerpo IgY - α - 6xHis-GdSir2.3 los extractos totales de trofozoítos asincrónicos y de células tras 24 horas de enquistación, se reconoce claramente una proteína de aproximadamente 44kDa durante todo el proceso, y su nivel se mantiene constante; dicho tamaño corresponde a lo esperado para la proteína endógena (43,9 kDa, Figura 7A). Como control de enquistación se detectaron las proteínas de la pared del quiste (CWP, 26 kDa) y se obtuvo el reconocimiento mostrando que aumenta a medida que pasan las horas de enquistación, lo cual indica que, efectivamente, el proceso de enquistación fue exitoso (Figura 7B)
Análisis del transcriptoma a lo largo del ciclo de vida del parásito han demostrado que el gen correspondiente al candidato GdSir2.3 aumenta significativamente su expresión a las 24 horas de enquistación, mientras que las sirtuinas ya caracterizadas, GdSir2.1 y GdSir2.2, junto a los otros candidatos, GdSir2.4 y GdSir2.5, disminuyen [31]. Lo anterior podría indicar la relevancia de GdSir2.3 en el proceso de enquistación. Por otra parte, Carranza y colaboradores (2016) encontraron que debido a la falta de mecanismos de regulación típicos, como factores de transcripción canónicos, para este organismo, la regulación de la expresión génica a nivel postranscripcional y traduccional, así como los factores epigenéticos, tendrían un papel muy importante durante el ciclo de vida, principalmente en la enquistación, como lo demuestra la disminución de los niveles de acetilación de histonas observados durante este proceso. Adicionalmente, en otros estudios se ha encontrado que al suplementar el medio de cultivo con inhibidores de HDCAs se produce una hipoacetilación de las histonas y se detiene la enquistación. El reconocimiento tanto en trofozoítos como durante la enquistación de GdSir2.3 y en extractos de quistes aporta evidencia de la importancia de esta proteína para la progresión del ciclo de vida del parásito [32].
Entre las sirtuinas citoplasmáticas se encuentra la SIRT2 de H. sapiens para la cual se han descrito diferentes funciones como el mantenimiento de la estabilidad genómica durante la mitosis [33], el control de la diferenciación celular mediante la deacetilación de factores de transcripción y tubulina [34] y la protección celular durante estrés oxidativo a partir de la regeneración de los niveles de NADPH mediante la vía de las pentosas fosfato [35]. De otra parte, en organismos evolutivamente más cercanos a G. duodenalis, como T. cruzi, se ha observado que la sobreexpresión de TcSir2RP1, una sirtuina citoplasmática, induce la diferenciación a tripamastigotes y aumenta la eficiencia del proceso infectivo de este organismo sobre células mamíferas [36]. Otro parásito protozoario que cuenta con una sirtuina citoplasmática caracterizada es Entamoeba histolytica (EhSir2a), y si bien su función específica no ha sido determinada, los ensayos de interacción de proteínas realizados por Dam y Lohia (2010) mostraron que esta sirtuina interactúa, entre otras proteínas, con la tubulina y además puede deacetilarla. Lo anterior indicaría que EhSir2a constituye un mecanismo de regulación adicional de procesos como la segregación de cromatina, la mitosis y la división celular [37].
Para G. duodenalis ya se ha identificado una sirtuina citoplasmática (GdSir2.1) que podría estar interactuando con estructuras ricas en tubulina como el cuerpo medio del parásito, y por lo tanto tendrían un papel importante en la organización estructural del trofozoíto [15]. Si bien GdSir2.3 también es citoplasmática, no habría una redundancia de funciones, ya que esta tiene una distribución diferente en el trofozoíto y además los niveles de expresión sugieren que, en esa etapa del ciclo de vida, GdSir2.1 sería la sirtuina activa.
En cuanto a la ubicación subcelular de GdSir2.3 en el quiste, también se encontró una localización citoplasmática; adicionalmente, se observa un fuerte reconocimiento sobre gránulos en la pared del quiste que podrían corresponder al cuerpo multivesicular del parásito (MVB). A lo largo del ciclo de vida de G. duodenalis existen estructuras vesiculares que este utiliza para procesos que van desde la endocitosis en trofozoítos hasta la regulación de la infección y la sincronización de la enquistación en estadios más avanzados de la enfermedad [38].
Es probable que GdSir2.3 se encuentre en estas vesículas, teniendo en cuenta lo encontrado por Ma'ayeh y colaboradores (2017) en sus análisis de las proteínas presentes en el secretoma de Giardia durante la interacción con las células del hospedero [39]. Las proteínas predominantes en dicho análisis corresponden a diferentes funciones metabólicas de carbohidratos, lípidos y salvamento de nucleótidos; esta última es una evidencia de la competencia por nutrientes entre las células del hospedero y el parásito.
Así mismo, las vesículas presentaron también proteínas, tanto ya caracterizadas como hipotéticas, con alto potencial antioxidante y cuya función está asociada a combatir las ROS producidas por el hospedero como respuesta a la infección [39]. Es importante resaltar que, además de regular el silenciamiento génico junto a las otras deacetilasas, las sirtuinas también constituyen un mecanismo de respuesta durante el estrés oxidativo [40] y, por lo tanto, la localización de GdSir2.3 en las vesículas secretadas indicaría una función adicional a la regulación del cambio de forma celular, que en este caso sería la defensa del parásito durante la infección.
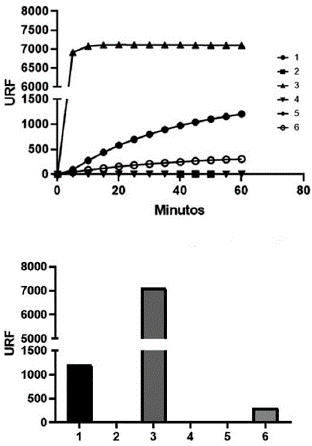
Figura 7. A. Evaluación de la actividad deacetilasa dependiente de NAD de 6xHis -GdSir2.3. A. Actividad deacetilasa en función del tiempo. 1. 6xHis - GdSir2.3 (300 ng) en buffer de deacetilación. 2. 6xHis - GdSir2.3 (250 ng) en buffer de elución. 3. SIRT1 de H sapiens (250 ng). 4. 6xHis - GdSir2.3 (250 ng) en ausencia de NAD (8mM). 5. Reacción de deacetilación sin 6xHis - GdSir2.3 en presencia de buffer de deacetilación. 6. Reacción de deacetilación sin 6xHis - GdSir2.3. B. Actividad deacetilasa medida en URF (unidades relativas de fluorescencia).

Figura 8. A. Extractos totales de parásitos de Giardia (1x106 parásitos) durante las 24 horas de la enquistación, tinción con coomassie. B. Detección de GdSir2.3 (43,9 kDa,) por western-blot en extractos totales de los parásitos enquistantes mostrados en la figura A. 1°: IgY-α-6xHis-GdSir2.3 (1:500), 2°: α - IgY peroxidasa (1:10000). C.Western blot de la proteína control de la enquistación CWP1 (26 kDa):2. 1°: α - CWP (1:500). 2°: α - IgG peroxidasa (1:10000). Revelado sistema quimioluminiscente.
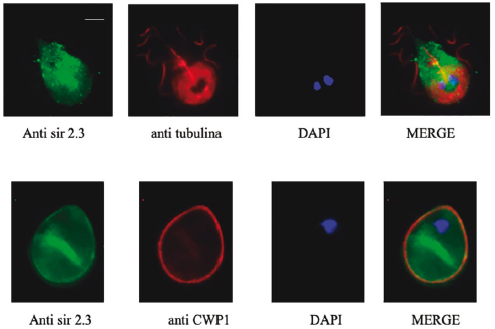
Figura 9 Identificación de GdSir2.3 endógena mediante inmunofluorescencia. A. Inmunofluorescencia sobre trofozoítos. En verde se presenta la localización de Gdsir2.3, en rojo la señal de tubulina, como control; en azul los núcleos y el merge de las tres imágenes. B. Inmunofluorescencia sobre quistes. En verde GdSir 2.3, en rojo anti CWP1, marcadora de la pared del quiste, en azul los núcleos y el merge de las tres imágenes. El anticuerpo IgY-α-6xHisGdSir2.3 permitió identificar a GdSir2.3 como una proteína citoplasmática en ambos estadios del ciclo de vida del parásito. Cabe mencionar que la proteína presenta una distribución vesicular y una concentración cerca de la pared del quiste.
Conclusiones
Se identificó, empleando herramientas computacionales y experimentales, al candidato GdSir2.3 de G. duodenalis como una deacetilasa dependiente de NAD que cuenta con características estructurales típicas de la familia sirtuina y presenta una localización subcelular citoplasmática en trofozoítos y quistes del parásito.














