Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.56 no.4 Palmira Oct./Dec. 2007
RESUMEN
Se utilizó la información generada por 29 microsatélites en 63 accesiones del acervo andino de P. vulgaris para elaborar agrupamientos y calcular índices de prevalencia. Las accesiones formaron grupos que correspondieron a las razas Nueva Granada, Chile y Perú. Se determinó la existencia de notoria introgresión en genotipos de las razas Chile y Perú
Palabras clave: Phaseolus vulgaris L.; acervo genético; introgresión; índice prevalencía; diferenciación genética; microsatélites.
ABSTRACT
We used the information generated by 29 microsatellite markers in the analysis of 63 common bean (Phaseolus vulgaris) accessions to evaluate clustering and prevalence indices and to determine the similarity and differentiation or introgression among races of the Andean gene pool. We identified three clusters of genotypes which corresponded to the races of the Andean gene pool, i.e. Nueva Granada, Chile and Peru and the existence of introgression between accessions of these three races. Introgression was more common for genotypes in races Chile and Peru, then for the race Nueva Granada.
Key words: Phaseolus vulgaris; genepool; introgression; marker prevalence index; genetic differentiation; microsatellites.
La detección de la variabilidad genética del ADN es más eficaz que la realizada mediante marcadores morfoagronómicos (Singh et al., 1991). El análisis de la relación entre accesiones se realiza mediante dendrogramas y agrupa las de parecido genético (Karp et al., 1997). Los marcadores microsatélite SSR se utilizaron por primera vez en fríjol por Yu et al. (1999), y el número se incrementó posteriormente por Gaitán et al. (2002) y Blair et al. (2003).
Los patrones de faseolina y del tamaño de la semilla de las accesiones de fríjol común proveen evidencias sobre el intercambio de germoplasma entre Mesoamérica y los Andes. En muestras de cultivares Mesoamericanos los únicos que tenían faseolina T (Tendergreen) eran de semilla grande y colores típicos del acervo Andino, las faseolinas B y S, típicas de los granos Mesoamericanos, se encuentran en algunos genotipos Andinos (Gepts, 1988).
Con el fin de establecer niveles de similitud y procesos de introgresión genética entre accesiones del acervo Andino de fríjol común, se analizó con marcadores SSR, una muestra de accesiones conservadas en la Colección Núcleo y en la Unidad de Recursos Genéticos del CIAT e identificadas como representativas de razas Andinas. También se comparó la distribución de patrones de faseolina con la agrupación dada por el análisis con los microsatélites.
Caracterización molecular y genotipos utilizados
El estudio se adelantó en el Centro Internacional de Agricultura Tropical (CIAT) con 63 accesiones seleccionadas dentro del grupo no caracterizado previamente por microsatélites y pertenecientes a razas del acervo Andino (Becerra and Gepts, 1994; Beebe et al., 2001; Singh et al., 1991; a, b y c); 25 de las cuales eran de la raza Nueva Granada, seis de la raza Chile y 28 de la raza Perú. También se incluyeron dos controles tipo Andino (Diacol Calima y Chaucha Chuga) y dos tipo Mesoamericano (DOR 364 e ICA Pijao). Dentro del total de accesiones, 44 variedades eran nativas, ocho Ñuñas, cuatro variedades comerciales y tres líneas mejoradas. Los controles se escogieron con base en el estudio previo de Blair et al. (2006).
En trifolios etiolados de cuatro plantas de ocho días de crecimiento en la oscuridad se extrajo el DNA (Afanador et al. 1993). Se almacenó a -20°C y se cuantificó en fluorómetro DyNA Quant (Hoefer Pharmacia Biotech Inc., 1995), con base en una solución de colorante Hoescht H 33258. La solución de trabajo de ADN se utilizó a 10ng/µl en buffer TE. Posteriormente se realizó el PCR para amplificar 29 marcadores SSR (18 genómicos y 11 génicos), escogidos a partir del nivel de polimorfismo encontrado en estudios anteriores (Blair et al., 2003; Gaitán et al., 2002; Yu et al., 1999; Yu et al., 2000).
La determinación de alelos se realizó a partir de la lectura de los geles de electroforesis y luego se generó una matriz de datos binaria que contenía la información de las accesiones y el genotipo observado a través de los locí examinados. Se consideraron como nulos los que no amplificaron después de repetir el procedimiento dos veces. Para determinar la diferenciación entre las accesiones se construyeron dendrogramas y se utilizó el índice de similaridad de huellas de ADN (Lynch, 1990), el cual se define como una fracción de bandas compartidas para los individuos X e Y, el número de bandas comunes (Nxy) se divide por el número de bandas promedio presentes en cada individuo. Se utilizaron los módulos Similarity/Genetic Distance y Clustering/SAHN del programa NTSYS V 2.11v (Rohlf, 2002). Con el índice de prevalencía (Beer et al., 1997) se cuantificó la relación entre la rareza de los marcadores utilizados y los genotipos en los cuales estaban presentes. Los genotipos con bajos valores indican composición alélica rara y posible producto de introgresión (Islam et al., 2004).
Índice de similaridad
El análisis a través de dendrograma diferenció dos grupos (Figura 1). El primero estuvo compuesto por genotipos de las razas Nueva Granada (75.9%), Perú (20.7%) y Chile (6.9%). El segundo por genotipos "Jalo" (G5024 y G2195) y los Mesoamericanos utilizados como controles. Los genotipos tipo "Jalo" indican procesos de introgresión entre el acervo Andino y el Mesoamericano, pues proceden de la zona suroriental de Brasil donde es frecuente encontrar cultivares tipo Andino y Mesoamericano (Maciel et al., 2003). También se pudieron delimitar 13 subgrupos (Figura 1) con una similitud de 0.58. Algunos de los grupos estaban compuestos por uno o dos genotipos. Sobresalieron dos grupos adyacentes, formados por genotipos de la raza Chile y otros grupos correspondientes a las razas Nueva Granada y Perú. La composición alélica de los genotipos que no formaron agrupaciones consistentes con la clasificación previa de razas andinas (Singh et al., 1991) pudo haber sido diferente por la introgresión con razas del acervo Andino o con materiales silvestres. La posición en el dendrograma del genotipo G2544 de raza Chile confirmó los resultados de Paredes y Gepts (1995), quienes reportaron por medio de isoenzimas la presencia de alelos de la raza Perú en la raza Chile.

Los genotipos G12407 (Bolón Bayo), G12709 (Mortiño), G11819 (Liborino) del norte de Perú, Colombia y Ecuador, pertenecientes a la raza Perú, que presentaron características morfoagronómicas Mesoamericanas, pero faseolina, aloenzimas y características de semillas andinas (Singh et al., 1991) se localizaron dentro del grupo denominado raza Perú. El genotipo G5708 (Sangretoro), clasificado como típico de la raza Perú, se agrupó con genotipos representativos de la raza Nueva Granada. Por medio de isoenzimas y marcadores moleculares tipo RFLP, Becerra y Gepts (1994), reclasificaron los genotipos G12587 (Ñuña Callashina) y G5702 (Cargamanto), de presunto origen en la raza Perú, en un grupo con características Mesoamericanas. Sin embargo, en el estudio G12587 se situó dentro del grupo raza Perú, mientras que los Cargamantos G5702 y G7231, considerados representativos de la raza Perú, se localizaron dentro del grupo raza Nueva Granada. Los genotipos G7930 (Alubia), G23604 (Ñuña) y G12644 (Morado), identificados mediante marcadores RAPD como introgresados (Islam et al. 2004), presentaron el mismo comportamiento mediante los marcadores SSR.
El análisis de la composición de las faseolinas evidencia que entre razas o acervos ha ocurrido introgresión genética. El proceso de flujo de genes fue más evidente para las razas Nueva Granada y Chile, pues la primera presentó genotipos con faseolinas (C, Ca, T/H1, H) no asignadas inicialmente a esta raza mientras que la raza Chile no presentó las faseolinas C y H, también asignadas inicialmente como propias de esta raza (Singh et al., 1991). Los tipos de faseolinas encontrados en la raza Perú se ajustaron a las formas ya reportadas para esa región. En breve, los resultados indicaron que la faseolina no fue un carácter determinante para la asignación de genotipos de P. vulgaris L.
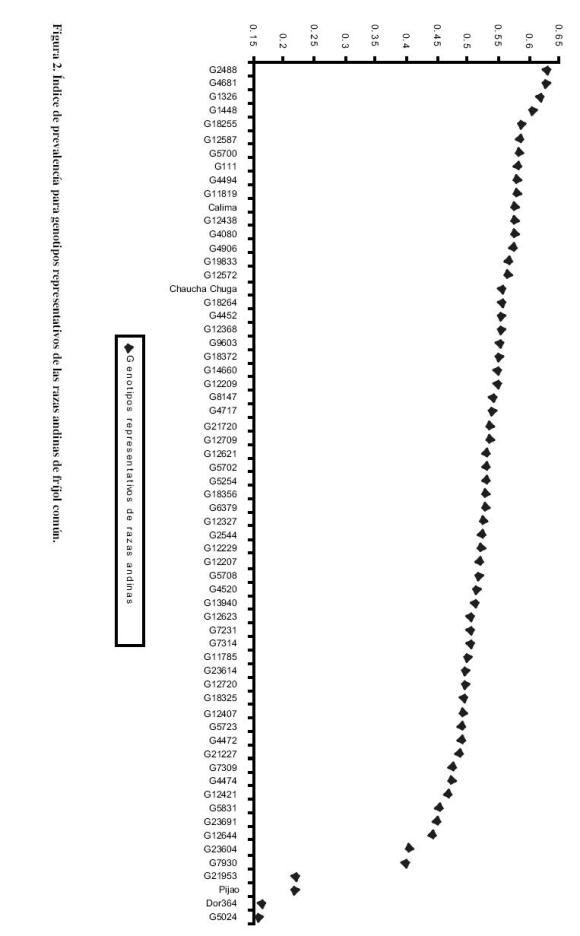
ÍNDICE DE PREVALENCIA
La pendiente suave de la gráfica para los índices de prevalencia (Figura 2) mostró los conjuntos de alelos comunes que se distribuyeron de manera continua; en el extremo de la curva se encontraron los genotipos que presentaron alelos raros o alto nivel de introgresiones (IP=0.51), lo que concuerda con datos de Islam et al., (2004). Los genotipos Mesoamericanos no formaron parte del grupo mayor Andino, presentaron bajos valores del índice de prevalencía y se situaron en el extremo de la curva. Los genotipos representantes de la raza Chile mostraron características que sugieren introgresión genética. Esto indica que el patrón de variabilidad de la raza Chile y los parámetros que la definen son diferentes del acervo Andino (Johns et al., 1997).
Los marcadores SSR determinaron con mayor nivel de detalle la estructura y procesos de flujo genético de las razas andinas superando los marcadores RAPD, RFLP y AFLP.
El índice de similaridad de huellas genéticas resultó útil en la individualización de conjuntos de genotipos; sin embargo, es recomendable la aplicación de procedimientos tipo multivariados que permitan obtener mayor resolución de la información obtenida por medio de marcadores SSR.
El concepto de razas andinas de fríjol es susceptible de revisión pues los rasgos originalmente establecidos no concuerdan en algunas de las accesiones estudiadas.
Fue evidente la existencia de flujo de genes entre los genotipos que representan razas andinas de P. vulgaris y entre los acervos
Los autores desean agradecer a Lucy Milena Díaz por la revisión del manuscrito, al personal del Laboratorio de Caracterización de Germoplasma de Fríjol y a la Unidad de Recursos Genéticos del CIAT, así como también a la Universidad Nacional de Colombia sede Palmira por ayudar a la realización de la tesis de Maestría de Juan Manuel Díaz y a "Generation Challenge Program" por la financiación del proyecto.
1. Afanador, L.; Hadley, S.; Kelly, J.D. 1993. Adoption of a mini-prep DNA extraction method for RAPD marker analysis in common bean (Phaseolus vulgaris L). Bean Improv. Coop 36:10–11. [ Links ]
2. Becerra, V.; Gepts, P. 1994. RFLP Diversity of Common Bean (Phaseolus vulgaris) in its centres of origin. Genome 37:256- 263. [ Links ]
3. Beebe, S.; Renjifo, J.; Gaitán-Solís, E.; Duque, M.C.; Tohme, J. 2001. Diversity and origin of Andean landraces of common bean. Crop Sci. 41:854-862. [ Links ]
4. Beer, S.; Siripoonwiwat W.; O´Donoughue L.; Souza E.; Matthews D.; Sorrells M. 1997. Associations between molecular markers and quantitative traits in an oat germplasm pool: Can we infer linkages? J. Agric. Genomics 3:1–16 www.probenalusdagov:8000/other.docs/jag [ Links ]
5. Blair, M.W.; Giraldo M.C.; Buendía H.F.; Tovar, E.; Duque M.C.; Beebe S. 2006. Microsatellite marker diversity in common bean (Phaseolus vulgaris L.). Theor. Appl. Genet. 113:100–109 [ Links ]
6. Blair, M.; Pedraza, F.; Buendía, H.; Gaitán, E.; Beebe, S.; Gepts, P.; Tohme, J. 2003. Development of a genome-wide anchored microsatellite for common bean (Phaseolus vulgaris L). Theor. Appl. Genet. 107:1362-1374. [ Links ]
7. Gaitán, E.; Duque, M.C.; Edwards, K.; Tohme, J. 2002. Microsatellite repeats in common bean (Phaseolus vulgaris L.): isolation, characterization, and cross-species amplification in Phaseolus spp. Crop. Sci. 42:2128-2136. [ Links ]
8. Gepts, P. 1988. Phaseolin as an evolutionary marker, p. 215-141, In: Gepts, P. (ed.) Genetic resources of Phaseolus beans. Kluwer Academic. [ Links ]
9. Islam, A.; Beebe, S.; Muñoz, M.; Tohme, J.; Redden, R.; Basford, K. 2004. Using molecular markers to assess the effect of introgression on quantitative attributes of common bean in the Andean gene pool. Theor. Appl. Genet. 108:243-252. [ Links ]
10. Johns, M.; Skroch, P.; Nienhuis, J.; Hinrichsen, P.; Bascur, G.; Muñoz-Schick, C. 1997. Gene pool classification of common bean landraces from Chile based on RAPD and Morphological data. Crop. Sci. 37:605-613. [ Links ]
11. Karp, A.; Kresovich, S.; Bhat, K.; Ayad, W.; Hodgkin, T. 1997. Molecular tools in plant genetic resources conservation: a guide to the technologies Rome: IPGRI. [ Links ]
12. Lynch, M. 1990. The similarity index and DNA fingerprinting. Mol. Biol. Evol. 5:478-484. [ Links ]
13. Maciel, F.; Echeverrigaray, S.; Gerald, L.; Grazziotin, F. 2003. Genetic relationship and diversity among Brazilian cultivar and of common beans (Phaseolus vulgaris L.) revealed by AFLP markers. Genet. Resourc. Crop. Evol. 50:887-893. [ Links ]
14. Paredes, O.; Gepts, P. 1995. Extensive introgression of middle American germplasm into Chilean common bean cultivars. Genet. Resourc Crop. Evol. 42:29-41. [ Links ]
15. Rohlf, F. 2002. NTSYS pc: Numerical taxonomy and multivariate analysis system Setauket, NY. Exeter Publishing. [ Links ]
16. Singh, S.; Gepts, P.; Debouck, D. 1991a. Races of common bean (Phaseolus vulgaris, Fabaceae). Econ. Bot. 45:379-396. [ Links ]
17. Singh, S.; Nodari, R.; Gepts, P. 1991c. Genetic diversity in cultivated common bean: I. Allozymes. Crop. Sci. 31:19-23. [ Links ]
18. Singh, S.; Gutiérrez, A.; Molina, A.; Urrea, C.; Gepts, P. 1991b. Genetic diversity in cultivated common bean: II. Marker-based analysis on morphological and agronomic traits. Crop. Sci 31:23-29. [ Links ]
19. Yu, K.; Park, S.; Poysa, V. 1999. Abundance and variation of microsatellite DNA sequences in beans (Phaseolus and Vigna). Genome 42:27-34. [ Links ]
20. Yu, K.; Park, S.; Poysa, V.; Gepts, P. 2000. Integration of simple sequence repeat (SSR) markers into a molecular linkage map of common bean (Phaseolus vulgaris). J. Hered .91:429-434. [ Links ]














