Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.57 no.1 Palmira Jan./Mar. 2008
RESUMEN
El Hartón del Valle (HV) hace parte de las siete razas criollas de ganado bovino colombiano y de acuerdo con los criterios de la FAO está considerado como vulnerable. Para estudiar su variabilidad genética fueron muestreados 33 individuos HV y tres animales de la raza Holstein, como control externo. Se extrajo ADN utilizando el método de Salting Out y las muestras fueron analizadas mediante la técnica molecular RAM (Randon amplified microsatellites). Se utilizaron los cebadores CGA, CCA, TG, y CT. El valor promedio de heterocigosidad esperada fue de 0.26 y el FST fue 0.39 ± 0.03. Con el índice de Dice-Nei Li y agrupando con el método UPGMA se distinguieron dos grupos: uno integrado por dos hatos de conservación y el otro por cuatro fincas que han compartido reproductores.
Palabras claves: Diversidad genética, bovinos criollos, microsatélites, Hartón del Valle, RAM.
ABSTRACT
Harton del Valle (HV) breed belong to the seven Creole breeds of the Colombian bovine livestock, according to FAO criteria it is considered as vulnerable. To study its genetical variability, 33 HV animals and three and 3 animals of Holstein breed as a external control were sampled. Simples of DNA were isolated using the Salting Out method and RAM (Random amplified microsatellites) technique was used. CGA, CCA, TG and CT primers were used. The mean value of expected heterocigozity was 0.26 and FST fue 0.39 ± 0.03. Using Dice-Nei Li index and UPGMA clustering method, two groups were distinguished: the first one integrated for two conservation herds and other one by tour faros that have been sharing breeding bulls.
Key words: Genetic diversity, Creole cattle, RAM, Harton del Valle.
Colombia cuenta con siete razas bovinas criollas cuya población es de 23.415, lo que representa el 0.09% del inventario nacional, que asciende a 24 millones de cabezas (Martínez, 1999). El Hartón del Valle, uno de estos grupos raciales, originado de los ganados introducidos por los conquistadores españoles en el siglo XV y sometido a un largo proceso de selección natural que le ha conferido gran adaptación que se refleja en su capacidad para sobrevivir en climas cálidos, húmedos o secos, con alimentos de baja calidad nutricional y escasez de agua, altas tasas reproductivas, así como menor susceptibilidad a enfermedades parasitarias y producción de carne y leche razonables (Casas y Valderrama, 1998)
La Universidad Nacional de Colombia Sede Palmira ha estudiado la raza desde hace más de cuarenta años, y es la única entidad estatal que actualmente tiene un programa de conservación, puesto que el HV no fue incluido en los programas de protección a razas criollas propiciados por el gobierno colombiano desde los años treinta, ni tampoco en el Plan Nacional de Fomento de Razas Criollas del Ministerio de Agricultura y Desarrollo Rural, para la protección, mejoramiento, multiplicación y repoblamiento de las razas bovinas criollas Costeño con cuernos, Blanco Orejinegro, San Martinero y Romosinuano (Resolución 007147 del 15 de marzo del 2004). Desde 1968, la sede Palmira cuenta con un pequeño núcleo de animales puros y desde hace 10 años con un banco de semen.
En el departamento del Valle del Cauca la población de HV está conformada por 5.531 animales puros y 4.227 cruzados, distribuidos en 81 fincas, y de acuerdo con los criterios de conservación de la FAO la población se encuentra en la categoría vulnerable (Martínez, 1999). Además, en los últimos años, se sospecha marcada reducción en el inventario, debido a la crisis económica, competencia con la agricultura y problemas de orden público en el norte del Valle.
La técnica molecular RAM descrita por Zietkiewicz, et al. (1994), es un novedoso método de medición de la diversidad genética en plantas y animales que combina el uso de cebadores que contienen secuencias microsatélites y un extremo 5' degenerado. Los marcadores obtenidos por los RAM se pueden utilizar para estudios de poblaciones, no requiere el conocimiento previo de secuencias ni uso de isótopos radiactivos (Hantula, et al. 1996).
Estudios de diversidad genética realizados en especies vegetales (Morillo et al., 2005) y animales (Muñoz, et al., 2007; Hernández, et al., 2007; Oslinger et al., 2006) en el Laboratorio de Biología Molecular de la Universidad Nacional de Colombia Sede Palmira sugieren que la técnica es una herramienta eficiente para este tipo de estudios. El objetivo de este estudio fue evaluar la variabilidad genética del ganado criollo Hartón del Valle mediante la técnica molecular RAM (Random Amplified Microsatélites).
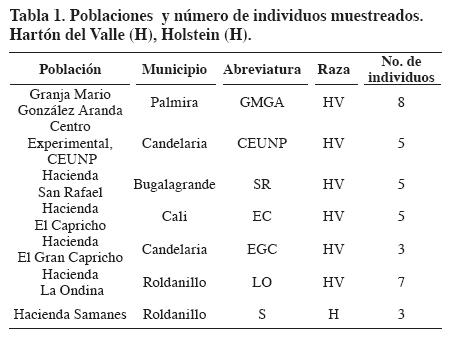
Las muestras se recolectaron en 33 individuos de HV provenientes de seis fincas representativas de la raza y tres individuos de la raza Holstein (H) como control externo (Tabla 1). El ADN se extrajo de sangre utilizando el protocolo Salting Out (Miller et al., 1988). La cantidad y calidad del ADN fueron evaluadas en geles de agarosa al 0.8%, teñidos con bromuro de etidio.
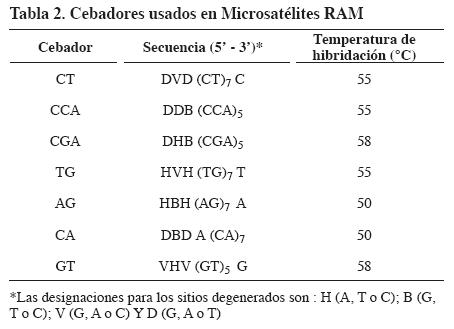
Para la amplificación se evaluaron siete cebadores RAM (Tabla 2) en un volumen total de 25 µl, que contenía tampón de Taq 1X, 2.5 mM de MgCl2, 0.2 mM de dNTP's, 4 µM de cebador, 1 U de Taq polimerasa y 20 ng de ADN, en un termociclador MJ Research PTC- 100. Las muestras fueron sometidas a 95°C por cinco minutos seguidas de 37 ciclos a 95°C por un minuto, 50-58°C, de acuerdo con el cebador (Tabla 2) por 45 segundos y 72°C por dos minutos. Se realizó una extensión final a 72°C por siete minutos. Los productos amplificados fueron visualizados en geles de agarosa al 1.2%, teñidos con bromuro de etidio.
Para evaluar la diversidad genética se estimaron las frecuencias alélicas, la heterocigosidad insesgada (He) (Nei, 1978) y el porcentaje de loci polimórfico mediante el programa TFPGA® Versión 1.3 (Miller, 1997). Se realizó un análisis de varianza molecular (AMOVA) con dos niveles jerárquicos (entre poblaciones y dentro de ellas). La diferenciación entre poblaciones se estimó a través de FST por pares de poblaciones mediante el programa Arlequín Versión 3.1 (Excoffier, 1996). El análisis de agrupamiento se realizó con el programa SAHN de NTSYS -pc y utilizó el método UPGMA, método gráfico de agrupamiento por parejas que emplea el promedio aritmético no ponderado. El dendrograma se construyó con el programa TREE de NTSYS-pc versión 1.80.
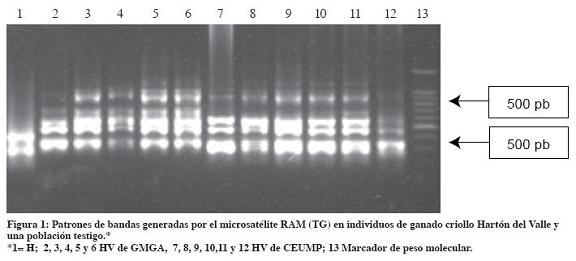
De los siete cebadores evaluados se seleccionaron los más polimórficos (CCA, CT, TG y CGA), que generaron un total de 52 patrones de bandas, distinguibles en un rango entre 80 y 1.200 pares de bases (Figura 1). La He fue 0.31, 0.35, 0.23 y 0.20, para cada cebador respectivamente. El valor promedio de He fue 0.26 (Tabla 3) con valores máximos de 0.32 para CEUNP y 0.25 para GMGA que también presentaron el mayor porcentaje de loci polimórfico (57.6% y 67.3%, respectivamente). El menor valor de He fue para la raza Holstein (He=0.09).
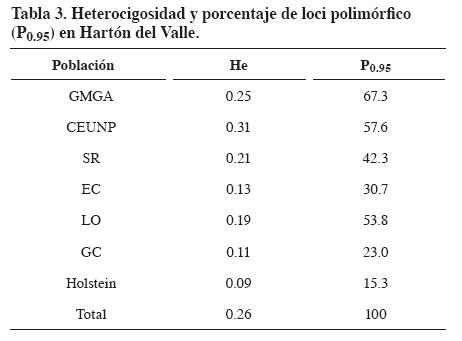
El valor de heterocigosidad obtenido en este estudio (He = 0.26) resultó ser similar al obtenido por Moreno et al. (2001) (He = 0.33) en ganado Hartón del Valle utilizando microsatélites; superiores a los hallados por Parejo, et al. (1998) en la raza Blanca Cacereña (H = 0.117) e inferiores a los encontrados en razas españolas (He = 0.72) (Arranz, et al., 1998), criollo de Argentina (He = 0.58) (Zamorano, et al.,1998), criollo de Uruguay (He = 0.487) (Postiglioni, et al., 1998) y criollo de Brasil (He=0.79) (Egyto, 2007). También los valores de heterocigosidad fueron notoriamente menores a los obtenidos con cinco microsatélites en siete razas de ganado criollo colombiano (Ho=0.67) y ocho individuos HV (Ho=0.7) (Bedoya et al., 2001) y a lo reportado por Barrera, et al. (2006) con 12 marcadores microsatélites en seis razas criollas colombianas (He=0.77) y 10 individuos HV (He=0.8). Es de aclarar que la mayoría de los reportes presentados anteriormente se basan en marcadores microsatélites autosómicos que son codominantes, mientras que los marcadores RAM, al ser dominantes, detectan menor variabilidad.
El porcentaje de variación entre fincas, estimado mediante el AMOVA, fue de 39.5% y dentro de fincas del 60.5%. El FST promedio en las siete poblaciones (0.39±0.03) mostró gran diferenciación genética entre poblaciones. Los valores de FST por pares de fincas mostraron diferencias significativas (P<0.05) entre ellas, con excepción de GC-HS y GC-LO (Tabla 4).
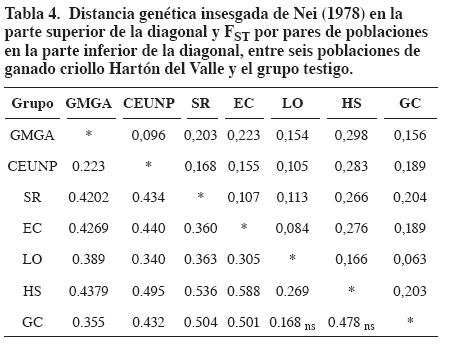
La matriz de distancias (Tabla 4) mostró alta similaridad entre los individuos de los hatos GC-LO y EC-LO y GMGA-CEUNP. El árbol de distancias (Figura 2) separó las dos razas HV y Holstein. En el HV se diferenciaron dos grupos: el primero con los individuos de LO, GC, SR y EC con bajo bootstrap (31%) y el segundo grupo lo conformaron los individuos de las poblaciones pertenecientes a la Universidad Nacional de Colombia (GMGA y CEUNP), con alto bootstrap (80%).
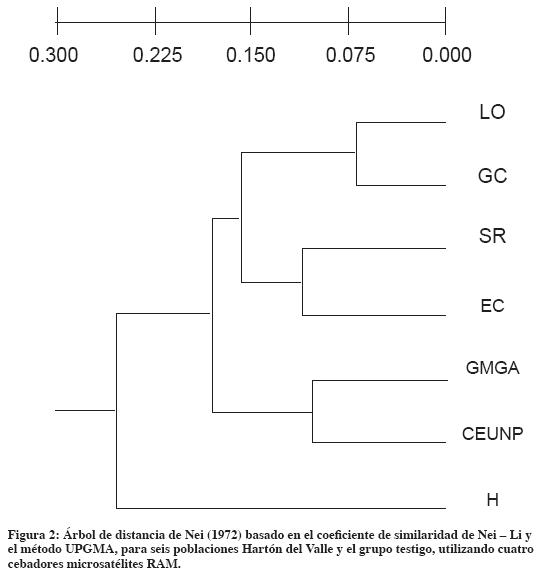
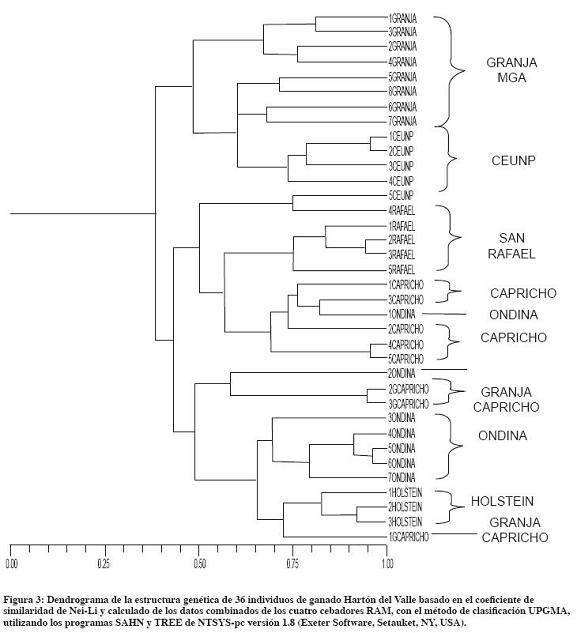
En el dendrograma construido con el análisis de polimorfismos (UPGMA), obtenidos por RAM (Figura 3), con base en el índice de similitud de Nei - Li, se observó, con contadas excepciones, que individuos de la misma población se agruparon juntos.
La diversidad genética (He=0.26) fue notoriamente menor a lo reportado con microsatélites autosómicos, lo cual era de esperarse, puesto que los marcadores RAM son dominantes. El valor encontrado para el FST (0.39) muestra estructura genética entre poblaciones de HV, que puede ocasionar reducción en la variación genética. La subdivisión entre poblaciones probablemente se deba a las diferencias en la orientación de los sistemas de producción y la escogencia de los reproductores, puesto que LO ha dirigido sus actividades hacia el doble propósito (carne y leche); SR, EC y GC son explotaciones dedicadas a la producción de leche y GMGA y CEUMP a la producción de carne.
La similaridad entre los individuos de las fincas pertenecientes a la Universidad Nacional (GMGA y CEUNP) fue alta, a pesar de que se suponía que estaban constituidos por material genético diferente. Las menores distancias entre GC-LO y EC-LO pueden ser explicadas por el intercambio de reproductores que ha ocurrido entre ellas. Igualmente, la baja distancia entre LO y SR se debió, posiblemente a que los animales fundadores de SR, eran de LO.
A pesar de que GC y EC pertenecen al mismo productor, presentaron baja similitud, debido a que en GC ha habido cruzamiento con Holstein, mientras que en EC se ha mantenido el núcleo criollo puro.
El hato GC presentó alta similaridad con Holstein, por lo tanto, los toros de esta finca pueden recomendarse para productores que cuenten con buen nivel tecnológico y necesiten incrementar la producción de leche.
Los resultados de este trabajo, a pesar de ser preliminares, muestran la fidelidad con que la técnica RAM reproduce la historia genética de los hatos y aclara las relaciones entre ellos; el polimorfismo encontrado sugiere que la técnica puede ser útil en programas de mejoramiento o manejo para identificar individuos lejanos para adoptar estrategias óptimas de cruzamientos. Es necesario investigar las razones de la alta estructuración genética entre fincas para tomar decisiones sobre la conservación de la raza y los métodos de apareamiento de los animales, y comprobar las relaciones genéticas entre poblaciones de Hartón del Valle.
La concordancia de los datos obtenidos con los marcadores RAM y la manera como se constituyeron los hatos permite que se sugieran estos marcadores para estudios de relaciones genéticas entre individuos y para estudios de diversidad genética en razas criollas, como una alternativa a los microsatélites clásicos.
A la Universidad Nacional de Colombia por la financiación del Trabajo de Grado de la zootecnista Ana. M. Piedrahíta de donde se derivó la información para el artículo. A los productores de ganado Hartón del Valle por el suministro de las muestras, especialmente a Marino Valderrama y a Eduaime Cárdenas.
1. Arranz, J. J.; Bayón, Y. 1998. San Primitivo. Genetic relationship among Spanish sheep using microsatellites. Anim. Genetic. 29 (6). 529 - 542. [ Links ]
2. Barrera, G. P.; Martínez R.; Pérez J. E.; Polanco N.; Ariza F. 2006. Evaluación de la variabilidad genética en Ganado Criollo Colombiano mediante 12 marcadores microsatélites. AGRI 38: 35-45 [ Links ]
3. Bedoya, G.,. Carvajal, L. G.; Bermúdez, N. R.; Moreno, F. L. 2001. Estructura Molecular y Poblacional del Ganado Criollo Colombiano (GCC). Rev. Col. Cienc. Pec. 14 (2): 107-118. [ Links ]
4. Casas, I.; Valderrama, M. 1998. El Bovino Criollo Hartón del Valle. Universidad Nacional de Colombia Sede Palmira - Asociación Nacional de Criadores de Ganado Hartón del Valle Asohartón. 60 p. [ Links ]
5. Egyto, A. 2007 Diversidade Genetica, ancestralidad individual e miscigenacao nas razas bovinas do brasil com base en microsatélites e haplotipos de DNA mitocondrial: subsidios para a conservacao. Tese Doutorado. Universidad de Brasilia. 282 p. [ Links ]
6. Excoffier L. 1996. Arlequín ver 3.11 Computational and molecular population genetic lab. CMPL. University of Berne http://cmpg.unibe.ch/software/arlequin3/ [ Links ]
7. Hantula, J.; Dusabenyagasani, M.; Hamelin, R. C. 1996. Randon Amplified Microsatellites (RAMS) a novel method for characterizing genetic variation within fungi. Eur J. Pathol. 26:159-166. [ Links ]
8. Hernández, D.; Muñoz, D.; Valencia, N. F.; Posso, A.; Muñoz, J. E. 2007. Caracterización molecular del pato criollo colombiano en cuatro departamentos. Acta Agron (Palmira) 56(3): 141-145 [ Links ]
9. Martínez, G. 1999. Memorias del Seminario Censo y Caracterización de los Sistemas de Producción del Ganado Criollo y Colombiano. Fondo Nacional del Ganado, ICA, Pronatta, ASOBON. Santafé de Bogotá. 158 p. [ Links ]
10. Miller, M. 1997. Tools for population genetics analices (TFPGA) 1.3: A Windows program for the analyse of allozyme and molecular population genetic data. [ Links ]
11. Miller, S. A.; Dikes, D.D.; Polesky, H. F. 1988. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acid Res., 16(3): 1215. [ Links ]
12. Moreno, F.; Bedoya, G.; Der., J.; Carvajal, L.; Bermúdez, N.; Zuluaga, F.; Ossa, J.; Verdugo, J.; Estrada, L.; Barrera, J.; Scoth, D.; Tobón, C.; Ruiz, L. 2001. Diversidad y relaciones filogenéticas del Ganado Criollo Colombiano. Rev Corpoica 3 (2): 17-23. [ Links ]
13. Morillo, A.; Morillo, Y.; Vásquez, H.; Muñoz, J. 2005. Caracterización molecular con microsatélites aleatorios RAM de la Colección de mora Rubus spp, de la Universidad Nacional de Colombia, Sede Palmira. Acta Agron (Palmira) 54 (2):15-24. [ Links ]
14. Muñoz, J. E.; Valencia, N. F.; Posso, A. 2007. Caracterización molecular en tipos de gallina criolla. Rev. Col. Cienc. Pec. 20:4. 532. [ Links ]
15. NEI, M. 1978 Genetic distance between populations. Am Nat 106: 283-292 [ Links ]
16. Oslinger, A.; Muñoz, J. E.; Alvarez, L. A.; Moreno, F.; Posso, A. 2006. Caracterización de cerdos criollos colombianos mediante la técnica molecular RAM. Acta Agron ( Palmira) 55 (4): 45- 50. [ Links ]
17. Parejo, J.; Sansinforiano, M.; Rabasco, A.; Martínez - Trancon, M.; Fernández - García, J.; Padilla, J. 1998. Utilización de la técnica RAPD para estudios poblacionales en la raza bovina Blanca Cacereña. Arch Zootec. 47: 279 - 286. [ Links ]
18. Postiglioni, A.; Rincón, G.; Kelly, L.; ángelo, D.; Gagliardi, R.; De Andre, D. 1998. Caracterización genética de bovinos criollos de Uruguay. II. Estudio de su variabilidad genética. Arch Zootec 47: 225 -231. [ Links ]
19. Zamorano, M.; Género, E.; Rodero, A.; Vega-Pla, J.; Rumiano, F. 1998. Caracterización genética de ganado bovino criollo argentino utilizando microsatélites. Arch Zootec 47: 273 - 277. [ Links ]
20. Zietkiewicz, E.; Rafalsky, A.; Labuda, D. 1994. Genome fingerprinting by simple sequence repeat (SSR) - anchored polymerase chain reaction amplification. Genomic 20: 176-183. [ Links ]














