Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.57 no.2 Palmira Apr./June 2008
RESUMEN
La técnica RAM (Random Amplified Microsatellites) se usó para el análisis de diversidad genética de 43 accesiones de uchuva recolectadas en seis regiones de Colombia (Nariño, Valle del Cauca, Cauca, Caldas, Quindío y Cundinamarca), pertenecientes a la colección establecida en la Universidad Nacional de Colombia sede Palmira. El análisis se realizó sobre 42 loci polimórficos obtenidos con siete cebadores, encontrándose baja diversidad para las introducciones de Nariño, Cauca, Quindío, Caldas y Cundinamarca. La población del Valle del Cauca reunió la mayor diversidad de los materiales recolectados.
Palabras claves: Solanaceae; loci polimórficos; cebadores; RAM.
ABSTRACT
The RAM's technique (Random Amplified Microsatellites) was used to assess the genetic diversity of 43 accesions of Cape gooseberry collected in six regions of Colombia (Nariño, Valle del Cauca, Cauca, Caldas, Quindío and Cundinamarca), property of the National University of Colombia at Palmira's campus collection. This analysis was based on the data from 42 polymorphic loci generated with seven primers. The results showed high genetic distance among accessions from Valle del Cauca and low genetic distance among accessions from Nariño, Cauca, Quindío, Caldas and Cundinamarca departments.
Key words: Solanaceae; Polymorphic loci; primers; RAM.
El género Physalis se encuentra distribuido desde Estados Unidos, México y Centro América hasta Sudamérica y las Antillas, con pocos representantes nativos del Viejo Mundo. La mayoría de las especies se encuentran en México, considerado como el centro de diversidad del género. P. peruviana L. ha sido la especie más utilizada en cultivos comerciales por su fruto azucarado (Quirós, 1984). La uchuva ha tenido gran auge en los mercados mundiales dadas sus características nutricionales y de fruta tropical exótica que la hacen llamar la fruta de oro (Fischer et al., 2000).
La caracterización y documentación de las introducciones de un banco de germoplasma permite evitar duplicados e identificar accesiones promisorias para procesos de selección, mejoramiento genético o procesos agroindustriales (Fisher, et al., 2000). Zietkiewicz, et al., 1994, propusieron un novedoso método de medición de la diversidad genética en plantas y animales utilizando una técnica molecular que combina el uso de cebadores que contienen secuencias microsatélites y un extremo 5' degenerado. Esta técnica combina los beneficios de los análisis microsatélites con el análisis RAPD (Random Amplified Polymorphic DNA) y se ha denominado microsatélites amplificados aleatoriamente (RAM). La técnica se basa en la reacción en cadena de la polimerasa (PCR), el método es reproducible y permite la detección de polimorfismo entre especies y dentro de ellas. Los marcadores obtenidos por los RAM se pueden usar para estudios de poblaciones, no requiere el conocimiento previo de secuencias ni el uso de isótopos radioactivos (Hantula et al., 1996). Los fragmentos de ADN amplificados están compuestos de dos microsatélites suficientemente cercanos para ser amplificados por PCR (Zietkiewicz et al., 1994). La fuente de variabilidad en los fragmentos obtenidos puede deberse a una inserción o supresión entre el fragmento amplificado que podría originar un amplio polimorfismo o ausencia de un producto, también la variabilidad en el número de repeticiones del microsatélite puede determinar el nivel de polimorfismo (Hantula, et. al., 1996).
Estudios de diversidad genética realizados en especies vegetales (Morillo et al., 2005) y animales (Oslinger et al., 2006) en el Laboratorio de Biología Molecular de la Universidad Nacional de Colombia sede Palmira sugieren que la técnica es una herramienta eficiente para este tipo de estudios. Muñoz et al. (2007) señalan entre las ventajas de la técnica RAM que no se necesita información previa, que es reproducible debido a la longitud del cebador, que es sencilla y de bajo costo, que se puede obtener gran número de bandas utilizando geles de alta resolución, que hay concordancia entre las características biológicas y los grupos genéticos formados y que diferencia al interior de las especies, entre especies y entre familias. El objetivo del trabajo fue evaluar la diversidad genética del Banco de Germoplasma de Uchuva de la Universidad Nacional de Colombia sede Palmira.
Semillas de 46 accesiones de uchuva (43 de P. peruviana y 3 de especies relacionadas) del Banco de Germoplasma de la Universidad Nacional de Colombia sede Palmira (Tabla 1), se sembraron en medio líquido (Murashige & Skoog 0.44 g L-1, tiamina 0.1g L-1, sacarosa 20g L-1), se incubaron a 18°C y humedad relativa de 70% durante tres meses.
La extracción de ADN de las hojas se realizó siguiendo la metodología descrita por Dellaporta et al. (1983). Las concentraciones se estimaron por comparación con patrones conocidos de ADN del bacteriófago lambda en un gel de agarosa al 0.8% corrido en buffer TBE 0.5X (Tris-borato 0045M; EDTA 0.001M) teñido con bromuro de etidio a una concentración final de 0.5 ug mL-1. El ADN cuantificado se diluyó en agua grado HPLC hasta una concentración de 5 ng uL-1 y se almacenó a –20°C.
Para la amplificación se seleccionaron siete cebadores (Tabla 2), teniendo en cuenta la habilidad para revelar polimorfismo, el número de bandas polimórficas y la reproducibilidad. Para estandarizar las condiciones de cada cebador se determinaron las concentraciones óptimas de cloruro de magnesio, cebador y buffer de Taq. También se determinó la temperatura de hibridación para cada cebador (Tabla 3). Para todos los cebadores se utilizaron 200 uM de cada DNTP, 1U de Taq polimerasa (Fermentas ®), en un volumen total de reacción de 12.5 uL. La amplificación se realizó en un termociclador PTC-100 Programmable Termal Controller (MJ Research Inc®), con las siguientes condiciones: 95°C por 5 minutos, seguido de 37 ciclos de 95°C por 30 segundos, 50-58°C por 45 segundos (de acuerdo con el cebador utilizado, Tabla 3), 72°C por 2 minutos y una extensión final de 72°C por 7 minutos.


Los productos amplificados se separaron por electroforesis en geles de agarosa al 1.2%, teñidos con bromuro de etidio a una concentración final de 0.5 ug mL-1, y se visualizaron bajo luz ultravioleta. La longitud de los productos de amplificación se estimó por comparación con un marcador de peso molecular de 100pb (Promega®).
La información suministrada se registró en una matriz binaria de presencia (1) o ausencia (0). Para la selección de bandas polimórficas se consideró como locus polimórfico aquel en el cual la frecuencia del alelo más común es menor al 99%. Para el registro no se tuvieron en cuenta aquellas bandas de difícil lectura. A partir de esta matriz, utilizando los programas SIMQUAL® del paquete Numerical Taxonomy System for Personal Computer NTSYS- pc versión 1.8 y el programa TFPGA® (Miller 1997), se realizaron los análisis estadísticos.
La similitud genética se calculó con el coeficiente de Nei- Li, S= 2a/ (2a+b+c) donde a = bandas compartidas por ambos individuos, b = bandas presentes en el individuo (1) pero no en (2), y C = bandas presentes en el individuo (2) pero no en (1). El análisis de agrupamiento se realizó con el programa SAHN de NTSYS –pc utilizando el método UPGMA, método gráfico de agrupamiento por parejas que usa el promedio aritmético no ponderado (Unweighted pair- group arithmetic mean). El dendrograma se construyó con el programa TREE de NTSYS –pc versión 1.80.
Se realizaron dos análisis de correspondencia múltiple (ACM), el primero para las 43 accesiones de P. peruviana de los diferentes departamentos y el segundo para las accesiones del Valle del Cauca. Este análisis permite asociar columnas y filas de la matriz binaria determinando el nivel de asociación o proximidad (Joseph et al., 1992).
Para estimar la diversidad genética se consideraron los parámetros de heterocigosidad promedio (H) y el porcentaje de loci polimórficos (P), los cuales se estimaron sobre todos los loci y el promedio de los mismos de acuerdo con la fórmula no sesgada de Nei (1973) así:
H= 1 -åf (i)2
f(i): Frecuencia del alelo i en la población.
åf (i)2 : Probabilidad de que dos individuos tomados al azar tengan el alelo i.
H: Probabilidad de que dos individuos tomados al azar tengan diferente alelo, H, es el valor con el que se representa la diversidad de la población.
Análisis descriptivo
Los siete cebadores RAM generaron 50 patrones de bandas distinguibles en un rango de longitud de 250 a 1.000 pares de bases. El número de loci polimórficos por cebador varió entre 1 para CCA y 10 para GT. El porcentaje de loci polimórficos fue 97.61%. El patrón de amplificación generado por el cebador CGA se muestra en la Figura 1.
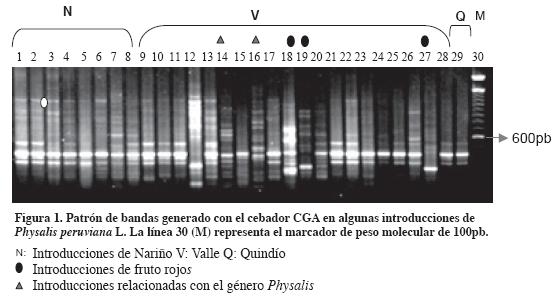
Las introducciones UNPU048, UNPU053, UNPU076, UNPU081, UNPU096 y UNPU077 procedentes del departamento del Valle presentaron mayor polimorfismo. Estos materiales presentaron fruto rojo (líneas 14 y 16 de la Figura 1) y corroboraron los resultados obtenidos en la caracterización morfológica (Bonilla et al., 2007). La introducción UNPU 018 (Juntas, Valle) presentó un patrón de bandas diferente; también las introducciones UNPU053 y UNPU048 (fruto rojo) presentaron cinco alelos únicos con los cebadores GT, CT, CGA y TG con tamaños de 680, 950, 420, 530 y 1.000 pares de bases, correspondiendo los dos últimos al cebador TG. Las diferencias morfológicas en color de fruto y semilla, en máculas, hojas, tricomas y la separación en un grupo genético diferente de los materiales mencionados, sugieren que deben separarse taxonómicamente de las otras accesiones. Para ello está en proceso un trabajo de investigación.
El coeficiente de similitud de Nei-Li, a nivel de 86%, diferenció cuatro grupos (A, B, C y D) y 24 haplotipos (individuos con estructura genética idéntica y que se diferencian de otro por lo menos en la presencia o ausencia de una banda al utilizar técnicas moleculares) (Figura 2). Los agrupamientos no se relacionaron estrictamente con el origen geográfico; sin embargo, los materiales de Cundinamarca tendieron a agruparse de la misma manera que los materiales de Nariño y Cauca, debido posiblemente a que fueron recolectados en sitios donde se encuentran cultivos comerciales a pequeña escala y es posible que haya ocurrido flujo de semilla entre regiones.
La localización en un mismo haplotipo de individuos de Cundinamarca (UNPU004, UNPU001, UNPU002), Cauca (UNPU110), Caldas (UNPU106) y Quindío (UNPU219) podría explicarse porque parte importante de la uchuva consumida en el departamento de Caldas proviene de Cundinamarca y al encontrarse al borde de carretera posiblemente puede provenir de semillas de frutos comerciales. De los materiales del Quindío y Cauca no se conoce con certeza su procedencia. Cinco materiales del Cauca (UNPU045, UNPU143, UNPU079, UNPU080 y UNPU042) tuvieron el mismo haplotipo, lo cual puede explicarse por el intercambio de semilla entre cultivadores comerciales (Figura 2).
Los materiales (UNPU021) del Valle y (UNPU113) de Nariño presentaron también el mismo haplotipo. En las cuatro agrupaciones hubo materiales del departamento del Valle, lo cual indica que la mayor parte de la variabilidad de la colección de germoplasma de la Universidad Nacional Sede Palmira está representada por materiales silvestres recolectados en un bosque secundario en la cuenca del río Guabas del municipio de Ginebra, Valle. Además, la muestra (17 accesiones) para este departamento fue la de mayor tamaño. Los materiales se encuentran en peligro de desaparecer debido a la deforestación que se presenta en esta zona.
La baja diversidad del resto de los materiales puede explicarse porque fueron recolectados cerca de cultivos comerciales, a orillas de carretera, en jardines y huertos.
El dendrograma, a nivel de similitud de 40%, diferenció dos grupos, el primero conformado por introducciones de P. peruviana y el segundo por especies relacionadas (Figura 3). Dentro de los grupos no se presentó clara diferenciación por procedencia geográfica.
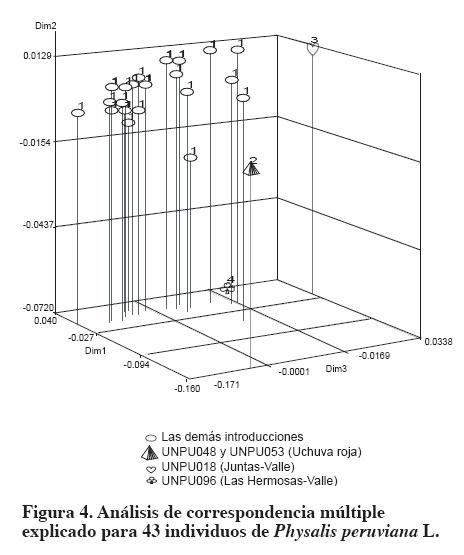
El análisis de correspondencia múltiple (ACM) (Figura 4) separó los materiales en cuatro grupos con coeficiente de similitud de 86%. El grupo 1 estuvo conformado por 39 introducciones, el grupo 2 lo integraron las introducciones de fruto rojo (UNPU048 y UNPU053), en los grupos 3 y 4 se encontraron las introducciones UNPU018 (Juntas-Valle) y UNPU096 (Las Hermosas-Valle) respectivamente.
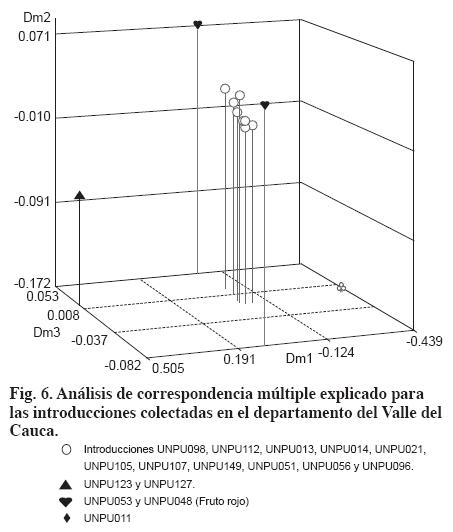
El análisis de similitud genética y de agrupamiento de las introducciones recolectadas en el departamento del Valle del Cauca (Figura 5), a nivel de similitud de 69%, formó dos grupos. El primero correspondió a las introducciones de P. peruviana y el segundo estuvo conformado por las introducciones UNPU048 y UNPU053 (fruto rojo).
UNPU096 (Las Mirlas), UNPU051 (La Selva) y UNPU018 (Juntas) se diferenciaron de las demás introducciones a nivel de similitud de 86%. Estas introducciones aportaron la mayor diversidad y se recolectaron en la cuenca del río Guabas (Valle). El análisis de correspondencia múltiple realizado para las accesiones del Valle del Cauca separó los individuos en cuatro grupos (Figura 6).
Diversidad genética
En las 43 introducciones se encontraron grupos de individuos con estructura genética idéntica que diferían de los otros por la presencia o ausencia de al menos una banda, identificándose 24 haplotipos.
El valor de heterocigosidad calculado para toda la población fue de 0.2559, valor no muy alto teniendo en cuenta que fluctúa entre 0 y 1. Sin embargo, en un estudio de Lycopersicon sculentum perteneciente a la misma familia se reportó un valor de heterocigosidad H=0.18 (Restrepo, 2003). Si se compara este valor con el obtenido en este estudio, éste fue considerablemente alto y puede estar asociado con la naturaleza alógama de la especie. Algunas introducciones recolectadas en estado silvestre en la cuenca del río Guabas y las introducciones UNPU048 y UNPU053 (de fruto rojo) están genéticamente más distantes de las demás introducciones. La poca diversidad que se observa en las introducciones de Cauca, Cundinamarca Nariño, Caldas y algunas del departamento del Valle, con similitud mayor de 90%, indican la posible homogenización de este recurso genético por la diseminación de materiales comerciales.
Se ha reportado que los estimativos de diversidad genética basados en marcadores de herencia dominante como los RAPD y los RAM están asociados con las estrategias de muestreo (número de plantas por población, distancia geográfica entre las poblaciones muestreadas y número de poblaciones estudiadas) y el número de bandas polimórficas (Nybom y Bartish, 2000).
La estructura genética poblacional de las especies se debe realizar con criterio de agrupamiento geográfico, edafo-climático o morfológico. En este estudio no se analizó la estructura poblacional debido a que en las introducciones el agrupamiento no se relacionó con la distribución geográfica, por lo cual este criterio no sería pertinente para calcular los estimativos de diferenciación poblacional. Para realizar este análisis se debe hacer un muestreo más amplio dirigido a zonas de bosques o lugares donde la influencia del componente antrópico sea mínimo y se encuentren materiales silvestres o establecer otro criterio de agrupamiento. Sin embargo, hubo un agrupamiento que discriminó entre especies y dentro de especies del género Physalis que permite concluir que la técnica RAM puede ser útil para caracterizar las poblaciones de Physalis aún no estudiadas.
La información hace parte del trabajo de grado de Ing. Agr. de M. Bonilla B. y K. Espinosa P. financiado por la Dirección de Investigaciones de la Universidad Nacional sede Palmira, DIPAL. Los autores expresan sus agradecimientos al Instituto Alexander von Humboldt, al laboratorio de Fitopatología de Fríjol del CIAT y a los integrantes del Laboratorio de Biología Molecular de la Universidad Nacional de Colombia sede Palmira.
1.Bonilla, M.; Espinosa, K.; Posso A., Vásquez, H., Muñoz J. 2007. Caracterización morfológica de 24 accesiones de uchuva del Banco de Germoplasma de la Universidad Nacional de Colombia sede Palmira. Acta Agron (Palmira) 57(2):101-108. [ Links ]
2.Dellaporta S. L.; Wood, J.; Hicks, J. B. 1983. A plant DNA minipreparation: Versión II. Plant Molec Biol Rep 14: 19-21. [ Links ]
3.Fischer, G.; Flórez, R.; ángel, D.; Sora, R. 2000. Producción, poscosecha y exportación de la uchuva Physalis peruviana L. Universidad Nacional de Colombia sede Santafé de Bogotá. 175p. [ Links ]
4.Hantula, J.; Dusabenyagasani, M.; Hamelin, R. C. 1996. Randon Amplified Microsatellites (RAM) a novel method for characterizing genetic variation within fungi. Eur J Path 26:159-166. [ Links ]
5.Joseph, H.; Anderson, R.; Tatham, R.; Black, W. 1992. Multivariate data Analysys with Readings. 3rd ed. Riverside. 554 p. [ Links ]
6.Miller, M. 1997 Tfpga (Tools for Population Genetics Analysis). Northern Arizona University, Flagstaff, Arizona. 102 p. [ Links ]
7.Morillo, A.; Morillo, Y.; Vásquez H.; Muñoz, J. 2005. Caracterización molecular con Microsatélites aleatorios RAM de la Colección de mora Rubus spp, de la Universidad Nacional de Colombia, Sede Palmira. Acta Agron (Palmira) 54 (2):15-24. [ Links ]
8.Muñoz, J. E.; Morillo, A.; Morillo, Y. 2007. Uso de la técnica Microsatélites Amplificados al Azar (RAM) para estudios de diversidad genética en vegetales. En prensa. Acta Agron (Palmira). [ Links ]
9.Nei, M. 1973. Analysis of genetic diversity in sub-divided populations. Proc Natl Acad Sci USA 70: 3321-3323. [ Links ]
10.Nybom H.; Bartish I. V. 2000 Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants. Persp Plant Ecol Evol Syst 3: 93-114. [ Links ]
11.Oslinger, A.; Muñoz J. E.; Alvarez L. A.; Moreno, F.; Posso, A. 2006. Caracterización de cerdos criollos colombianos mediante la técnica molecular RAM. Acta Agron (Palmira) 55 (4): 45-50. [ Links ]
12.Quirós, C. 1984. Overview of the genetics and breeding of husk tomato. Hort Sci 19(6):872-874. [ Links ]
13.Restrepo, S. E. 2003. Estudio de la diversidad genética del tomate cultivado tipo chonto Lycopersicon esculentum en las zonas productoras de Colombia. Trabajo de grado (M. Sc.). Universidad Nacional de Colombia sede Palmira. 182p. [ Links ]
14.Zietkiewicz, E.; Rafalski, A.; Labuda, D. 1994. Genome Fingerprinting by Simple Sequence Repeat (SSR-Anchored) Polymerase Chain Reaction Amplification. Genomics 20:176-183. [ Links ]














